水稻糖代谢相关酶和糖类转运蛋白编码基因的鉴定和表达分析
王义杰 张绍杰 赖艳 胡永峰
摘要:水稻(Oryza sativa L.)子粒淀粉的积累是产量形成的基础,灌浆期叶片通过光合作用合成蔗糖,并运输到子粒为淀粉合成提供原料,该过程中所涉及的代谢相关酶和运输相关蛋白已有研究报道。对卡尔文循环、蔗糖合成与降解、淀粉合成与降解等糖代谢相关的酶以及糖类转运蛋白编码基因进行了系统分析,共发现糖代谢相关蛋白编码基因148个和糖类转运蛋白编码基因102个,其中有228个基因已有文献报道,新鉴定基因22个。表达谱分析发现其中的部分基因表达具有组织特异性,与其功能有密切的关系。同时还发现许多基因受逆境胁迫影响表达发生变化,表明这些基因可能在水稻抗逆境胁迫中具有重要的作用。
关键词:水稻(Oryza sativa L.);糖代谢;糖类转运蛋白;产量形成
中图分类号:S511;Q78 文献标识码:A
文章编号:0439-8114(2019)22-0185-09
DOI:10.14088/j.cnki.issn0439-8114.2019.22.043 开放科学(资源服务)标识码(OSID):
Identification and expression analysis of genes encoding carbohydrate metabolism enzymes and sugar transporters
WANG Yi-jie,ZHANG Shao-jie,LAI Yan,HU Yong-feng
(Jingchu University of Technology/Institute of Plant Germplasm Resources Exploitation and Utilization,Jingmen 448000,Hubei,China)
Abstract: Starch accumulation in rice(Oryza sativa L.) seeds is the basis of rice yield formation. In filling stage sucrose is synthesized in rice leaves by photosynthesis, and then transported to seeds providing materials for starch synthesis. Several enzymes and transporters in this process have been reported. In this paper the genes encoding enzymes involved in carbohydrate metabolism including Calvin cycle, sucrose synthesis and degradation, starch synthesis and degradation and genes encoding sugar transporters were analyzed systematically. 148 genes encoding carbohydrate metabolism enzymes and 102 genes encoding sugar transporters were identified. 228 of these genes have been reported previously and 22 genes were found for the first time. Expression profile analysis indicates that some genes showed tissue specific expression pattern, which is closely related to their function. Expression of many genes was affected by abiotic stresses, demonstrating the important role of these genes in resistance to stress.
Key words: rice(Oryza sativa L.); carbohydrate metabolism; sugar transporter; yield formation
水稻(Oryza sativa L.)是中國主要的粮食作物之一,提高水稻产量是育种学家主要的育种目标,水稻子粒淀粉的积累是水稻的产量形成的基础,子粒淀粉合成的原料来源于叶片光合作用的产物,将叶片等可输出光合产物的器官称为“源”器官,而子粒等消耗或者储藏光合产物的器官称为“库”器官,连接源库器官的系统称为“流”,源流库协同作用是提高水稻产量的重要生理基础[1]。
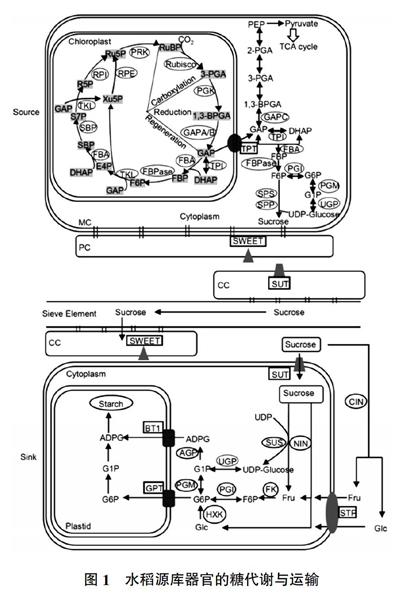
植物体内光合产物的形成、运输以及在子粒中淀粉合成的过程已有一定的研究基础。通过光合作用的卡尔文循环在叶片叶绿体中形成磷酸丙糖,磷酸丙糖被运输至细胞质中用于合成蔗糖[2]。在双子叶植物中磷酸丙糖可在叶绿体中暂时合成淀粉[3],但在水稻中淀粉的暂时储存是发生在叶鞘和茎中,在抽穗之前蔗糖被运输至叶鞘和茎中用于暂时合成淀粉,抽穗之后淀粉分解用于合成蔗糖,然后运输至子粒合成淀粉[4]。蔗糖是植物运输光合产物的主要形式,叶肉细胞产生的蔗糖通过共质体和质外体两种途径短距离运输到源端韧皮部,装载到筛管-伴胞复合体中,然后在筛管中进行长距离运输,在库端韧皮部卸载,并通过共质体和质外体两种途径进入库器官[1]。蔗糖可通过蔗糖转运蛋白直接进入细胞质分解,也可在细胞外分解为单糖后由单糖转运蛋白转运进入细胞质,单糖最终被运输至子粒的质体中用于淀粉的合成[2](图1)。
部分参与糖代谢的酶和糖类转运蛋白的编码基因已相继被鉴定出来。其中,参与卡尔文循环的编码基因有:5个编码Rubisco小亚基的RBCS基因(OsRBCS1-5)[5],4个编码磷酸甘油酸激酶(PGK)的编码基因(Ospgk1-4)[5],7个3-磷酸甘油醛脱氢酶(GAPDH)编码基因,包括OsGAPC1-3、OsGAPCp1、OsGAPCp2以及OsGAPA和OsGAPB,分别定位在细胞质、非绿色质体和叶绿体中[6],3个丙糖磷酸异构酶(TPI)编码基因(OscTPI1、OscTPI2和OspTPI)[5],3个1,6-二磷酸果糖酯酶(FBPase)编码基因[5],1个1,7-二磷酸景天庚酮糖酯酶(SBPase)编码基因(OsSBP)[7],2个磷酸戊糖异构酶(RPE)编码基因(OsRPEcyt和OsRPEchl)[8],1个5-磷酸核酮糖在磷酸核酮糖激酶(PRK)编码基因[9]。参与蔗糖合成与降解的编码基因有:2个6-磷酸葡萄糖异构酶(PGI)编码基因[10],2个葡萄糖磷酸变位酶(PGM)编码基因(OscPGM和OspPGM)[5],2个UDP-葡萄糖焦磷酸化酶(UGP)编码基因(OsUGP1和OsUGP2)[5],5个磷酸蔗糖合酶(SPS)编码基因(OsSPS1、OsSPS2、OsSPS6、OsSPS8和OsSPS11)[5],3个磷酸蔗糖磷酸酶(SPP)编码基因(OsSPP1、OsSPP2和OsSPP3)[11],7個蔗糖合酶(SUS)编码基因(OsSUS1~OsSUS7)[5],9个细胞壁转化酶(OsCIN1~OsCIN9)、2个液泡转化酶(OsVIN1和OsVIN2)和8个碱性转化酶(OsNIN1~OsNIN8)[12]。参与淀粉合成编码基因有:ADP-葡萄糖焦磷酸化酶大亚基由4个基因编码(OsAGPL1-4),小亚基由2个基因编码(OsAGPS1~OsAGPS2),淀粉合酶由7个OsSS和2个OsGBSS基因编码,淀粉分支酶由3个基因编码,淀粉去分支酶由4个基因编码,淀粉歧化酶由2个基因编码[13]。淀粉的降解主要依赖葡聚糖-水双激酶(GWD)、磷酸葡聚糖-水双激酶(PWD)、磷酸葡聚糖磷酸酶(SEX4)α淀粉酶(Amy)、β淀粉酶(BAM),在水稻中OsGWD1、OsPWD和OsSEX4已被鉴定出来[5],α淀粉酶由11个基因编码[14],有7个基因编码β淀粉酶[15]。另外,在淀粉和蔗糖的合成过程中也具有非常重要作用的己糖激酶(HXK)和果糖激酶(FK)编码基因也已经被鉴定出来,水稻中的己糖激酶由10个基因编码(OsHXK1~OsHXK10)[5],有2个果糖激酶编码基因(OsFKI和OsFKII)[16]。
糖类转运蛋白已鉴定的基因有:2个磷酸丙糖转运蛋白(TPT)编码基因(OsTPT1和OsTPT2)[5],4个磷酸葡萄糖转运蛋白(GPT)编码基因(OsGPT1、OsGPT2-1、OsGPT2-2和OsGPT2-3)[17],3个ADP-葡萄糖转运蛋白编码基因(OsBT1-1、OsBT1-2和OsBT1-3)[18],但只有OsBT1-1具有转运ADP-葡萄糖的活性,而其他两个蛋白只能转运AMP、ADP和ATP,而不具备转运ADP-葡萄糖的活性。功能分析表明,OsBT1-1参与淀粉的合成,而OsBT1-3与叶片中叶绿体的发育有关[5]。5个蔗糖转运蛋白编码基因:OsSUT1~OsSUT5[5],21个SWEET蛋白编码基因[19],1个麦芽糖转运蛋白编码基因(OsMEX1)[20]。但仍有部分参与糖代谢的酶和糖类转运蛋白的编码基因没有鉴定,本研究对水稻源流库器官中糖类的合成与降解相关的酶以及转运蛋白编码基因进行鉴定,包含卡尔文循环、淀粉合成与降解、蔗糖合成与降解、己糖激酶和果糖激酶以及糖类转运蛋白等,并对这些基因的表达谱以及响应非生物胁迫表达变化进行了分析,为进一步弄清楚水稻子粒淀粉积累的分子机制、提高水稻产量奠定基础。
1 材料与方法
1.1 水稻糖代谢相关酶和转运蛋白编码基因的鉴定
用小麦(Triticum aestivum)、拟南芥(Arabidopsis thaliana)或水稻中已报道的蛋白序列作为query分别在RGAP数据库(http://rice.plantbiology.msu.edu/)、RAPDB数据库(http://rapdb.dna.affrc.go.jp/)和NCBI数据库中(https://blast.ncbi.nlm.nih.gov/Blast.cgi)进行搜索,进行序列比对,并将序列在SMART数据库(http://smart.embl-heidelberg.de/)中进行预测,确定蛋白结构域。
1.2 进化树分析
将要进行进化树分析的蛋白质序列用ClustalX 程序进行序列比对,然后用MEGA 3.1软件邻近连接法(Neighborhood-joining)进行进化树分析,使用如下参数:Model:Amino:Poisson correction,Gaps/Missing Data:Pairwise Deletion,phylogeny Test and options:Bootstrap,Replications:1000,Random seed,其他参数默认。
1.3 表达分析
在RiceChip注释网站上(http://www.ricechip.org/)获取每个基因的AFFY Probe Set IDs,基因表达谱分析用基因的AFFY Probe Set IDs作为query在CREP数据库(http://crep.ncpgr.cn/crep-cgi/home.pl)中搜索[5],获取的基因表达值用Cluster 3.0软件进行聚类分析,并用Treeview软件进行可视化。从GEO数据库中下载芯片数据(GSE6901)来分析基因响应胁迫的表达变化[5]。
1.4 水稻生长和胁迫处理
本试验所用水稻品种为Hwayoung,水稻种子经表面消毒以后,在生根培养基中发芽,在光照14 h/黑暗10 h光周期条件下生长14 d,将水稻全苗放在200 mmol/L的NaCl溶液和PEG 6000溶液中处理,在0、1、3、6 h取样,放置于超低温冰箱中保存。
1.5 RNA的抽提、反转录和实时荧光定量PCR
总RNA用TRIzol试剂进行抽提,4 μg RNA用于反转录,首先用1单位的DNase进行处理,然后在20 μL包含0.5 μg Oligo(dT)、0.75 mmol/L的dNTPs和200 U的M-MLV反转录酶的反应体系中进行反转录。
取反转录产物2 μL在10 μL体系中(引物终浓度为0.3 mmol/L、SYBR Green 5 μL)、在Stepone PCR仪上根据操作手册进行实时定量荧光PCR,所有引物在60 ℃退火1 min,总共40个循环。水稻Actin基因作为内参基因,所有目标基因的表达水平用Actin的表达进行平衡。
2 结果与分析
2.1 水稻糖代谢相关酶编码基因的鉴定
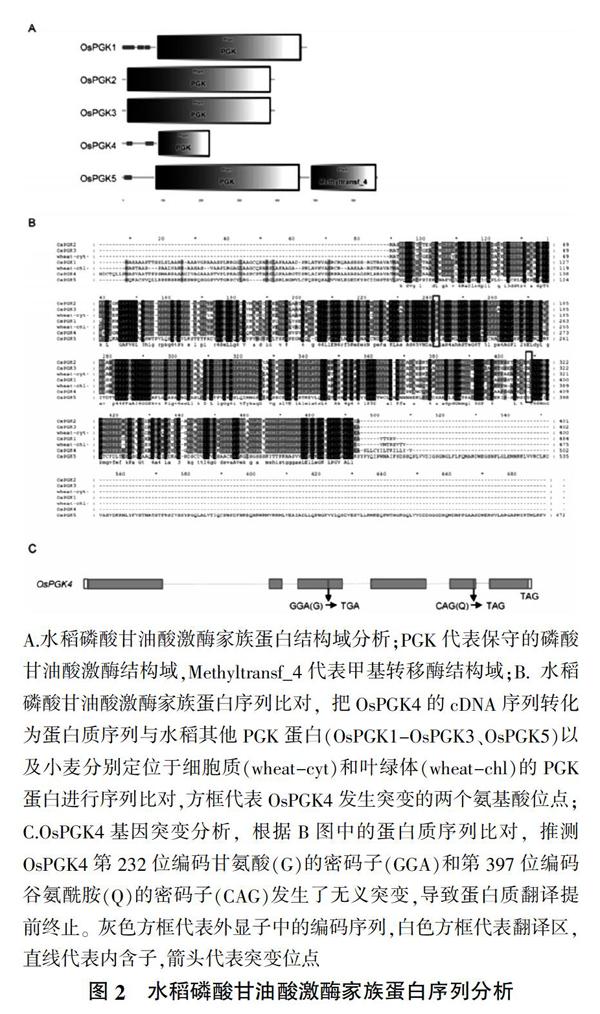
2.1.1 卡尔文循环 用小麦的PGK蛋白质序列在水稻数据库(MSU)中搜索发现[5],水稻中有5个同源性较高的蛋白,依次命名为OsPGK1~OsPGK5,通过序列比对和结构域分析发现OsPGK5除了含有PGK结构域以外,还含有一个甲基转移酶结构域(图2A),而且它的蛋白质序列与其他蛋白同源性较低,因此认为它不属于磷酸甘油酸激酶家族的基因。将所有蛋白质序列在GenBank数据库中搜索,发现OsPGK4蛋白序列与MSU预测序列不一致,由于GenBank数据库中有RNA-seq试验数据支持基因的预测,因此其预测结果更加可靠。GenBank数据库中OsPGK4预测蛋白仅有231个氨基酸,通过将OsPGK4的cDNA序列转化为蛋白质序列然后再与其他PGK蛋白序列进行比对,推测OsPGK4在进化的过程中发生了突变导致翻译提前终止,使得PGK结构域发生了缺失而失去了功能(图2B、图2C)。
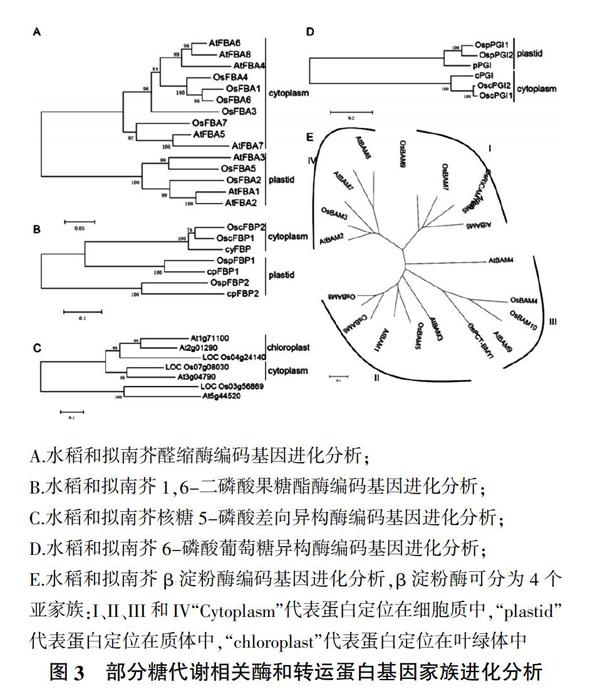
综上所述,水稻中共有3个有功能的磷酸甘油酸激酶编码基因(OsPGK1~OsPGK3)。由于PGK既可定位在叶绿体中也可定位在细胞质中,在小麦中这两种PGK均已克隆[5],将水稻3个PGK蛋白质序列与不同细胞定位的小麦PGK蛋白序列对比,结果表明,OsPGK1编码蛋白定位于叶绿体,而另外2个基因编码的蛋白定位于细胞质中。利用拟南芥的醛缩酶蛋白序列在水稻数据库中搜索,发现水稻中共有7个FBA基因,命名为OsFBA1~OsFBA7。进化树分析表明OsFBA2和OsFBA5与拟南芥预测的定位在质体中的蛋白属于同一类,另外5个蛋白则与定位在细胞质中的蛋白属于同一类(图3A)。利用拟南芥的FBPase蛋白序列进行搜索,在水稻数据库中找到了4个同源序列,通过序列比对进化树分析将与拟南芥cyFBP同源的2个蛋白分别命名为OscFBP1和OscFBP2,而与拟南芥cpFBP1和cpFBP2同源的2个蛋白分别命名为OspFBP1和OspFBP2(图3B)。在拟南芥中有2个TKL编码基因(AtTKL1和AtTKL2),两个蛋白均定位在叶绿体中,但表达分析表明AtTKL1在绿色组织中表达较高,推测它是参与卡尔文循环的主要转酮酶[5]。用拟南芥AtTKL1蛋白序列进行同源搜索,在水稻中找到2个同源蛋白,分别命名为OsTKL1和OsTKL2。拟南芥中有3个核糖-5-磷酸差向异构酶(RPI)编码基因:At1g71100、 At2g01290 和 At3g04790,另外1个基因At5g44520编码的蛋白序列虽然与这3个蛋白有一定的相似性,但缺少酶活关键的氨基酸,因此它可能催化其他反应[21]。根据预测At1g71100和At2g01290定位在细胞质中,而At3g04790定位在叶绿体中,可能参与卡尔文循环[21]。通过蛋白序列同源搜索,在水稻基因组中找到3个基因:LOC_Os03g56869、LOC_Os07g
08030和LOC_Os04g24140,通过序列比对和进化树分析发现LOC_Os03g56869与拟南芥At5g44520同源,因此它不是水稻的RPI编码基因,而其他2个基因编码的蛋白与拟南芥RPI有很高的同源性(图3C),根据它们与拟南芥两类细胞定位不同的蛋白的同源性将LOC_Os07g08030和LOC_Os04g24140分别命名为OsRPIcyt和OsRPIchl。水稻中已发现了1个PRK编码基因[9],用这个蛋白序列进行同源搜索,在水稻基因组中发现了另外1个PRK编码基因,并将这两个基因分别命名为OsPRK1和OsPRK2。
2.1.2 蔗糖合成 在水稻中早有报道至少有2个定位于细胞质中的PGI[10],通过同源搜索发现水稻基因组共有4个PGI编码基因,序列对比和进化树分析表明2个定位于细胞质(OscPGI1和OscPGI2),另外2个定位于质体(OspPGI1和OspPGI2)(图3D)。
2.1.3 淀粉合成与水解 水稻中已经鉴定有7个基因编码β淀粉酶[15],通過拟南芥的β淀粉酶蛋白质序列进行同源搜索,发现水稻中至少有10个编码基因,进化树分析表明与拟南芥类似[15],水稻的β淀粉酶也分属4个亚家族:OsRICAMYBA和OsBAM7属于亚家族I,OsPCT-BMYI、OsBAM5、OsBAM6和OsBAM8属于亚家族II,OsBAM4和OsBAM10属于亚家族III,OsBAM3和OsBAM9属于亚家族IV(图3E)。
2.1.4 果糖激酶 水稻中已鉴定了2个果糖激酶编码基因:OsFKI和OsFKII,分别属于I和II类果糖激酶[16],但通过同源序列搜索发现水稻中还存在1个果糖激酶基因,将其命名为OsFKIII。
2.2 糖类转运蛋白编码基因
2.2.1 SWEET家族转运蛋白 水稻中已鉴定有21个SWEET蛋白,通过同原序列搜索发现水稻中还有另外2个基因编码SWEET蛋白:Os01g0314600和Os09g0508250,在MSU数据库中没有这2个基因的注释,因此水稻共有23个SWEET蛋白编码基因。
2.2.2 单糖转运蛋白家族 对拟南芥的单糖转运蛋白家族基因的序列分析表明单糖转运蛋白家族可分为7个亚家族:STP、VGT-like、PLT、INT、TMT、pGlcT和ERD6-like[22]。STP是一种细胞质膜蛋白,将质外体的己糖转运进入细胞,VGT-like蛋白可定位在液泡膜和叶绿体膜上转运葡萄糖,PLT蛋白定位于细胞质膜上转运单糖和多糖,INT蛋白在细胞质膜上负责肌醇的转运,TMT定位在液泡膜上负责己糖的运输,pGlcT蛋白在叶绿体膜上将淀粉水解产生的葡萄糖转运至细胞质中,ERD6-like蛋白可能定位在液泡膜上转运糖类物质。在水稻中单糖转运家族共有64个基因[23],其中STP亚家族包含28个基因(OsMST1~OsMST28),OsMST1~OsMST6和OsMST8均有文献报道[5],其他基因则按照他们在染色体上的位置顺序来进行命名。VGT-like亚家族包含2个基因(OsVGT1和OsVGT2),OsVGT1与拟南芥的AtVGT1和AtVGT2亲缘关系较近,可能定位于液泡膜上,而OsVGT2与拟南芥的At5g59250较同源,可能定位在叶绿体膜上[22,23]。PLT亚家族包含15个基因(OsPLT1~OsPLT15),根据他们在染色体上的位置顺序来进行命名。INT亚家族包含3个基因(OsINT1~OsINT3),根据与他们同源的拟南芥基因来进行命名。TMT亚家族包含6个基因(OsTMT1~OsTMT6),其中OsTMT1~OsTMT4已有文献报道[5]。pGlcT包含4个基因(OspGlcT1~OspGlcT4),ERD6-like亚家族包含6个基因(OsERD6-1~OsERD6-6),均根据他们在染色体上的位置来进行命名。
2.3 表达谱分析
基因的表达特异性可间接地反映其功能特异性,因此为了分析糖代谢相关酶和转运蛋白编码基因的表达谱,利用基因的AFFY芯片探针号从CREP提取了部分基因在22个组织器官中的表达数据(部分基因没有搜索到探针号),分别将组织器官分为源(全苗、叶片、叶鞘、开花之前的稃和浸种72 h的种子)和库(根、茎、雄蕊、穗、小穗以及胚乳),并分析了基因的表达规律。
分析表明,在卡尔文循环中部分基因具有很高的表达特异性,包括:OsPGK1、OsFBA2、OsGAPA、OsGAPB、OsRBCS4、OsPRK1、OsSBP、OspFBP2、OsTKL1、OsRPEchl和OsRPIchl(图4A)。这些基因在源器官中除了种子以外在其他组织中的表达量均较高,而在库器官中的表达相对较低,但发现这些基因在穗和小穗中表达量与源器官中相近,这可能是由于穗和小穗中均包含了可进行光合作用的稃片,因此推测这些基因可能是参与卡尔文循环的主要基因。同一家族的基因表达谱差异表达,这可能与其功能有一定的关系,例如甘油脱氢酶编码基因OsGAPA和OsGAPB主要在绿色细胞中表达参与光合作用,OsGAPCp1和OsGAPCp2在除胚乳以外的库器官中表达较高,在非绿色质体中参与糖代谢,而OsGAPC1~OsGAPC3之间差异更为明显,它们可能分别在不同的组织中参与糖酵解或糖异生。与拟南芥中的AtTKL1和AtTKL2表达相似,OsTKL1在绿色细胞中表达较高,而OsTKL2在胚乳中表达较高,表明OsTKL2不参与卡尔文循环。与OsPGK1在主要绿色细胞中表达相反,OsPGK2和OsPGK3在库器官中表达较高,而且这2个蛋白主要定位在细胞质中,表明他们主要在库器官中参与糖酵解或糖异生(图4A)。
蔗糖合成与降解相关酶编码基因的表达没有规律性,大部分基因呈现组成性表达,仅有少部分基因表达呈现组织特异性,例如:OsCIN1、OsCIN5和OsNIN7在源器官中表达相对较高,OsCIN6和OsCIN7仅在种子和根中表达较高,由于蔗糖的降解产物还可用于合成其他糖类物质,推测这些基因在部位的表达可能与这些糖类物质的合成有关(图4B)。没有发现在源器官中表达相对较高的与蔗糖合成相关酶编码基因,表明这些基因在其他组织器官中也有很重要的作用(图4B)。与小麦中相似,水稻OsSPS1和OsSPP1随着胚乳的发育表达逐渐升高(图4B),推测这些酶可能与蔗糖分解与再合成形成的无效循环有关,而这个无效循环可增加对碳水化合物代谢调控的灵活性[24]。
淀粉合成与降解相关酶编码基因中有一部分表达呈现很强的组织特异性,例如:OsAGPS1、OsAGPS2、OsSSI、OsAGPL2、OsSSIIa、OsGBSSI、OsBEI、OsBEIIb、OsSSIIIa、OsISA1和OsISA2,这些基因在胚乳中表达较高,表明这些基因产物主要在胚乳发育的过程中催化淀粉的合成(图4C)。α淀粉酶編码基因大部分在种子中表达较高与淀粉的降解有关,但有部分基因也在胚乳中表达,目前还不知道它们在胚乳的发育中有何作用,有部分淀粉降解酶编码基因如OsAmy3D、OsRICAMYBA、OsBAM7、OsBAM8、OsGWD1和OsSEX4在叶片中表达也较高,表明水稻叶片可能暂时储藏淀粉,OsGWD1和OsSEX4在茎中表达水平也比较高,他们可能参与茎中暂时储藏淀粉的分解,但他们的积累是在抽穗以前,抽穗以后表达明显下降(图4C)。除此之外,OsBAM4、OsBAM6、OsBAM6、OsGWD1和OsSEX4在雄蕊中表达水平很高,他们可能在雄配子体发育过程中降解淀粉合成其他碳水水化合物(图4C)。
己糖激酶和果糖激酶中精油OsHXK6在源器官中表达比库器官高,OsHXK4、OsHXK8和OsFKI在种子中表达较高,OsHXK1、OsHXK2、OsHXK5和OsHXK10在雄蕊中表达较高,表明不同的果糖激酶在不同的组织器官中发挥作用(图4D)。单糖转运蛋白中仅有部分STP类编码基因具有一定的表达特异性,在源器官表达较高,同时在根中有很高的表达,另外还有部分单糖转运蛋白在雄蕊中表达较高,其他基因表达没有规律性(图5A)。磷酸丙糖转运蛋白编码基因(OsTPT1和OsTPT2)在源器官表达水平较高,进一步表明他们主要在叶绿体中转运磷酸丙糖(图5B)。磷酸葡萄糖转运蛋白编码基因OsGPT1和OsGPT2的表达具有一定的互补性,OsGPT1在叶片中表达较低,在胚乳发育早期表达较高,OsGPT2则在叶片和胚乳发育的晚期表达较高,表明他们可能在不同的组织器官转运磷酸葡萄糖(图5B)。ADP-葡萄糖转运蛋白编码基因(OsBT1-1)主要在胚乳中表达,麦芽糖转运蛋白编码基因OsMEX1在雄蕊和穗中表达较高,在叶片中表达较低,与之前报道的表达谱相似(图5B)。SWEET转运蛋白家族中OsSWEET1b在源器官和胚乳中表达较高,OsSWEET11和OsSWEET15倾向于在库器官中表达,OsSWEET5与蔗糖转运蛋白编码基因OsSUT3表达谱非常相似,在根、雄蕊和穗中表达量最高。其他蔗糖转运蛋白编码基因的表达没有呈现很强的组织特异性(图5B)。
2.4 响应非生物胁迫的表达分析
有研究表明有些参与糖代谢的酶参与植株的抗逆性。在水稻中3-磷酸甘油醛脱氢酶编码基因OsGAPC3可被盐胁迫诱导表达,超表达OsGAPC3可提高植株的抗盐性[5],在烟草中超表达磷酸甘油酸激酶编码基因OsPgk2a-P可在盐胁迫下提高烟草的产量[5],还有研究表明在缺水的胁迫下,蔗糖合酶和淀粉分支酶的活性会显著升高,从而提高子粒灌浆的速率[25]。为了分析糖代谢相关酶编码基因和糖类转运蛋白编码基因受非生物胁迫的表达变化,从GEO数据库中提取了干旱和盐胁迫处理的表达芯片数据,并用AFFY芯片探针号进行搜索,共得到120个糖代谢相关酶编码基因和66个糖类转运蛋白编码基因的表达数据。
结果表明,糖代谢相关酶编码基因中,受干旱胁迫影响有33个基因上升表达,32个基因下降表达,受盐胁迫影响16个基因上升表达,15个基因下降表达(表1)。糖类转运蛋白编码基因中,受干旱胁迫影响有21个基因上升表达,22个基因下降表达,受盐胁迫影响10个基因上升表达,12个基因下降表达(表2)。选取了几个具有显著性差异的基因,利用RT-qPCR技术进行了验证(图6),结果与芯片表达数据一致。进一步分析芯片表达结果发现,在源器官特异表达的参与卡尔文循环的基因除了OsFBA2,其他均受干旱影响下降表达,表明逆境胁迫下光合速率下降的其中一个原因是参与卡尔文循环的酶的合成受到了抑制,致使其碳同化的能力下降(表1)。许多参与蔗糖降解的蔗糖合酶和转化酶参与淀粉降解的酶编码基因受逆境胁迫诱导表达,这可能与植物在受逆境脅迫以后需要大量的小分子可溶性糖来进行渗透调节有关(表1)。还发现同一种酶的不同编码基因受逆境胁迫影响不同,例如转酮酶编码基因OsTKL1受干旱胁迫影响下降表达,而OsTKL2上升表达,由于2个基因的表达模式具有明显的差异,因此推测2个基因可能参与不同的生化途径,并在抗逆境胁迫中起不同的作用(表1)。糖类转运蛋白编码基因中大部分蔗糖转运蛋白编码基因受逆境胁迫诱导表达,表明在逆境条件下需要加快蔗糖的转运来供应能量(表2)。其他家族的基因受逆境胁迫影响既有上升表达基因也有下降表达基因,推测不同的转运蛋白在逆境胁迫中起不同的作用。
参考文献:
[1] 李合生.现代植物生理学[J].第3版.北京:高等教育出版社,2012,
[2] BUCHANAN B B,GRUISSEM W,JONES R L. Biochemistry & molecular biology of plants,2nd edition[M].Hoboken:Wiley,2015.
[3] LLOYD J R,KOSSMANN J,RITTE G. Leaf starch degradation comes out of the shadows[J].Trends in plant science,2005,10(3):130-137.
[4] HIROSE T,ENDLER A,OHSUGI R. Gene expression of enzymes for starch and sucrose metabolism and transport in leaf sheaths of rice (Oryza sativa L.) during the heading period in relation to the sink to source transition[J].Plant Prod Sci,1999, 2:178-183.
[5] WANG L,XIE W B,CHEN Y,et al. A dynamic gene expression atlas covering the entire life cycle of rice[J].The plant journal,2010,61:752-766.
[6] ZHANG X H,RAO X L,SHI H T,et al. Overexpression of a cytosolic glyceraldehyde-3-phosphate dehydrogenase gene OsGAPC3 confers salt tolerance in rice[J].Plant cell tissue & organ culture,2011,107:1-11.
[7] CHEN X,XIONG J,YU T,et al. Molecular cloning and characterization of rice sedoheptulose-1,7-bisphosphatase gene that is regulated by environmental stresses[J].Journal of plant biochemistry & biotechnology,2004,13:93-99.
[8] KOPRIV,S,KOPRIVOVA A,S?譈SS K H. Identification,cloning, and properties of cytosolic D-Ribulose-5-phosphate 3-Epimerase from higher plants[J].J Biol Chem,2000,275:1294-1299.
[9] CHEN X,YU T,XION,J, et al. Molecular cloning and expression analysis of rice phosphoribulokinase gene that is regulated by environmental stresses[J].Mol Biol Rep,2004,31:249-255.
[10] NOZUE F,UMEDA M,NAGAMURA Y,et al. Characterization of cDNA encoding for phosphoglucose isomerase of rice(Oryza sativa L.)[J].DNA Seq,1996,6:127-135.
[11] LUNN J E. Sucrose-phosphatase gene families in plants[J].Gene,2003,303:187-196.
[12] JI X,ENDE W V D,LAERE A V,et al. Structure,evolution,and expression of the two invertase gene families of rice[J].J Mol Evol,2005,60:615-634.
[13] FU F F,XUE H W. Coexpression analysis identifies rice starch regulator 1,a rice AP2/EREBP family transcription factor,as a novel rice starch biosynthesis regulator[J].Plant physiol,2010,154:927-938.
[14] 廖登群,張洪亮,李自超,等.水稻(Oryza sativa L.) α-淀粉酶基因的进化及组织表达模式[J].作物学报,2010,36(1):17-27.
[15] FULTON D C,STETTLER M,METTLER T,et al. β-AMYLASE4,a noncatalytic protein required for starch breakdown,acts upstream of three active b-amylases in Arabidopsis chloroplasts[J].The plant cell,2008,20:1040-1058.
[16] JIANG H,DIAN W,LIU F,et al. Isolation and characterization of two fructokinase cDNA clones from rice[J].Phytochemistry,2003,62(1):47-52.
[17] TOYOTA K,TAMURA M,OHDAN T,et al. Expression profiling of starch metabolism-related plastidic translocator genes in rice[J].Planta,2006,223(2):248-257.
[18] HU D,LI Y,JIN W,et al. Identification and characterization of a plastidic adenine nucleotide uniporter(OsBT1-3) required for chloroplast development in the early leaf stage of rice[J].Scientific reports,2017,7:41355.
[19] YUAN M,ZHAO J,HUANG R,et al. Rice MtN3/saliva/SWEET gene family:Evolution,expression profiling,and sugar transport[J].J Integr Plant Biol,2014,56:559-570.
[20] RYOO N,EOM J S,KIM H B,et al. Expression and functional analysis of rice plastidic maltose transporter,OsMEX1[J].J Korean Soc Appl Biol Chem,2013,56:149-155.
[21] KUNZ H H,ZAMANI-NOUR S,H?魧USLER R E,et al. Loss of cytosolic phosphoglucose isomerase affects carbohydrate metabolism in leaves and is essential for fertility of Arabidopsis[J].Plant physiol,2014,166:753-765.
[22] B?譈TTNER M. The monosaccharide transporter(-like) gene family in Arabidopsis[J].FEBS Letters,2007,581:2318-2324.
[23] JOHNSON D A,THOMAS M A. The monosaccharide transporter gene family in Arabidopsis and rice:A history of duplications,adaptive evolution,and functional divergence[J].Mol Biol Evol,2007,24:2412-2423.
[24] CASTLEDEN C K,AOKI N,GILLESPIE V J,et al. Evolution and function of the sucrose-phosphate synthase gene families in wheat and other grasses[J].Plant physiol,2004,135:1753-1764.
[25] YANG J,ZHANG J,WANG Z,et al. Activities of enzymes involved in sucrose-to-starch metabolism in rice grains subjected to water stress during filling[J].Field crops research,2003,81:69-81.

