巨蛎属牡蛎DNA 序列的比较及鉴定
彭 敏,罗 邦,朱威霖,杨春玲,赵永贞,李强勇,刘青云,曾地刚,李 旻,陈秀荔
(1.广西壮族自治区水产科学研究院// 2.广西水产遗传育种与健康养殖重点实验室,广西 南宁 530021)
牡蛎属双壳纲(Bivalvia )翼形亚纲(Pterimorphia)珍珠贝目(Pterioida)牡蛎科(Ostreidae),是分布广、种类多、产量高的一种经济贝类[1-2],在近岸生态系统中扮演重要角色[3]。牡蛎具有广栖息性,外部形态常随生活环境不同而变化,在所有海洋无脊椎动物中,牡蛎是最常见的、形态变异大的一类[4],通过形态来确定牡蛎的分类地位往往会造成错误和困惑[5-9]。因此,寻找快速有效的种类鉴定方法,对牡蛎的遗传改良、种质资源管理与保护等具有重要意义。目前,线粒体及核糖体DNA序列是鉴定和分类巨砺属牡蛎的常用方法,例如Klinbunga等[10]用线粒体DNA标记对泰国的Crassostrea belcheri、艾氏牡蛎(Crassostrea iredalei)和僧帽牡蛎(Saccostrea cucullata)进行分类。Mazon-Suastegui等[11]报道称28S rDNA是商业上重要的牡蛎鉴定的替代标记。Cordes等[12]报道了基于线粒体DNA标记区分9种巨蛎属牡蛎物种。Diaz等[13]根据5S rDNA基因扩增实现对Crassostrea和Ostrea oysters的物种鉴定;Lam等[14-15]基于线粒体DNA方法鉴定出一种新的巨蛎属牡蛎和印度西太平洋岩牡蛎;Wang等[7]基于线粒体16S rRNA和核28S rRNA基因的DNA序列对近江牡蛎进行分类;杨叶欣等[16]采用RAPD分子标记和rDNA-ITS1序列分析对近江牡蛎两个野生种群的遗传多样性分析;李咏梅等[17]进行了钦州湾牡蛎线粒体16S rRNA和COⅠ基因片段的序列变异分析。此外,还有非基因方法来研究鉴定和分类,如Wang等[18]采用高分辨率熔解分析法研究了来自中国的五种巨蛎属牡蛎的鉴定。DNA序列分析是DNA多态分析技术中最有效的方法之一,是各类生物的遗传、进化和生态等领域研究的重要工具。核糖体DNA转录间隔子序列(Internal transcribed spacer,ITS)进化速度快、在种和种下水平变异丰富,是重要的分子标记,在无脊椎动物的分子系统学研究中,ITS 区序列已得到了广泛的应用,为解决一些长期存在的分类学问题提供了新的重要标记[19-25]。目前关于采用线粒体DNA片段(COI、12S、16S和tRNAs)以及核糖体RNA基因转录间隔子序列ITS-1、ITS-2和ITS对巨蛎属牡蛎进行鉴定和分类鲜有报道。针对牡蛎外壳形态的多变性,为寻找合适的用于区分牡蛎的DNA条形码(DNA barcodes),本研究通过对线粒体DNA片段(COI、12S、16S和tRNAs)以及核糖体RNA基因转录间隔子序列(ITS-1、ITS-2和ITS)的比对分析,对我国沿海分布的香港牡蛎(Crassostreahongkongensis)、有明牡蛎(Crassostrea ariakensis)、葡萄牙牡蛎(Crassostrea gigas angulata)、太平洋牡蛎(Crassostrea gigas gigas)、熊本牡蛎(Crassostrea sikamea)、岩牡蛎(Crassostrea nippona)和艾氏牡蛎(Crassostrea iredalei)等7种巨砺属牡蛎进行分子系统学研究,旨在寻找出快速有效的种类鉴定和分类方法,以期阐明其系统关系和种类区分,进而为牡蛎的遗传改良、种质资源管理与保护提供依据和指导。
1 材料与方法
1.1 供试材料
本研究分别在广西钦州、防城、广东阳江、山东莱州湾和福建等地收集香港牡蛎、有明牡蛎、葡萄牙牡蛎、太平洋牡蛎和熊本牡蛎等5种牡蛎样本各10个,分别对其进行扩增测序。
利用GenBank公布的与本研究同属或同种的牡蛎线粒体全基因序列33条及全长ITS序列25条比对分析,这些序列划属于7个巨砺属种(表1)。
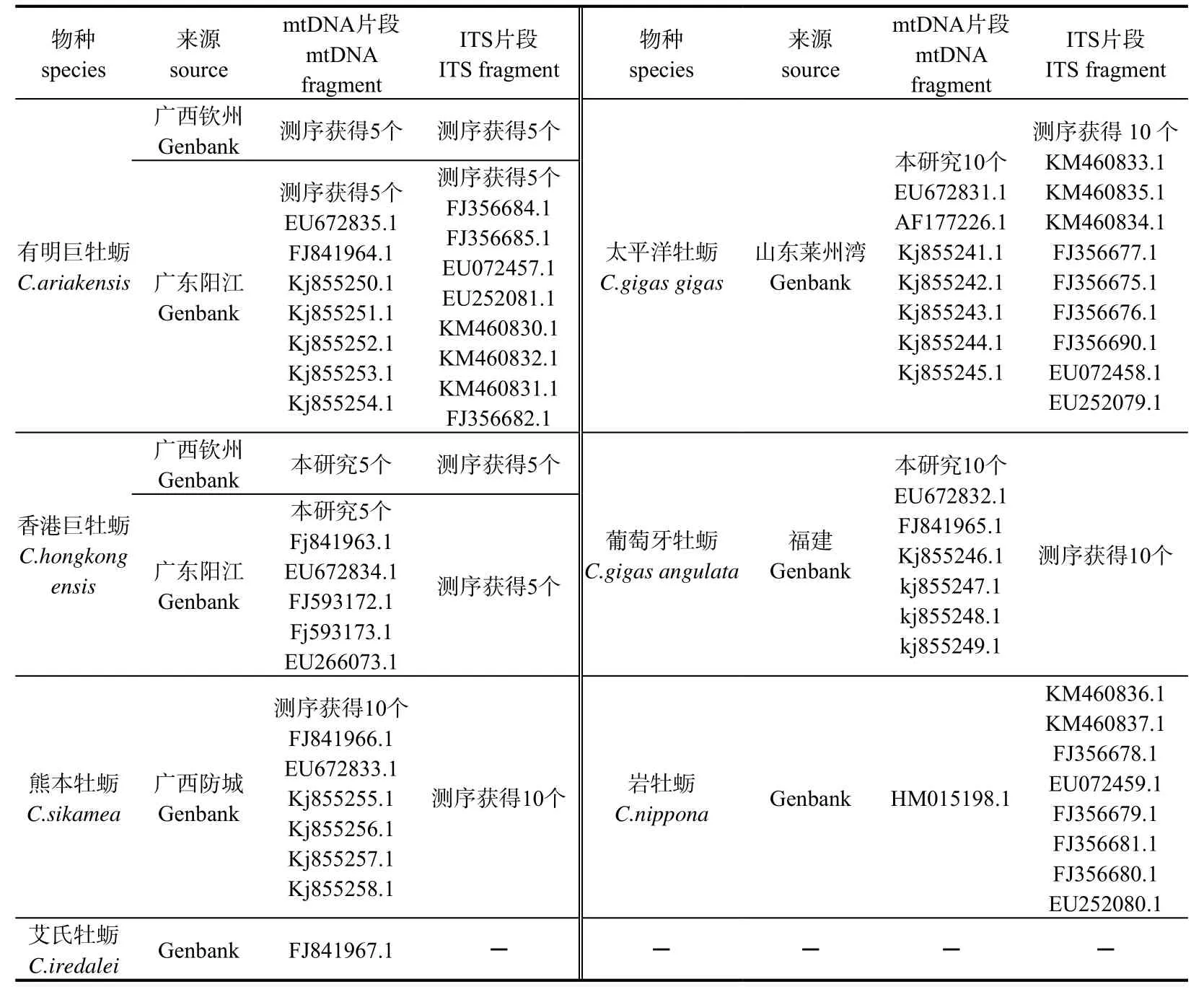
表1 所用样品的相关信息Table 1 Related information on the samples used in this study
1.2 DNA 的提取、扩增与测序
采集牡蛎闭壳肌按醋酸铵法提取牡蛎总DNA[26]。本研究选用4段线粒体DNA片段和3段核DNA片段对我国采集的香港牡蛎、有明牡蛎、葡萄牙牡蛎、太平洋牡蛎和熊本牡蛎样本进行PCR扩增,tRNAs片段扩增界于12S和16S之间,其引物根据Genbank中巨砺属线粒体DNA序列设计相应的扩增引物,而COI、16S、12S、ITS1、ITS2和ITS均采用贝类基因通用引物,本研究所采用的引物见表2。
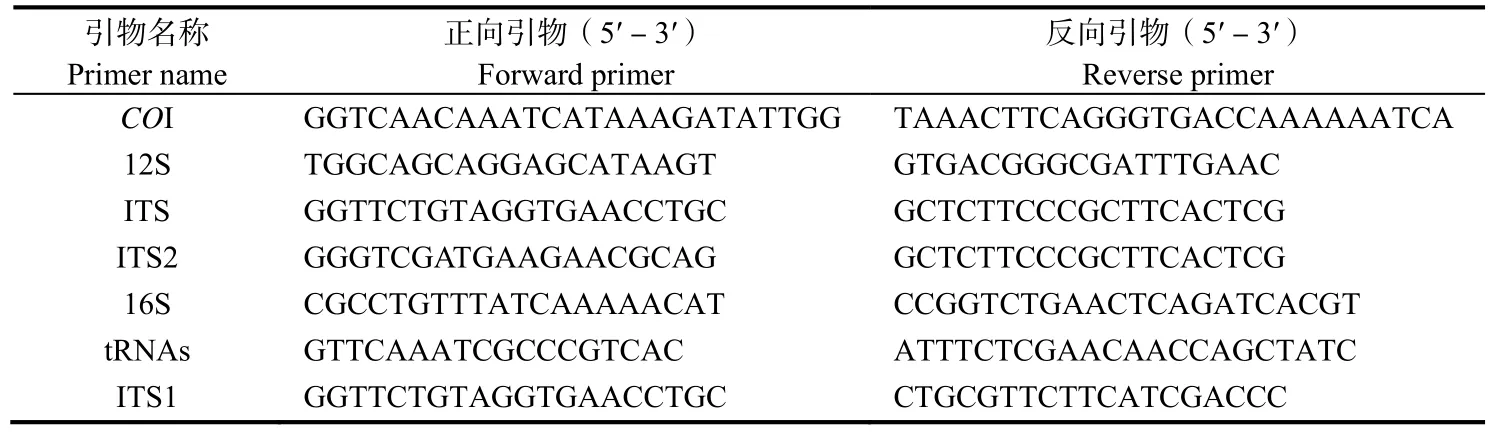
表2 PCR扩增K片段及引物序列Table 2 Primer sequence for PCR
1.3 DNA序列处理及数据分析
采用SeqMan程序,COI、16S、12S片段拼接后去除引物区及不稳定区域,tRNAs、ITS片段测序拼接后去除两端序列,ITS1和ITS2序列直接用ITS序列切除获得;在GenBank下载的序列与本实验样品的测序结果比对后获得相应的序列。应用MEGA4.1软件计算基于Kimura双参数距离模型(K2P)的序列种间和种内遗传距离[27];通过Wilcoxon检验分析不同序列之间的变异性[28];通过比较序列种内和种间变异的分布评估条形码间隙(barcoding gap);采用BLAST和Nearest Distance考察序列的鉴定成功率。
2 结果与分析
2.1 7个基因片段的序列分析
由表3可知,50个样本的7个片段均全部PCR扩增成功,COI、16S、12S和tRNAs PCR扩增产物均可直接测序成功,ITS1、ITS2和ITS直接测序成功率极低,故ITS PCR产物电泳切胶回收后克隆,所得的克隆子均测序成功,ITS1和ITS2序列直接用ITS序列切除而得。
各条序列的长度从277~ 1 203 bp不等,去除两端12S和16S部分序列后获得的tRNAs序列最短,为277~ 306 bp,ITS序列最长,为1101~ 1 203 bp。tRNAs序列变异位点最多,为51.4%,12S变异位点最少,为10.3%。各序列的GC含量也不相同,ITS1序列的GC含量最高,达到58.0%,tRNAs序列的GC含量最低,A+T含量高达71.4%。
2.2 7个基因片段序列的差异分析
对获得的巨砺属牡蛎不同序列的平均种间距离、平均种内距离、最小种间距离和最大种内距离进行分析。结果如表4所示,在种间最小距离中,tRNAs序列最大,COI序列次之,16S序列最小,种内最大距离中,同样是tRNAs序列最大,COI序列次之,12S序列最小,ITS1的种内最大距离过了种间最小距离。
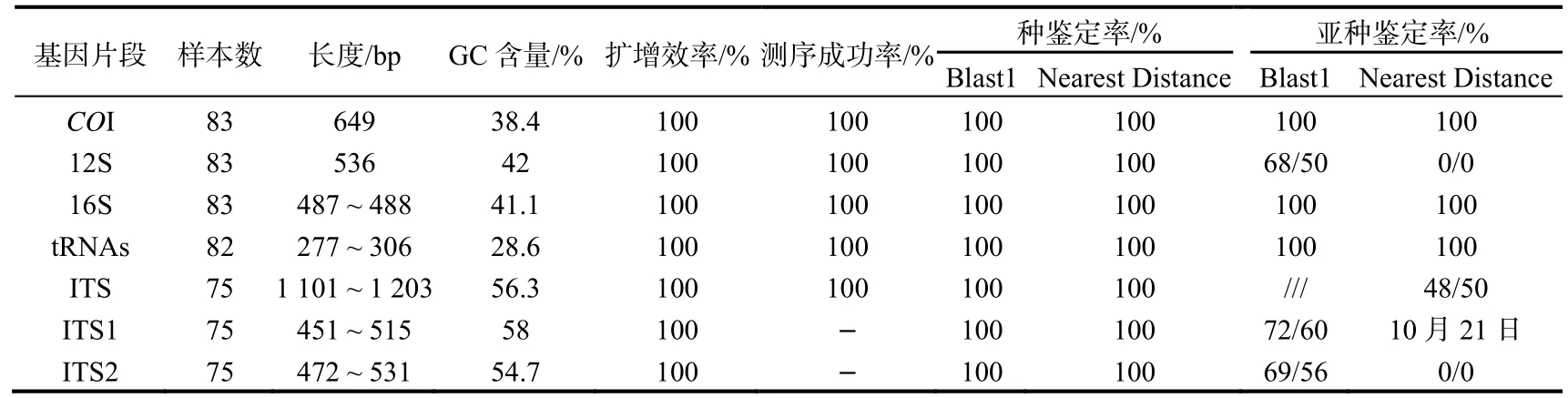
表3 7个基因片段的序列分析结果Table 3 Sequence analysis results of seven DNA fragment
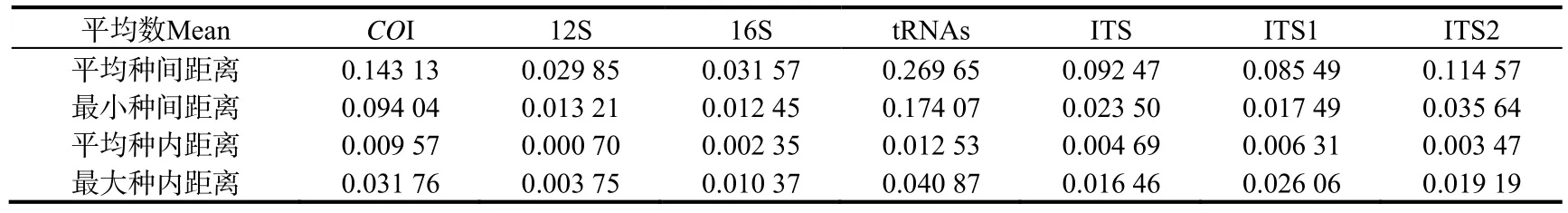
表4 7个基因片段在巨蛎属种内种间变异Table 4 Inter-specific variation and Intra-specific variation in seven gene fragments in Crassostrea
亚种间及亚种内不同序列的差异分析结果如表5,亚种间最小距离序列差异对比发现,tRNAs序列差异最大,COI次之,而12S、ITS、ITS1、ITS2亚种间最小距离的序列差异为0;亚种内最大距离对比发现,ITS1和ITS差异最大,12S、ITS、ITS1和ITS2的亚种内最大距离序列差异变化均超过了亚种间最小距离的序列变化差异。
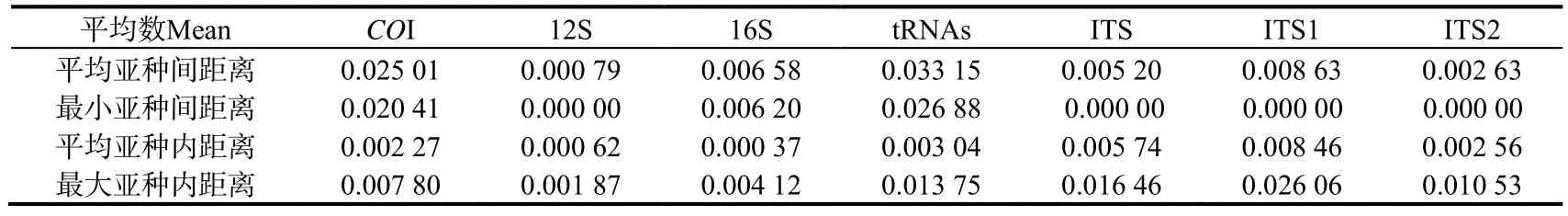
表5 7个基因片段在巨蛎属亚种内亚种间变异Table 5 Inter-subspecific variation and Intra-subspecific variation in seven gene fragments in Crassostrea
2.3 7个基因片段序列变异程度比较
Wilcoxon秩和检验结果(表6、7)表明,无论是种间变异还是种内变异,最大的都是tRNAs序列,COI序列次之,12S序列最小。在亚种间变异,最大的是tRNAs序列,COI次之,12S序列最小,而在亚种内变异,ITS1序列最大,ITS次之,12S序列最小。tRNAs和COI序列较其它序列具有极显著性差异,反映出各序列(亚)种间的变异情况是tRNAs和COI较其它序列变异更快,所得结果与表5中(亚)种间距离值结果一致。
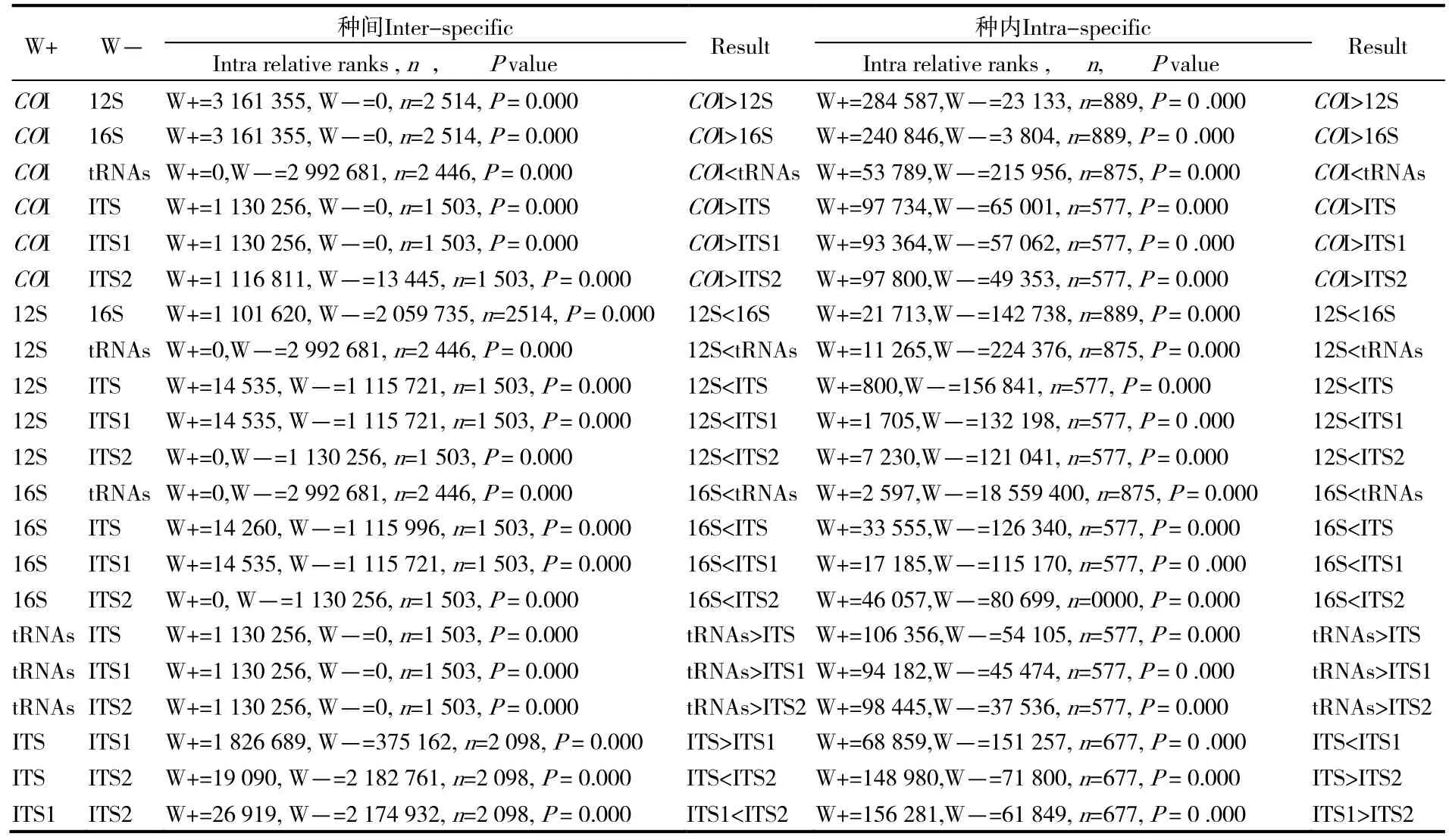
表6 7个基因片段在种间种内差异的Wilcoxon秩和检验Table 6 Wilcoxon Rank and Test of Inter-specific variation and Intra-specific variation in seven gene fragments
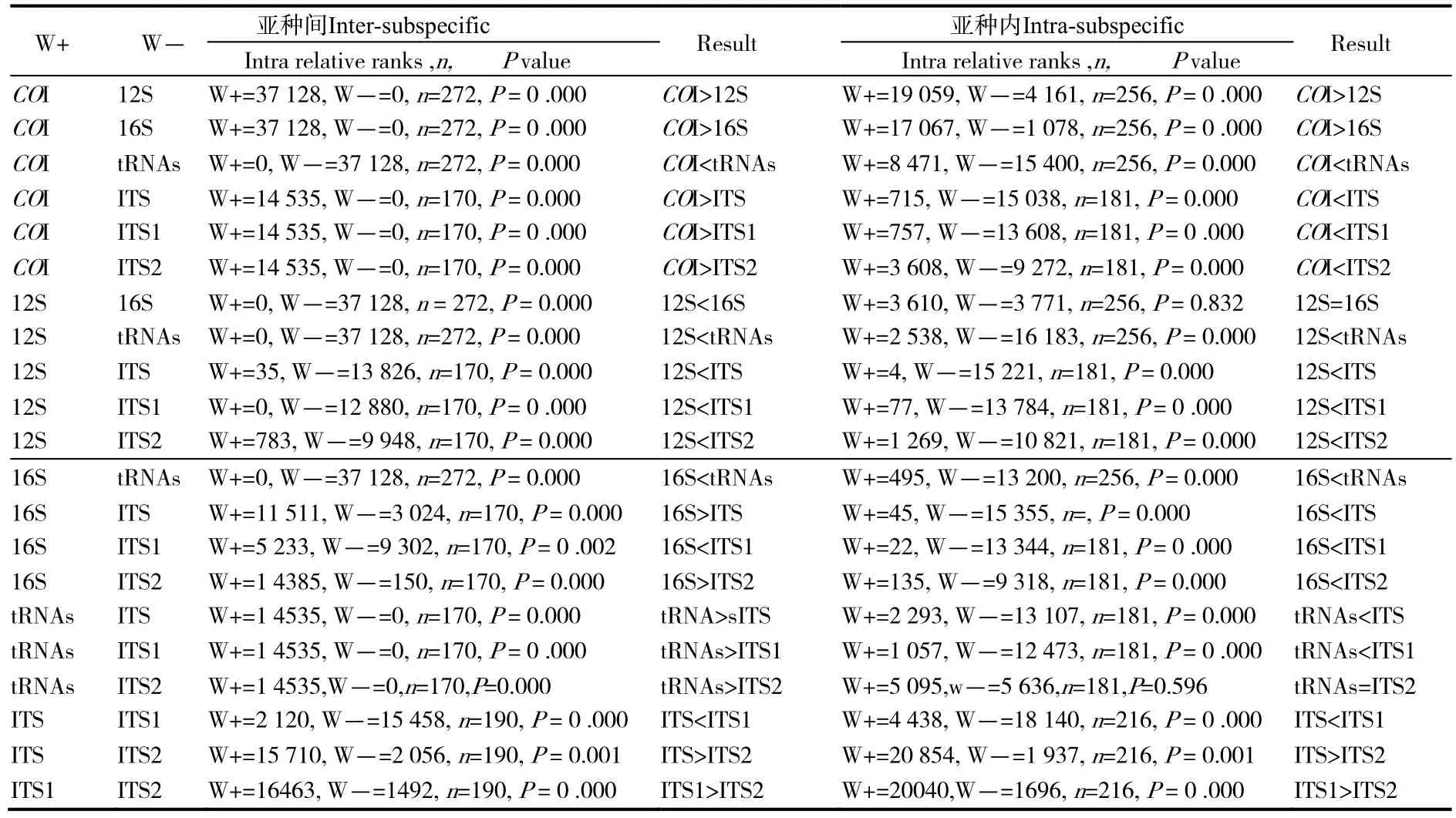
表7 7个基因片段在亚种间亚种内差异的Wilcoxon秩和检验Table 7 Wilcoxon Rank and Test of Inter-subspecific variation and Intra-subspecific variation in seven gene fragments
2.4 7个基因片段在种内变异和种间变异分布
巨砺属不同序列种内变异和种间变异分布图(图1)显示,在种层面上,tRNAs和COI序列种间变异与种内变异之间有明显的条形码间隙,12S、16S、ITS和ITS2的种间变异与种内变异没有重叠,但也不存在条形码间隙,而ITS1的种间变异与种内变异出现重叠,不利于区分物种。在亚种层面上,12S、ITS、ITS1和ITS2的亚种间变异与亚种内变异出现重叠,16S的亚种间变异与亚种内变异虽没有重叠,但也不存在条形码间隙,而tRNAs和COI序列亚种间变异与亚种内变异之间有明显的条形码间隙,有利于区分亚种。
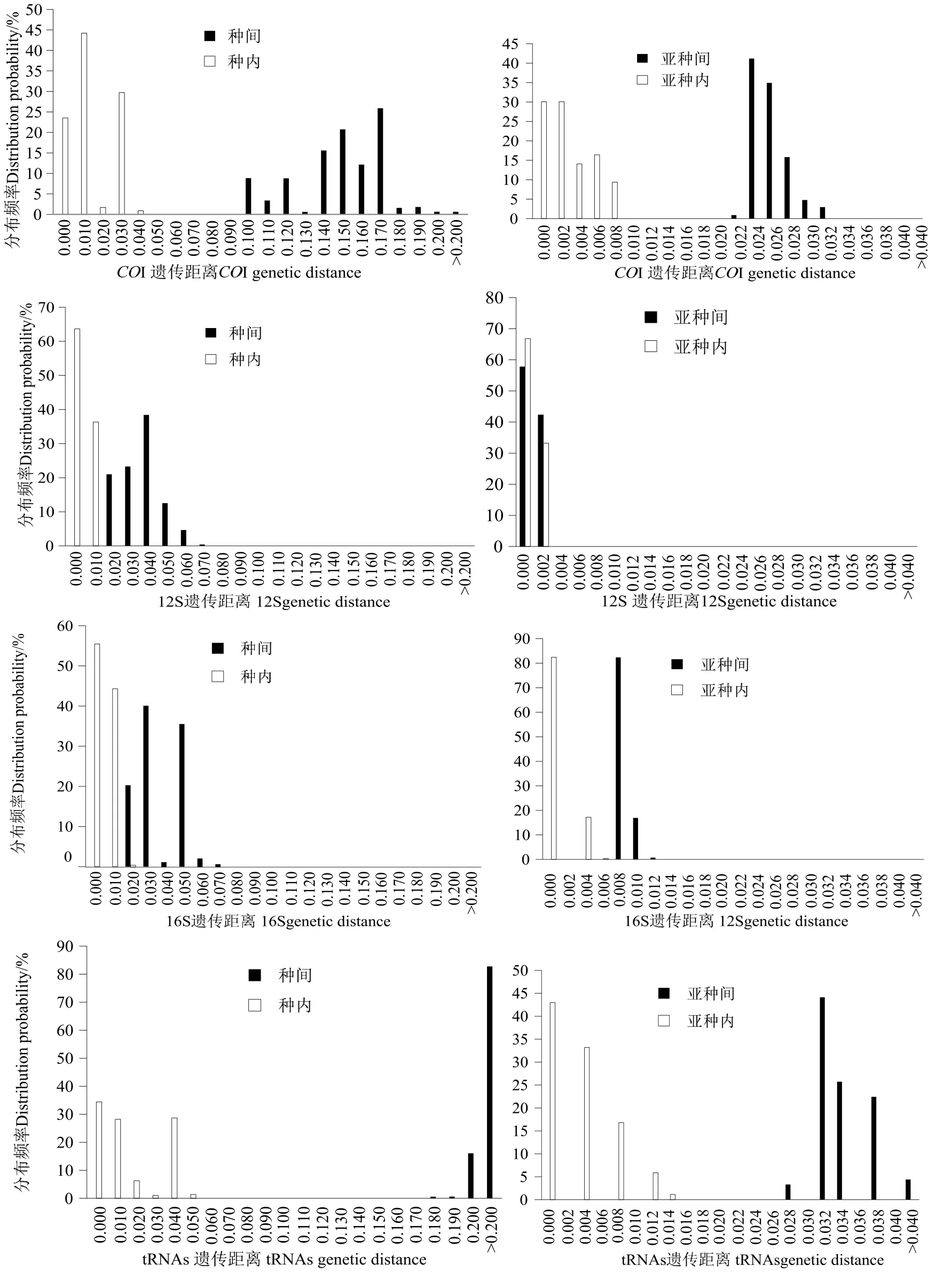
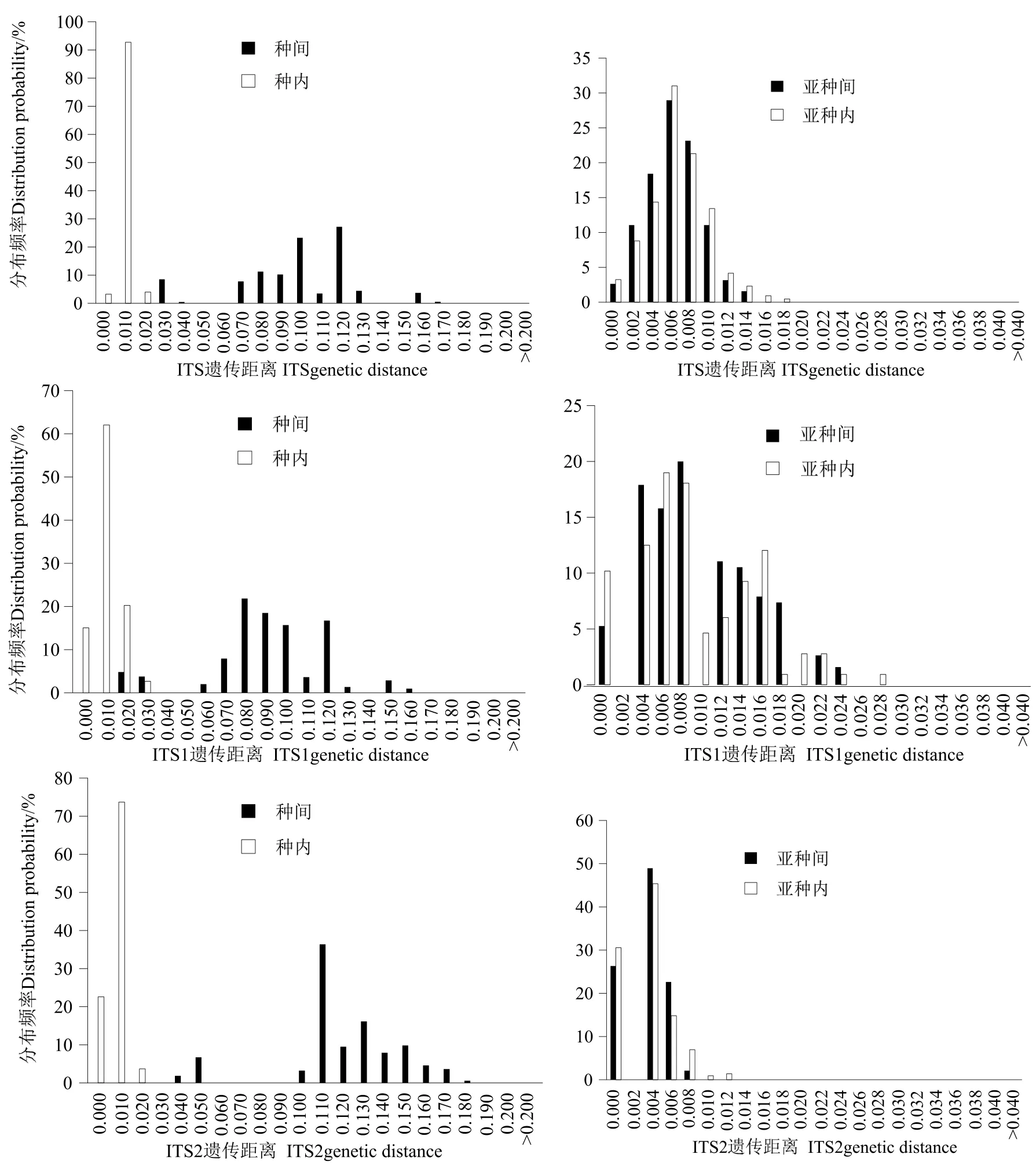
图1 (亚)种内和(亚)种间变异分布Fig.1 genetic variation distribution of species and subspecies
3 讨论
巨牡蛎属的分类已经研究了很多年,尽管已引入分子鉴别方法,但是争论仍然不断,其中1 个典型例子就是C.gigas和C.angulata的分类关系。一些研究者认为这2 个是不同的种,但是遗传上近缘关系很近[29],另外有人则表示这2 个应该为1 个种[30-31],还有人将福建等南方海域广泛分布并大量养殖的葡萄牙牡蛎命名为福建长牡蛎(C.gigas angulata),并将其与我国长江口以北海域广泛分布并大量养殖的长牡蛎(C.gigas gigas)由种间关系修订为亚种关系[32]。本研究采用第三种观点对我国巨砺属牡蛎进行分析。
理想的DNA条形码序列应具备明显的种间变异,同时种内变异要足够小,并在两者间形成一个明显的间隔区,即条形码间隙[33],并且应该能够使用单引物对其进行扩增,通过双向测序得到高质量的序列[34-35]。Hebert等认为种内与种间的标准差异应为种内平均遗传距离的10倍(10倍法则)[36]。另外有一些研究者则建议当计算条形码间隙时应用最小种间距离取代平均种间距离[37]。本研究对7个不同牡蛎品种的tRNAs、COI、16S、12S、ITS、ITS1和ITS2序列进行分析。分析结果表明,在种层面上,7种序列的种内种间平均距离均超过种内平均距离10倍以上,但若用最大种内距离和最小种间距离取代平均种内距离和平均种间距离,最大倍率为tRNAs(4.25)随后是12S(3.52)和COI(2.96)最低为ITS1,仅为0.67,10倍距离法是不能满足。在亚种层面上,tRNAs、COI、16S的种内种间平均距离均超过种内平均距离10倍以上,而最小种间距离与最大种内距离最大倍率为COI(2.62),随后是tRNAs(1.95),12S、ITS、ITS1和ITS2最小种间距离与最大种内距离比值为0,无法区分亚种。在植物鉴定中,表现较好的ITS、ITS1、ITS2常作为各种植物鉴定的候选DNA条形码序列[38-39]。然而,在本研究中巨蛎属牡蛎种内与种间遗传差异中ITS1存在重叠,而ITS、ITS2与12S无条形码间隙,即使C.gigas gigas和C.gigas angulata有共同序列也难以区分这些种,因此这些基因片段不适合用作种水平的物种鉴定。16S尽管也有较高的遗传分化率,但在(亚)种间与(亚)种内距离分布图中没有明显的条形码间隙,而16S和12S遗传变异在本研究中7种序列中最小,随着采样范围的扩大和样品数量的增加,(亚)种内的遗传距离很可能也随之增大而出现重叠。相较而言,tRNAs、COI比12S和16S更适合巨砺属牡蛎物种鉴别,与王青[40]的研究结果认为COI优于16S一致,与刘凯凯等[41]认为COI可以快速鉴定岩牡蛎和长牡蛎结果一致,但他们认为16S与COI均可快速鉴定岩牡蛎和长牡蛎。
4 结论
在鉴别巨蛎属牡蛎种以及亚种方面本研究中表现最好的是tRNAs,其次为COI 序列,tRNAs比COI 序列拥有更高的变异,但从巨砺属牡蛎物种来说COI 序列更适合物种鉴定,COI 引物具有更广的通用性[42]。在鉴别巨砺属牡蛎方面tRNAs 具有更高的变异性和更大的条形码间隙可作为COI 序列的辅助序列。本研究证实了tRNAs 和COI 序列不仅可用于种间的鉴定,也可用于亚种间的鉴定。该研究可用于阐明牡蛎系统关系和种类区分,进而为其遗传改良、种质资源管理与保护提供依据和指导。
——和田盘羊

