基于混沌分组编码的DNA存储技术研究
彭 源, 石晓龙
(1.华中科技大学 人工智能与自动化学院, 湖北 武汉 430074; 2.广州大学 计算科技研究院, 广东 广州 510006)
计算机科学技术在飞速发展的同时,信息量的增长速度也不容小觑.根据Stasista公司的统计,到2025年全球信息的总量将达到2018年的5倍.现在的存储介质如硬盘和硅制半导体的增长速度远远没有达到信息量的增长速度[1-3],因此,寻找下一代信息存储介质迫在眉睫.
脱氧核糖核酸(DNA)作为遗传信息的载体,具有存储密度大、并行性较高及低能耗等好处,是一种天然的数据存储介质[4].随着第二代DNA测序技术的出现,结合人工合成技术,研究人员期望用DNA作为下一代数据存储介质.
近年来DNA存储技术逐渐成为研究人员关注的焦点[5].哈佛大学医学院的Church团队在2012年首次将650 kb的一本图书存储在DNA上[6],实现了体外DNA的数据存储,在国际尚属首次,五年后利用大肠杆菌DNA存储了一段视频文件[7], 实现了DNA数据的快速复制.同年,哥伦比亚大学和纽约基因组中心的研究人员发明了一种DNA喷泉系统,该系统在达到较高的香农信息量的同时,还可以有较高的鲁棒性[8].2018年,Organick等[9]提出一种编码方法,该方法显著减少了测序冗余,并实现了文件的随机访问.微软计划于2020年在数据中心建立基于DNA的数据存储系统.DNA存储取得革命性进展的同时还存在难题需要攻克.
本文研究一种基于混沌分组编码的DNA存储技术,该方法具备一定的抗数据损坏的稳健性,具有较高的数据存储容量,并缩短编码时间,提高存储性能.
1 方法设计与原理
基于混沌分组码的DNA存储技术步骤如图1所示,先将待存储的文件压缩为二进制文件,随后对二进制文件进行编码使其变为含有ATCG四种核苷酸的DNA序列.在序列两端加入DNA扩增所需的引物片段和底物片段,随后将这些碱基序列合成为DNA片段,这样就完成了信息的存储.在进行信息读取时,为了获得多段相同的DNA,对DNA库进行聚合酶链反应技术(PCR)进行扩增,再对DNA序列进行测序,然后解码恢复所存储的信息.
整个存储技术中关键部分为DNA编码部分,该部分主要分为编码和筛选两个环节.
1.1 编码
本文编码部分分为两个步骤,第一步将二进制文件进行LT编码得到中间数据,然后将中间数据进行混沌分组编码,进行混乱和扩散.因为DNA链的生化特性,不能含有较长的均聚物长度以及需要稳定的GC含量.混沌分组编码的混乱和扩散特性刚好可以降低均聚物长度,以及稳定GC含量,这样可以降低程序运行时间以及提升DNA存储的正确性.
首先,将二进制文件预处理为一系列一定长度的非重叠段,然后,迭代计算三个步骤,即Luby变换、混乱扩散和筛选.Luby变换为喷泉码奠定了基础.基本上,它通过使用鲁棒孤波分布从文件中选择随机片段并将其异或变为新数据,这样将二进制数据打包为任意数量的短消息(小滴).流程如图2所示.小滴包含两条信息:保存数据异或过程结果和固定长度的短种子.该种子对应于液滴创建过程中变换的随机数生成器的状态,并允许解码器算法推断液滴中各段的身份.从理论上讲只要液滴的累积大小略大于原始文件的大小,就可以使用高效算法,通过收集液滴的任何子集来逆向Luby变换.本算法在每次迭代中利用一轮计算来创建单个液滴.
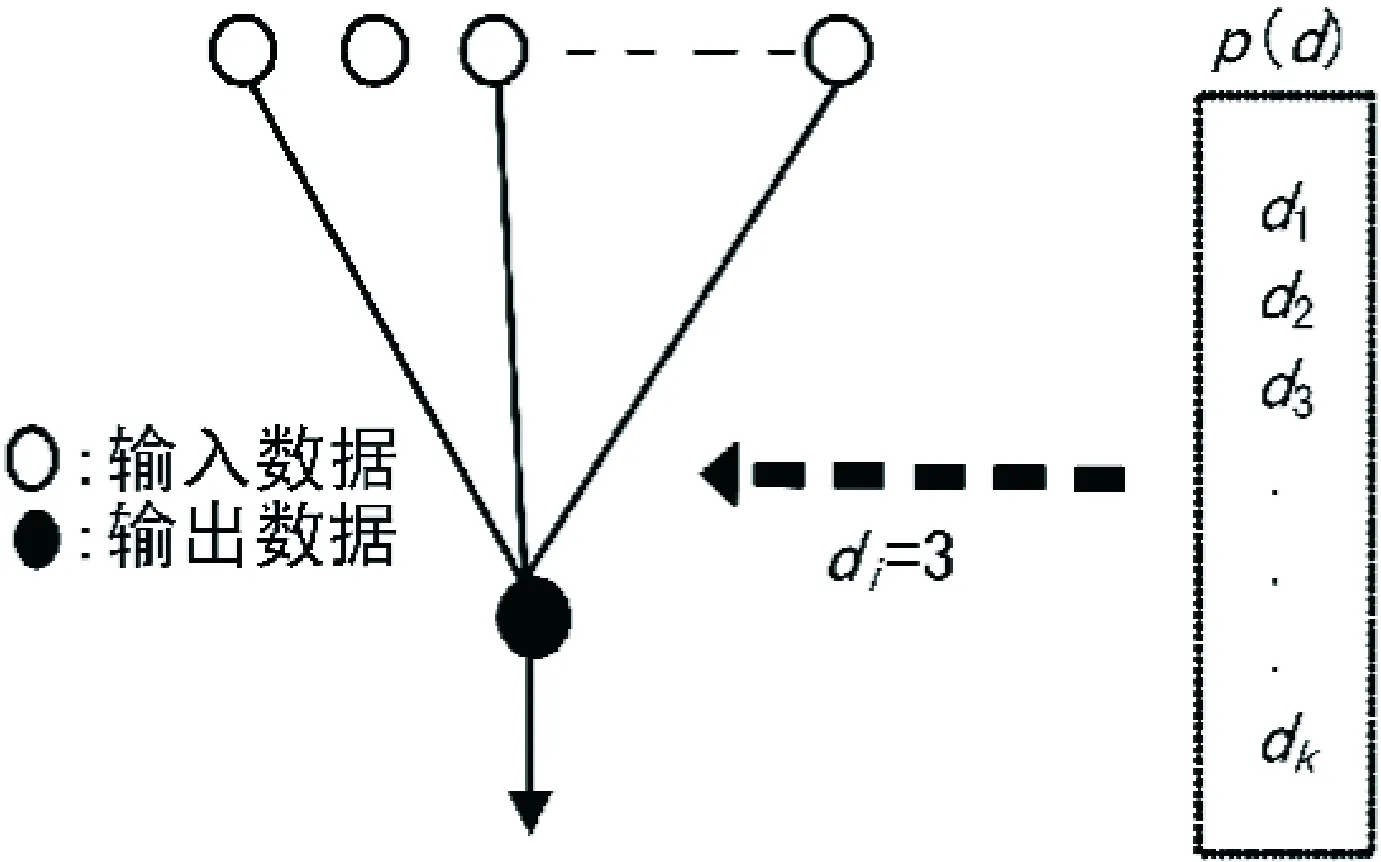
图2 Luby变换流程图Fig.2 Flowchort of Luby transformation
下面利用混沌分组编码来对单个液滴进行混乱扩散重排,使单个液滴转化碱基序列的GC含量稳定以及不含有较长的均聚物长度,满足DNA的生化特性.利用logistic映射,本文提出一种基于Feistel结构的混沌分组密码.记Bi为长度8字节64位的小液滴,即Bi是一个长为8字节xi,0,xi,1,xi,2,xi,3,xi,4,xi,5,xi,6,xi,7的分组,Bi=xi,0xi,1xi,2xi,3xi,4xi,5xi,6xi,7.作用过程由k轮相同计算循环作用于同一个明文分组构成,其变换为
xi,n+1=xi-1,n⊕fn-1[xi-1,1,…,xi-1,n-1,zi-1,n-1,z].
其中,i表示当前是循环次数i=1,…,k.n=1,…,8,f0=z,z是密钥,为简化计算,密钥取相同值,其流程如图3所示.

图3 混沌分组编码流程图Fig.3 Flowchart of chaotic block coding
函数f1,…,f7的形式为fj=fj(x1,…,xj,z),其中,j=1,…,7,f是由logistic映射产生的一个函数.函数的输出xk,0xk,1xk,2xk,3xk,4xk,5xk,6xk,7是下一轮函数的输入.所以,k轮的输出xk,0xk,1xk,2xk,3xk,4xk,5xk,6xk,7长度为8字节64位的密文分组,长度与明文分组相同.本文的函数fj=g(x1⊕x2⊕…⊕xj⊕z),其中,g是通过离散化具有混合和扩散的非线性logistic映射.
(1)改变logistic映射的系数,使其输入输出值在0到256之间.
(2)离散化标准化后的logistic映射.
DNA存储技术中的信息在生成DNA链,以及DNA链复制,和DNA测序等众多过程中,碱基极易发生替换、丢失等问题,这样在DNA信息解码时,因为某个碱基的错误而导致信息解码错误,进而数据恢复出错.为了保证DNA存储数据的正确性,加入纠错码尤为重要.RS(Reed-Solomon)纠错码因为其性能优良,被广泛应用于DNA存储技术中,确保信息存储的正确性.
1.2 筛选
与计算机和手机等基于信息论信道通信过程不同的是,DNA存储技术过程中生成的DNA序列需要满足一定的生化特性.在双链DNA中,如果其中胞嘧啶C,鸟嘌呤G的含量越高,决定双链DNA稳定性的氢键越多.DNA越稳定则测序时需要的条件越高.因此,为了降低DNA测序的条件,需要将DNA序列中的胞嘧啶C和鸟嘌呤G控制在一定范围.研究发现均聚物长度也是引起DNA合成,及测序错误的一个重要原因.所以为了确保DNA合成和测序时的准确性,除了引入RS纠错码外还需对候选碱基序列进行胞嘧啶C,鸟嘌呤G含量和均聚物长度过滤.
本文选取DNA碱基序列中GC含量在45%~55%之间,以及最长均聚物长度不超过3的序列进行DNA存储.流程如图4所示.对不满足条件的碱基序列去除重新计算,满足条件的碱基序列保留生成DNA序列.

图4 筛选流程Fig.4 Screening plan process
2 实验结果及分析
实验过程:本文对一张12 KB的图片进行压缩后得到的10 KB压缩文件作为本文DNA存储技术的输入,后经编码、筛选后生成碱基序列实现DNA储存.为了全方位评价本算法的优越性,加入了近年来主流的DNA[4-10]存储方案进行对比实验.对比结果如表1所示.

表1 不同存储技术对比实验结果Table 1 Performance of different storage plans
从表1可以看出,本文提出的方法具有1.57位/nt的信息密度,并且DNA存储的信息中冗余信息占比较低,并可以将信息还原,可以看出本方案的各项评价指标皆优于其他方案,与DNA喷泉方案相同,故更详细地对比这两种方案.
本文在DNA喷泉码的基础上对生成的中间文件进行混沌编码,使其GC含量稳定并使均聚物长度降低.为了对比的效果更加明显,本文固定信息冗余量为0.1.统计编码碱基序列需要尝试的次数,在设定最长均聚物长度不超过3的条件下对比结果如图5(a)所示,可以看出,本算法具有一定的混乱和扩散特性,编码尝试的次数降低了20%.图5(b)表示均聚物长度不超过4的编码尝试次数,可以看出编码尝试的次数主要与最长均聚物长度有关.本文提出的DNA存储技术方案可以有效降低编码所需尝试次数,增加编码成功率.

图5 DNA喷泉与本文编码次数Fig.5 DNA fountain and the coding times of this artide
与DNA喷泉码相比,本文提出的方案在相同条件下可以降低编码尝试的次数,提升编码成功率,并具有相同的信息密度.
3 结 论
本文提出一种基于混沌分组编码的DNA存储技术,在DNA喷泉码的基础上将混沌分组编码对原文的混乱与扩散性应用于中间文件中,克服生成的DNA序列中GC含量差别较大以及均聚物长度过长的问题.实验使用压缩的图片验证提出方案的编码效率及编码成功率,同时与DNA喷泉系统进行对比.实验结果说明,所提方案在不影响DNA恢复准确率的情况下,编码尝试次数减少,提升了编码成功率.

