CYP79B2/B3基因在吲哚族硫苷与IAA代谢中的功能研究进展
孙夏莉 臧运祥 徐义康 许双双
摘要:吲哚族硫苷与吲哚-3-乙酸(indole-3-acetic acid,简称IAA)在十字花科植物生长发育和防御过程中发挥了重要作用。CYP79B2/B3基因催化生成的吲哚乙醛肟(IAOx)是这2条代谢途径的共同前体物,因此,阐明CYP79B2/B3基因在吲哚族硫苷与IAA代谢平衡中的作用,对于十字花科植物生长发育调控及病虫害防御研究具有重要意义。本文对吲哚族硫苷合成途径、IAA合成途径及CYP79B2/B3基因在2条代谢途径中的功能研究进展进行了综述。
关键词: CYP79B2/B3;吲哚族硫苷;IAA;代谢途径;功能分化
中图分类号:Q945.18;S184 文献标志码: A
文章编号:1002-1302(2020)14-0050-05
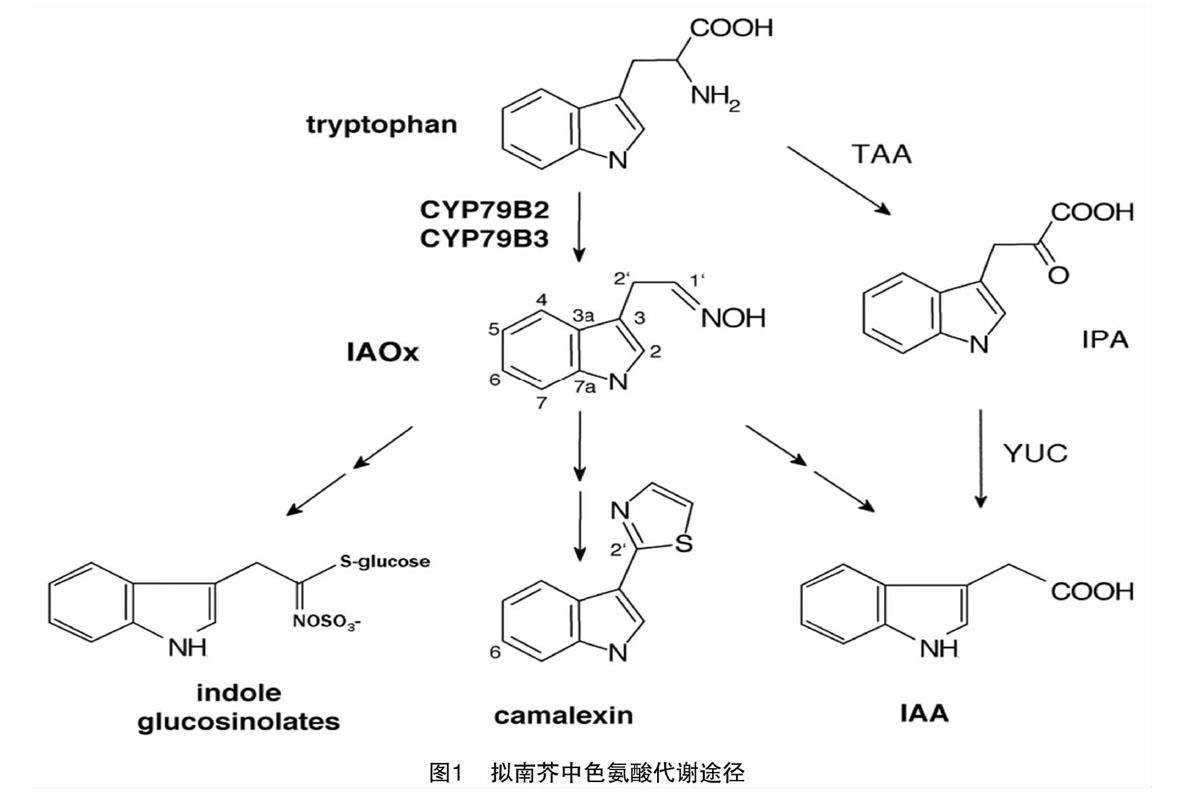
十字花科植物富含一类具有抗病虫功能的重要次生代谢物质——硫代葡萄糖苷(简称硫苷),其水解产物具有较强的抗病虫能力[1]。硫苷由硫代葡萄糖基、磺酸肟和源于不同氨基酸的R侧链3个部分组成。根据侧链基团的种类和性质不同,可将其分为脂肪族、芳香族和吲哚族硫苷3类[2]。吲哚族硫苷与吲哚-3-乙酸(indole-3-acetic acid,简称IAA)的合成具有共同的前体物——吲哚乙醛肟(IAOx),由CYP79B2/B3催化形成(图1)[3]。模式植物拟南芥中有CYP79B2、CYP79B3等2个基因,而白菜等十字花科蔬菜中含有多个拷贝的CYP79B2/B3基因。因此,阐明CYP79B2/B3基因参与吲哚族硫苷与IAA代谢平衡的分子机制,可为十字花科植物生长发育调控及其对害虫、病原菌的防御研究提供理论基础。本文拟围绕吲哚族硫苷与IAA代谢,对CYP79B2/B3基因在2条代谢途径中的功能研究进展进行简要综述,以便为他人开展相关研究提供参考。
1 CYP79B2/B3基因在吲哚族硫苷代谢途径中的功能
1.1 硫苷的作用
硫代葡萄糖苷是源于氨基酸的含氮硫阴离子的植物次级代谢产物,目前自然界中已发现200多种硫代葡萄糖苷,其中模式植物拟南芥中约有40种硫代葡萄糖苷[4-5]。硫苷水解产物,如异硫氰酸酯、腈类和硫代氰酸酯等对草食动物和多种微生物具有抵御作用,近年来又因个别组分的强效抗癌、抗氧化作用而引起植物学、医学、生物学、食品科学等众多学科研究者的关注。前人的研究结果表明,吲哚族硫苷对草食性动物具有威慑作用,并且有助于预防害虫和影响昆虫产卵,是构成植物防御体系不可或缺的一部分[6-8]。此外,吲哚族硫苷水解产物吲哚-3-甲醇可以通过控制Ⅰ相酶与Ⅱ相酶活性来预防前列腺癌、肺癌、膀胱癌、食道癌等,能通过多条途径预防肿瘤发生,促进机体抗氧化物的生成[9-11]。
1.2 吲哚族硫苷的合成途径
吲哚族硫苷的合成底物源于色氨酸。拟南芥中含有4种吲哚族硫苷,分别是吲哚-3-甲基硫苷、1-甲氧基-吲哚-3-甲基硫苷、4-羟基-吲哚-3-甲基硫苷和4-甲氧基-吲哚-3-甲基硫苷[12-14]。
目前拟南芥中吲哚族硫苷的合成途径已经较为清楚。先由分支酸盐经邻氨基苯甲酸酶(AS)催化反应形成邻氨基苯甲酸,经过多步反应后形成吲哚,再经色氨酸合成酶B(TSB)催化形成色氨酸;然后由细胞色素P450酶CYP79B2和CYP79B3催化生成吲哚-3-乙醛肟(IAOx)[15];再然后,在单加氧酶CYP83B1的催化作用下形成不稳定的吲哚-3-甲基酸式硝基化合物,进入吲哚族硫苷合成途径;随后,通过非酶促反应或谷胱甘肽硫转移酶(GSTF9和GSTF10)的作用获得半胱氨酸或由谷胱甘肽(GSH)提供的硫,生成S-吲哚-3-甲基-硫代氧肟,经γ-谷氨酰多肽合成酶GGP1和C-S裂解酶SUR1(C-S lyase)催化生成硫代肟基酸[16-17];之后,在葡萄糖基转移酶UGT74B1的催化作用下发生糖基化而形成脱硫硫苷[18];最后,经硫酸根转移酶AtST5a酸化生成完整的吲哚族硫苷,其核心结构再经过羟基化、甲基化、葡萄糖基化等次级修饰而形成不同的吲哚族硫苷[19]。
硫苷的合成涉及许多因素,转录调控网络复杂。研究发现,MYB类转錄因子(MYB34、MYB51和MYB122)在参与硫苷生物合成的过程中能特异地调控吲哚族硫苷的合成。MYB34被认为是色胺代谢的正向调节因子,在过量表达突变体中发现CYP79B2、CYP79B3和CYP83B1的表达量均显著增加,吲哚族硫苷的含量增加了约10倍[20-21]。当合成基因突变导致吲哚族硫苷缺失时,MYB34的转录水平大幅提升,说明MYB34在植物吲哚族硫苷含量缺失时被激活。机械损伤可以诱导MYB51过量表达,从而调控CYP79B2、CYP79B3、CYP83B1、UTG74B1、At ST5a等表达,引起植物体内吲哚族硫苷大量积累[22]。
MYB34、MYB51和MYB122能促进植物根、芽中吲哚族硫苷的生物合成,MYB122在此过程中可能协助MYB34起辅助作用。在myb34、myb51、myb122三重突变体中没有检测到吲哚族硫苷,说明这3个MYB因子对于吲哚族硫苷的合成是必不可少的[23]。有研究发现,IQD1和OBP2可能是间接调控因子,IQD1对吲哚族硫苷、脂肪族硫苷都具有正向调节作用,OBP2则是通过提高CYP83B1的表达量来增加吲哚族硫苷含量[24-26]。
1.3 CYP79B2/B3基因在代谢途径中的功能
Hull等将拟南芥CYP79B2和CYP79B3基因转入酵母后发现,这2个基因均能将色氨酸转化为吲哚-3-乙醛肟,并且CYP79B2基因的过表达使得拟南芥产生抵抗毒性的色氨酸类似物[27]。Zhao等通过在拟南芥中过表达CYP79B2和CYP79B3基因发现,转基因植株中吲哚族硫苷和生长素含量均显著提高,而拟南芥CYP79B2/B3双突变体中则完全缺失吲哚族硫苷,存在较低含量的生长素,说明这2个酶都能催化吲哚-3-乙醛肟合成生长素和吲哚族硫苷[28]。CYP79B2在氨基酸水平上与CYP79B3有85%的同源性。一般来说,CYP79B2在氨基酸水平上与其他CYP[STBX]79[STBZ]显示出44%~67%的相似性,这可能解释了两者存在功能上的冗余[29]。研究发现,参与色氨酸代谢的CYP79B2、CYP79B3基因在 1 μmol/L 2,4-D上生长的拟南芥中被诱导表达,导致吲哚硫苷积累量增加。在生长素水平高的sur1中,CYP[STBX]79B2、CYP79B3[STBZ]被诱导,从而增加了IAOx产量,并增强了超根性表型,这说明CYP79B2、CYPB3酶通过催化生成IAOx进入吲哚族硫苷和IAA代谢途径[30]。
Mikkelsen等指出,CYP79B2不仅在植物叶片、茎、花和根部可诱导,而且在植物根中的表达量最高;另外关于CYP79B2的GUS报告基因表达研究发现,GUS报告基因在植物幼苗期根系中有较高表达量,在子叶中部分表达,而在老叶中的表达很弱,CYP79B2的表达模式与拟南芥中吲哚族硫苷的分布非常对应[29]。拟南芥cyp79b2突变体中吲哚族硫苷含量比cyp79b3突变体高[28]。以上研究结果说明,与CYP79B2相比,在吲哚族硫苷合成过程中,CYP79B3可能发挥更重要的作用。
2 CYP79B2/B3[STHZ]基因在IAA代谢途径中的功能
2.1 IAA合成途径
吲哚-3-乙酸是植物体内最主要的天然活性生长素,调节、参与植物的生长发育和多种生理过程,特别是植物细胞生长和分裂、植株向地性和向光性的形成、主侧根及下胚轴生长、维管束发育和花器官形成等形态构建过程[31]。按照合成底物的不同,IAA合成途径被分为依赖于色氨酸的途径和不依赖于色氨酸的途径[32-33]。在十字花科植物中,IAA合成主要依赖色氨酸途径,依据中间产物又可分为吲哚丙酮酸(indole-3-pyruvic acid,简称IPA)途径、吲哚乙醛肟(indole-3-acetaldoxime,简称IAOx)途径、色胺(tryptamine)途径和吲哚乙酰胺(indole-3-acetamide,简称IAM)途径4条支路。遗传学和酶学研究发现,TAA和YUC基因在合成植物IAA的过程中是必需的,TAA[STBX]1[STBZ]是吲哚丙酮酸合成途径中的关键基因,其编码蛋白催化色氨酸转化为吲哚-3-丙酮酸,最后生成IAA,YUCCA是色胺途径中的限速酶,可经羟基化形成N-羟基色胺,脱羧后形成IAOx,最后由多步反应转化为生长素。IAA的合成途径复杂,且途径之间存在交叉。IPA途径被证实是拟南芥中主要的IAA生物合成途径[34]。吲哚乙醛肟途径被研究人员普遍认为不是植物体内主要的IAA合成途径,却是十字花科植物防御体系中的一个重要调控环节,与吲哚族硫苷的合成存在相关性。
2.2 CYP79B2/B3基因在吲哚乙醛肟代谢途径中的功能
前人的研究结果表明,CYP[STBX]79B2[STBZ]基因的过表达会使植物体内IAA含量明显提高,但在cyp79B2和cyp79B3双突变体中,植物则表现出幼苗下胚轴变短、长势低矮的表型,体内IAA含量较低,合成速率降低[35]。拟南芥中吲哚乙醛肟途径中IAA的生物合成过程已经明确,即CYP79B2/B3蛋白将色氨酸转化为吲哚-3-乙醛肟,再经过吲哚-3-乙醛生成IAA,或者生成吲哚-3-乙腈,在腈水解酶作用下生成IAA[21,36]。
此外,研究还发现CYP79B2/B3双突变体在 26 ℃ 条件下时IAA活性显著下降,突变体植株呈现生长素缺乏表型[37];当温度升高至29 ℃时,在剥离的胚轴中IAA的合成速率大幅度提高。这说明在拟南芥中,吲哚乙醛肟途径的IAA合成还受到特定温度的影响[38]。
有研究发现,将拟南芥CYP79B2基因与CYP79B3基因分别转入大白菜中后,在转CYP79B2基因的试验中由于IAA的过量积累而未能获得转基因植株,而在转CYP[STBX]79B3[STBZ]的试验中获得了转基因植株,但表现出IAA过量合成的性状[39-41]。以上结果说明,在IAA合成的过程中,与CYP79B3基因相比,CYP79B2基因可能发挥更重要的作用。而当这2个基因与吲哚族硫苷合成途径中的CYP83B1基因一起被转入大白菜中时,获得的转基因植株积累的吲哚族硫苷和表现型与对照相似。以上结果表明,CYP83B1基因的高效表达可以将大量IAOx引入吲哚族硫苷合成途径,从而避免IAA的过量合成。
3 吲哚族硫苷与IAA代谢的关系
IAA的另一条合成途径是色氨酸经由TAAs(色氨酸氨基转移酶)与YUCCAs催化形成[34,40-41]。它与CYP79B2/CYP79B3催化的IAA合成途径互补,所以在拟南芥cyp[STBX]79b2/cyp79b3[STBZ]双突变体中仍能检测到较高含量的IAA。然而,在拟南芥cyp[STBX]79b2/cyp79b3[STBZ]双突变体中检测不到吲哚族硫苷或亚麻荠素,表明由CYP79B2/B3催化的吲哚族硫苷或亚麻荠素的合成途径是唯一的[28,42]。
吲哚族硫苷合成后大部分储存在植物细胞组织液泡中,由于不具有挥发性,又与黑芥子酶存在空间上的隔离,因此结构较为稳定。当遭遇动物啃食或机械外力造成细胞破碎时,会与特定蛋白体中的黑芥子酶发生不可逆的水解反应,产生不同种类的生物活性物质。温度、压强、酸碱性和时间等多个因子对硫苷降解都有影响[43-44]。有研究发现,在酸性条件下(如pH值为2~5时)且存在亚铁离子时,主要产生腈类物质,吲哚-3-乙腈经腈水解酶水解反应,最后生成吲哚-3-乙酸。
在生物合成途徑中,IAOx是主要代谢产物和次级代谢产物的重要分支点,其下游基因(CYP83B1、CS-裂合酶基因或UGT74B1基因)的丢失会导致IAA过量产生[45-46]。目前在拟南芥中已经鉴定出生长素含量增加的突变体,并且发现sur1可以编码C-S裂解酶,参与吲哚族硫苷的合成[39]。而与之相似的sur2则可编码CYP83B1。在sur2cyp83B1突变体中,IAOx转化吲哚族硫苷的途径被阻断,导致IAOx更多地向IAA转化[47]。以上分析说明,在吲哚族硫苷和IAA的合成过程中存在竞争反应底物的关系。近年来的生化分析结果表明,在拟南芥中大部分吲哚-3-乙醛肟是在CYP[STBX]79[STBZ]B途径中产生的,推测吲哚-3-乙醛肟的合成与转变为生长素的过程是耦合在一起的,而与吲哚-3-乙醛肟转变为硫代葡萄糖苷的过程不是耦合在一起的[48]。色胺途径产生的色胺以及植物体内形成的N-羟基色胺可抑制CYP83B1酶的活性,降低吲哚族硫苷的生成量,于是吲哚-3-乙醛肟参与到生长素合成过程中。吲哚族硫苷在Fe2+存在的条件下可水解生成吲哚-3-乙腈,经腈水合酶催化生成IAA,并诱导CYP83B1/sur2和UGT74B1基因的表达,说明IAA与吲哚族硫苷合成有协同调节作用[49]。
4 展望
亞麻荠素只存在于拟南芥、荠菜等少数十字花科植物中,在大白菜植株中尚未检测到亚麻荠素。因此,大白菜可以作为研究吲哚族硫苷与IAA代谢平衡调控的模式植物。以吲哚族硫苷与IAA代谢的共同前体物吲哚乙醛肟为切入点,阐明CYP79B2/B3基因参与吲哚族硫苷(参与植物的病虫害防御反应)与IAA(调控植物的生长发育过程)代谢平衡的分子机制,可为十字花科植物生长发育调控及其对害虫、病原菌的防御研究提供理论基础。
参考文献:
[1]Grubb C D,Abel S. Glucosinolate metabolism and its control[J]. Trends in Plant Science,2006,11(2):89-100.
[2]Klopsch R,Witzel K,Brner A,et al. Metabolic profiling of glucosinolates and their hydrolysis products in a germplasm collection of Brassica rapa turnips[J]. Food Research International,2017,100(3):392-403.
[3]Bende J,Celenza J L. Indolic glucosinolates at the crossroads of tryptophan metabolism[J]. Phytochemistry Reviews,2009,8(1):25-37.
[4]Ishida M,Hara M,Fukino N,et al. Glucosinolate metabolism,functionality and breeding for the improvement of Brassicaceae vegetables[J]. Breeding Science,2014,64(1):48-59.
[5]Kliebenstein D J,Kroymann J,Brown P,et al. Genetic control of natural variation in Arabidopsis glucosinolate accumulation[J]. Plant Physiology,2001,126(2):811-825.
[6]Kim J H,Jander G. Myzus persicae (green peach aphid) feeding on Arabidopsis induces the formation of a repellent indole glucosinolate[J]. The Plant Journal,2007,49(6):1008-1019.
[7]Bednarek P,Pislewska-Bednarek M,Svatos A,et al. A glucosinolate metabolism pathway in living plant cells mediates broad-spectrum antifungal defense[J]. Science,2009,323(5910):101-106.
[8]Clay N K,Adio A M,Denoux C,et al.Glucosinolate metabolism required for an Arabidopsis innate immune response[J]. Science,2009,323(5910):95-101.
[9]Murillo G,Mehta R G. Cruciferous vegetables and cancer prevention[J]. Nutrition & Cancer,2001,41(1/2):17-28.
[10]Mithen R F,Dekker M,Verkerk R,et al.Review:the nutritional significance,biosynthesis and bioavailability of glucosinolates in human food[J]. Journal of the Science of Food & Agriculture,2000,80(7):967-984.
[11]Fahey J W,Zhang Y,Talalay P,et al. Broccoli sprouts:an exceptionally rich source of inducers of enzymes that protect against chemical[J]. PNAS,1997,94(19):10367-10372.
[12]Chen I,Mc Dougal A,Wang F,et al. Aryl hydrocarbon receptor rmediated antiestrogenic and antitumorigenic activity of diindolylmethane[J]. Carcinogenesis,1998,19:1631-1639.
[13]Kliebenstein D J,Gershenzon J,Mitchell-Olds T,et al.Comparative quantitative trait loci mapping of aliphatic,indolic and benzylic glucosinolate production in Arabidopsis thaliana leaves and seeds[J]. Genetics,2001,159(1):359-370.
[14]Brown P D,Tokuhisa J G,Reichelt M,et al.Variation of glucosinolate accumulation among different organs and developmental stages of Arabidopsis thaliana[J]. Phytochemistry,2003,62(3):471-481.
[15]李一蒙,陳亚州,阎秀峰. 植物中的吲哚族芥子油苷与生长素代谢途径的关系[J]. 植物生理学报,2009,45(2):195-201.
[16]Mikkelsen M D,Naur P,Halkier B A,et al. Arabidopsis mutants in the C-S lyase of glucosinolate biosynthesis establish a critical role for indole-3-acetaldoxime in auxin homeostasis[J]. Plant Journal,2004,37(5):770-777.
[17]Bak S,Feyereisen R.The involvement of two P450 enzymes CYP83B1 and CYP83A1 in auxin homeostasis and glucosinolate biosynthesis[J]. Plant Physiology,2001,127(1):108-118.
[18]Petersen B L,Andréasson E,Bak S,et al. Characterization of transgenic Arabidopsis thaliana with metabolically engineered high levels of p-hydroxybenzylglucosinolate[J]. Planta,2001,212(4):612-618.
[19]Snderby I E,M Burow,Rowe H C,et al. A complex interplay of three R2R3-MYB transcription factors determines the profile of aliphatic glucosinolates in Arabidopsis[J]. Plant Physiology,2010,153(1):348-368.
[20]Bender J,Fink G R.A Myb homologue,ATR1,activates tryptophan gene expression in Arabidopsis[J]. PNAS,1998,95(10):5655-5660.
[21]Celenza J L,Quiel J A,Smolen G A,et al. The Arabidopsis ATR1 Myb transcription factor controls indolic glucosinolate homeostasis[J]. Plant Physiology,2005,137(1):253-262.
[22]Gigolashvili T,Berger B,Mock H,et al.The transcription factor HIG1/MYB51 regulates indolic glucosinolate biosynthesis in Arabidopsis thaliana[J]. Plant Journal,2007,50(5):886-901.
[23]Hai D U,Ran F,Liu J,et al. Genome-wide expression analysis of glucosinolate biosynthetic genes in Arabidopsis across diverse tissues and stresses induction[J]. Scientia Acricultura Sinica,2016,49(15):2879-2897.
[24]Schweizer F,Fernándezcalvo P,Zander M,et al. Arabidopsis basic helix-loop-helix transcription factors MYC2,MYC3,and MYC4 regulate glucosinolate biosynthesis,insect performance,and feeding behavior[J]. Plant Cell,2013,25(8):3117-3132.
[25]Skirycz A,Reichelt M,Burow M,et al.DOF transcription factor AtDof1.1 (OBP2) is part of a regulatory network controlling glucosinolate biosynthesis in Arabidopsis[J]. Plant Journal,2006,47(1):10-24.
[26]Levy M,Wang Q,Kaspi R,et al. Arabidopsis IQD1,a novel calmodulin-binding nuclear protein,stimulates glucosinolate accumulation and plant defense[J]. Plant Journal,2005,43(1):79-96.
[27]Hull A K,Vij R,Celenza J L,et al. Arabidopsis cytochrome P450s that catalyze the first step of tryptophan-dependent indole-3-acetic acid biosynthesis[J]. PNAS,2000,97(5):2379-2384.
[28]Zhao Y,Hull A K,Gupta N R,et al. Trp-dependent auxin biosynthesis in Arabidopsis:involvement of cytochrome P450s CYP79B2 and CYP79B3[J]. Genes Dev,2002,16(23):3100-3112.
[29]Mikkelsen M D,Hansen C H,Wittstock U,et al. Cytochrome P450 CYP79B2 from Arabidopsis catalyzes the conversion of tryptophan to indole-3-acetaldoxime,a precursor of indole glucosinolates and indole-3-acetic acid[J]. Journal of Biological Chemistry,2000,275(43):33712-33717.
[30]Mikkelsen M D,Halkier B A. Modulation of CYP[STBX]79[STBZ] genes and glucosinolate profiles in Arabidopsis by defense signaling pathways[J]. Plant Physiology,2003,131(1):298-308.
[31]Bandurski R S,Cohen J D,Slovin J P,et al. The plant hormones:auxin biosynthesis and metabolism[M]. Berlin,Heidelberg:Springer-Verlag,1995:39-65.
[32]Bartel B,Leclere S,Magidin M,et al. Inputs to the active indole-3-acetic acid pool:de novo synthesis,conjugate hydrolysis,and indole-3-butyric acid β-oxidation[J]. Journal of Plant Growth Regulation,2001,20(3):198-216.
[33]Normanly J,Bartelt B. Redundancy as a way of life:IAA metabolism[J]. Current Opinion in Plant Biology,1999,2(3):207-213.
[34]Mashiguchi K,Tanaka K,Sakai T,et al. The main auxin biosynthesis pathway in Arabidopsis[J]. PNAS,2011,108(45):18512-18517.
[35]Quittenden L J,Davies N W,Smith J A,et al. Auxin biosynthesis in pea:characterization of the tryptamine pathway[J]. Plant Physiology,2009,151(3):1130-1138.
[36]Tam Y Y,Normanly J. Overexpression of a bacterial indole-3-acetyl-l-aspartic acid hydrolase in Arabidopsis thaliana[J]. Physiologia Plantarum,2010,115(4):513-522.
[37]Nemoto K,Hara M,Suzuki M,et al. The NtAMI[STBX]1[STBZ] gene functions in cell division oftobacco BY-2 cells in the presence of indole-3-acetamide[J]. Febs Letters,2009,583(2):487-492.
[38]Pollmann S,Düchting P,Weiler E W. Tryptophan-dependent indole-3-acetic acid biosynthesis by‘IAA-synthase proceeds via indole-3-acetamide[J]. Phytochemistry,2009,70(4):523-531.
[39]Tao Y,Ferrer J L,Ljung K,et al. Rapid synthesis of auxin via a new tryptophan-dependent pathway is required for shade avoidance in plants[J]. Cell,2008,133(1):164-176.
[40]Won C,Shen X,Mashiguchi K,et al. Conversion of tryptophan to indole-3-acetic acid by TRYPTOPHAN AMINOTRANSFERASES OF ARABIDOPSIS and YUCCAs in Arabidopsis[J]. PNAS,2011,108(45):18518-18523.
[41]Zhao Y D. Auxin biosynthesis:a simple two-step pathway converts tryptophan to indole-3-acetic acid in plants[J]. Molecular Plant,2012,5(2):334-338.
[42]Glawischnig E,Hansen B G,Olsen C E,et al. Camalexin is synthesized from indole-3-acetaldoxime,a key branching point between primary and secondary metabolism in Arabidopsis[J]. PNAS,2004,101(21):8245-8250.
[43]Wittstock U,Halkier B A. Glucosinolate research in the Arabidopsis era[J]. Trends in Plant Science,2002,7(6):263-270.
[44]陳凌云,余芳洁,陈君杰,等. 植物硫代葡萄糖苷二次修饰及调控的研究进展[J]. 江苏农业科学,2019,47(4):22-29.
[45]Bttcher C,Westphal L,Schmotz C,et al. The multifunctional enzyme CYP71B15 (PHYTOALEXIN DEFICIENT3) converts cysteine-indole-3-acetonitrile to camalexin in the indole-3-acetonitrile metabolic network of Arabidopsis thaliana[J]. Plant Cell,2009,21(6):1830-1845.
[46]Barlier I,Kowalczyk M,Marchant A,et al. The SUR[STBX]2[STBZ] gene of Arabidopsis thaliana encodes the cytochrome P450 CYP83B1,a modulator of auxin homeostasis[J]. PNAS,2000,97(26):14819-14824.
[47]张海峰,袁 晶,汪俏梅. 植物激素与芥子油苷在生物合成上的相互作用[J]. 细胞生物学杂志,2005,27(4):423-426.
[48]王家利,刘冬成,郭小丽,等. 生长素合成途径的研究进展[J]. 植物学报,2012,47(3):292-301.
[49]阮 颖,周朴华,刘春林. 硫代葡萄糖苷合成核心途径与植物生长素微调[J]. 生命科学研究,2006(增刊3):25-29.
收稿日期:2019-08-20
基金项目:国家自然科学基金(编号:31572130、31000916);浙江省自然科学基金(编号:LY14C150005、Y3090538)。
作者简介:孙夏莉(1990—),女,浙江舟山人,硕士研究生,研究方向为蔬菜品质调控与分子机理。E-mail:1367201782@qq.com。
通信作者:臧运祥,博士,教授,硕士生导师,研究方向为园艺作物品质生理、生物技术与分子育种。E-mail:yxzang@zafu.edu.cn。

