蔓花生AP2基因家族的生物信息学分析
王玲 刘晓伟 江纳 付春
摘要:AP2作为一类植物转录因子,在花的发育和营养器官的形成过程中都起着非常重要的作用。旨在对蔓花生(Arachis duranensis)AP2蛋白的理化性质及分子进化关系等进行详细的生物信息学分析。结果表明,蔓花生AP2基因家族成员编码蛋白亚细胞定位预测均在细胞核中,有11个成员的理论等电点小于7,推测该家族蛋白多为酸性蛋白,有8个成员存在2个保守结构域;该家族成员均无信号肽;除了Aradu.SE6Q0與Aradu.42K79成员中存在跨膜现象外,其他成员鲜少出现跨膜现象。亲疏水性预测结果表明,蔓花生AP2基因家族编码蛋白的亲疏水性系数均小于0,推测其为亲水蛋白;磷酸化位点主要集中在丝氨酸上;其二级结构以无规则卷曲和α-螺旋为主。研究还得出,蔓花生AP2基因家族编码蛋白的主要保守基序是Motif1、Motif2和Motif4;Motif3、Motif4都含有YRG结构域,Motif5含有RAYD结构域;构建的系统发育树也有力地说明了AP2基因家族的进化程度。
关键词:蔓花生;AP2基因;基因家族;生物信息学
中图分类号: S541+.901 文献标志码: A
文章编号:1002-1302(2020)14-0065-13
蔓花生(Arachis duranensis),别称遍地黄金,是豆科蝶形花亚科蔓花生属的多年生宿根草本植物,原产于亚洲热带及南美洲地区,其根系发达,抗逆性强,病虫害少,是动物的良好牧草[1-6]。蔓花生有一定的耐热耐旱性、抗寒性、耐阴性、耐贫瘠性[2-3,7-13],对有害气体有较强的抗性[14-15]。APETALA2(AP2)是一类植物转录因子,其结构首次被发现于拟南芥中,并证实在花的发育过程中和营养器官中均有表达[16-20]。该类因子由60~70个氨基酸残基组成,并参与DNA的结合过程[18,21-22]。AP2基因家族是AP[STBX]2[STBZ]/ERF超级基因家族下的1个分支,其家族蛋白包含2个重复的AP2结构域,即AP2-R1和AP2-R2,根据2个结构域是否含有插入序列,将AP2基因家族分为ANT组、euAP2组,此外,AP2基因家族还含有较长的识别序列GCAC(A/G)N(A/T)TCCC(A/G)ANG(C/T)元件[23-28]。AP[STBX]2/ERF超级基因家族中除AP2[STBZ]家族外,还包括ERF家族和RAV家族,ERF家族编码蛋白含有1个AP2结构域,RAV家族编码蛋白含有1个AP2结构域和1个B3结构域,其中B3结构域在其他植物的特异转录因子中是1个保守的DNA结合结构域[21]。AP[STBX]2[STBZ]/ERF亚家族成员通过与CC、CRT/DRE、JERE或VWRE等作用元件相互作用而诱导下游基因的表达[29-32]。AP2参与花发育、小穗分生组织的形成、叶表皮细胞识别和胚胎发育等过程的调控[33]以及花器官的识别,如花瓣、萼片[34-38]。在AP[STBX]2[STBZ]家族编码蛋白中,拟南芥蛋白AP213(AP2)、AP2-14(TOE1)、AP2-8(AIL6)、AP2-12(AIL7)含有EAR-like基序[17]。AP2的CAP2在鹰嘴豆(Cicer arietinum Linn.)中具有抗盐、抗干旱胁迫的功能[39],EgAP2-1在油棕(Elaeis guineensis Jacq.)中调控合子和体细胞胚的发育[40]。AP2/ERF转录因子基因家族的结构和进化在拟南芥、水稻和大豆中是相对保守的[41],许多亚家族和亚类存在于这3个物种中,这说明绝大多数AP2/ERF亚家族成员存在于物种分化之前[18]。AP[STBX]2[STBZ]转录基因在花生种子发育的4个时期的表达存在差异[33],其通过影响胚、胚乳和种皮的发育来控制种子的大小,同时通过对糖代谢的影响来影响种子的质量[18,39,42]。目前尚未见关于蔓花生AP2基因家族理化特性及其进化关系的报道,因此,本研究以蔓花生AP2蛋白序列为材料,利用生物信息学工具对蔓花生AP2基因家族的理化特性、保守基序和系统进化关系进行详细研究,以期为进一步研究和揭示AP2基因家族在蔓花生生长发育、基因调控中的作用提供参考依据。
1 材料与方法
1.1 材料
从PlantTFDB数据库[43](http://planttfdb.cbi.pku.edu.cn/)中下载蔓花生、野花生、花生3个落花生属和拟南芥的AP2蛋白序列,以及二穗短柄草(Brachypodium distachyon)AP2家族成员Bradi2g37800.13.p、鹰嘴豆AP2家族成员XP 004506004.1、黄花蒿(Artemisia annua)AP2家族成员Aan013559、木豆(Cajanus cajan)AP2家族成员C.cajan 21449、大豆(Glycine max)AP2家族成员Glyma.12G073300.1.p、绿豆(Vigna radiata)AP2家族成员Vradi03g03260.1、芸豆(Phaseolus vulgaris)AP2家族成员Phvul.011G071100.1、向日葵(Helianthus annuus)AP2家族成员Han007343、芝麻(Sesamum indicum)AP2家族成员XP 011089852、蓖麻(Ricinus communis)AP2家族成员28752.m000339、玉米(Zea mays)AP2家族成员GRMZM2G700665 P03、印度水稻(Oryza sativa ssp. indica)AP2家族成员BGIOSGA018911-PA、小麦(Triticum urartu)AP2家族成员TRIUR3 14819-P1的蛋白序列。
1.2 方法
1.2.1 蔓花生AP2基因家族理化性质的分析及亚细胞定位 利用在线程序PrtoParam[44-47](https://web.expasy.org/protparam/)统计蔓花生AP2基因家族的编码氨基酸数量、理论等电点、分子量、带正电荷氨基酸残基数、带负电荷氨基酸残基数和脂肪族氨基酸指数及总平均亲疏水性等理化性质信息,并在PlantTFDB数据库中以拟南芥基因序列为探针,进行BLAST对比。利用在线软件CELLOv2.5(http://cello.life.nctu.edu.tw/)[48]对17条蔓花生的AP2蛋白序列进行亚细胞定位。
1.2.2 氨基酸保守结构域的预测分析 利用NCBI Consered Domains[49](https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)对蔓花生AP2基因家族编码蛋白进行氨基酸保守结构域的预测,对该家族各个成员的保守结构域数量和位置进行统计。
1.2.3 氨基酸信号肽及跨膜结构的预测分析 使用SignalP 3.0 Server[44,46](http://www.cbs.dtu.dk/services/SignalP-3.0/)预测蔓花生AP2基因家族的17个成员的信号肽位置;在TMHMM Severv.2.0[44,46-47,49](http://www.cbs.dtu.dk/services/TMHMM/)在线程序中输入AP2蛋白序列,分析该蛋白是否存在跨膜现象以及是否含有跨膜结构域。
1.2.4 氨基酸亲疏水性的预测分析 利用在线工具Protscale[44](https://web.expasy.org/protscale/)对蔓花生AP2蛋白进行亲疏水性预测分析,对其各个成员的最大亲疏水性所在位点和分值进行统计。
1.2.5 磷酸化位点的预测分析 使用在线工具NetPhos 3.1 Serve[44,47](http://www.cbs.dtu.dk/services/NetPhos/)对AP2蛋白序列的潜在磷酸化位点进行预测分析,找出各个成员最可能的磷酸化位点及其磷酸化评分值,以及可能的磷酸化位点数量与占比。
1.2.6 蛋白质二级结构和三级结构的预测分析 使用SOPMA[44](https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)程序对蔓花生AP2蛋白的二级结构进行预测分析,对该家族各个成员的二级结构类型在各成员中的占比和分布进行统计;借助Phyre2网站[44,46-47,50](http://www.sbg.bio.ic.ac.uk/phyre2)对AP2蛋白进行三级结构的预测。
1.2.7 蔓花生AP2基因家族的蛋白基序分析 利用MEME在线工具[50-51]对蔓花生AP2基因家族成员进行motif查找,设置查找的motif数量为10个,其余参数设为默认值。
1.2.8 系统进化树的构建 用MEGA软件[44-46,49-50]分别构建蔓花生AP2基因家族的系统发育进化树,落花生属蔓花生(Arachis duranensis)、野花生(Arachis ipaensis)和花生(Arachis hypogaea L.)的系统发育进化树与蔓花生AP2基因家族、拟南芥AP2基因家族的系统发育进化树,以及蔓花生AP2家族成员Aradu.387PF、二穗短柄草AP2家族成员Bradi2g37800.13.p、拟南芥AP2家族成员AT2G28550.3、鹰嘴豆AP2家族成员XP 004506004.1、黄花蒿AP2家族成员Aan013559、木豆AP2家族成员C.cajan 21449、大豆AP2家族成员Glyma.12G073300.1.p、绿豆AP2家族成员Vradi03g03260.1、芸豆AP2家族成员Phvul.011G071100.1、向日葵AP2家族成员Han007343、芝麻AP2家族成员XP 011089852、蓖麻AP2家族成员28752.m000339、玉米AP2家族成员GRMZM2G700665 P03、印度水稻AP2家族成员BGIOSGA018911-PA、小麦AP2家族成员TRIUR3_14819-P1的系统发育进化树。
2 结果与分析
2.1 蔓花生AP2基因家族编码蛋白理化性质的分析及亚细胞定位
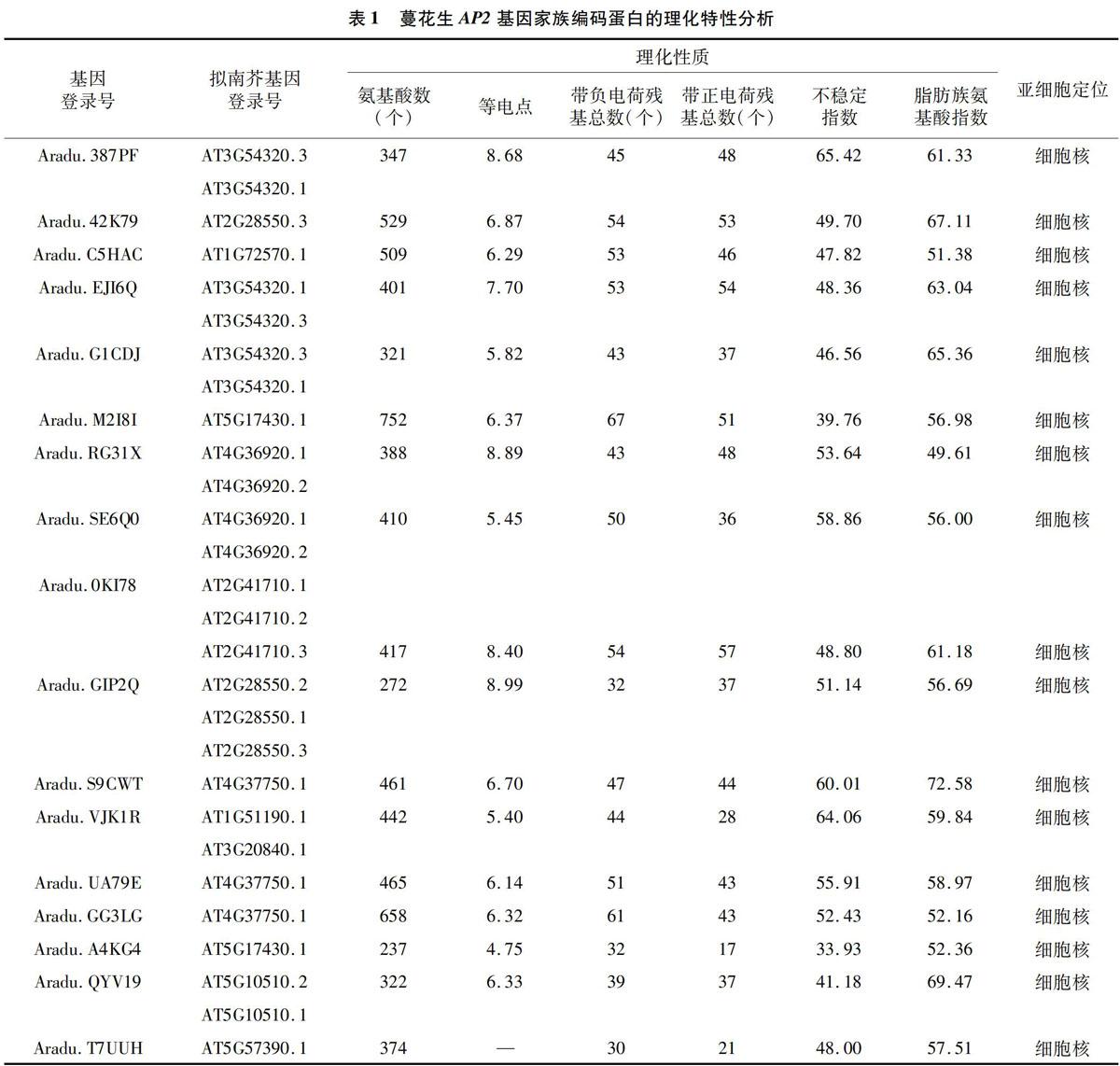
对蔓花生AP2基因家族成员编码蛋白的理化性质进行分析发现,该家族各蛋白的氨基酸数量范围为237~752个,氨基酸数量最多的是 Aradu.M2I8I 家族成员(752个),氨基酸数量最少的是Aradu.A4KG4家族成员(237个);蛋白质的理论等电点除Aradu.T7UUH成员尚存在争议外,其余16个成员的等电点都能分析得出,其中,除 Aradu.387PF、Aradu.RG31X、Aradu.0KI78和Aradu.GIP2Q成員的等电点大于8,Aradu.EJI6Q成员的等电点为7.70外,其余11个成员的等电点均小于7,说明该家族含有大量酸性氨基酸;蛋白不稳定指数分析结果显示,除Aradu.M2I8I、Aradu.A4KG4 这2个成员的不稳定指数小于40外,其余15个成员的不稳定指数均大于40,说明该家族成员多为不稳定蛋白;该家族的脂肪族氨基酸指数分布范围为4961(Aradu.RG31X)~7258(Aradu.S9CWT),脂肪族氨基酸指数反映了蛋白质的热稳定性[52],由此说明该家族蛋白质间的热稳定性差异较大。由表1中AP2基因的亚细胞定位结果看出,蔓花生AP2蛋白成员均分布在细胞核中。在蔓花生AP2基因家族成员中,可按对应氨基酸组成分为Ⅲ类,其中第Ⅰ类以丝氨酸占比最高,第Ⅱ类的丝氨酸和天冬酰胺占比一样,第Ⅲ类以天冬酰胺占比最高(图1)。
2.2 氨基酸保守结构域分析
对蔓花生AP2基因家族编码蛋白的氨基酸保守结构域进行预测分析,结果显示,Aradu.387PF、Aradu.42K79、Aradu.EJI6Q、Aradu.G1CDJ、Aradu.M2I8I、Aradu.RG31X、Aradu.GIP2Q、Aradu.SE6Q0这8个成员均存在2个保守结构域,其余9个成员均含有1个保守结构域。由表2、图2可以看出,蔓花生AP2基因家族编码蛋白成员保守结构域所含氨基酸数量大于100个的有7个;所含氨基酸数量最多的保守结构域在Aradu.M2I8I成员上,共有135个氨基酸;所含氨基酸数量最少的保守结构域在Aradu.A4KG4成员上,共有59个氨基酸。
2.3 氨基酸信号肽及跨膜结构分析
对蔓花生AP2基因家族编码蛋白各氨基酸信号肽进行预测,分析发现,蔓花生AP2基因家族编码蛋白的17个成员均无信号肽,因此推测蔓花生AP2基因家族编码蛋白成员均为非分泌蛋白。
对蔓花生AP2基因家族编码蛋白成员进行跨膜结构域预测分析发现,只有Aradu.SE6Q0和Aradu.42K79这2个成员存在跨膜现象。成员Aradu.SE6Q0在第236个氨基酸与第258个氨基酸之间存在跨膜现象,但并不形成跨膜结构域;成员Aradu.42K79在第349个氨基酸与第370个氨基酸之间存在跨膜现象,也并不存在跨膜结构域。经SignalP3.0 Server程序分析可知,蔓花生AP2基因家族编码蛋白成员均不存在跨膜结构区域,因此推断该家族蛋白为非跨膜蛋白。
2.4 氨基酸亲疏水性预测分析
对蔓花生AP2基因家族编码蛋白的17个成员进行亲疏水性预测分析发现,该家族成员Aradu.387PF的第17位精氨酸的亲水性最强,为-3.756;成员Aradu.42K79的第262位甘氨酸的疏水性最强,为3.789;总平均亲疏水性均小于0,说明AP2基因家族编码蛋白成员均为亲水性蛋白。最高亲水性值为-1.004,在该基因家族编码蛋白成员Aradu.A4KG4上;最低亲水性值为-0.449,在成员Aradu.T7UUH上(图3)。
2.5 磷酸化位点分析
对蔓花生AP2基因家族编码蛋白的潜在磷酸化位点进行预测分析发现,在该家族同一成员之间, 丝氨酸含量总是最高的,其次是苏氨酸,最低的是酪氨酸;丝氨酸最可能的磷酸化位点数最多,且磷酸化评分值往往在0.997~0.998之间,只有Aradu.A4KG4、Aradu.S9CWT的磷酸化评分值低于0.997,分别为0.925、0.994;苏氨酸、酪氨酸最可能的磷酸化位点数都只有1个,且磷酸化评分值都低于0.990;在蔓花生AP2蛋白家族成员中,Aradu.387PF具有的潜在磷酸化位点数最多,磷酸化位点数占该家族氨基酸总数的14.99%;成员Aradu.A4KG4含有的潜在磷酸化位点数最少,磷酸化位点数占该家族氨基酸总数的8.02%;在AP2蛋白家族成员中,成员Aradu.SE6Q0的丝氨酸、苏氨酸含量都是较高的,占比分别为8.65%、5.19%;成员Aradu.A4KG4的丝氨酸、苏氨酸含量都是较低的,占比分别为3.38%、2.53%,酪氨酸含量最高,占比为2.11%;成员Aradu.42K79的酪氨酸含量最低,占比为0.19%(图4)。
2.6 蛋白质二级结构和三级结构的预测分析
对蔓花生AP2蛋白家族成员进行二级结构分析,结果显示,17个蔓花生AP2基因家族编码蛋白成员的二级结构均表现为无规则卷曲最多,β-转角最少,有14个成员的α-螺旋数>延伸链数,Aradu.GG3LG、Aradu.QYV19和Aradu.T7UUH这3个成员的延伸链数>α-螺旋数。总体而言,蔓花生AP2基因家族编码蛋白的二级结构以无规则卷曲和α-螺旋为主,而延伸链和β-转角则分散在各个氨基酸链中(图5)。
对蔓花生AP2基因家族编码蛋白的17个成员进行三级结构预测并对预测结果进行比对,根据各成员三级结构是否相似,可将该家族分为Ⅶ大类,分别用Ⅰ、Ⅱ、Ⅲ、Ⅳ、Ⅴ、Ⅵ、Ⅶ类表示。由图6可以看出,第Ⅳ类所含成员数最多,一共有5个,分别是Aradu.RG31X、Aradu.M2I8I、Aradu.SE6Q0、Aradu.G1CDJ和Aradu.42K79;其次是第Ⅰ类,含有Aradu.0KI78、Aradu.387PF和Aradu.VJK1R这3个成员;第Ⅱ、Ⅲ、Ⅴ和Ⅵ类均含有2个成员,Aradu.GIP2Q、Aradu.QYV19属于第Ⅱ类成员,第Ⅲ类成员有Aradu.A4KG4和Aradu.GG3LG,第Ⅴ类成员有Aradu.C5HAC和Aradu.UA79E,第Ⅵ类成员有Aradu.T7UUH和Aradu.S9CWT;第Ⅶ类只有1个成员,为Aradu.GIP2Q。
2.7 蔓花生AP2基因家族编码蛋白基序分析
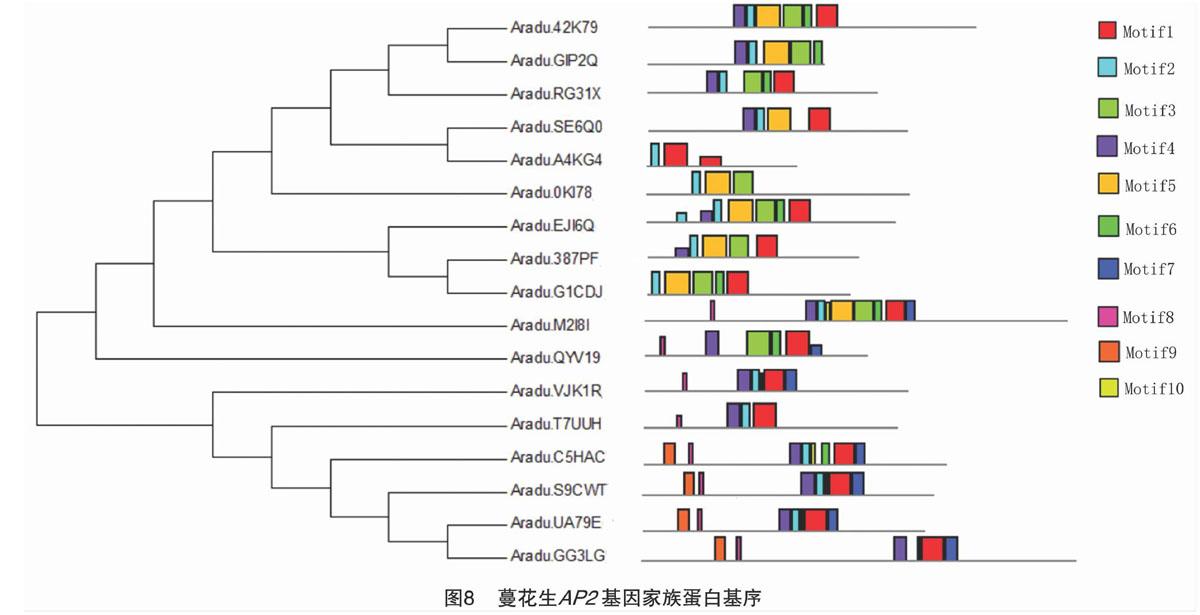
对蔓花生AP2基因家族编码蛋白成员进行Motif分析发现,Motif3、Motif4都含有1个共同的结构域,即YRG;Motif5含有RAYD结构域(图7)。YRG结构域有利于DNA的结合,RAYD结构域主要调节转录因子与DRE顺式元件结合[53],说明蔓花生AP2基因家族编码蛋白的Motif3、Motif4和Motif5属于AP2结构域。根据蔓花生AP2蛋白序列所含Motif类型的不同,将该家族分为Ⅴ大类,第Ⅰ类主要含有Motif1、Motif4和Motif8,其成员有Aradu.GG3LG、Aradu.UA79E、Aradu.S9CWT、Aradu.C5HAC、Aradu.T7UUH 和Aradu.VJK1R;第Ⅱ类包括成员Aradu.M2I8I、Aradu.QYV19,都含有Motif1、Motif3、Motif4、Motif6、Motif7、Motif8;第Ⅲ类主要含有Motif1、Motif2、Motif3和Motif5,成员有Aradu.EJI6Q、Aradu.387PF和Aradu.G1CDJ;第Ⅳ类主要含有Motif1和Motif2,成员有Aradu.A4KG4和Aradu.SE6Q0;第Ⅴ类主要含有Motif2、Motif3、Motif4、Motif5、Motif6,成員有Aradu.42K79和Aradu.GIP2Q。在蔓花生AP2蛋白序列中,含有2个AP2保守结构域的蛋白质序列均含有Motif5,在蔓花生AP2基因家族编码蛋白的17个成员中,除Aradu.GIP2Q、Aradu.A4KG4不含有Motif1外,其他15个成员均含有Motif1;Motif2在该家族的15个成员中均存在,只有Aradu.QYV19、Aradu.GG3LG中不存在;有9个成员含有Motif3,14个成员含有Motif4,8个成员含有Motif5,说明Motif1、Motif2和Motif4是蔓花生AP2基因家族编码蛋白的主要保守基序(图8)。
2.8 系统进化关系分析
通过对蔓花生AP2基因家族编码蛋白系统发育进化树的分组比对发现,该家族成员Aradu.GG3LG、Aradu.UA79E、Aradu.S9CWT、Aradu.C5HAC、Aradu.T7UUH和Aradu.VJK1R的亲缘关系比该家族其他成员的亲缘关系近。其中Aradu.GG3LG和Aradu.UA79E、Aradu.387PF和Aradu.G1CDJ、Aradu.A4KG4和Aradu.SE6Q0、Aradu.42K79和Aradu.GIP2Q这4组的组内成员亲缘关系更近(图8),这与以拟南芥为探针的BLAST比对结果基本吻合(表1)。
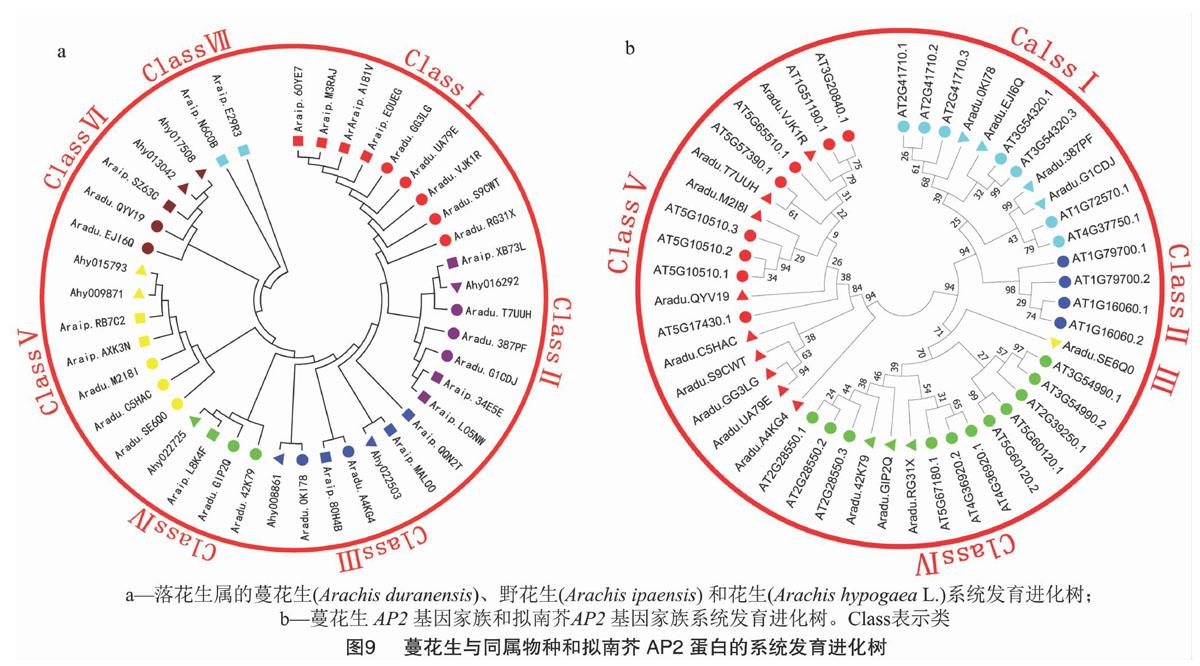
落花生属3个物种的系统进化关系可以分为Ⅶ大类,第Ⅰ类有9个成员,主要是蔓花生AP2基因家族编码蛋白的5个成员和野花生的4个成员;第Ⅱ类有7个成员,主要是野花生AP2基因家族编码蛋白的3个成员,蔓花生AP2基因家族编码蛋白的3个成员及花生AP2基因家族编码蛋白的成员Ahy016292,其中Araip.XB73L与Ahy016292的亲缘关系相对较近;第Ⅲ类有7个成员,野花生有3个, 蔓花生有2个,花生有2个,其中Araip.QQN2T与Ahy022503的亲缘关系相对较近,Aradu.A4KG4与Araip.80H4B的亲缘关系相对较近,Aradu.0KI78与Ahy008861的亲缘关系相对较近;第Ⅳ类有4个成员,分别是蔓花生的Aradu.42K79、Aradu.GIP2Q,野花生的Araip.L8K4F,花生的Ahy022725,其中Araip.L8K4F与Ahy022725的亲缘关系相对较近;第Ⅴ类有7个成员,分别是蔓花生的Aradu.M2I8I、Aradu.SE6Q0、Aradu.C5HAC,野花生的Araip.AXK3N、Araip.RB7C2,花生的Ahy015793、Ahy009871;第Ⅵ类有5个成员,分别是蔓花生的Aradu.EJI6Q、Aradu.QYV19,野花生的Araip.SZ63C,花生的Ahy013042、Ahy017508;第Ⅶ类与其他类别的亲缘关系相对较远,有2个成员都是野花生AP2蛋白家族成员,分别是Araip.N6C0B、Araip.E29R3(图9-a)。
蔓花生AP2基因家族编码蛋白和拟南芥AP2基因家族编码蛋白的系统进化关系可分为Ⅴ类,第Ⅰ类有11个成员的亲缘关系相对较近,其中有7个是蔓花生的成员,4个是拟南芥的成员;第Ⅱ类全是蔓花生的4个成员; 第Ⅲ类只有1个成员,是蔓花生的Aradu.SE6Q0;第Ⅳ类有14个成员,其中蔓花生有3个,拟南芥有11个;第Ⅴ类有17个成员,其中蔓花生有9个,拟南芥有8个,成员Aradu.77UUH与成员AT5G57390.1的亲缘关系相对较近(图9-b)。
对蔓花生与二穗短柄草等15个物种AP2蛋白的系统进化关系分析发现,蔓花生的Aradu.42K79与大豆的Glyma.12G073300.1.p亲缘关系最近,与木豆的C.cajan.21449、芸豆的Phvul.011G071100.1亲缘关系较近,这与其同属于豆科有关,但同为豆科的绿豆Vradi03g03260.1与其亲缘关系较远,说明绿豆该蛋白的进化速度更快;二穗短柄草的Bradi2g37800.13.p与蔓花生的Aradu.42K79亲缘关系最远,可能与其分别为单双子叶植物有关(图10)。
3 讨论与结论
目前,关于AP2转录因子的相关研究显示,AP2亚家族主要参与植物的生长发育过程,对花发育的调控研究得最多[37,54-58],对根的生长、果实和种子发育的调控作用也有研究[25,59]。吴彦庆等通过研究芍药(Paeonia lactiflora Pall.)分生组织决定基因AP[STBX]2[STBZ]发现,plAP2蛋白为亲水性不稳定蛋白,无跨膜结构和信号肽,非分泌蛋白[60]。阮先乐等对甘薯[Dioscorea esculenta (Lour.) Burkill]AP2蛋白的研究发现,AP2蛋白为不稳定的、无跨膜结构域的亲水性非分泌蛋白[61]。邓浪等通过对‘红阳猕猴桃(Actinidia chinensis)AP2/EREB转录因子的研究发现,该家族成员多为酸性氨基酸,是无信号肽的非分泌亲水性蛋白[62],这与本研究中蔓花生AP2蛋白的性质基本吻合。芍药的AP2蛋白主要在胞质中起着生物学功能[60],甘薯的大多数AP2成员亚细胞定位在细胞核[61]。甘蓝(Brassica oleracea L.)AP2蛋白的亚细胞定位结果显示,其AP2蛋白多出现于细胞核中[63];乔永刚等通过对金银花(Lonicera japonica Thunb.)AP2蛋白的亚细胞定位分析发现,其蛋白多在细胞核中[64];此外,新疆紫草[Arnebia euchroma (Royle) Johnst]的AP2蛋白也定位于细胞核中[65]。而本研究结果显示,蔓花生AP2蛋白均定位在细胞核中,说明AP2蛋白在不同植物中具有的作用和承担的功能可能并不相同,但其最可能在细胞核中起着生物学作用。蓖麻的16个AP2成员以α-螺旋、无规则卷曲为其主要的二级结构[66]。郭慧等通过对甘蓝AP2/ERF转录因子的研究发现,无规则卷曲、α-螺旋和延伸链是甘蓝AP2蛋白的主要二级结构[63]。文冠果(Xanthoceras sorbifolium Bunge)AP2蛋白的二级结构主要由无规则卷曲、α-螺旋和延伸链组成[67]。杨树(Populus trichocarpa)AP2蛋白以无规则卷曲为主[68]。在本研究中,蔓花生AP2蛋白也是以无规则卷曲、α-螺旋和延伸链为主,说明这3种二级结构是AP2蛋白的主要二级结构。
本研究结果表明,蔓花生AP2蛋白为不稳定的亲水性非分泌蛋白,亚细胞定位在细胞核中,二级、三级结构以无规则卷曲、α-螺旋和延伸鏈为主,含有YRG、RAYD结构域,花生属的Araip.XB73L和Ahy016292、Araip.QQN2T和Ahy022503、Aradu.A4KG4和Araip.80H4B、Aradu.0KI78和Ahy008861、Araip.L8K4F和Ahy022725这5组组内成员的亲缘关系相对较近,蔓花生AP2成员Aradu.77UUH与拟南芥AP2成员AT5G57390.1的亲缘关系最为接近。物种进化关系分析结果显示,蔓花生的Aradu.42K79与大豆的Glyma.12G073300.1.p亲缘关系最近,与二穗短柄草的Bradi2g37800.13.p亲缘关系最远。这些研究结果为进一步研究和揭示AP2基因家族在蔓花生生长发育、基因调控中的作用提供了一定的参考依据。
参考文献:
[1]周 立.蔓花生(Arachis duranensis Krap.et Greg)在低温胁迫下的生理反应研究[D]. 重庆:西南大学,2008.
[2]张 利. Pb2+、Hg2+胁迫下蔓花生逆境生理及田间不定根发生研究[D]. 重庆:西南大学,2008.
[3]张振霞,郑玉忠. 两种蔓花生坪用性状的比较[J]. 湖北农业科学,2008,47(9):1053-1055.
[4]郑 荷. 地被植物蔓花生再生体系建立及多倍体诱导[D]. 重庆:西南大学,2008.
[5]蔚冬红,乔善义,赵毅民.中药合欢皮研究概况[J]. 中国中药杂志,2004,29(7):619-621.
[6]唐湘梧. 新的牧草栽培品种[J]. 草原与牧草,1991(3):41-42.
[7]罗 丹,陈红跃,刘 乾. 5种屋顶绿化植物抗旱性研究[J]. 广东林业科技,2009,25(6):81-85.
[8]应朝阳,吕亮雪,刘国道,等. 干旱胁迫对多年生落花生生长和活性氧代谢的影响[J]. 热带作物学报,2006,27(1):17-21.
[9]黄毅斌,应朝阳,柯碧南,等. 两种多年生花生品种低温胁迫下的质膜透性研究[J]. 四川草原,2005(10):4-5,12.
[10]余高镜,应朝阳,黄毅斌,等. 2个多年生花生品种低温胁迫下活性氧代谢特性研究[J]. 草业科学,2006(4):29-32.
[11]周 立,郑 荷,张 利,等. 低温胁迫下磷酸二氢钾对蔓花生的生理影响研究[J]. 现代农业科学,2008(2):27-28,31.
[12]彭建宗,陈兆平. 遮阴对多年生花生(Arachis pintoi)生长的影响[J]. 华南师范大学学报(自然科学版),1999(2):92-94.
[13]彭建宗. 优良地被植物多年生花生[J]. 广东园林,2002(4):41-42.
[14]陈伟俊. 一种优良的园林地被植物——金花生[J]. 热带农业科技,2006,29(1):39.
[15]谭光营,潘志权. 优良植被——多年生花生的栽培及其应用[J]. 广西热带农业,2004(1):46-47.
[16]张 妍,刘 瀛,孙丰宾,等. 白桦APETALA2(AP2)转录因子基因的分离及其表达[J]. 林业科学研究,2012,25(2):254-260.
[17]高春艳,吴 芮,袁 玉,等. 植物AP2/ERF转录因子及其在非生物胁迫应答中的作用[J]. 江汉大学学报(自然科学版),2017,45(3):236-240.
[18]张计育,王庆菊,郭忠仁. 植物AP2/ERF类转录因子研究进展[J]. 遗传,2012,34(7):835-847.
[19]季爱加,罗红梅,徐志超,等. 藥用植物转录因子AP2/ERF研究与展望[J]. 科学通报,2015,60(14):1272-1284.
[20]Jofuku K D,Boer B,Montagu M V,et al. Control of Arabidopsis flower and seed development by the homeotic gene APETALA[STBX]2[STBZ][J]. Plant Cell,1994(6):1211-1225.
[21]Toshitsugu N,Kaoru S,Tatsuhito F,et al. Genome-wide analysis of the ERF gene family in Arabidopsis and rice[J]. Plant Physiol,2006,140(2):411-432.
[22]闻可心,刘雪梅. AP[STBX]2[STBZ]功能基因在植物花发育中的重要作用[J]. 生物技术通报,2010(2):1-7.
[23]Houston K,McKim S M,Comadran J,et al. Variation in the interaction between alleles of HvAPETALA2 and microRNA172 determines the density of grains on the barley inflorescence[J]. Proceedings of the National Academy of Sciences of the United States of America,2013,110(41):16675-16680.
[24]刘 强,张贵友,陈受宜. 植物转录因子的结构与调控作用[J]. 科学通报,2000,45(14):1465-1474.
[25]Yan X H,Zhang L,Chen B,et al. Functional identification and characterization of the Brassica napus transcription factor gene BnAP[STBX]2[STBZ],the ortholog of Arabidopsis thaliana APETALA[STBX]2[STBZ][J]. PLoS One,2012,7(3):e33890.
[26]Shigyo M,Ito M. Analysis of gymnosperm two-AP2-domain-containing genes[J]. Dev Genes Evol,2004,214(3):105-114.
[27]Kim S,Soltis P S,Wall K,et al. Phylogeny and domain evolution in the APETALA[STBX]2[STBZ]-like gene family[J]. Mol Biol Evol,2006,23(1):107-120.
[28]Nole-Wilson S,Krizek B A. DNA binding properties of the Arabidopsis floral development protein AINTEGUMENTA[J]. Nucleic Acids Res,2000,28:4076-4082.
[29]Ohme-Takagi M,Shinshi H. Ethylene-inducible DNA binding proteins that interact with anethylene-responsive element[J]. Plant Cell,1995,7(2):173-182.
[30]Fits L V D,Memelink J. The jasmonate-inducible AP2/ERF-domain transcription factor ORCA3 activates gene expression via interaction with a jasmonate-responsive promoter element[J]. The Plant Journal,2001,25(1):43-53.
[31]Gu Y Q,Wildermuth M C,Chakravarthy S,et al. Tomato transcription factors pti4,pti5,and pti6 activate defense responses when expressed in Arabidopsis[J]. Plant Cell,2002,14(4):817-831.
[32]Sasaki K,Mitsuhara I,Seo S,et al. Two novel AP2/ERF domain proteins interact with cis-element VWRE for wound-induced expression of the Tobacco tpoxN[STBX]1[STBZ] gene[J]. The Plant Journal,2007,50(6):1079-1092.
[33]宋佳靜. 花生突变体库的构建及APETALA[STBX]2[STBZ]基因的表达分析[D]. 郑州:河南农业大学,2013.
[34]Haughn G W,Somerville C R. Genetic control of morphogenesis in Arabidopsis[J]. Dev Genet,1988,9(2):73-89.
[35]Bowman J L,Smyth D R,Meyerowitz E M. Genes directing flower development in Arabidopsis[J]. Plant Cell,1989,1(1):37-52.
[36]Bowman J L,Drews G N,Meyerowitz E M. Expression of the Arabidopsis floral homeotic gene agamous is restricted to specific cell types late in flower development[J]. Plant Cell,1991,3(8):749-758.
[37]Kunst L,Klenz J E,Martinezzapater J,et al. Ap[STBX]2[STBZ] gene determines the identity of perianth organs in flowers of Arabidopsis thaliana[J]. Plant Cell,1989,1(12):1195-1208.
[38]Huala E,Sussex I M. Leafy interacts with floral homeotic genes to regulate Arabidopsis floral development[J]. Plant Cell,1992,4(8):901-913.
[39]Ohto M A,Fischer R L,Goldberg R B,et al. Control of seed mass by APETALA[STBX]2[STBZ][J]. Proceedings of the National Academy of Sciences,2005,102(8):3123-3128.
[40]Morcillo F,Gallard A,PilloT M,et al. EgAP2-1,an AINTEGUMENTA-like (AIL) gene expressed in meristematic and proliferating tissues of embryos in oil palm[J]. Planta,2007,226:1353-1362.
[41]Zhuang J,Anyia A,Vidmar J,et al. Discovery and expression assessment of the AP2-like genes in Hordeum vulgare[J]. Acta Physiol Plant,2011,33(5):1639-1649.
[42]Jofuku K D,Omidyar P K,Gee Z,et al. Control of seed mass and seed yield by the floral homeotic gene APETALA[STBX]2[STBZ][J]. Proceedings of the National Academy of Sciences,2005,102(8):3117-3122.
[43]李 宁,高升华,王 飞,等. 辣椒CONSTANS-like转录因子家族的生物信息学分析[J]. 北方园艺,2019(15):1-7.
[44]王 双,李 跃,李孟建,等. 蓖麻APs基因克隆及生物信息学分析[J]. 分子植物育种,2019,17(22):7344-7349.
[45]闫 艳,王 希,周珂辉,等. 西洋参bHLH转录因子家族生物信息学分析[J]. 吉林农业大学学报,2019,41(3):316-323.
[46]李孟建,李国瑞,黄凤兰,等. 蓖麻RcGASA[STBX]9[STBZ]基因克隆及生物信息学分析[J]. 分子植物育种,2019,17(21):6981-6986.
[47]狄建軍,孙佳欣,杨智慧,等. 蓖麻PLA[STBX]2[STBZ]α基因启动子的克隆及生物信息学分析[J]. 分子植物育种,2019,17(15):4961-4966.
[48]Yu C S,Chen Y C,Lu C H,et al. Prediction of protein subcellular localization[J]. Proteins:Structure,Function,and Bioinformatics,2006,64(3):643-651.
[49]刘承圆,何其光,林春花,等. 巴西橡胶树(Hevea brasiliensis) HbMlo2基因克隆与表达分析[J]. 分子植物育种,2019,17(5):1512-1518.
[50]李惠敏,李璐璐,陈玉梅,等. 罗汉果几丁质酶基因家族的生物信息学分析[J]. 生物信息学,2019(3):167-174.
[51]陈湘瑜,徐日荣,陈 昊,等. 杜兰落花生球蛋白基因家族的生物信息学分析[J]. 中国油料作物学报,2019,41(3):391.
[52]徐志军,刘 洋,徐 磊,等. 玉米转录因子NF-YB基因家族的生物信息学分析[J]. 分子植物育种,2019,17(12):3807-3816.
[53]周 凡,李 丹,周 军,等. 梨DREB基因家族全基因组鉴定及分析[J]. 基因组学与应用生物学,2018,37(9):3889-3902.
[54]Drews G N,Bowman J L,Meyerowitz E M. Negative regulation of the Arabidopsis homeotic gene AGAMOUS by the APETALA2 product[J]. Cell,1991,65(6):991-1002.
[55]Shannon S,Meeks-Wagner D R. Genetic interactions that regulate inflorescence development in Arabidopsis[J]. Plant Cell,1993,5(6):639-655.
[56]Houston K,McKim S M,Comadran J,et al. Variation in the interaction between alleles of HvAPETALA[STBX]2[STBZ] and microRNA172 determines the density of grains on the barley inflorescence[J]. Proc Natl Acad Sci USA,2013,110(41):16675-16680.
[57]Jofuku K D,den Boer B G,van Montagu M,et al. Control of Arabidopsis flower and seed development by the homeotic gene APETALA2[J]. Plant Cell,1994,6:1211-1225.
[58]Zhang P,Yang P Z,Zhang Z Q,et al. Isolation and characterization of a buffalograss (Buchloe dactyloides) dehydration responsive element binding transcription factor,BdDREB2[J]. Gene,2014,536:123-128.
[59]Aya K,Hobo T,Sato-Izawa K,et al. A novel AP2-type transcription factor,SMALL ORGAN SIZE1,controls organ size downstream of an auxin signaling pathway[J]. Plant Cell Physiol,2014,55(5):897-912.
[60]吴彦庆,成梦琳,赵大球,等. 芍药分生组织决定基因APETALA[STBX]2(AP2[STBZ])的克隆及生物信息学分析[J]. 华北农学报,2017,32(3):58-64.
[61]阮先乐,张 杰,张福丽,等. 甘薯AP2基因家族的生物信息学分析[J]. 分子植物育种,2017,15(6):2066-2072.
[62]邓 浪,沈兵琪,王连春,等. ‘红阳猕猴桃全基因组AP[STBX]2[STBZ]/EREBP转录因子生物信息学分析[J]. 果树学报,2017,34(7):790-805.
[63]郭 慧,金司阳,刘 寒,等. 甘蓝AP2/ERF转录因子的克隆和生物信息学分析[J]. 中国药师,2017,20(1):6-10.
[64]乔永刚,陈 亮,崔芬芬,等. 基于转录组金银花AP2基因家族的生物信息学及表达分析[J]. 核农学报,2019,33(9):1698-1706.
[65]谢 腾,王 升,周良云,等. 新疆紫草AP2/ERF转录因子的电子克隆和生物信息学分析[J]. 中国中药杂志,2014,39(12):2251-2257.
[66]闫星伊,风 兰,韩雯毓,等. 蓖麻AP2/ERF转录因子家族的生物信息学分析[J]. 分子植物育种,2020,18(4):1088-1102.
[67]刘晓辉,马静怡,罗 媛,等. 木本油料文冠果APETALA[STBX]2[STBZ]基因全长cDNA序列与生物信息学分析[J]. 现代农业科技,2018(2):145-146.
[68]赵金玲,姚文静,王升级,等. 杨树AP2/ERF转录因子家族生物信息学分析[J]. 东北林业大学学报,2015,43(10):21-29.
收稿日期:2019-09-15
基金项目:四川省科技厅项目(编号:17ZZ019);乐山师范学院人才启动项目(编号:XJR17005)。
作者简介:王 玲(1999—),女,四川南充人,主要从事植物分子生物学研究。E-mail:1981887804@qq.com。
通信作者:付 春,博士,讲师,主要从事经济植物的生物信息学研究。E-mail:fuchun421@aliyun.com。

