基于肿瘤基质评分的胃癌预后基因分析
罗 安,朱欣彦,胡晔东,刘雁冰,冉晨曦,刘 菲
(同济大学附属东方医院消化内科,上海 200120)
胃癌(gastric cancer, GC)是消化系统常见的恶性肿瘤之一。我国每年新发病例约40万,占世界总发病例的42%[1]。胃癌的发生和进展与多种因素有关,如饮食习惯、幽门螺旋杆菌感染、环境因素、遗传因素等。治疗效果及预后与肿瘤的浸润、转移密切相关[2]。胃癌患者早期主要通过内镜下治疗和手术治疗,5年生存率可以达到90%[3]。但是大多数患者确诊时已为晚期且多已发生远处转移,总体治疗效果差,患者的生活质量低。目前对于胃癌的预后预测是临床医师和科研工作者面临的一个难题。根据肿瘤病理(T)、淋巴结活检(N)、远处器官转移(M)来判断患者的预后被普遍认为是用于预测胃癌的治疗实践的主要工具[4]。然而,通过TNM系统预测患者的预后,往往存在很大的差异。因此,需要一些其他的手段去补充。
肿瘤微环境是指肿瘤所在的细胞环境,基质细胞和免疫细胞是两种主要的非肿瘤成分,其中肿瘤基质细胞被报道在对肿瘤的诊断和预后评估有价值[5]。基质免疫评估数据库(estimation of stromal and immune cells in malignant tumor tissues using expression data, ESTIMATE)可通过分析肿瘤微环境中基质细胞和免疫细胞的特异性基因表达特征,计算免疫和基质评分来预测非肿瘤细胞的浸润程度[6]。基于算法的科学性,研究者们很快将这种算法运用在乳腺癌[7]和结肠癌[8]中,说明该算法的实用价值。Wang等[9]将此类评分方法运用到胃癌中,通过分析在胃癌肿瘤基质和肿瘤免疫中均差异表达的基因,来判断这些基因在胃癌预后预测上的作用,具有一定的指导作用。本研究通过癌症基因组图谱数据库(the cancer genome atals, TCGA)保存的大规模多中心的胃癌数据,获取胃癌患者的临床资料和相关组织的转录组测序(ribonucleic acid sequencing, RNAseq)表达数据,筛选出胃癌组织中与基质评分最为相关的基因,建立一个立足于胃癌肿瘤基质评分的胃癌基因预后模型,筛选出预示胃癌预后不良的相关基因,提示胃癌肿瘤基质与预后关系相关研究的进一步方向。
1 材料与方法
1.1 数据提取
从TCGA数据库(http:∥cancergenome.nih.gov/)下载胃癌患者的临床信息(包括性别、年龄、生存时间、肿瘤组织类型、TNM等)和组织RNAseq表达谱数据。从ESTIMATA网站(https:∥bioinformatics.mdanderson.org/estimate/)下载对TCGA数据库中胃癌患者的肿瘤基质评分。本研究获取了胃癌患者的临床数据、ESTIMATE基质评分、RNAseq表达谱3类信息,保证数据的单一性、完整性。将这些患者按照ESTIMATE基质评分分为高基质评分组(H1组:0~1000;H2组: 1000~2000)和低基质评分组(L1组:-1000~0;L2组:-2000~-1000)。其中低基质组130例(其中L1组102例,L2组28例),高基质组140例(其中H1组105例,H2组35例)。
1.2 观察基质评分与预后的关系
根据270例患者的临床信息,分析不同胃癌lauren分型(肠型和弥漫型)、不同胃癌分期上(Ⅰ期和Ⅱ+Ⅲ+Ⅳ期)的基质评分差异。用R语言survival包比较高低基质评分组的患者在生存时间上的差异。
1.3 差异表达和共表达网络
DEseq2程序包对高基质评分组和低基质评分组患者组织RNAseq数据进行标准化处理和差异表达分析筛选阈值(cut off)为P<0.05,|log2FC|>2。对筛选出来的差异基因重新构建表达矩阵,WGCNA包进行权重共表达分析,找出其中与胃癌基质评分最为相关的基因群,为后续构建模型的初始基因。
1.4 基因功能和基因临床模型的构建
构建整合模型初始基因与患者生存状态、生存时间的表达矩阵。单因素COX回归模型初步筛选出与患者生存周期具有相关性的基因(P<0.05)。LASSO模型通过惩罚机制筛选出其中与患者生存周期相关的关键基因。多因素COX回归模型以关键基因为建模基础,构建关键基因的基因临床预测模型。计算模型C指数,绘制校准图、ROC曲线以评价模型的灵敏度和特异度。生成列线图量化基因表达与生存周期的关系。在模型内部总结出生存分析曲线,评价基因作为单个独立危险因素,对患者生存周期的影响。
1.5 外部数据库验证
Oncomine(https:∥www.oncomine.org/resource/login.html)数据库验证基因在胃癌组织中的表达情况。km-plotter数据库(http:∥kmplot.com/analysis/)验证大数据中基因的临床预后。
1.6 统计学处理
统计学分析使用GraphPad Prism 7.0软件,进行独立样本t检验。P<0.05为差异有统计学意义。生物信息学分析均使用R语言(version 3.6.1)(http:∥www.R-project.org)及其相应的包。基质和免疫评分使用默认参数的estimate包计算。使用DEseq2包进行矩阵标准化处理和差异表达分析(cut off |log2FC|>2,P<0.05)。使用WGCNA包进行加权共表达分析。使用survival包(单因素COX,以P<0.05为差异有统计学意义)、glmnet包、rms包、timeROC包构建最佳COX回归模型并绘制基因生存分析曲线。
2 结 果
2.1 基质评分和患者预后的相关性分析
根据患者临床信息将270例患者分为两组,肠型胃癌组和弥漫性胃癌组。按照胃癌lauren分级,弥漫性胃癌患者表现为低分化且预后更差[10]。比较两组胃癌的基质评分,弥漫性胃癌的基质评分更高(P<0.001),见图1A。用同样的方法分析早期胃癌(Ⅰ期)和进展期胃癌(Ⅱ+Ⅲ+Ⅳ期)的基质评分差异,进展期胃癌表现为更高的基质评分(P<0.001),见图1B。不同基质评分的4个组中,即基质评分L2组(-2000~-1000)、L1组(-1000~0)、H1组(0~1000)、H2组(1000~2000),生存分析表明随着基质评分的升高,患者的生存期呈现下降趋势(P<0.05),见图1C。
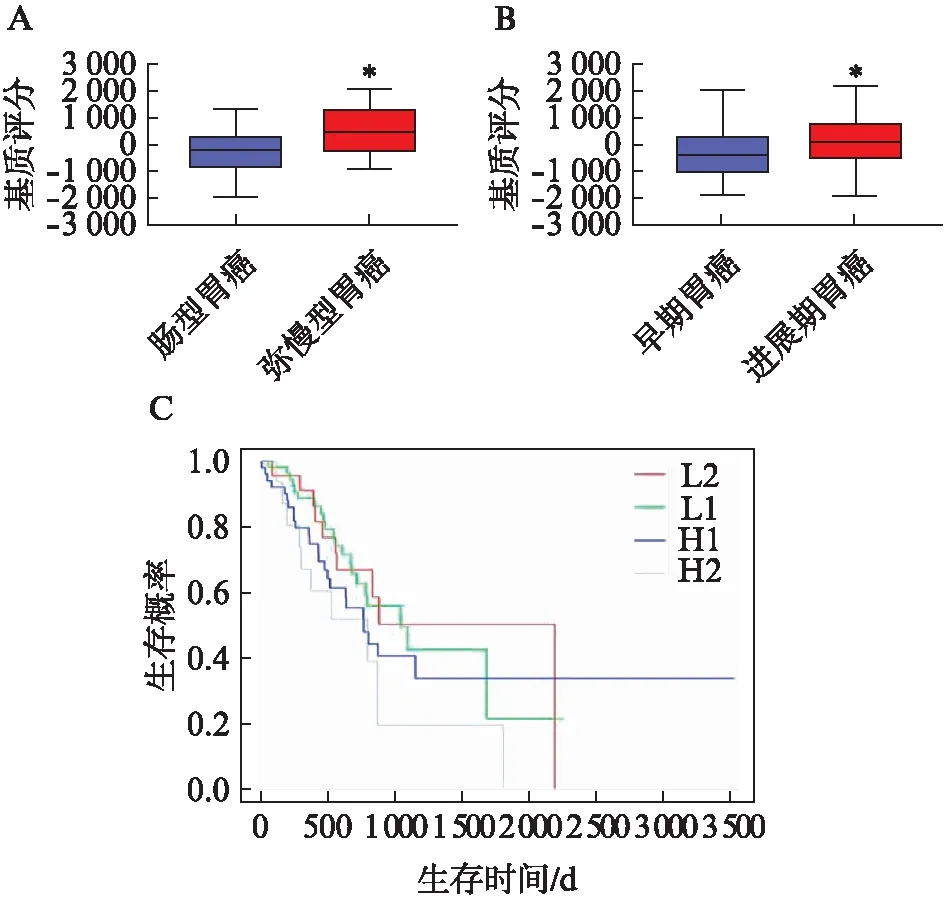
图1 基质评分的高低与胃癌患者预后的关系
2.2 与胃癌患者基质评分密切相关的基因筛查分析
DEseq2程序包中用于RNAseq表达矩阵的分析[11],对比低基质组与高基质组的差异,标准化处理后得到1581个差异表达基因。将|log2FC|变化最大的20个基因做了热图,见图2A。WGCNA程序包中分析1581个基因表达矩阵与基质评分的相关性[12],仅关注与胃癌基质评分关系最为密切的基因群,其中蓝绿色相关性最高为0.7(P<0.001),该基因群共计1015个基因,为后续建模的初始基因。将各个颜色的基因群在树状图和热图中显示,见图2B。

图2 1581个差异表达基因中筛选与基质评分密切相关的基因
2.3 胃癌预后相关基因的筛选,及临床预测模型的构建
利用单因素COX回归模型初步定位出胃癌预后相关的基因,共计377个(P<0.05)。LASSO回归模型筛选377个基因中影响胃癌预后的关键基因,当纳入模型的基因变量为12个时,模型为最佳(λ最小为12),见图3A,此12个基因为与胃癌患者预后相关的关键基因,即ACAT1、ADAMTS12、LINCO614、MATN3、MTUS2、PLCL1、MEGF10、POSTN、SERPINE1、TPTEP1、GAD1、MMP16。构建这12个基因多因素COX回归模型[13],C指数[14]为0.68,显示具有较强的预测能力。预测值贴近实际值,见图3B、C。受试者工作曲线(receiver operator curve, ROC)表明3年生存时间曲线下面积(area under curve, AUC)为0.693,5年生存时间AUC为0.725,模型预测的准确性较强,见图3D。本研究大致量化了这12个基因的表达量与生存时间的关系,其中ACAT1、ADAMTS12、LINCO1614、MATN3、MTUS2、PLCL1、POSTN、SERPINE1、TPTEP1、MEGF10相对表达量越高,患者生存时间越短;GAD1和MMP16相对表达量越低,患者生存期越短,见图3E。在模型中(n=270)绘制了这12个基因的生存曲线,得到6个与胃癌预后相关性较强(P<0.05)的基因:ADAMTS12、MATN3、MEGF10、PLCL1、POSTN、SERPINE1。同时作为差异表达基因,相对于低基质组患者,这6个基因均在胃癌患者高基质组中高表达,作为独立危险因素,6个基因均与胃癌患者预后呈负相关,见图4。

图3 回归分析构建的基因预后模型
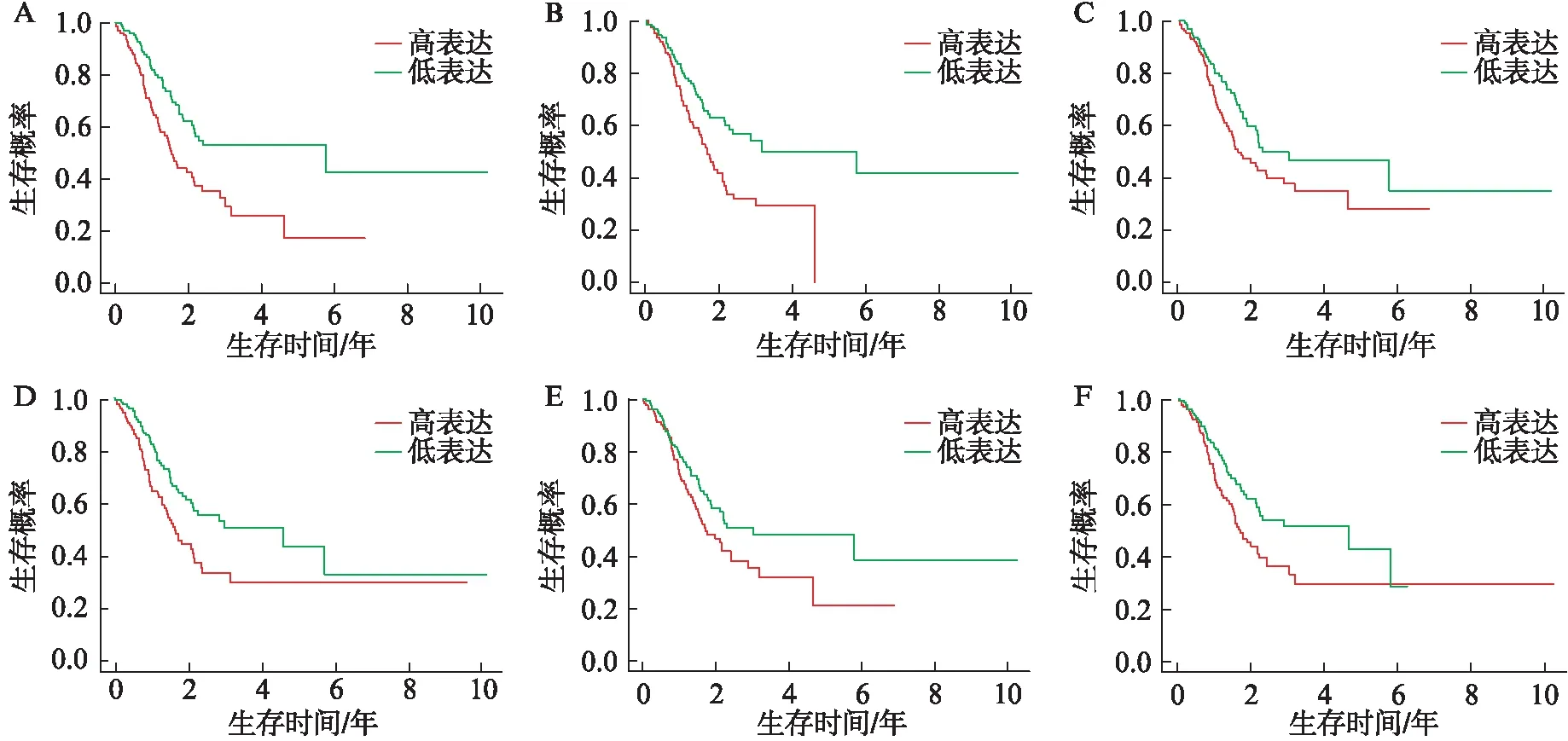
图4 预测模型内部6个最佳预测基因与患者的生存分析曲线
2.4 临床预测模型的大数据验证
KM-plotter数据库(http:∥kmplot.com/analysis/)显示这6个基因与本研究模型所预测的一致(n=375),均表现为表达量越高,预后越差,生存周期越短(P=0.0037、0.000091、0.0047、0.00019、0.00003、0.0000066),见图5。获取Oncomine数据库(https:∥www.oncomine.org/resource/login.html)数据集GSE27342(n=160)中这6个基因的相对表达量,ADAMTS12、MATN3、POSTN、SERPINE和本研究的结果相符。MEGF10数据差异无统计学意义(P>0.05)。PCLC1与预期结果相反,见图6。

图5 6个最佳预测潜力基因在KM-plotter数据库中的生存分析

图6 6个最佳预测潜力基因在GSE27342数据集中的验证与本研究预测模型一致
3 讨 论
肿瘤的进展不仅受其肿瘤本身特征的影响,还受肿瘤基质细胞的影响。越来越多的证据表明肿瘤基质在预测肿瘤进展和预后中的重要作用[15-16]。因此,本研究提出了一个结合肿瘤基质评分和胃癌预后的模型,筛选出与胃癌预后密切相关的基因。本研究中采取了很多“筛选”的步骤:(1)差异表达筛选出差异基因;(2)共表达网络筛选差异基因中与基质关系最为密切的基因;(3)单因素COX回归去掉了与胃癌患者预后无相关性的基因(P>0.05);(4)LASSO回归给出了最小λ,纳入12个基因时模型的预测功能最佳,此12个基因为该模型下预测胃癌预后的关键基因;(5)得到一个较为准确的临床预测模型(c指数=0.68),同时将单个基因作为独立危险因素,绘制基因在模型内部的生存分析曲线,进一步优化了与胃癌预后密切相关的基因(P<0.05)。最终得到6个基因,MATN3、MEGF10、ADAMTS12、PLCL1、POSTN、SERPINE。作为基于基质评分的胃癌独立危险因素的临床预后分析,患者肿瘤基质评分越高,6个基因均表达量越高,患者生存期越短,预后越差。6个基因各自具有良好的胃癌预后预测能力。
与同类型文章相比[9,17]本文的创新点在于将RNAseq数据差异表达分析后,并未直接以差异基因作为后续分析的初始基因,而是用WGCNA分析差异基因的表达矩阵。固然本研究是以低基质评分组为对照组,高基质评分组为实验组,但单纯的差异表达并不能直接说明差异基因与基质评分之间的相关性,通过WGCNA可以找到与基质评分密切相关的基因群。
本研究得到的这6个基因,MATN3作为一种常见于细胞外基质的蛋白,Wu等[18]收集病例并分析总结出MATN3可用来预测胃癌预后,其在胃癌组织中表达越高,患者预后越差;目前对MEGF10的研究局限于胶质瘤。研究表明MEGF10的甲基化水平与胶质瘤常见的IDH突变相关。其甲基化水平越高,患者的预后越差[19]。ADAMTS12是金属蛋白酶家族一员,其在上皮恶性细胞周围组织中的表达明显高于正常配对组织[20],提示其可能参与到肿瘤的发生发展中。Wang等[21]通过收集112例结肠癌患者,用免疫组化染色的方法检测ADAMTS12的表达量,发现ADAMTS12的表达与结肠癌的病例分级、肿瘤浸润、淋巴结转移具有明显的相关性。表达越高,预示着结肠癌患者的预后越差。目前对PLCL1的研究较少,仅发现其可以通过介导脂质褐变而抑制肿瘤进程,具体机制未被阐明[22]。OH等[23]用免疫组化的方法检测了结肠癌患者的肿瘤基质,发现高表达POSTN的患者的预后明显更差。一项基础研究表明SERPINE可以促进SGC-7901细胞的侵袭性,提示SERPINE可以作为胃癌的一种新的预后因子和抗癌靶点[24]。结合本研究所构建的多因素COX回归模型以及基因表达与生存状态、生存时间之间的相关性,从肿瘤基质预后基因分析的角度讲,本研究预测的6个基因,基本符合现有的临床和基础研究事实,具有一定的临床预测能力。
本研究选择了两个数据库:Oncomine数据库和KM-plotter数据库。前者病例资料来源于GEO(gene expression omnibus)数据库,选择的数据集为GSE 27342(n=160),作为一个独立的病例资料,与本文研究的TCGA数据库病例资料(n=270)不存在数据重叠。后者数据来源于包括GEO、TCGA、EGA在内的多个肿瘤学数据库,此数据库设计的目的即为对目的基因进行生存分析验证,从而佐证实验结果,本文选择其中的胃癌模块(n=375),本研究无法判断此375例病患资料是否与前述GSE27342以及本文TCGA数据库270例病患资料存在重叠性,但是可以肯定的是KM-plotter数据库设计的科学性。因此,作为验证,两个数据库是合理的。
本研究发现大数据库的生存分析与本研究的预测结果是完全一致的。在胃癌组织内的表达和本研究的预测基本一致,显示本研究的模型具有很高的准确性。但本研究从肿瘤基质评分的角度出发,经过差异表达分析、WGCNA“纯化”、COX回归模型预测得到的这6个基因,均是从生信分析的角度得到的结果,尚有待后续实验研究进一步验证。

