鸡BRD2基因及其剪接体的克隆测序与亚细胞定位分析
周磊 袁超 韩一帆 高洪波 王燕碧 赵采芹 唐宏 段志强


摘要:【目的】克隆雞含溴结构域蛋白2(BRD2)基因及其剪接体,并对其进行序列分析和亚细胞定位,为深入研究鸡BRD2基因及其剪接体在细胞转录调控中的作用机理打下基础。【方法】通过RT-PCR扩增鸡BRD2基因及其剪接体的编码区(CDS)序列,采用BioEdit、ProtParam、SOPMA、SWISS-MODEL、CDART等在线软件进行生物信息学分析;鸡BRD2基因及其剪接体经Xho I和BamH I双酶切后,分别亚克隆至真核表达载体pEGFP-C1多克隆位点上构建重组真核表达载体,通过转染HEK-293T细胞进行亚细胞定位分析。【结果】鸡BRD2基因CDS序列全长2340 bp,编码779个氨基酸残基,分子量为85.66 kD,理论等电点(pI)为8.74,其编码蛋白二级结构主要由无规则卷曲(60.59%)和α-螺旋(28.50%)组成,含有3个明显的功能结构域(2个BD结构域和1个ET结构域)。与鸡BRD2基因相比,其剪接体BRD2-X1、BRD2-X2和BRD2-X3的CDS序列长分别为2301、2283和1983 bp,编码766、760和660个氨基酸残基。以鸡BRD2氨基酸序列为参照, BRD2-X1表现为第526~537位氨基酸缺失,BRD2-X2表现为第1~19位氨基酸缺失,BRD2-X3表现为第1~119位氨基酸缺失;与鸡BRD2蛋白相比,鸡BRD2基因剪接体BRD2-X1和BRD2-X2并未影响鸡BRD2蛋白的功能结构域,而剪接体BRD2-X3导致鸡BRD2蛋白第一个BD结构域部分缺失。鸡BRD2基因融合蛋白EGFP-BRD2及其剪接体融合蛋白EGFP-BRD2-X1、EGFP-BRD2-X2和EGFP-BRD2-X3均定位在细胞核,EGFP-BRD2和EGFP-BRD2-X1在细胞核中呈均匀分布,而EGFP-BRD2-X2和EGFP-BRD2-X3在细胞核中呈不均匀的点状分布。【结论】鸡BRD2蛋白及其剪接体均定位在细胞核,但BD结构域缺失可引起BRD2剪接体BRD2-X3细胞核定位模式的变化。
关键词: 鸡;BRD2基因;剪接体;BD结构域;ET结构域;亚细胞定位
中图分类号: S831.2 文献标志码: A 文章编号:2095-1191(2020)08-1806-10
Cloning, sequence analysis and subcellular localization of chicken BRD2 gene and its splices
ZHOU Lei, YUAN Chao, HAN Yi-fan, GAO Hong-bo, WANG Yan-bi,
ZHAO Cai-qin, TANG Hong, DUAN Zhi-qiang*
(College of Animal Sciences, Guizhou University/Key Laboratory of Animal Genetics, Breeding and Reproduction in the Plateau Mountains Region, Ministry of Education/Key Laboratory of Animal Genetics, Breeding and
Reproduction in Guizhou, Guiyang 550025, China)
Abstract:【Objective】This study aimed to clone chicken bromodomain-containing protein 2(BRD2) gene and its splices, and analyze their sequences and subcellular localization ,which laid a foundation for further study of mechanism of chicken BRD2 gene and its splice in cellular transcriptional regulation. 【Method】The coding region(CDS) region sequence of chicken BRD2 gene and its splice were amplified by RT-PCR and then analyzed by online softwares including BioEdit, ProtParam, SOPMA, SWISS-MODEL, CDART, etc. The PCR products of chicken BRD2 gene and its splices were digested by Xho I and BamH I, and subcloned into eukaryotic expression vector pEGFP-C1 multiple cloning site to construct recombinant eukaryotic expression vectors. The subcellular localization of chicken BRD2 gene and its splices were analyzed by transfecting the constructed plasmids into HEK-293T cells. 【Result】The results revealed that the full-length CDS region sequence of chicken BRD2 gene was 2340 bp and encoded 779 amino acids with a molecular weight of 85.66 kDa, and the theoretical pI was 8.74, The secondary structure of chicken BRD2 protein was mainly composed of random coil(60.59%) and α-helix(28.50%), and contained three obvious functional domains(2 BD domains and 1 ET domain). Compared with chicken BRD2 gene, the splices BRD2-X1, BRD2-X2 and BRD2-X3 were 2301, 2283 and 1983 bp in length, and encoded 766, 760 and 660 amino acid residues, respectively. Taking the amino acid sequence of chicken BRD2 as reference, BRD2-X1, BRD2-X2 and BRD2-X3 showed amino acid deletion at positions 526-537, 1-19 and 1-119, respectively. In addition, BRD2-X1 and BRD2-X2 did not affect the functional domain of chicken BRD2 protein, while the splice BRD2-X3 caused partial deletion of the first BD domain of chicken BRD2 protein. Moreover, the fusion protein EGFP-BRD2,and its splices fusion proteins EGFP-BRD2-X1, EGFP-BRD2-X2 and EGFP-BRD2-X3 were mainly located in the nucleus. However, the fusion proteins EGFP-BRD2 and EGFP-BRD2-X1 showed even distribution in the nucleus, but EGFP-BRD2-X2 and EGFP-BRD2-X3 exhibited unevenly spotty pattern distribution in the nucleus. 【Conclusion】The chicken BRD2 protein and its different splices locate in the nucleus, but the deletion of BD domain lead to the nuclear localization pattern changes of BRD2 splice BRD2-X3.
Key words: chicken; BRD2 gene; splice; BD domain; ET domain; subcellular localization
Foundation item: National Natural Science Foundation of China(31960698,31760732); Guizhou Basic Research Project(Science and Technology Fund)(QKHJC〔2020〕1Y134); Guizhou Science and Technology Planning Project(QKHPTRC〔2017〕5788)
0 引言
【研究意義】含溴结构域蛋白2(Bromodomain-containing protein 2,BRD2)又称RING3,是BET (Bromodomain and extra-terminal domain)蛋白家族的一员。BET蛋白家族成员主要包括BRD2、BRD3、BRD4和睾丸组织特异性表达的BRDT,均具有2个串联的N-末端溴结构域(Bromodomain,BD)和1个保守的C-末端外结构域(Extra-terminal domain,ET)(Belkina and Denis,2012)。BRD2作为表观遗传阅读器,参与基因转录、染色质重塑及细胞周期增殖和凋亡,且BRD2基因异常表达还与炎症、癫痫和肿瘤等疾病的发生有关(Ferri et al.,2016;Pathak et al.,2018)。因此,开展BRD2基因及其剪接体的亚细胞定位研究,对揭示BRD2在细胞中的生物学功能具有重要意义。【前人研究进展】BD结构域首次发现于果蝇的Brahma蛋白,是一种广泛分布于多种生物中的高度保守结构域,一般由60~110个氨基酸残基组成(Dyson et al.,2001);BD结构域能结合核小体组蛋白赖氨酸的ε-氨基乙酰基,特别是乙酰化组蛋白H4,通过招募转录因子、转录辅激活因子及转录辅抑制因子而调节基因转录(Leroy et al.,2008)。ET结构域是由约80个氨基酸残基组成的保守区域(Jeanmougin et al.,1997;Dyson et al.,2001;Rahman et al.,2011),其通过招募特定效应蛋白(组蛋白甲基转移酶)来实现调节基因转录功能(Rahman et al.,2011)。BRD2蛋白作为转录调节因子,是通过细胞骨架蛋白招募不同的蛋白分子而调控相关细胞周期,如Cyclin A、Cyclin E、Cyclin D及激活转录因子E2F-1(Zeng and Zhou,2002;Sinha et al.,2005)。在神经发育方面,BRD2基因在神经系统增殖的前体细胞和分化的神经元中均有表达(Crowley et al.,2004),但过量表达会损害神经元分化,可能是由于BRD2刺激了细胞周期进程(Garcia-Gutierrez et al.,2012)。在疾病研究方面,模仿乙酰化赖氨酸的药物通过阻断BRD2蛋白BD结构域而发挥作用,如BRD2抑制剂JQ1能有效抑制人类髓母细胞瘤细胞系增殖,诱导癌细胞凋亡(Venataraman et al.,2014)。近年来的研究发现,在我国26个优质鸡种中,BRD2基因具有一定保守性,但不同鸡种间相似性较低(吴允等,2017);BRD2蛋白ET结构域能与猪内源性逆转录病毒A/C整合酶相互作用,介导病毒基因组整合到宿主基因组中(Gallay et al.,2019);鸡BRD2蛋白ET结构域还能与新城疫病毒M蛋白相互作用,可能与M蛋白抑制细胞基因转录有关(Duan et al.,2018);BRD2蛋白在逆转录病毒与宿主相互作用中同样发挥重要作用(Sharma et al.,2013)。基因进行转录时首先形成包含所有内含子和外显子的前体mRNA(pre-mRNA),而pre-mRNA经酶切内含子和连接外显子方式被加工成含有同一基因不同序列的成熟mRNA的过程称为基因选择性剪接(Alternative splicing,AS)(Modrek and Lee,2002)。即单个基因可经过AS获得更多的转录异构体,从而产生多种发挥不同功能的蛋白(Kelemen et al.,2013)。【本研究切入点】目前,有关BRD2基因的功能研究主要集中在人类和小鼠上,可作为肾脏疾病新的表观遗传学靶点(Morgado-Pascual et al.,2019)及角质形成细胞炎症反应的细胞因子(Slivka et al.,2019),或通过ERK/HSL信号通路促进小鼠白色脂肪组织的脂解(Zong et al.,2019);但针对鸡BRD2基因及其剪接体克隆、序列特征及亚细胞定位等情况尚不明确。【拟解决的关键问题】根据GenBank已公布的鸡BRD2基因及其3个剪接体进行克隆和生物信息学分析,进一步构建鸡BRD2基因及其剪接体的重组真核表达载体,转染细胞后进行亚细胞定位分析,以期为深入研究鸡BRD2基因及其剪接体在细胞转录调控中的作用机理打下基础。
1 材料与方法
1. 1 试验材料
真核表达载体pEGFP-C1、大肠杆菌DH5α感受态细胞、鸡胚成纤维细胞系(DF-1细胞)和人胚胎肾细胞(HEK-293T细胞)均由高原山地动物遗传育种与繁殖教育部重点实验室保存提供。TRIzol Rea-gent、pCR? 2.1载体购自美国Invitrogen公司;Revert-Aid First Strand cDNA Synthesis Kit、T4 DNA连接酶、DNA Maker、Pfu Taq DNA高保真聚合酶、限制性内切酶Xho I和BamH I购自Thermo Fisher公司;DNA凝胶回收试剂盒和质粒小量提取试剂盒购自AxyGen公司;4%多聚甲醛和Trition X-100购自碧云天生物技术研究所;FuGENE? HD Transfection Reagent购自Promega公司;胎牛血清和DMEM基础培养基购自Gibco公司;其他试剂均为国产分析纯。
1. 2 引物设计与合成
根据GenBank已公布的鸡BRD2基因序列,利用Primer Premier 5.0设计扩增鸡BRD2基因开放阅读框(ORF)及其3个不同剪接体的特异性引物(表1),并在上、下游引物中分别插入Xho I和BamH I酶切位点(下划线部分),所有引物委托昆泰锐(武汉)生物技術有限责任公司合成。
1. 3 鸡BRD2基因及其剪接体克隆与测序分析
使用TRIzol法从DF-1细胞中提取总RNA并进行反转录,以特异性引物BRD2、BRD2-X1、BRD2-X2和BRD2-X3分别扩增鸡BRD2基因及其剪接体的编码区(CDS)序列。PCR扩增程序:94 ℃预变性5 min;94 ℃ 40 s,62 ℃ 50 s,72 ℃ 90 s,进行30个循环;72 ℃延伸10 min,4 ℃保存。PCR扩增产物经1.0%琼脂糖凝胶电泳检测及回收纯化后连接至pCR?2.1载体,并转化DH5α感受态细胞,挑取单菌落扩大培养后进行菌液PCR鉴定,将阳性菌液送至英潍捷基(上海)贸易有限公司测序。
鸡BRD2基因及其剪接体的测序结果用BioEdit进行序列拼接,并与GenBank已公布的鸡BRD2基因序列进行BLAST比对分析。利用ProtParam(http://expasy.org/tools/protparam.html)、SOPMA(http://np-sapbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_so-pma.html)和SWISS-MODEL(http://swissmodel.expasy.org/)分别对鸡BRD2基因及其剪接体编码蛋白进行理化性质及其二、三级结构预测;使用NCBI服务器上的CDART工具预测鸡BRD2蛋白功能结构域。另外,从GenBank搜索下载不同物种的BRD2基因,采用MegAlign进行核苷酸序列相似性分析,并以MEGA 5.0中的邻接法(Neighbor-joining,NJ)构建系统发育进化树。
1. 4 鸡BRD2基因及其剪接体重组真核表达载体构建
使用Xho I和BamH I限制性内切酶分别对真核表达载体pEGFP-C1和鸡BRD2基因及其剪接体克隆质粒进行双酶切,经1.0%琼脂糖凝胶电泳检测后进行切胶纯化,然后将目的基因片段与pEGFP-C1进行连接并转化DH5α感受态细胞。以含卡那霉素的LB培养基进行抗性筛选,将菌液PCR鉴定和双酶切鉴定均呈阳性的重组质粒送至英潍捷基(上海)贸易有限公司测序。
1. 5 细胞培养与转染
HEK-293T细胞使用含10%胎牛血清、100 U/mL青霉素和100 μg/mL链霉素的DMEM培养基进行培养,在37 ℃、5% CO2的细胞培养箱中培养。转染前,将处理好的HEK-293T细胞平铺在35 mm细胞培养皿中培养过夜,待细胞完全贴壁生长至80%左右,更换培养基,然后将提纯后的鸡BRD2基因及其剪接体重组真核表达载体和脂质体按3 μg∶10 μL的比例加入到100 μL无抗无血清DMEM基础培养基中,轻轻吹打混匀后室温静置20 min,再分别加入细胞培养皿中,置于37 ℃、5% CO2的细胞培养箱中继续培养24 h。
1. 6 鸡BRD2基因及其剪接体融合蛋白荧光观察
在鸡BRD2基因及其剪接体重组真核表达载体转染HEK-293T细胞24 h后,PBS洗涤细胞2次,加入预冷的4%多聚甲醛,室温固定20 min;弃液体,PBS洗涤细胞2次;加入0.25% TritonX-100室温作用5 min;PBS洗涤细胞2次,加入DAPI染细胞核10 min;再以PBS洗涤细胞2次,置于倒置荧光显微镜下观察。获得的融合蛋白荧光图片与对应的细胞核荧光图片用Photoshop CS3进行Merge处理,通过荧光共定位判断融合蛋白的亚细胞定位。
2 结果与分析
2. 1 鸡BRD2基因及其剪接体的克隆和重组真核表达载体构建
以DF-1细胞总RNA反转录合成的cDNA为模板,使用特异性引物扩增鸡BRD2基因及其不同剪接体,结果获得BRD2(2340 bp)、BRD2-X2(2283 bp)和BRD2-X3(1983 bp)3条目的条带(图1-A),经重叠PCR扩增获得BRD2-X1(2301 bp)目的条带(图1-B),片段大小均与预期结果相符。获得的目的片段经克隆测序后分别亚克隆至真核表达载体pEGFP-C1多克隆位点上,构建鸡BRD2基因及其剪接体重组真核表达载体pEGFP-BRD2、pEGFP-BRD2-X1、pEGFP-BRD2-X2和pEGFP-BRD2-X3。使用Xho I和BamH I进行双酶切鉴定,结果均获得真核表达载体片段及相应的目的基因片段(图2)。测序结果显示,鸡BRD2基因及其剪接体已按正确的位置插入到真核表达载体pEGFP-C1上,且未发生核苷酸突变和ORF移码,说明鸡BRD2基因及其剪接体重组真核表达载体构建成功。
2. 2 鸡BRD2基因及其剪接体的生物信息学分析结果
2. 2. 1 鸡BRD2基因及其剪接体编码蛋白理化性质和功能结构域预测分析结果 运用BioEdit对鸡BRD2基因及其剪接体进行推导氨基酸序列比对分析,以鸡BRD2氨基酸序列为参照,结果发现BRD2-X1表现为第526~537位氨基酸缺失,BRD2-X2表现为第1~19位氨基酸缺失,BRD2-X3表现为第1~119位氨基酸缺失(图3-A)。鸡BRD2基因编码779个氨基酸残基,分子量为85.66 kD,理论等电点(pI)为8.74;BRD2-X1编码766个氨基酸残基,分子量为84.23 kD,pI为8.8;BRD2-X2编码760个氨基酸残基,分子量为83.69 kD,pI为8.8;BRD2-X3编码660个氨基酸残基,分子量为72.45 kD,pI为7.62。蛋白功能结构域预测分析结果(图3-B)表明,鸡BRD2蛋白主要含有2个BD结构域和1个ET结构域。与鸡BRD2蛋白相比,BRD2基因剪接体BRD2-X1和BRD2-X2并未影响鸡BRD2蛋白的功能结构域,而剪接体BRD2-X3导致鸡BRD2蛋白第一个BD结构域部分缺失。
2. 2. 2 鸡BRD2蛋白结构预测和功能结构域保守性分析结果 使用SOPMA和SWISS-MODEL分别对鸡BRD2蛋白进行二、三级结构预测,结果显示,鸡BRD2蛋白含有丰富的二级结构,其中无规则卷曲占60.59%、α-螺旋占28.50%、延伸链占7.45%、β-转角占3.47%;鸡BRD2蛋白三级结构(图4)与其二级结构相符,主要结构为α-螺旋和无规则卷曲。鸡BRD2蛋白含有3个明显的功能结构域(2个BD结构域和1个ET结构域),其中,2个BD结构域分别位于第90~162和361~433位氨基酸,ET结构域位于第610~692位氨基酸(图5)。对鸡、猪、小鼠和人类的BRD2蛋白功能结构域进行保守性分析,结果发现猪、小鼠和人类的BRD2蛋白功能结构域氨基酸位点一致,而鸡BRD2蛋白BD结构域氨基酸变异位点主要位于G137A、A139S、E385D、I396V和H406R,ET结构域氨基酸变异位点主要位于K618R、T621S和S649A。说明哺乳动物的BRD2蛋白功能结构域高度保守,而鸡BRD2蛋白功能结构域氨基酸位点的变异可能会导致其功能改变。
2. 2. 3 鸡BRD2蛋白的互作蛋白预测结果 运用STRING对鸡BRD2蛋白的互作蛋白进行预测,发现可能存在的互作蛋白有BRD3、TAP1、TCLEC2D、BLB2、CCND1、KRAS、CBFB、EP300、RNUNX3和HDAC4(图6)。其中,BRD2和BRD3同属于BET蛋白家族,参与基因转录及调节细胞生长;TAP1和BLB2位于组织相容复合体中,TAP1作为一种转运蛋白,具有肽段转运和抗原加工功能(Cresswell et al.,1999);CCND1调节细胞周期的不同阶段,并通过与周期素依赖性蛋白激酶4(CDK4)或周期素依赖性蛋白激酶6(CDK6)结合形成复合体的方式,促进细胞从G1期进入S期(Chen et al.,2012;Lin et al.,2015);KRAS参与细胞内的信号传递,控制细胞正常生长,发生异常时会导致细胞持续增殖而引发癌变(Fernández-Medarde and Santos,2011;Simanshu et al.,2017);EP300作为转录辅激活物,参与控制基因组稳定性、发育、记忆形成、神经元可塑性和细胞生长(Dancy and Cole,2016);RUNX3在T细胞发育过程中发挥重要作用,尤其是在初始双阳性T细胞分化为CD4+或CD8+单阳性细胞的阶段(Grueter et al.,2005)。
2. 2. 4 鸡BRD2基因核苷酸序列相似性和遗传进化分析结果 采用MegAlign对鸡与火鸡、日本鹌鹑、孟加拉雀等禽类的BRD2基因核苷酸序列进行相似性分析,结果(图7)显示,禽类BRD2基因核苷酸序列相似性在70.8%~95.7%,其中,鸡与火鸡的相似性最高(95.7%),而与长尾金雀的相似性最低(70.8%)。鸡与牛、马、猪、家鼠及人类等非禽类的BRD2基因核苷酸序列相似性分析结果表明,鸡与绵羊的相似性最高(72.7%),而与斑马鱼的相似性最低(64.9%)。基于BRD2基因核苷酸序列相似性构建的系统发育进化树(图8)也显示,鸡和火鸡聚类在同一分支,其遗传距离最近,与斑马鱼的遗传距离最远。
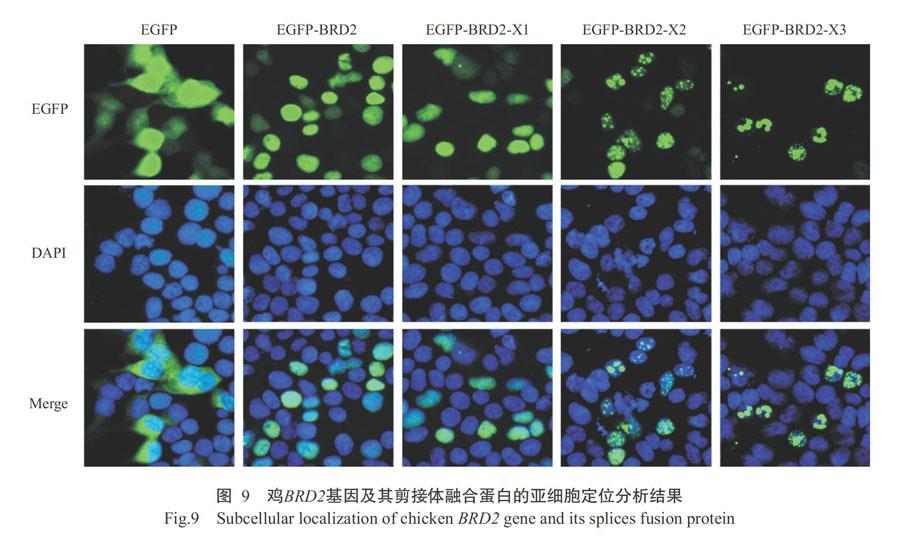
2. 3 鸡BRD2蛋白及其剪接体亚细胞定位分析结果
运用PSORT Ⅱ对鸡BRD2蛋白进行亚细胞定位预测,结果表明细胞核定位占69.60%、细胞质定位占26.10%、分泌系统囊泡定位占4.30%,即主要定位在细胞核,且鸡BRD2蛋白可能存在的细胞核定位信号位于500KKKKKKSEKHK510。以构建的鸡BRD2基因及其剪接体重组真核表达载体分别转染HEK-239T细胞24 h后,观察融合蛋白的细胞荧光,发现鸡BRD2基因融合蛋白(EGFP-BRD2)主要定位在细胞核,与预测结果一致;鸡BRD2基因剪接体融合蛋白EGFP- BRD2-X1、EGFP-BRD2-X2和EGFP-BRD2-X3同样定位在细胞核(图9)。虽然鸡BRD2蛋白及其剪接体均定位在细胞核,但EGFP-BRD2和EGFP-BRD2-X1在细胞核中呈均匀分布,而EGFP-BRD2-X2和EGFP-BRD2-X3在细胞核中呈不均匀的点状分布。
3 讨论
BRD2作为一种转录激活因子,参与基因转录、染色质重塑、抗原呈递加工、调控免疫应答、诱导免疫反应及抑制细胞活性等免疫调节(Taniguchi,2016;Pathak et al.,2018)。在真核细胞中,组蛋白赖氨酸乙酰化导致染色质的转录活性变为开放状态,从而促进基因转录(Dhalluin et al.,1999)。BD结构域是BRD2蛋白通过与染色质内核小体相互作用,而对基因转录进行表观遗传调控的主要功能结构域(Kanno et al.,2004;Jang et al.,2005)。已有大量研究表明,BRD2基因在哺乳动物体内各组织均有表达,且在B细胞和T细胞中高表达,暗示BRD2基因可能参与机体免疫功能调节(Wilhelm et al.,2014;Kim et al.,2014;Fujisawa and Filippakopoulos,2017)。BRD2蛋白ET結构域能与卡波西肉瘤疱疹病毒的主要潜伏相关核抗原1(LANA-1)相互作用,介导LANA-1与其他蛋白或核酸的相互作用(Ottinger et al.,2006)。Choi等(2015)研究发现,在脂多糖(LPS)诱导的小鼠原代星形胶质细胞免疫应答中,BRD2基因及其编码蛋白水平升高;敲除BRD2基因后,能抑制LPS诱导小鼠原代星形胶质细胞纤溶酶原激活物抑制物-1(PAI-1)的表达和分泌,表明BRD2参与细胞免疫应答。
AS在细胞内普遍存在,且大多数AS涉及蛋白结构和功能的改变(章国卫等,2004)。周扬(2017)研究发现,牛脂肪沉积相关基因TUSC5的2种剪接体在细胞质中的主要分布位置存在明显差异,进而导致其组织表达谱、在牛原代脂肪细胞分化过程中的表达规律及在293T细胞内的时序表达均存在差异。此外,小鼠BRD2基因能表达出不同的组织特异性转录本,且这些转录本启动子5'非翻译区(5'-UTR)的长度明显不同(Shang et al.,2011)。本研究成功克隆获得鸡BRD2基因的选择性剪接体BRD2-X1(第526~537位氨基酸缺失)、BRD2-X2(第1~19位氨基酸缺失)和BRD2-X3(第1~119位氨基酸缺失),提示鸡BRD2基因剪接体也可能存在组织表达特异性。BRD2蛋白功能结构域氨基酸位点变异分析结果表明,猪、小鼠和人类的BRD2蛋白功能结构域氨基酸位点一致,但在鸡BRD2蛋白中存在多个氨基酸位点变异,说明BRD2蛋白功能结构域在哺乳动物中具有高度保守性,而氨基酸位点变异可能会导致鸡BRD2蛋白在细胞中的转录调控机制不同。此外,BRD2作为一种重要的转录调节因子,可直接激活或作为辅激活因子调节基因转录。本研究对BRD2蛋白的互作蛋白预测结果显示,可能存在的互作蛋白有BRD3、TAP1、TCLEC2D、BLB2、CCND1、KRAS、CBFB、EP300、RNUNX3和HDAC4,分别涉及转录激活、控制细胞周期和调节神经系统发育等功能,但具体作用机理还需进一步探究。
本研究通过构建鸡BRD2基因及其剪接体重组真核表达载体并转染HEK-239T细胞,证实鸡BRD2基因及其剪接体融合蛋白均定位在细胞核,但EGFP-BRD2和EGFP-BRD2-X1在细胞核中呈均匀分布,而EGFP-BRD2-X2和EGFP-BRD2-X3在细胞核中呈不均匀的点状分布。何佳谨(2007)也曾研究发现,含有2个BD结构域的小鼠BRD2蛋白N端(第1~435位氨基酸)和含有ET结构域的小鼠BRD2蛋白C端(第428~798位氨基酸)均定位在细胞核,且2个BD结构域间存在1个非典型的核定位信号序列。由此推测,BRD2蛋白BD结构域缺失并不会影响其细胞核定位。BRD2蛋白同家族成员BRD4蛋白的BD结构域缺失突变后,其在细胞核内的分布模式明显改变(Dey et al.,2003),说明BD结构域在BRD4蛋白的细胞核内分布模式中发挥关键作用。本研究中,剪接体BRD2-X3导致鸡BRD2蛋白第一个BD结构域部分缺失,虽然其亚细胞定位仍位于细胞核,但定位模式由均匀分布变为不均匀的点状分布,与Dey等(2003)的研究结果一致。值得注意的是,未缺失BD结构域的剪接体BRD2-X2在细胞核中也呈不均匀的点状分布,因此鸡BRD2基因及其剪接体的细胞核定位差异还有待进一步研究。
4 结论
鸡BRD2蛋白及其剪接体均定位在细胞核,但BD结构域缺失可导致BRD2剪接体BRD2-X3在细胞核中呈不均匀的点状分布,即引起细胞核定位模式的变化。
参考文献:
何佳瑾. 2007. BRD2核定位信号的核定位功能和细胞凋亡始动功能的初步研究[D]. 长沙:中南大学. [He J J. 2007. Primary studies on fuctions of BRD2 nuclear localization signal and initiation fuction of cellular apoptosis of BRD2[D]. Changsha:Central South University.]
吴允,毕榆林,张扬,郭晓敏,李志腾,万方,朱鹏飞,徐璐,常国斌,徐琪,陈国宏. 2017. 部分雞种Brd2基因SNP和INDEL分析[J]. 安徽农业大学学报,44(2):203-207. [Wu Y,Bi Y L,Zhang Y,Guo X M,Li Z T,Wan F,Zhu P F,Xu L,Chang G B,Xu Q,Chen G H. 2017. SNP and INDEL analysis of Brd2 gene in indigenous chicken breeds[J]. Journal of Anhui Agricultural University,44(2):203-207.]
章国卫,宋怀东,陈竺. 2004. mRNA选择性剪接的分子机制[J]. 遗传学报,31(1):102-107. [Zhang G W,Song H D,Chen Z. 2004. Molecular mechanism of mRNA alternative splicing[J]. Acta Genetica Sinica,31(1):102-107.]
周扬. 2017. 秦川牛脂肪沉积相关基因筛选及可变剪接对基因表达和细胞定位的影响研究[D]. 杨凌:西北农林科技大学. [Zhou Y. 2017. Identifying genes related to adipogenesis in Qinchuan cattle and studying the effects of alternative splicing on gene expression and cellular loca-lization[D]. Yangling:Northwest A & F University.]
Belkina A C,Denis G V. 2012. BET domain co-regulators in obesity,inflammation and cancer[J]. Nature Reviews. Cancer,12(12):465-477.
Chen X M,Zhao T S,Li L,Xu C H,Zhang X L,Tse V,Zhang T,Liu X Q,Lu F M. 2012. CCND1 G870A polymorphism with altered cyclin D1 transcripts expression is associated with the risk of glioma in a Chinese population[J]. DNA and Cell Biology,31(6):1107-1113.
Choi C S,Hong S H,Sim S,Cho K S,Kim J W,Yang S M,Jeon S J,You J S,Shin C Y. 2015. The epigenetic reader BRD2 as a specific modulator of PAI-1 expression in lipopolysaccharide-stimulated mouse primary astrocytes[J]. Neurochemical Research,40(11):2211-2219.
Cresswell P,Bangia N,Dick T,Diedrich G. 1999. The nature of the MHC class I peptide loading complex[J]. Immunological Reviews,172(1):21-28.
Crowley T E,Brunori M,Rhee K,Wang X Y,Wolgemuth D J. 2004. Change in nuclear-cytoplasmic localization of a double-bromodomain protein during proliferation and di-fferentiation of mouse spinal cord and dorsal root ganglia[J]. Developmental Brain Research,149(2):93-101.
Dancy B M,Cole P A. 2016. Protein lysine acetylation by p300/CBP[J]. Chemical Reviews,115(6):2419-2452.
Dey A,Chitsaz F,Abbasi A,Misteli T,Ozato K. 2003. The double bromodomain protein Brd4 binds to acetylated chromatin during interphase and mitosis[J]. Proceedings of the National Academy of Sciences of the United States of America,100(15):8758-8763.
Dhalluin C,Carlson J E,Zeng L,He C,Aggarwal A K,Zhou M M. 1999. Structure and ligand of a histone acetyltransferase bromodomain[J]. Nature,399(6735):491-496.
Duan Z Q,Xu H X,Ji X Q,Zhao J F,Xu H Q,Hu Y,Deng S S,Hu S L,Liu X F. 2018. Importin α5 negatively regulates importin β1-mediated nuclear import of Newcastle disease virus matrix protein and viral replication and pathogenicity in chicken fibroblasts[J]. Virulence,9(1):783-803.
Dyson M H,Rose S,Mahadevan L C. 2001. Acetyllysine-binding and function of bromodomain-containing proteins in chromatin[J]. Frontiers in Bioscience,6:D853-D865.
Fernández-Medarde A,Santos E. 2011. Ras in cancer and developmental diseases[J]. Genes & Cancer,2(3):344-358.
Ferri E,Petosa C,Mckenna C E. 2016. Bromodomains:Structure,function and pharmacology of inhibition[J]. Bioche-mical Pharmacology,106:1-18
Fujisawa T,Filippakopoulos P. 2017. Functions of bromodomain-containing proteins and their roles in homeostasis and cancer[J]. Nature Reviews. Molecular Cell Biology,18(4):246-262.
Gallay K,Blotd G,Chahpazoffe M,Yajjou-Hamalian H,Confort M P,de Boisséson C,Leroux A,Luengo C,Fiorini F,Lavigne M,Chebloune Y,Gouet P,Moreau K,Blanchard Y,Ronfor C. 2019. In vitro,in cellulo and structural characterizations of the interaction between the integrase of Porcine Endogenous Retrovirus A/C and proteins of the BET family[J]. Virology,532:69-81.
Garcia-Gutierrez P,Mundi M,Garcia-Dominguez M. 2012. Association of bromodomain BET proteins with chromatin requires dimerization through the conserved motif B[J]. Journal of Cell Science,125(15):3671-3680.
Grueter B,Petter M,Egawa T,Laule-Kilian K,Aldrian C J,Wuerch A,Ludwig Y,Fukuyama H,Wardemann H,Waldschuetz R,M?r?y T,Taniuchi I,Steimle V,Littman D R,Ehlers M. 2005. Runx3 regulates integrin alpha E/CD103 and CD4 expression during development of CD4-/CD8+ T cells[J]. Journal of Immunology,175(3):1694-1705.
Jang M K,Mochizuki K,Zhou M S,Jeong H S,Brady J N,Ozato K. 2005. The bromodomain protein Brd4 is a positive regulatory component of P-TEFb and stimulates RNA polymerase II-dependent transcription[J]. Molecular Cell,19(4):523-534.
Jeanmougin F,Wurtz J M,Douarin B L,Chambon P,Losson R. 1997. The bromodomain revisited[J]. Trends in Biochemical Sciences,22(5):151-153.
Kanno T,Kanno Y,Siegel R M,Jang M K,Lenardo M J,Ozato K. 2004. Selective recognition of acetylated histones by bromodomain proteins visualized in living cells[J]. Molecular Cell,13(1):33-43.
Kelemen O,Convertini P,Zhang Z Y,Wen Y,Shen M L,Falaleeva M,Stamm S. 2013. Function of alternative splicing[J]. Gene,514(1):1-30.
Kim M S,Pinto S M,Getnet D,Nirujogi R S,Manda S S,Chaerkady R,Madugundu A K,Kelkar D S,Isserlin R,Jain S,Thomas J K,Muthusamy B,Leal-Rojas P,Kumar P,Sahasrabuddhe N A,Balakrishnan L,Advani J,George B,Renuse S,Selvan L D N,Patil A H,Nanjappa V,Radhakrishnan A,Prasad S,Subbannayya T,Raju R,Kumar M,Sreenivasamurthy S K,Marimuthu A,Sathe G J,Chavan S,Datta K K,Subbannayya Y,Sahu A,Yelamanchi S D,Jayaram S,Rajagopalan P,Sharma J,Murthy K R,Syed N,Goel R,Khan A A,Ahmad S,Dey G,Mudgal K,Chatterjee A,Huang T C,Zhong J,Wu X Y,Shaw P G,Freed D,Zahari M S,Mukherjee K K,Shankar S,Mahadevan A,Lam H,Mitchell C J,Shankar S K,Satishchandra P,Schroeder J T,Sirdeshmukh R,Maitra A,Leach S D,Drake C G,Halushka M K,Keshava Prasad T S,Hruban R H,Kerr C L,Bader G D,Iacobuzio-Donahue C A,Gowda H,Pandey A. 2014. A draft map of the human proteome[J]. Nature,509(7502):575-581.
Leroy G,Rickards B,Flint S J. 2008. The double bromodomain proteins Brd2 and Brd3 couple histone acetylation to transcription[J]. Molecular Cell,30(1):51-60.
Lin J T,Li H Y,Chang N S,Lin C H,Chen Y C,Lu P J. 2015. WWOX suppresses prostate cancer cell progression through cyclin D1-mediated cell cycle arrest in the G1 phase[J]. Cell Cycle,14(3):408-416.
Modrek B,Lee C. 2002. A genomic view of alternative spli-cing[J]. Nature Genetics,30(1):13-19.
Morgado-Pascual J L,Rayego-Mateos S,Tejedor L,Suarez-Alvarez B,Ruiz-Ortega M. 2019. Bromodomain and extraterminal proteins as novel epigenetic targets for renal di-seases[J]. Frontiers in pharmacology,10:1315. doi:10. 3389/fphar.2019.01315.
Ottinger M,Christalla T,Nathan K,Nathan K,Brinkmann M M,Viejoborbolla A,Schulz T F. 2006. Kaposi?s sarcoma-associated herpesvirus LANA-1 interacts with the short variant of BRD4 and releases cells from a BRD4- and BRD2/RING3-induced G1 cell cycle arrest[J]. Journal of Virology,80(21):10772-10786.
Pathak S,Miller J,Morris E C,Stewart W C L,Greenberg D A. 2018. DNA methylation of the BRD2 promoter is associated with juvenile myoclonic epilepsy in Caucasians[J]. Epilepsia,59(5):1011-1019.

