基于PARMS技术的绿豆抗叶斑病基因VrTAF5分子标记的开发
李灵慧,吴然然,陈景斌,崔晓艳,袁星星,朱月林,陈新
摘要:叶斑病是绿豆生产上主要的真菌性病害,利用抗性品种是防治该病害最经济、安全、有效的途径。为筛选抗病资源,根据抗性基因VrTAF5的突变位点,利用五引物扩增受阻突变体系(Penta-primer amplification refractory mutation system,PARMS)开发分子标记。并利用抗病性具有差异的164份绿豆资源对所开发的分子标记的可靠性进行检验和评价。结果表明,VrTAF5-1517分子标记在7个分子标记中的检出率最高,基因分型为AA、AC、CC,可以有效区分基因碱基序列的SNP差异。根据叶斑病抗性评价结果,该标记对抗病材料的选择效率为75.00%、感病材料的選择效率为93.42%。VrTAF5-1517分子标记对抗病资源中检出抗病基因型的比例、对高感和感病资源中检出感病基因型比例均为100%,说明本研究开发的分子标记可用于绿豆抗叶斑病分子标记辅助育种。
关键词:绿豆;叶斑病;VrTAF5基因;SNP分子标记;PARMS技术
中图分类号:S522.035.3文献标识码:A文章编号:1000-4440(2021)06-1386-07
Development of molecular markers of mung bean leaf spot disease resistance gene VrTAF5 based on PARMS technology
LI Ling-hui1,2,WU Ran-ran2,CHEN Jing-bin2,CUI Xiao-yan2,YUAN Xing-xing2,ZHU Yue-lin1,CHEN Xin2
(1.College of Horticulture, Nanjing Agricultural University, Nanjing 210095, China;2.Institute of Industrial Crops, Jiangsu Academy of Agricultural Sciences, Nanjing 210014, China)
Abstract:Cercospora leaf spot (CLS) disease is the main fungal disease in mung bean production. Using resistant varieties is the most economical, safest and effective way to control CLS disease. For selection of disease-resistant resources, molecular markers were developed based on the mutation sites of the resistance gene VrTAF5 by penta-primer amplification refractory mutation system (PARMS) technology. In addition, 164 mung bean resources with different disease resistance were used to test and evaluate the reliability of the developed molecular markers. The results showed that the detection rate of VrTAF5-1517 was the highest among the seven molecular markers, and the genotypes were AA, AC and CC. Moreover, SNP differences in gene base sequences could be effectively distonguished. According to the evaluation of leaf spot resistance, the selection efficiency of VrTAF5-1517 for resistant and susceptible accessions was 75% and 93.42%, respectively. The proportions of resistant genotypes detected in resistant resources and susceptoble genotypes detected in highly susceptible and susceptible resources by VrTAF5-1517 were 100%, suggestingt that the molecular markers developed in this study could be used in molecular marker-assisted breeding for resistance to CLS disease of mung bean.
Key words:mung bean;Cercospora leaf spot disease;VrTAF5 gene;SNP molecular marker;PARMS technology
叶斑病是中国绿豆生产上的主要真菌性病害,其病原菌为变灰尾孢菌(Cercosporacanescens Ell.etMartin),为害植株的叶片,以开花结荚期为害严重,可导致叶片早落、豆荚数目减少及种子变小[1-3]。该病害在高温高湿环境下发生与传播迅速,造成严重的产量损失,绿豆减产可超过50%[4]。
叶斑病抗性品种能够有效减轻病害的发生[5],但抗病育种的效率受到抗病种质资源较少的制约。Hartman等对近4 000份绿豆资源进行抗病性鉴定,得到的抗叶斑病资源不足4%[6],李怡琳和李淑英对200份绿豆资源进行田间抗病性鉴定,表现抗性的资源仅占6%[7]。由于绿豆叶斑病的抗病性鉴定费时费力,田间鉴定还易受到环境的影响,因此相较于表型鉴定,分子标记具有准确、经济、快速的优势。
抗性基因的发掘为分子标记的开发提供了理论依据。南海洋等通过大豆胞囊线虫病抗性候选基因rhg1开发InDel标记,对抗病资源与感病资源的选择效率分别为88.2%和100.0%[8]。在绿豆叶斑病抗性基因的研究中,Chankaew等利用抗病品种V4718与感病品种KPS1杂交,在所得的F2与BC1F1群体中发现了1个主效QTL——qCLS,可以解释65.5%-80.5%的抗病表型变异[9]。在本实验室的前期工作中,Yundaeng等通过F2群体与BC8F2群体将该QTL精细定位到绿豆6号染色体上,候选基因VrTAF5[编码TATA结合蛋白相关因子5(TAF5),注释基因ID为LOC106765332],是转录起始因子IID(Transcription initiation factor IID, TFIID)和Spt-Ada-Gcn5乙酰转移酶(SAGA)复合物的亚基[10]。通过抗病亲本V4718与感病亲本KPS1的VrTAF5基因碱基序列比对发现,这两份材料在外显子区域存在7个单核苷酸的突变[10]。五引物扩增受阻突变体系(Penta-primer amplification refractory mutation system,PARMS)是一种新型SNP分型技术,属于第三代分子标记[11],具有准确性高、成本低廉、高通量等优势,近年来在农学和医学领域被广泛应用[12-13]。
目前,将PARMS技术应用于绿豆抗叶斑病资源筛选的分子标记开发尚未见报道。本研究中,基于定位亲本V4718和KPS1在VrTAF5基因外显子上7个单核苷酸的差异,利用PARMS技术开发分子标记,并对164份绿豆种质资源进行抗性检测,以期为绿豆抗叶斑病分子标记辅助育种提供依据。
1材料与方法
1.1试验材料
供试的绿豆[Vigna radiata (L.)Wilczek]资源共164份,均来自江苏省农业科学院经济作物研究所,其中V4718与KPS1分别作为抗病对照和感病对照。接种所用的变灰尾孢菌为江苏省农业科学院经济作物研究所保存。
1.2接种方法及抗病性评价标准
接种于2020年6月至10月在江苏省农业科学院光温可控的温室进行,采用菌丝块微伤法接种复叶期绿豆植株,接种后保湿36 h,之后于温度25 ℃、湿度60%的环境中正常生長,接种15 d后统计发病情况。每份绿豆资源3次重复,每个重复6株,抗感评价方法参考刘振兴等的方法[14]。病情分级标准为:0级:叶片上无病斑;1级:叶片上仅有小点状病斑,病斑面积占叶面积不足2%;3级:病斑直径1.0~2.0 mm,无褪绿晕圈,病斑面积占叶面积2%~25%;5级:病斑较大,直径2.1~5.0 mm,有褪绿晕圈,病斑面积占叶面积26%~50%;7级:病斑直径5.1 mm以上,病斑面积占叶面积51%~75%;9级:病斑相连成片,病斑面积占叶面积76%以上,严重落叶。根据病情指数划分抗病等级:高抗(HR):病情指数0~2.0;抗病(R):病情指数2.1~20.0;中抗(MR):病情指数20.1~40.0;中感(MS):病情指数40.1~60.0;感病(S):病情指数60.1~80.0;高感(HS):病情指数80.1~100.0。
病情指数=∑(各级病株数×该病级值)/(调查总株数×最高病级值)×100
1.3绿豆DNA的提取
采用改良的CTAB法提取164份绿豆资源的DNA,利用KAIAO超微量分光光度计检测DNA浓度和质量,DNA于-20 ℃保存备用。
1.4PARMS引物设计与反应
利用Primer 5.0软件根据7个SNP位点的侧翼序列信息设计PARMS引物,采用高通量SNP检测平台GeneMatrix进行样品检测,两次重复。PCR反应体系为10.00 μl:5.00 μl 2×PARMSmaster mix,Allele X primer(10 μmol/L)与Allele Y primer(10 μmol/L)各0.15 μl,0.40 μl Common primer(10 μmol/L),1.00 μl模板DNA(50 ng/μl),3.30 μl ddH2O。PCR反应扩增程序:94 ℃热激活15 min,94 ℃变性20 s,57~65 ℃退火和延伸1 min,10个循环;94 ℃变性20 s,57 ℃退火和延伸1 min,30个循环。使用TECAN infinite M1000酶标仪读取荧光信号,在软件Snpdecoder(http://www.snpway.com/snpdecoder/)中解析转换荧光信号,得到清晰直观的分型图,并根据颜色不同,输出基因型结果。
1.5PARMS标记的验证
选择4份绿豆材料对PARMS分型结果进行测序验证,PCR扩增体系为50 μl:25 μl 2×Phanta Max Buffer, 1 μl dNTP Mix ,正、反向引物各2 μl, 1 μl模板DNA , 1 μl Phanta Max Super-Fidelity DNA Polymerase , 18 μl ddH2O。反应程序:95 ℃预变性3 min,95 ℃变性15 s,56~60 ℃退火15 s,72 ℃延伸45 s,72 ℃终延伸5 min,35个循环。扩增产物切胶回收,送至上海生工生物工程有限公司南京部测序(测序引物参照参考文献[10])。
1.6数据分析
采用Excel 2019进行数据分析与制表作图,采用SPSS 22.0进行聚类分析与方差分析,Chromas2输出测序峰图,BioXM 2.6进行序列拼接与比对。
2结果与分析
2.1VrTAF5分子标记开发
通过抗病对照V4718和感病对照KPS1中VrTAF5基因碱基序列比对,对得到的7个SNP及侧翼序列设计了7组PARMS引物(表1)。每组引物包含2条携带不同荧光信号的等位基因特异引物,1条通用反向引物。抗病对照V4718与感病对照KPS1均可被7个标记明确区分,表明标记开发成功。
2.2VrTAF5分子标记的通用性
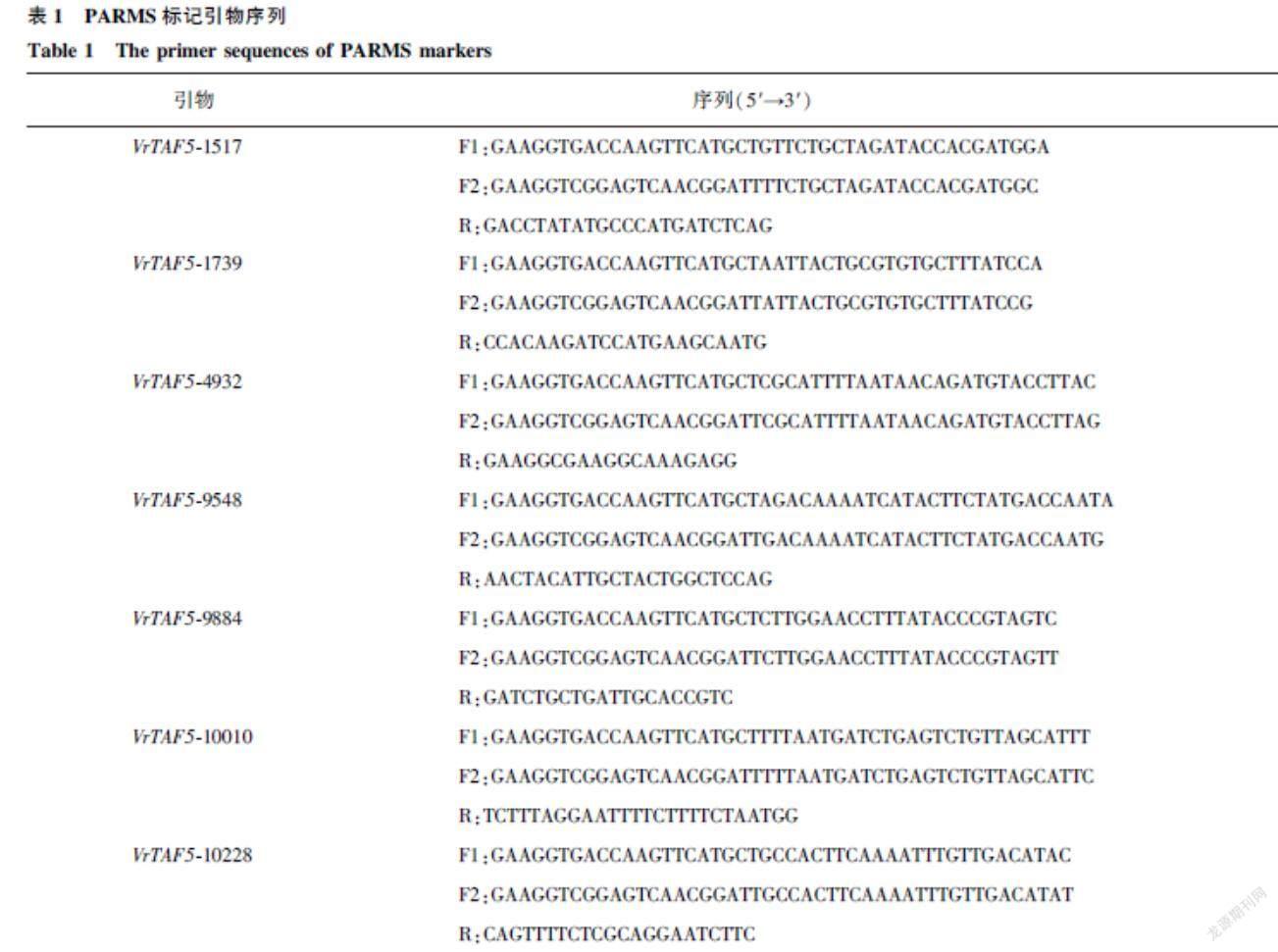
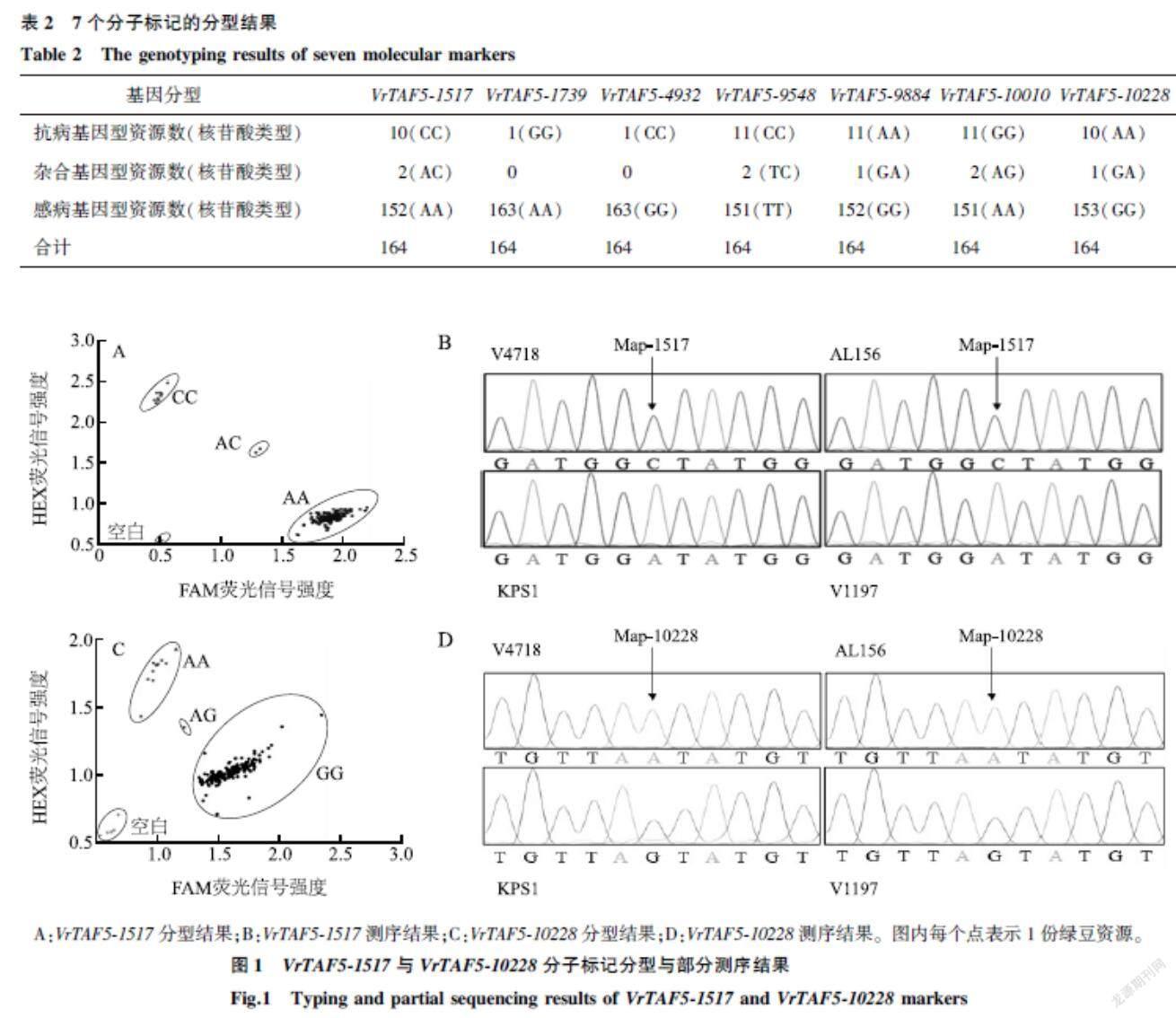
为验证分子标记的通用性与准确性,将开发的标记对164份绿豆资源进行基因分型,并通过测序方法验证其可靠性。表2结果显示,在供试绿豆资源中7个分子标记均能有效区分对应的SNP差异。VrTAF5-1517分型结果:10份绿豆资源为抗病基因型,2份为杂合基因型,152份为感病基因型;VrTAF5-10228分型结果:10份绿豆资源为抗病基因型,1份为杂合基因型,153份为感病基因型;VrTAF5-9884分型结果:152份感病基因型,1份杂合基因型,11份抗病基因型。VrTAF5-9548与VrTAF5-10010的分型结果:11份为抗病基因型,151份为感病基因型,2份杂合基因型。而在VrTAF5-1739和VrTAF5-4932分型结果中,只有抗性对照V4718显示出与其他资源不同的基因分型,结合抗感情况,这两个标记的分型效率极低。同时取10份绿豆材料测序验证VrTAF5-1517分子标记的特异性及准确性,取4份材料测序验证剩余6个分子标记,测序结果与分型结果完全吻合,说明7个分子标记的分型结果可靠。以VrTAF5-1517和VrTAF5-10228分子标记为例,基因分型图与部分品種的测序峰图见图1。
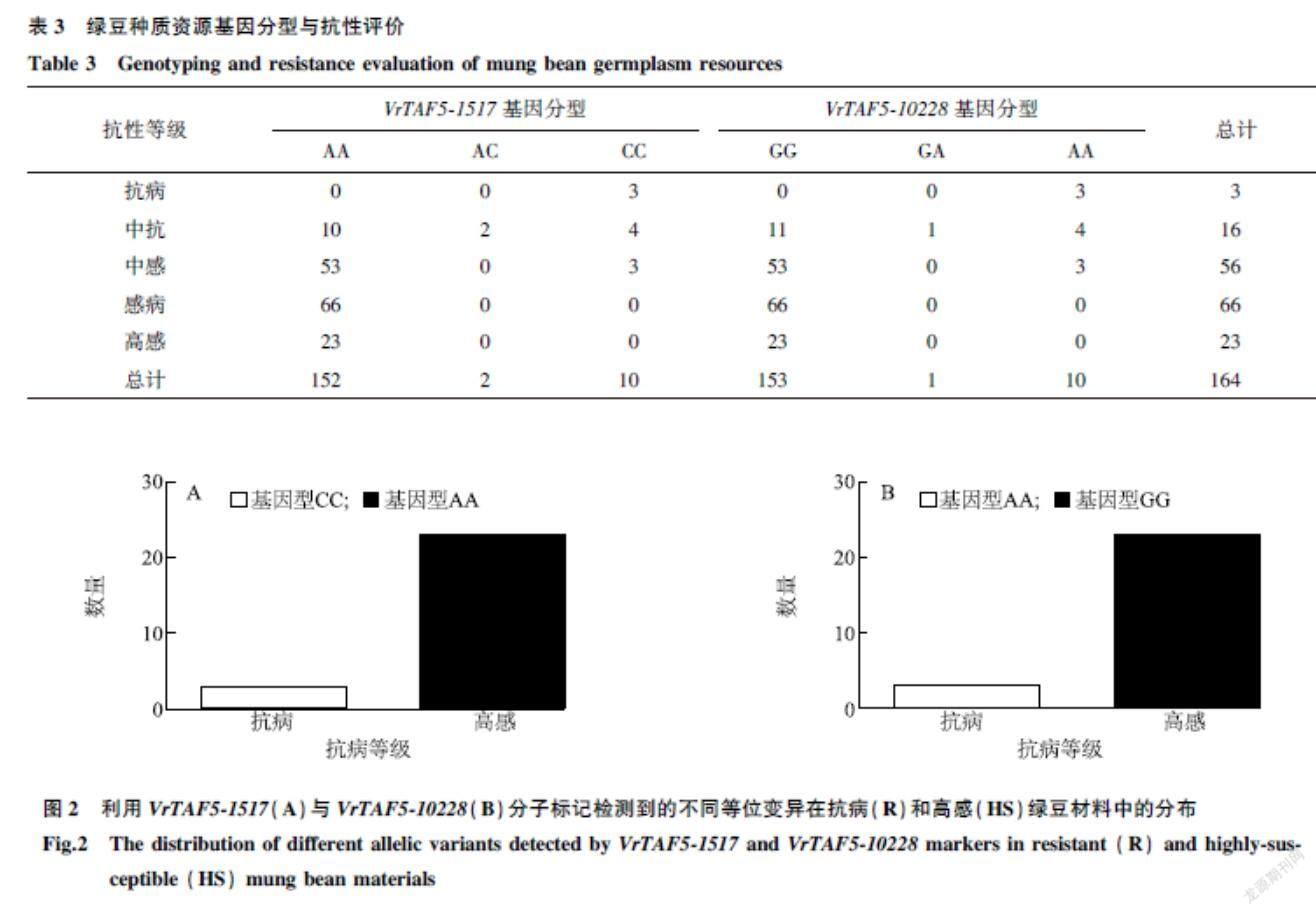
2.3VrTAF5分子标记评价
根据7个分子标记基因分型结果与抗病性鉴定结果的比对,发现VrTAF5-1517分子标记最佳,可以作为叶斑病抗性资源的筛选分子标记。VrTAF5-10228分子标记次之,可作为VrTAF5-1517分子标记的补充。具体结果如表3所示,VrTAF5-1517对抗病材料的选择效率为75.00%,对感病材料的选择效率为93.42%,感病基因型与剩余基因型(杂合及抗病)在病情指数上存在极显著差异(F=37.928,P<0.01)。从不同抗性群体的基因分型来看,抗病、中抗绿豆资源中抗病基因型的比例分别为100.00%、37.50%,高感、感病、中感绿豆资源中感病基因型比例分别为100.00%、100.00%、94.64%。VrTAF5-10228对抗病材料的选择效率为72.73%,对感病材料的选择效率为92.81%。以VrTAF5-1517和VrTAF5-10228分子标记为例,它们能够准确对绿豆抗病材料(R)与高感材料(HS)进行分型(图2)。
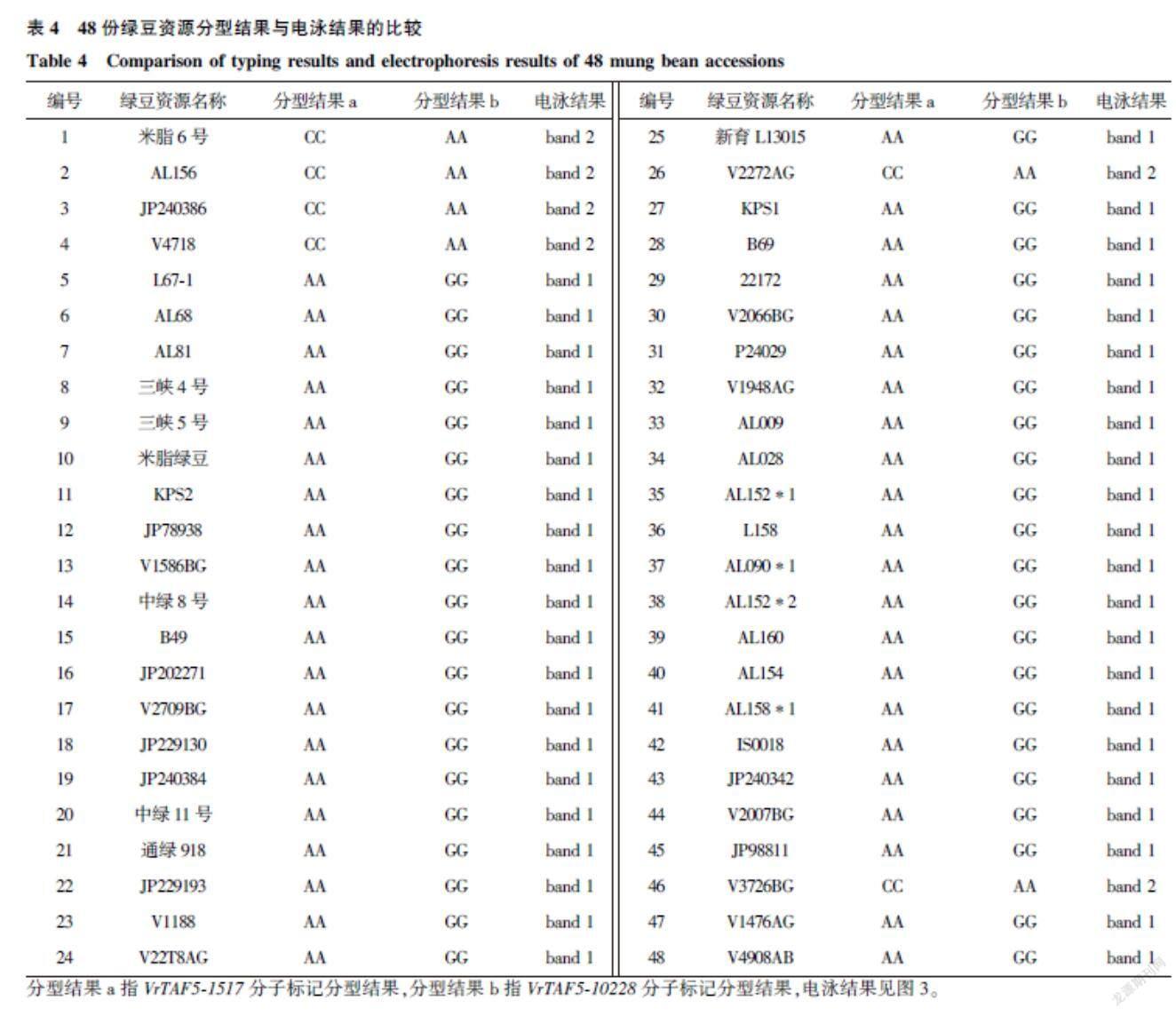
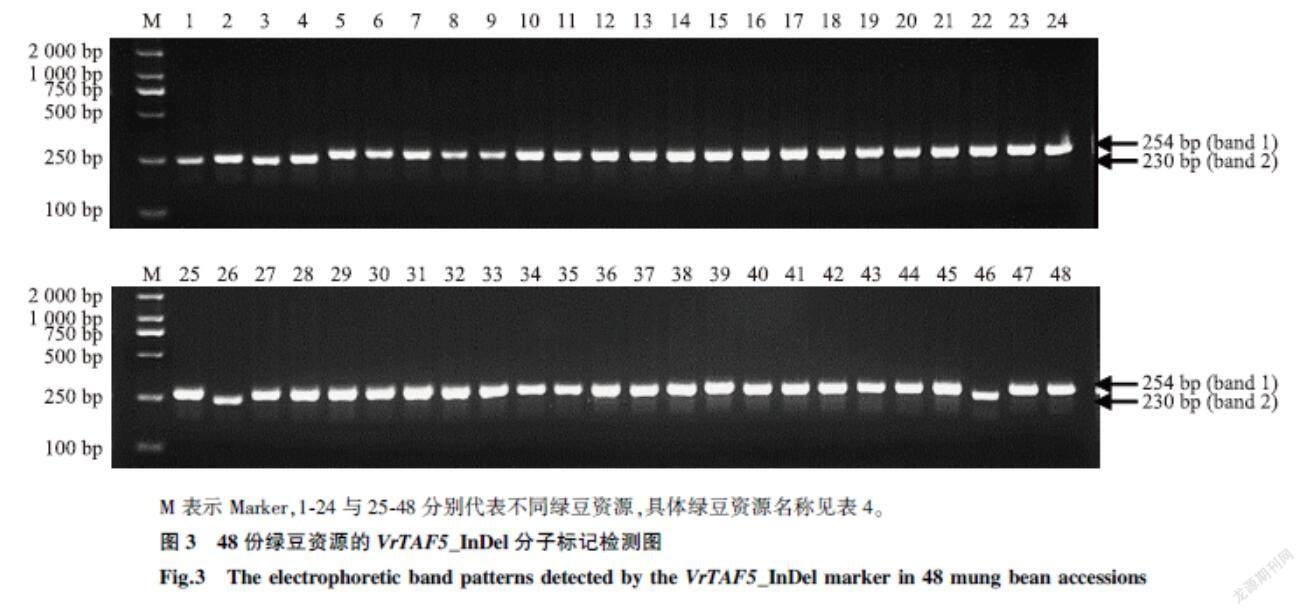
在本实验室的前期工作中,Yundaeng等发现在定位群体(F2群体与BC8F2群体)内VrTAF5_InDel标记(F:5′-CTCATGAAACCTGGAGAACT-3′,R:5′-CCCAGTGTACTCAGTTTGACTT-3′)与叶斑病抗性完全连锁[10]。在本研究中的供试材料中选取48份绿豆资源,利用VrTAF5_InDel标记进行检测,结果显示VrTAF5_InDel分子标记与VrTAF5-1517、VrTAF5-10228分子标记得到的结果完全一致(表4、图3),进一步验证了所开发的分子标记的准确性。
3讨论
绿豆叶斑病的主要致病菌为尾孢菌属的变灰尾孢菌[3],感染的最佳温度为25~30 ℃[15]。同时由于变灰尾孢菌为死体营养型[16],需要穿过叶片表皮的物理屏障才可以引发叶斑病。故本研究中在人工接种前,利用无菌牙签对叶片的表皮造成微伤口,有效规避了由于不同绿豆资源的表皮硬度差异引起的表型鉴定偏差,适用于抗病绿豆资源的筛选[17]。
分子标记技术不仅在基因定位方面有着重要作用,同时可以作为优良品种的选育工具[18-20]。如魏广辉等在小麦富硒基因QTL定位基础上开发出AX-1标记,该标记显示高硒基因型在高硒材料中占比81% [21];卿冬进等开发出水稻抗稻瘟病基因Pigm的分子标记,该分子标记显示的1份抗病基因型材料表现为抗病,47份感病基因型材料中有36份表现为感病[22]。王文辉等开发出1个SSR标记Satt309,与抗大豆胞囊线虫病的主效基因rhg1紧密连锁,该分子标记对抗病材料的鉴定效率达72.5%[23]。PARMS技术为新型的SNP分型技术[24],被广泛应用在油菜[25]、小麦[26]、水稻[27]等研究中,在绿豆中应用此技术的研究鲜有报道。且绿豆叶斑病抗性基因研究难度较大,在很大程度上限制了抗绿豆叶斑病优良品种的选育。本研究开发的VrTAF5-1517分子标记为绿豆叶斑病抗性相关的高通量分子标记,从操作便捷程度和经济成本上均优于传统分子标记。在实际应用中,可以利用该标记先筛选出抗病基因型材料,再结合抗病性鉴定筛选出叶斑病抗性资源,可有效减少抗病性鉴定的工作量。值得注意的是VrTAF5-1517分型结果,152份感病基因型的绿豆种质资源中,有10份绿豆资源表现为抗叶斑病,暗示了绿豆叶斑病的抗性机制非常复杂,可能存在其他抗病途径。因此,在后续研究工作中仍需进一步挖掘绿豆抗叶斑病的其他基因。
参考文献:
[1]ABBAS H, IQBAL M A, KAMRAN M, et al. Evaluation of advanced mungbean germplasm against Cercospora leaf spot and its in-vitro management by different fungicides[J]. Pakistan Journal of Agricultural Research, 2020, 33(4): 872-877.
[2]CHAND R, SINGH V, PAL C, et al. First report of a new pathogenic variant of Cercospora canescens on mungbean (Vigna radiata) from India[J]. New Disease Reports, 2012, 26: 6.
[3]刘昌燕,肖炎农,吴小微,等. 绿豆叶斑病病原鉴定及生物学特性研究[J]. 植物保护, 2015, 41(6): 83-87.
[4]KUMAR N, KUMAR S, PRAJAPATI S, et al. Cercospora leaf spot disease of green gram and its management: a review[J]. Journal of Pharmacognosy and Phytochemistry, 2020, 9(1): 1574-1576.
[5]YADAV D L, PANDEY R N, JAISANI P, et al. Sources of resistance in mungbean genotypes to Cercospora leaf spot disease and its management[J]. African Journal of Agricultural Research, 2014, 9(41): 3111-3114.
[6]HARTMAN G L, WANG T C, KIM D. Field evaluation of mungbeans for resistance to Cercospora leaf spot and powdery mildew[J]. International Journal of Pest Management, 1993, 39(4): 418-421.
[7]李怡琳,李淑英. 綠豆品种抗叶斑病鉴定研究[J]. 作物品种资源, 1987, 8(4): 12-14.
[8]南海洋,李英慧,常汝镇,等. 基于大豆胞囊线虫病抗性候选基因rhg1的InDel标记开发与鉴定[J]. 作物学报, 2009, 35(7): 1236-1243.
[9]CHANKAEW S, SOMTA P, SORAJJAPINUN W, et al. Quantitative trait loci mapping of Cercospora leaf spot resistance in mungbean, Vigna radiata (L.) Wilczek[J]. Molecular Breeding, 2011, 28(2): 255-264.
[10]YUNDAENG C, SOMTA P, CHEN J B, et al. Fine mapping of QTL conferring Cercospora leaf spot disease resistance in mungbean revealed TAF5 as candidate gene for the resistance[J]. Theoretical and Applied Genetics, 2020, 134(2): 701-714.
[11]YE S, DHILLON S, KE X, et al. An efficient procedure for genotyping single nucleotide polymorphisms[J]. Nucleic Acids Research, 2001, 29(17): e88.
[12]LIJAVETZKY D, CABEZAS J A, IBANEZ A, et al. High throughput SNP discovery and genotyping in grapevine (Vitis vinifera L.) by combining a re-sequencing approach and SNPlex technology[J]. BMC Genomics, 2007, 8: 424.
[13]SIMS D, SUDBERY I, ILOTT N E, et al. Sequencing depth and coverage: key considerations in genomic analyses[J]. Nature Reviews Genetics, 2014, 15(2): 121-132.
[14]刘振兴,周桂梅,陈健,等. 几种生物农药对绿豆叶斑病的防治效果[J]. 作物杂志, 2018, 6(187): 154-157.
[15]WINDELS C E, LAMEY H A, HILDE D, et al. A Cercospora leaf spot model for sugar beet: in practice by an industry[J]. Plant Disease, 1998, 82(7): 716-726.
[16]张海涛. 绿豆响应变灰尾孢菌侵染的防卫酶活性变化及抗性基因表达模式分析[D]. 大庆:黑龙江八一农垦大学, 2017.
[17]贺闽,尹俊杰,冯志明,等. 水稻稻瘟病和纹枯病抗性鉴定方法[J]. 植物学报, 2020, 55(5): 577-587.
[18]孙正文,黄兴奇,李维蛟,等. 分子标记技术及其在水稻基因定位上的应用[J]. 基因组学与应用生物学, 2011, 30(1): 78-86.
[19]黄其椿,卢东长城,陈东奎,等. 沃柑SSR分子标记筛选及其在品种鉴定上的应用[J].江苏农业科学,2020,48(1):75-79.
[20]田孟祥,张时龙,何友勋,等. 水稻耐低温基因bZIP73分子标记的开发与验证[J].江苏农业学报,2019,35(6):1265-1270.
[21]魏广辉,李执,陈强,等. 人工合成小麦SHW-L1高硒含量KASP分子标记开发及其应用[J]. 中国农业科学, 2020, 53(20): 4103-4112.
[22]卿冬进,刘开强,杨燕宇,等. 基于PARMS技术的抗稻瘟病基因Pigm分子标记的开发[J]. 西南农业学报, 2018, 31(8): 1617-1621.
[23]王文辉,邱丽娟,常汝镇,等. 中国大豆种质抗SCN基因rhg1位点SSR标记等位变异特点分析[J]. 大豆科学, 2003, 22(4):246-250.
[24]LU J, HOU J, OUYANG Y D, et al. A direct PCR-based SNP marker-assisted selection system(D-MAS)for different crops[J]. Molecular Breeding, 2020, 40(1): 9.
[25]CHAI L, FENG B, LIU X , et al. Fine mapping of a locus underlying the ectopic blade-like outgrowths on leaf and screening its candidate genes in rapeseed (Brassica napus L.)[J]. Frontiers in Plant Science, 2021, 11: 616844.
[26]JIA M J, YANG L J, ZHANG W, et al. Genome-wide association analysis of stripe rust resistance in modern Chinese wheat[J]. BMC Plant Biology, 2020, 20(1): 491.
[27]卿冬進,刘开强,邓国富,等. 基于PARMS技术的水稻粒形基因GW8分子标记的开发[J]. 西南农业学报, 2019, 32(3): 463-469.
(责任编辑:蒋永忠)
收稿日期:2020-03-25
基金项目:国家重点研发计划项目(2019YFD1001301、2019YFD1001300);国家食用豆产业技术体系生物防治与综合防控岗位科学家项目(CARS-08-G15);江苏特粮特经产业技术体系集成创新中心项目[JATS(2019)399];江苏高校优势学科建设工程资助项目
作者简介:李灵慧(1995-),男,山东德州人,硕士研究生,从事植物分子育种研究。(E-mail)2582355776@qq.com
通讯作者:朱月林,(E-mail)ylzhu@njau.edu.cn;陈新,(E-mail)cx@jaas.ac.cn

