凡纳滨对虾进口亲虾子一代群体的遗传变异分析
徐煜 黄小帅 胡晓娟 徐武杰 苏浩昌 文国樑 曹煜成 杨铿






摘要:【目的】明确凡纳滨对虾(Litopenaeus vannamei)进口亲虾直接繁殖子一代种苗群体的遗传变异情况,旨在评估遗传特征与养殖生产性能的关联性,为我国养殖对虾优质种苗大数据建设及质量评估体系的完善提供基础数据支持。【方法】以来源于正大神湾虾苗场的Stock 1~Stock 5群体、正大漳浦虾苗场的Stock 6群体、海尚种苗培育基地的Stock 7和Stock 8群體及中海水产种苗科技有限公司的Stock 9群体等9个不同批次凡纳滨对虾进口亲虾子一代种苗为研究对象,利用11个微卫星分子标记(M1、Pvan1758、Pvan1815、HLJN-008、HLJN-023、TUMXLv7.121、TUMXLv8.256、TUMXLv9.43、TUMXLv10.312、TUYFLvL16.1a和TUDGLv1-3.224)对不同凡纳滨对虾种苗群体进行遗传变异分析。【结果】9个凡纳滨对虾种苗群体的等位基因数(Na)、有效等位基因数(Ne)、观测杂合度(Ho)、期望杂合度(He)、Shannon’s信息指数(I)和多态信息含量(PIC)的平均值分别在5.5455~8.8182、3.2795~5.4735、0.3769~0.5182、0.6189~0.6948、1.2823~1.6092和0.5863~0.6646,即各凡纳滨对虾种苗群体均具有较高的遗传多样性,具体排序为Stock 3>Stock 2>Stock 4>Stock 5>Stock 7>Stock 6>Stock 9>Stock 8>Stock 1。在9个凡纳滨对虾种苗群体中有91.82%的遗传变异源自群体内,仅有8.18%的变异源自群体间。Stock 2、Stock 3、Stock 4和Stock 5等4个群体间及Stock 7群体与Stock 8群体间的遗传分化系数(Fst)均小于0.05(0.0079~0.0371),即群体间无遗传分化,而其他群体间表现为轻度遗传分化(0.05<Fst <0.15)。基于Nei’s遗传距离,9个凡纳滨对虾种苗群体大致可划分为三大分支,第一分支包括Stock 1~Stock 6群体,第二分支由Stock 7群体和Stock 8群体组成,而Stock 9群体独立为第三分支。【结论】不同批次凡纳滨对虾进口亲虾子一代种苗群体的遗传特征存在明显差异,各群体间的遗传距离随着采样时间和地点的改变而发生明显变化,且这些遗传背景差异可能会造成不同群体在养殖生产性能方面产生差异。
关键词: 凡纳滨对虾;进口亲虾;子一代群体;遗传变异;微卫星分子标记
中图分类号: S945.49 文献标志码: A 文章编号:2095-1191(2021)11-3139-08
Genetic variation analysis among first filial generation of introduced Litopenaeus vannamei stocks
XU Yu1,2, HUANG Xiao-shuai1, HU Xiao-juan1,2, XU Wu-jie1,2, SU Hao-chang1,2,
WEN Guo-liang1, CAO Yu-cheng1,2*, YANG Keng1*
(1South China Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences/Key Laboratory of South China Sea Fishery Resources Exploitation & Utilization, Ministry of Agriculture and Rural Affairs/Guangdong Provincial Key Laboratory of Fishery Ecology and Environment, Guangzhou 510300, China; 2 Shenzhen Base of South China Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Shenzhen, Guangdong 518121, China)
Abstract:【Objective】The main purpose of this study was to analyze and assess the genetic diversity of first filial ge-neration of Litopenaeus vannamei, in order to evaluate the correlation between its genetic characteristics and aquaculture production performance, and provide basic data support for the big data construction and establishment of quality evaluation system of high-quality post-larvae in China. 【Method】The objects were L. vannamei Stock 1-Stock 5 from Zhengda Shenwan shrimp larvae farm, Stock 6 from Zhengda Zhangpu shrimp larvae farm, Stock 7 and Stock 8 from Haishang shrimp larvae farm, Stock 9 from Zhonghai aquatic larvae culture company. The genetic variation of nine first filial gene-ration stocks were investigated using eleven microsatellite DNA markers(M1, Pvan1758, Pvan1815, HLJN-008, HLJN-023, TUMXLv7.121, TUMXLv8.256, TUMXLv9.43, TUMXLv10.312, TUYFLvL16.1a and TUDGLv1-3.224). 【Result】This paper showed that the average values of the number of alleles (Na), number of effective alleles (Ne), observed heterozygosity (Ho), expected heterozygosity (He), Shannon’s diversity index (I), and polymorphism information content (PIC) were among 5.5455-8.8182, 3.2795-5.4735, 0.3769-0.5182, 0.6189-0.6948, 1.2823-1.6092 and 0.5863-0.6646, respectively. That presented a high genetic diversity of these filial generation of introduced L. vannamei stocks, and according to the richness, they ranked as follows: Stock 3>Stock 2>Stock 4>Stock 5>Stock 7>Stock 6>Stock 9>Stock 8>Stock 1. AMOVA analysis indicated that the genetic variation within stock was 91.82% and the remaining 8.18% of the variation occurred among stocks. The genetic differentiation among Stocks 2-5 and among Stocks 7-8 showed a low level based on their Fst values (0.0079-0.0371), which were lower than 0.05, whereas the other stocks had no genetic differentiation (0.05<Fst <0.15). Based on Nei’s genetic distance, the nine stocks could be roughly divided into three branches: the first branch including Stock 1 - Stock 6 stocks, the second branch being composed of Stock 7 and Stock 8, and the third branch only included Stock 9. 【Conclusion】These findings indicated that different batches of first filial generations have distinct genetic characteristics. Dramatically, genetic distance of first filial generations are clearly fluctuated with the breeding farm and breeding time. Changes of genetic background might directly affect the shrimp traits in subsequent farming.
Key words: Litopenaeus vannamei; exotic parents; first filial generation; genetic variation; microsatellite molecular marker
Foundation item: Central Public-interest Scientific Institution Basal Research Fund of Chinese Academy of Fishery Sciences(2020TD54); China Agriculture Research System of MOF and MARA(CARS-48); Guangdong Provincial Special Fund for Modern Agriculture Industry Technology Innovation Teams (2019KJ149)
0 引言
【研究意義】凡纳滨对虾(Litopenaeus vannamei)又称南美白对虾,隶属于十足目(Decapoda)游泳亚目(Natantia)对虾科(Penaeidae),是当今最主要的对虾养殖品种。凡纳滨对虾自1988年引入后逐步发展成为我国对虾养殖产业的主导品种,近年来我国的凡纳滨对虾养殖产量稳居世界首位(代平等,2018)。凡纳滨对虾为热带型种类,原产自中南美洲太平洋沿岸水域,其优良种质资源长期受到国外育种机构的垄断(王兴强等,2004;代平等,2018;彭敏等,2020)。当前全球凡纳滨对虾种虾资源主要分布在美国的夏威夷、迈阿密和关岛,以及泰国、新加坡和印尼,我国每年需从境外进口大量亲虾,由进口亲虾直接繁育子一代种苗具有生长速度快、养成群体个头均匀等优点,在我国对虾养殖业中占有较高的市场份额。但受种质资源退化及养殖环境恶化等因素的影响,对虾养殖业面临病害频发、养殖风险剧增等突出问题(王丽花等,2012;文国樑等,2015;Qiu et al.,2017)。从不同批次进口亲虾子一代种苗的养殖实践来看,虾苗的成活率、抗逆性及生长周期等表现良莠不齐,严重影响了养殖经济效益。因此,针对凡纳滨对虾进口亲虾直接繁殖子一代种苗群体开展遗传变异分析,对评估其遗传特征与养殖生产性能的关联性具有重要意义。【前人研究进展】微卫星分子标记分析具有简便、快捷及稳定性好等优点,已广泛应用于种质资源评价和遗传育种等研究领域(孙效文等,2008;赵志英等,2018;周大颜等,2019)。近年来,微卫星分子标记也常应用于水生生物遗传多样性分析。刘连为等(2014)利用8个微卫星位点对北太平洋柔鱼(Ommastrephes bartramii)的6个群体进行分析,结果发现群体间具有较高的遗传多样性。毛守康等(2016)利用14个微卫星位点对4个野生梭鱼(Liza haematocheila)地理群体进行遗传多样性分析,结果表明,4个群体遗传多样性处于中等多态水平,且按其地理位置由北向南依次聚为一类。Wang等(2016)利用8个微卫星位点分析中国对虾亲本和回捕群体的遗传多样性,发现这2个种群拥有丰富遗传多样性,但存在较高程度的近亲繁殖,且在中国对虾种群中呈现一定的近交衰退。黄小芳等(2020)利用8对微卫星引物对广西不同地区克氏原螯虾群体的遗传多样性进行分析,结果发现各克氏原螯虾群体具有一定的遗传多样性,但其杂合子缺失现象普遍存在,应通过适当引种及加强不同地区群体间的基因交流,保护好优良种质资源。在凡纳滨对虾方面,童馨等(2007,2009)在研究不同世代凡纳滨对虾养殖群体生长性状变异的基础上,利用微卫星分子标记分析种群遗传多样性的变化,发现随着繁育世代增加,凡纳滨对虾的生长性状逐代分化,生长速度变缓,群体遗传多样性降低,种质发生退化;吴怡迪等(2016)基于微卫星分子标记对凡纳滨对虾选育系间杂交的生长性状及遗传多样性进行分析,结果表明,凡纳滨对虾的生长性能与群体遗传多样性水平呈显著正相关,选育系间杂交可使杂交后代的生长性状和遗传多样性水平得到改善;李强勇等(2020)基于第三代高通量测序技术——单分子实时(SMRT)测序开发出凡纳滨对虾微卫星分子标记,有助于开展凡纳滨对虾遗传育种研究工作;彭敏等(2020)从GenBank中筛选11对能有效扩增的微卫星引物对2016—2018年桂海1号凡纳滨对虾连续3个世代选育群体进行遗传多样性分析,监测相邻2个世代选育群体间的遗传变异情况,结果显示桂海1号凡纳滨对虾各世代选育群体尚具有较丰富的遗传变异,但群体遗传结构处于不稳定状态。【本研究切入点】遗传变异是影响对虾养殖性能表现的重要因素,但近年来鲜见从遗传变异水平研究进口亲虾子一代种苗养殖性能差异的问题。【拟解决的关键问题】选取不同批次凡纳滨对虾进口亲虾子一代种苗群体,利用微卫星分子标记分析不同群体的遗传变异及其多样性水平,旨在评估遗传特征与养殖生产性能的关联性,为我国养殖对虾优质种苗大数据建设及质量评估体系的完善提供基础数据支持。
1 材料与方法
1. 1 试验材料
供试凡纳滨对虾种苗群体为2017—2018年收集于广东中山、茂名、汕尾及福建漳州等地的虾苗场或养殖场,共9个群体,分别命名为Stock 1~Stock 9。各凡纳滨对虾种苗群体样品具体信息如表1所示。
1. 2 DNA提取
取凡纳滨对虾种苗腹部组织10~30 mg,参照E.Z.N.A. Tissue DNA Kit (OMEGA Bio-tek)试剂盒说明提取各样品基因组DNA。采用1.0%琼脂糖凝胶电泳检测DNA完整性,以NanoDrop 2000紫外分光光度计测定其浓度,-80 ℃保存备用。
1. 3 微卫星位点
根据相关文献报道,选取凡纳滨对虾中多态性高且扩增效果好的11个微卫星位点:M1(Garcia et al.,1996;Alcivar-Warren et al.,2007a)、Pvan1758(Cruz et al.,2002)、Pvan1815(Cruz et al.,2002)、HLJN-008(Jia et al.,2006)、HLJN-023(Jia et al.,2006)、TUMXLv7.121(Meehan et al.,2003)、TUMX Lv8.256(Meehan et al.,2003;Alcivar-Warren et al.,2007b)、TUMXLv9.43(Meehan et al.,2003;Alcivar-Warren et al.,2007b)、TUMXLv10.312(Meehan et al.,2003;Alcivar-Warren et al.,2007b)、TUYFLvL16.1a(Alcivar-Warren et al.,2007b)和TUDGLv1-3.224(Alcivar-Warren et al.,2007b;Garcia et al.,2007),在各位点上游引物5'端分别进行6-FAM、ROX、HEX或TAMRA荧光标记,引物序列及荧光标记类型详见表2,并委托北京睿博兴科生物技术有限公司合成。
1. 4 PCR扩增及产物检测
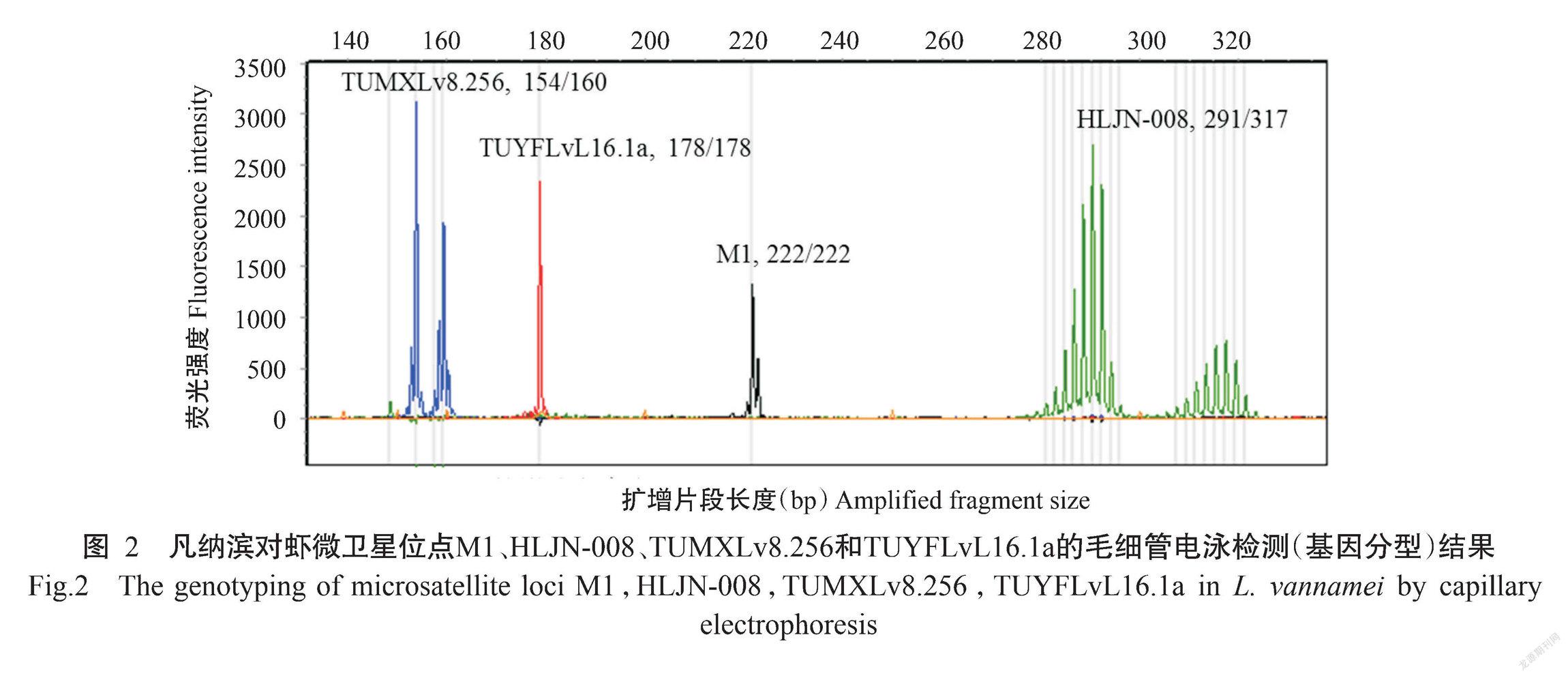
PCR反应体系15.0 μL:DNA模板(50 ng/μL)1.0 μL,Premix TaqTM Mix(TaKaRa)7.5 μL,上、下游引物(0.5 μmol/L)各0.5 μL,双蒸水5.5 μL。扩增程序:95 ℃预变性3 min;95 ℃ 30 s,50 ℃ 30 s,72 ℃ 20 s,进行32个循环;72 ℃延伸6 min。以1.0%琼脂糖凝胶电泳检测PCR扩增产物,并委托北京睿博兴科生物技术有限公司,根据产物荧光特性进行毛细管电泳混检,测序结果使用Gene Marker 2.2.0读取。
1. 5 数据分析
根据各凡纳滨对虾种苗群体在各微卫星位点的基因型,利用GENE Stock 1.32进行统计分析(Yeh and Boyle,1997),计算各微卫星位点的等位基因数(Na)、有效等位基因数(Ne)、观测杂合度(Ho)、期望杂合度(He)、Shannon’s信息指数(I)及Nei’s遗传距离;采用PIC_CALC 0.6(https://github.com/luansheng/PIC_CALC)计算多态信息含量(PIC);运用Arlequin 3.1计算群体间遗传分化系数(Fst)及进行分子变异分析(AMOVA) (Excoffier et al.,2005);同时基于Nei’s遗传距离,通过MEGA 5.0以UPGMA法进行聚类分析(Tamura et al.,2011)。
2 结果与分析
2. 1 微卫星位点多态性分析结果
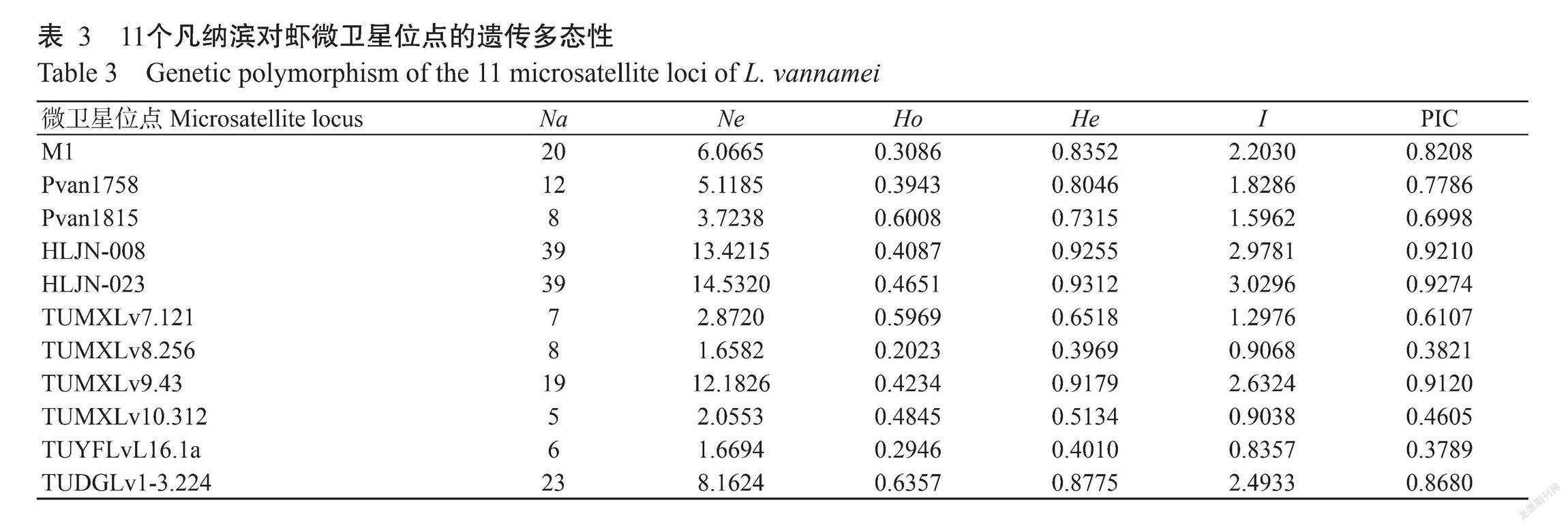
利用11对微卫星引物对9个凡纳滨对虾种苗群体共计258个个体的基因组进行扩增,结果(图1和图2)显示,11个微卫星位点均能进行有效扩增,其Na、Ne、Ho、He、I及PIC等信息见表3。各微卫星位点的Na为5~39,Ne为1.6582~14.5320,Ho为0.2023~0.6357,He为0.3969~0.9312,I为0.8357~3.0296,PIC为0.3789~0.9274。其中,TUMXLv8.256、TUMXLv10.312和TUYFLvL16.1a位点的PIC分别为0.3821、0.4605和0.3789,而其余8个微卫星位点的PIC均大于0.6000。
2. 2 凡纳滨对虾种苗群体遗传多样性分析结果
9个凡纳滨对虾种苗群体的Na、Ne、Ho、He、I和PIC平均值分别在5.5455~8.8182、3.2795~5.4735、0.3769~0.5182、0.6189~0.6948、1.2823~1.6092和0.5863~ 0.6646(表4)。其中,Stock 3群体的Na、Ne和I平均值最高,Stock 2群体的He和PIC平均值最高,而Stock 1群体的Na、Ne、Ho、PIC和I平均值均最低。在9个凡纳滨对虾种苗群体中,He平均值以Stock 6群体最低,而Ho平均值以Stock 7群体最高。可见,各凡纳滨对虾种苗群体均具有较高的遗传多样性,遗传多样性水平排序为Stock 3>Stock 2>Stock 4>Stock 5>Stock 7>Stock 6>Stock 9>Stock 8>Stock 1。
2. 3 凡纳滨对虾种苗群体遗传分化及遗传距离分析结果
9个凡纳滨对虾种苗群体的AMOVA分析结果如表5所示,有91.82%的变异来自于群体内,仅有8.18%的变异来自于群体间。两两群体间的Fst范围在0.0079~0.1494(表6),即整体遗传分化表现为中低水平。其中,Stock 6群体与Stock 9群体间的Fst最高(0.1494),而Stock 2群体与Stock 4群体间的Fst最低(0.0079)。Stock 2、Stock 3、Stock 4和Stock 5等4个群体间及Stock 7群体与Stock 8群体间的Fst均小于0.05(0.0079~0.0371),即群体间无遗传分化,而其他群体间表现为轻度遗传分化(0.05<Fst <0.15)。
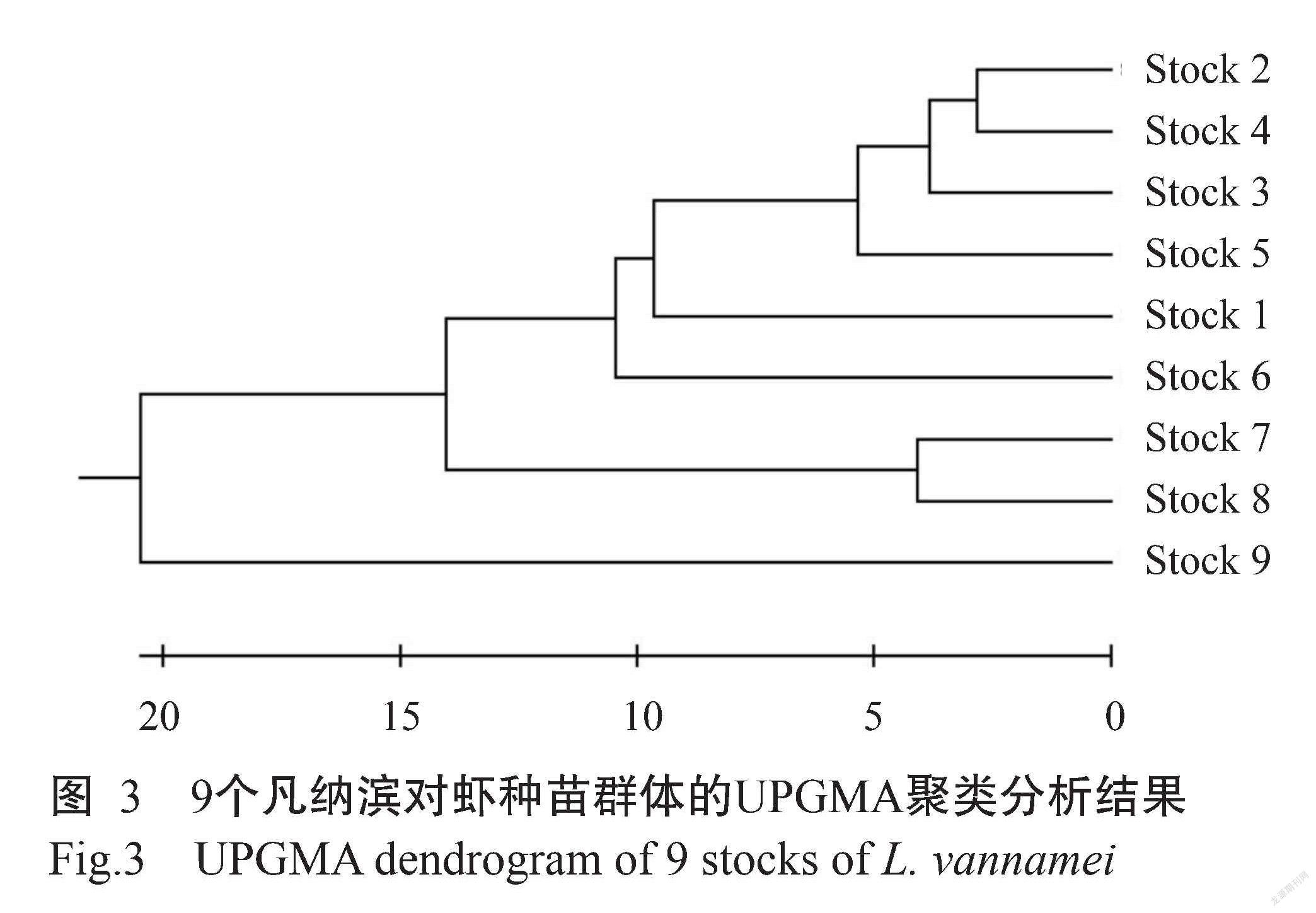
9个凡纳滨对虾种苗群体间的Nei’s遗传距离为0.0568~0.5084(表6),以Stock 2群体与Stock 4群体的Nei’s遗传距离最近,而Stock 8群体与Stock 9群体的Nei’s遗传距离最远。根据Nei’s遗传距离构建的UPGMA聚类图(图3)显示,9个凡纳滨对虾种苗群体大致可划分为三大分支,第一分支包括Stock 1~Stock 6群体,其中Stock 2群体和Stock 4群体先聚为一簇,然后依次同Stock 3、Stock 5、Stock 1和Stock 6等4个群体聚为一支;第二分支由Stock 7群体和Stock 8群体组成,而Stock 9群体独立为第三分支。
3 讨论
微卫星分子标记是物种群体遗传多样性分析的重要手段,而基因型分析是微卫星分子标记研究的关键环节。本研究采用基于毛细管电泳的荧光标记引物进行基因分型,选取11个具有多态性的微卫星位点,针对不同批次的凡纳滨对虾进口亲虾一代苗群体开展遗传多样性分析,结果显示各微卫星位点的PIC为0.3789~0.9274。PIC能反映等位基因的多態性水平,PIC在0.25~0.50为中度多态,在0.50之上则为高度多态(Shete et al.,2000;Nagy et al.,2012)。说明本研究选取的11个微卫星分子标记均呈中高度多态,是评估群体遗传多样性的有效分子标记。
遗传多样性是种质资源评价的重要依据,Na、Ho、He和PIC等均是衡量群体遗传多样性的重要指标。Zhang等(2014)选用7个微卫星分子标记对由新加坡和美国引进的7个亲虾群体遗传多样性进行分析,结果显示,各群体的Na平均值为4.286~11.143,Ne平均值为3.090~6.407,Ho平均值为0.526~0.754,He平均值为0.600~0.802。本研究中,由引进亲虾直接繁育的9个子一代苗群体的Na、Ne和He平均值均在以上相应范围内,仅Ho平均值小于上述指标,推测是由于各凡纳滨对虾群体幼体来源相同,其遗传多样性差异相对较小。包秀凤(2014)针对亲虾源于正大集团的养殖种苗群体开展微卫星分析,发现受试群体的Na、Ne、He和PIC平均值分别为3.571、2.215、0.509和0.448,各项指标均低于本研究结果。此外,于2018年5月在正大神湾虾苗场采集的4个批次种苗群体(Stock 2、Stock 3、Stock 4和Stock 5)的I分别为1.5908、1.6092、1.4919和1.4874,其遗传多样性相近,且均高于其余5个群体(Stock 1、Stock 6、Stock 7、Stock 8和Stock 9),故推测遗传多样性水平能反映后续对虾养殖生产性能的差异。
Fst是衡量群體间遗传分化程度的重要参数(黄小芳等,2020;彭敏等,2020)。Fst为0~0.05时表示群体间无遗传分化,Fst为0.05~0.15时表示群体间存在轻度遗传分化,Fst为0.15~0.25表示群体间存在中度遗传分化,Fst大于0.25时表示群体间存在高度遗传分化。谢丽等(2009)研究指出Molokai、OI、SIS和Kona Bay等4个养殖凡纳滨对虾群体间的遗传分化占15.83%;包秀凤(2014)对国内外7个选育凡纳滨对虾群体的遗传分析结果表明,群体间的Fst平均值为0.2155;Zhang等(2014)研究发现7个国外引进凡纳滨对虾群体间的遗传分化水平为11.30%。在本研究中,9个凡纳滨对虾种苗群体间的Fst平均值为0.0818,即8.18%的变异来自于群体间,表现为中低水平的遗传分化;但两两群体间的遗传分化水平存在明显差异,Stock 2、Stock 3、Stock 4和Stock 5等4个群体间及Stock 7群体与Stock 8群体间的Fst均小于0.05(0.0079~0.0371),即群体间无遗传分化,而其他群体间表现为轻度遗传分化,与各群体间的Nei’s遗传距离基本对应。一般情况下,群体间的遗传距离越小,其亲缘关系越近。基于Nei’s遗传距离的聚类分析结果也表明,2018年5月采集自正大神湾虾苗场的4个批次群体(Stock 2、Stock 3、Stock 4和Stock 5)间及2017年8月采集自海尚种苗培育基地的2个批次群体(Stock 7和Stock 8)间的亲缘关系较接近,而较早采集自正大神湾虾苗场的Stock 1群体与Stock 2~Stock 5群体的亲缘关系相对较远,其遗传分化达中等水平。
本研究利用微卫星分子标记分析不同批次进口亲虾子一代种苗的遗传变异特征,结果发现不同群体间存在遗传多样性差异,且表现出明显的遗传分化;由同一种苗培育(基地)企业提供的虾苗在短期内各群体间的遗传分化保持在较低水平,但随着时间的推移可能会发生较大变化;在同一时期由不同种苗培育(基地)企业培育获得的虾苗即使其亲虾来源相同,也会存在明显的遗传分化。这可能与近年来进口亲虾子一代种苗在生长性能和养殖成活率等方面的性能变化有关联。因此,有必要进一步系统分析凡纳滨对虾进口亲虾子一代种苗的遗传变异状况,并通过与之对应的养殖实践以获得不同群体的养殖生产性能信息,建立各群体遗传特征与养殖生产性能的关联数据库,为今后我国养殖对虾优质种苗的大数据建设及其推广应用提供基础数据支撑。
4 结论
不同批次凡纳滨对虾进口亲虾子一代种苗群体的遗传特征存在明显差异,各群体间的遗传距离随着采样时间和地点的改变而发生明显变化,且这些遗传背景差异可能会造成不同群体在养殖生产性能方面产生差异。
参考文献:
包秀凤. 2014. 凡纳滨对虾选育群体遗传多样性分析[D]. 湛江:广东海洋大学. [Bao X F. 2014. Analysis of genetic diversity of selected stocks of Litopenaeus vannamei[D]. Zhanjiang:Guangdong Ocean University.] doi:10.7666/d.D522247.
代平,孔杰,栾生. 2018. 我国凡纳滨对虾种质资源引进与分析[J]. 科学养鱼,(1):3-5. [Dai P,Kong J,Luan S. 2018. Introduction and analysis of germplasm resources of Litopenaeus vannamei in China[J]. Scientific Fish Farming,(1):3-5.] doi:10.14184/j.cnki.issn1004-843x.2018.01. 002.
黄小芳,唐章生,刘俊丹,张宏燕,钟一治,卢智发,侯树鉴,王大鹏,陆专灵. 2020. 广西不同地区克氏原螯虾群体遗传多样性微卫星分析[J]. 南方农业学报,51(2):437-444. [Huang X F,Tang Z S,Liu J D,Zhang H Y,Zhong Y Z,Lu Z F,Hou S J,Wang D P,Lu Z L. 2020. Genetic diversity microsatellite analysis of Procambarus clarkii po-pulations in different regions of Guangxi[J]. Journal of Southern Agriculture,51(2):437-444.] doi:10.3969/j.issn. 2095-1191.2020.02.025.
李强勇,李旻,曾地刚,朱威霖,彭敏,杨春玲,刘青云,赵永贞,陈秀荔,陈晓汉. 2020. 凡纳滨对虾微卫星分子标记的开发及不同养殖家系遗传多态性分析[J]. 南方农业学报,51(2):429-436. [Li Q Y,Li M,Zeng D G,Zhu W L,Peng M,Yang C L,Liu Q Y,Zhao Y Z,Chen X L,Chen X H. 2020. Development of microsatellite markers of Litopenaeus vannamei and genetic polymorphism analy-sis of different cultured families[J]. Journal of Southern Agriculture,51(2):429-436.] doi:10.3969/j.issn.2095-1191. 2020.02.024.
刘连为,陈新军,许强华,李伟文. 2014. 北太平洋柔鱼微卫星标记的筛选及遗传多样性[J]. 生态学报,34(23):6848-6854. [Liu L W,Chen X J,Xu Q H,Li W W. 2014. Isolation and genetic diversity of microsatellite DNA of Ommastrephes bartramii in the North Pacific Ocean[J]. Acta Ecologica Sinica,34(23):6848-6854.] doi:10.5846/stxb 201303030334.
毛守康,马爱军,丁福红,闫喜武,罗海忠,李伟业,徐宝荣,王怀忠,王宝义,王广宁. 2016. 梭鱼(Liza haematocheila)4个野生地理群体遗传多样性的微卫星分析[J]. 渔业科学进展,37(2):68-75. [Mao S K,Ma A J,Ding F H,Yan X W,Luo H Z,Li W Y,Xu B R,Wang H Z,Wang B Y,Wang G Y. 2016. Analysis of genetic structures of four wild geographic populations of mullet Liza haematocheila by using microsatellite marker technique[J]. Progress in Fishery Sciences,37(2):68-75.] doi:10.11758/yykxjz.20150407001.
彭敏,陈慧芳,李强勇,杨春玲,曾地刚,刘青云,赵永贞,陈晓汉,林勇,陈秀荔. 2020. 凡纳滨对虾连续 3 个世代选育群体的遗传多样性分析[J]. 南方农业学报,51(6):1442-1450. [Peng M,Chen H F,Li Q Y,Yang C L,Zeng D G,Liu Q Y,Zhao Y Z,Chen X H,Lin Y,Chen X L. 2020. Genetic diversity of three consecutive generations of Litopenaeus vannamei[J]. Journal of Southern Agriculture,51(6):1442-1450.] doi:10.3969/j.issn.2095-1191. 2020.06.026.
孙效文,张晓锋,赵莹莹,张研,贾智英,常玉梅,鲁翠云,梁利群. 2008. 水产生物微卫星标记技术研究进展及其应用[J]. 中国水产科学,15(4):689-703. [Sun X W,Zhang X F,Zhao Y Y,Zhang Y,Jia Z Y,Chang Y M,Lu C Y,Liang L Q. 2008. Development and application of microsatellite markers in aquatic species[J]. Journal of Fishery Sciences of China,15(4):689-703.] doi:10.3321/j.issn:1005-8737.2008.04.022.
童馨,龚世圆,喻达辉,杜博,黄桂菊,李莉好,郭奕惠,李色东. 2007. 凡纳滨对虾不同世代生长性状的变异[J]. 南方水产科学,3(6):30-33. [Tong X,Gong S Y,Yu D H,Du B,Huang G J,Li L H,Guo Y H,Li S D. 2007. Variation of growth traits at different generations of Pacific white shrimp(Litopenaeus vannamei)[J]. South China Fisheries Science,3(6):30-33.] doi:10.3969/j.issn.2095-0780.2007.06.005.
童馨,龚世圆,喻达辉,黄桂菊,杜博,李色东. 2009. 凡纳滨对虾(Litopenaeus vannamei)不同世代养殖群体的遺传多样性分析[J]. 海洋与湖沼,40(2):214-220. [Tong X,Gong S Y,Yu D H,Huang G J,Du B,Li S D. 2009. Genetic diversity of cultured Pacific white shrimp(Litopenaeus vannamei) stocks of different generations in China[J]. Oceanologia et Limnologia Sinica,40(2):214-220.] doi:10.3321/j.issn:0029-814X.2009.02.017.
王丽花,曹煜成,李卓佳. 2012. 溶藻细菌控藻作用及其在对虾养殖池塘中的应用前景[J]. 南方水产科学,8(4):76-82. [Wang L H,Cao Y C,Li Z J. 2012. Algae-constrai-ning effect of algae-lysing bacteria and its application prospect in shrimp ponds[J]. South China Fisheries Science,8(4):76-82.] doi:10.3969/j.issn.2095-0780.2012.04.012.
王兴强,马甡,董双林. 2004. 凡纳滨对虾生物学及养殖生态学研究进展[J]. 海洋湖沼通报,(4):94-100. [Wang X Q,Ma S,Dong S L. 2004. Studies on the biology and cultural ecology of Litopenaeus vannamei:A review[J]. Transactions of Oceanology and Limnology,(4):94-100.] doi:10.3969/j.issn.1003-6482.2004.04.016.
文国樑,曹煜成,徐煜,胡晓娟,徐武杰,李卓佳. 2015. 养殖对虾肝胰腺坏死综合症研究进展[J]. 广东农业科学,42(11):118-123. [Wen G L,Cao Y C,Xu Y,Hu X J,Xu W J,Li Z J. 2015. Review on hepatopancreas necrosis syndrome of shrimp[J]. Guangdong Agricultural Sciences,42(11):118-123.] doi:10.3969/j.issn.1004-874X.2015.11. 022.
吴怡迪,骆轩,杨章武,黄永春,游伟伟. 2016. 凡纳滨对虾选育系间杂交的生长性状及遗传多样性分析[J]. 廈门大学学报(自然科学版),55(5):646-653. [Wu Y D,Luo X,Yang Z W,Huang Y C,You W W. 2016. Growth performance and genetic diversity analysis for hybrids between selective lines of Litopenaeus vannamei[J]. Journal of Xiamen University (Natural Science),55(5):646-653.] doi:10.6043/j.issn.0438-0479.201511010.
谢丽,陈国良,叶富良,栗志民. 2009. 凡纳滨对虾4个选育群体遗传多样性的SSR分析[J]. 广东海洋大学学报,29(4):5-9. [Xie L,Chen G L,Ye F L,Li Z M. 2009. Genetic diversity of four selected stocks of Litopenaeus vannamei as revealed by SSR marker[J]. Journal of Guangdong Ocean University,29(4):5-9.] doi:10.3969/j.issn. 1673-9159.2009.04.002.
赵志英,梁丽运,白丽蓉. 2018. 斑节对虾3个野生群体遗传多样性的微卫星标记分析[J]. 热带海洋学报,37(3):65-72. [Zhao Z Y,Liang L Y,Bai L R. 2018. Analysis of genetic diversity among three wild populations of Penaeus monodon using microsatellite marker[J]. Journal of Tropical Oceanography,37(3):65-72.] doi:10.11978/2017084.
周大颜,张志新,黄彩林,招志杰,莫飞龙. 2019. 3种白鲫杂交子代的转录组学分析[J]. 南方农业学报,50(6):1328-1338. [Zhou D Y,Zhang Z X,Huang C L,Zhao Z J,Mo F L. 2019. Transcriptomic analysis of three Carassius auratus cuvieri hybrids[J]. Journal of Southern Agriculture,50(6):1328-1338.] doi:10.3969/j.issn.2095-1191.2019. 06.24.
Alcivar-Warren A,Meehan-Meola D,Park S W,Xu Z K,Dela-ney M,Zuniga G. 2007a. ShrimpMap:A low-density,microsatellite-based linkage map of the pacific white leg shrimp,Litopenaeus vannamei:Identification of sex-linked markers in linkage group 4[J]. Journal of Shellfish Research,26(4):1259-1277. doi:10.2983/0730-8000(2007)26[1259:SALMLM]2.0.CO;2.
Alcivar-Warren A,Song L S,Meehan-Meola D,Xu Z K,Xiang J H,Warren W. 2007b. Characterization and mapping of expressed sequence tags isolated from a subtrac-ted cDNA library of Litopenaeus vannamei injected with white spot syndrome virus[J]. Journal of Shellfish Resear-ch,26(4):1247-1258. doi:10.2983/0730-8000(2007)26[1247:CAMOES]2.0.CO;2.
Cruz P,Mejia-Ruiz C H,Perez-Enriquez R,Ibarra A M. 2002. Isolation and characterization of microsatellites in Pacific white shrimp Penaeus(Litopenaeus) vannamei[J]. Molecular Ecology Notes,2(3):239-241. doi:10.1046/j.1471- 8286.2002.00211.x-i2.
Excoffier L,Laval G,Schneider S. 2005. Arlequin (version 3.0):An integrated software package for population genetics data analysis[J]. Evolutionary Bioinformatics,1:47-50. doi:10.1177/117693430500100003.
Garcia D K,Alcivar-Warren A. 2007. Characterization of 35 new microsatellite genetic markers for the pacific whiteleg shrimp,Litopenaeus vannamei:Their usefulness for studying genetic diversity of wild and cultured stocks,tracing pedigree in breeding programs,and linkage mapping[J]. Journal of Shellfish Research,26(4):1203-1216. doi:10.2983/0730-8000(2007)26[1203:CONMGM]2.0.CO;2.
Garcia D K,Dhar A K,Alcivar-Warren A. 1996. Molecular analysis of a RAPD marker(B20) reveals two microsatellites and differential mRNA expression in Penaeus vannamei[J]. Molecular Marine Biology and Biotechnology,5(1):71-83.
Jia Z Y,Sun X W,Liang L Q,Li D Y,Lei Q Q. 2006. Isolation and characterization of microsatellite markers from Pacific white shrimp(Litopenaeus vannamei)[J]. Molecular Ecology Notes,6(4):1282-1284. doi:10.1111/j.1471-8286.2006.01515.x.
Meehan D,Xu Z K,Zuniga G,Alcivar-Warren A. 2003. High frequency and large number of polymorphic microsatellites in cultured shrimp,Penaeus(Litopenaeus) vannamei [Crustacea:Decapoda][J]. Marine Biotechnology,5:311-330. doi:10.1007/s10126-002-0092-z.
Nagy S,Poczai P,Cernák I,Gorji A M,Hegedűs G,Taller J. 2012. PICcalc:An online program to calculate polymorphic information content for molecular genetic studies[J]. Biochemical Genetics,50:670-672. doi:10.1007/s10528-012-9509-1.
Qiu L,Chen M M,Wan X Y,Li C,Zhang Q L,Wang R Y,Cheng D Y,Dong X,Yang B,Wang X H,Xiang J H,Huang J. 2017. Characterization of a new member of Iridoviridae,Shrimp hemocyte iridescent virus(SHIV),found in white leg shrimp(Litopenaeus vannamei)[J]. Scienti-fic Reports,7:11834. doi:10.1038/s41598-017-10738-8.
Shete S,Tiwari H,Elston R C. 2000. On estimating the he-terozygosity and polymorphism information content value[J]. Theoretical Population Biology,57(3):265-271. doi:10.1006/tpbi.2000.1452.
Tamura K,Peterson D,Peterson N,Stecher G,Nei M,Kumar S. 2011. MEGA5:Molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J]. Molecular Biology and Evolution,28(10):2731-2739. doi:10.1093/molbev/msr121.
Wang M S,Wang W J,Xiao G X,Liu K F,Hu Y L,Tian T,Kong J,Jin X S. 2016. Genetic diversity analysis of spaw-ner and recaptured populations of Chinese shrimp(Fenneropenaeus chinensis) during stock enhancement in the Bohai Bay based on an SSR marker[J]. Acta Oceanologica Sinica,35(8):51-56. doi:10.1007/s13131-016-0830-0.
Yeh F C,Boyle T J B. 1997. Population genetic analysis of co-dominant and dominant markers and quantitative traits[J]. Belgian Journal of Botany,129(2):157.
Zhang K,Wang W J,Li W Y,Zhang Q Q,Kong J. 2014. Analysis of genetic diversity and differentiation of seven stocks of Litopenaeus vannamei using microsatellite mar-kers[J]. Journal of Ocean University of China,13(4):647-656. doi:10.1007/s11802-014-2208-2.
收稿日期:2020-05-26
基金項目:中国水产科学研究院基本科研业务费专项(2020TD54);国家现代农业产业技术体系建设专项(CARS-48);广东省现代农业产业技术体系创新团队建设专项(2019KJ149)
通讯作者:曹煜成(1979-),https://orcid.org/0000-0002-0995-1131,博士,研究员,主要从事水产健康养殖及养殖生态环境调控与修复研究工作,E-mail:cyc_715@163.com;杨铿(1975-),https://orcid.org/0000-0002-6970-0668,副研究员,主要从事水产健康养殖技术及产品推广应用研究工作,E-mail:yangkeng66@163.com
第一作者:徐煜(1987-),https://orcid.org/0000-0003-2364-6659,主要从事对虾分子生理及健康养殖研究工作,E-mail:xuyublq@163.com
1443501186395

