南瓜SWEET蛋白家族的全基因组鉴定与进化分析
申长卫 袁敬平

摘 要: SWEET(sugar will eventually be exported transporter)家族是一种新型的糖转运体,该家族基因在碳水化合物运输、发育、环境适应性和寄主-病原相互作用等多个过程中发挥着重要作用。为更好地了解南瓜发育的分子机理,该研究基于已知的南瓜基因组数据库,利用生物信息学方法对中国南瓜SWEET基因(CmSWEET)的系统发育树、基因结构、跨膜结构、保守基序、启动子预测、共线性预测和基因复制等进行综合分析。结果表明:共鉴定到21个CmSWEET 基因,通过系统发育分析将21个CmSWEET基因分为4个亚族(I,II,III和IV),分别包含3、5、10和3個基因。此外,通过基因结构、跨膜结构域和保守基序发现CmSWEET在进化过程中是非常保守的。染色体定位结果显示,CmSWEET基因不均匀地分布在21条中国南瓜染色体中的13条染色体上,且在染色体Cm00、Cm01、Cm03、Cm05、Cm07、Cm09、Cm19和Cm20上不存在。启动子顺式作用元件分析显示,CmSWEET基因与植物激素(脱落酸、茉莉酸甲酯、水杨酸和生长素)响应有关,也可能参与各种环境胁迫的响应。从系统进化发育树和基因共线性方面揭示了CmSWEET基因与印度南瓜SWEET(CmaSWEET)之间的进化关系。该研究在全基因组水平上系统地鉴定了中国南瓜中SWEET基因家族,为进一步了解中国南瓜和其他葫芦科作物SWEET基因提供了基础,也为进一步的功能分析提供了重要的候选基因。
关键词: 南瓜, SWEET, 系统发育分析, 共线性, 顺式作用元件, 生物信息学
Abstract: SWEET (sugar will eventually be exported transporter) is a new type of sugar transporter, which plays important roles in carbohydrate transport, development, environmental adaptability and host pathogen interaction. In order to better understand the molecular mechanism of pumpkin development, we used bioinformatics methods to comprehensively analyzed the phylogenetic tree, gene structure, transmembrane structure, conserved motif, promoter prediction, collinearity prediction and gene replication of the Cucurbita moschata SWEET gene (CmSWEET), based on the known pumpkin genome database. The results were of follows: 21 CmSWEET genes were identified and divided into four subfamilies (I, Ⅱ, Ⅲ and IV) by phylogenetic analysis, including 3, 5, 10 and 3 genes respectively. In addition, the gene structure, transmembrane domain and conserved motif showed that CmSWEET genes were very conserved in evolution. The results of chromosome mapping showed that CmSWEET genes were unevenly distributed on 13 of 21 chromosomes and did not exist on chromosomes Cm00, Cm01, Cm03, Cm05, Cm07, Cm09, Cm19 and Cm20. Analysis of promoter cis-acting element showed that the CmSWEET genes were related to plant hormone response (abscisic acid, methyl jasmonate, salicylic acid and auxin), and may also participate in response to various environmental stresses. Finally, the phylogenetic relationship was revealed from phylogenetic tree and gene collinearity. This study systematically identified the SWEET genes family in C. moschata at the whole genome level, which provided a basis for further understanding the SWEET gene of C. moschatas and other Cucurbitaceae crops, and also provided an important candidate gene for further functional analysis.
Key words: pumpkin, SWEET, phylogenetic analysis, collinearity, cis-acting elements, bioinformatics
可溶性糖,如蔗糖、葡萄糖和果糖是光合作用的主要产物,可作为碳骨架的来源,用于许多其他细胞化合物、信号、渗透压细胞和运输分子的生物合成,并可作为瞬时能量储存(Feng & Frommer, 2015)。在植物中,糖在源叶中合成,并且可以通过韧皮部从叶片运输到根、茎、花、果实和种子,为新细胞的生长和发育提供营养(Ruan, 2014;Miao et al., 2017)。糖类化合物不能独立跨植物生物膜系统进行运输,而需要相应糖转运蛋白介导细胞或亚细胞间隔的吸收或释放。迄今为止,三种糖转运蛋白家族已被鉴定,即单糖转运蛋白(monosaccharide transporters, MSTs)(Slewinski, 2011)、蔗糖转运蛋白(sucrose transporters, SUTs)(Kühn & Grof, 2010;Ayre, 2011)和糖外排转运蛋白(sugars will eventually be exported transporters, SWEETs)(Chen et al., 2010),它们决定着作物的产量和质量(Eom et al., 2015)。
MSTs和SUTs为MFS超家族,具有12个跨膜结构域(Kühn & Grof, 2010;Ayre, 2011;Slewinski, 2011)。SWEETs是最近发现的一类糖外流转运蛋白,选择性地在细胞内或质膜上转运单糖或双糖,它们属于MtN3家族(Chen, 2014;Eom et al., 2015)。SWEET广泛分布于各种生物中,包括原核生物、动物和植物(Patil et al., 2015)。真核SWEET蛋白在结构上具有7次跨膜α-螺旋(Trans Membrane Helix, TMH/TM),由2个具有3次重复的TMH组成的MtN3基序和位于其中起连接作用的1个TMH构成(陈慧敏等,2017;Liu et al., 2019)。与真核SWEET蛋白相反,原核SWEET蛋白(即半SWEET蛋白)仅含有一个3-TM,这可能表明真核SWEET蛋白是通过复制和融合原核半SWEET蛋白中存在的基本3-TM单元而进化来的(Xuan et al., 2013)。
目前,对SWEET的功能研究在短柄草、高粱、番茄、葡萄、大豆、玉米和茉莉花等多个物种中进行了部分报道(Yuan & Wang, 2013;Chong et al., 2014;Feng et al., 2015;Patil et al., 2015;Sosso et al., 2015;胡丽萍等,2017;王盼盼,2019),研究较深的主要集中在拟南芥和水稻上(Chen et al., 2010;Xuan et al., 2013)。相关研究发现SWEET基因参与对胁迫的响应(Seo et al., 2011)、宿主与病原菌互作(Slewinski, 2011;Cong et al., 2014;Hu et al., 2014)和植物衰老的调控(Zhou et al., 2014)等生理过程,也参与糖的运输、分配和贮藏及生殖生长的发育(Le et al., 2015;李安琪,2017;冯超阳,2018)。越来越多的研究表明,植物SWEET基因可能在糖转运方面起广泛作用。
中国南瓜(Cucurbita moschata)是世界上重要的经济和营养蔬菜作物之一。甜度是南瓜口感的关键指标,而南瓜SWEET基因家族在糖转运方面的功能及作用机理仍不清楚。据我们所知,目前还没有对葫芦科南瓜作物SWEET基因家族进行系统的研究报道。此外,南瓜基因组的测序(Sun et al., 2017)促进了南瓜SWEET基因家族成员的研究。本文对中国南瓜SWEET基因的系统发育、染色体分布、基因结构、结构域、顺式作用调控元件和系统进化关系进行了详细分析,鉴定了印度南瓜SWEET基因,为瓜类SWEET基因家族的研究以及进一步探讨南瓜SWEET基因家族对糖分转运的功能提供有价值的信息。
1 材料与方法
1.1 中国南瓜SWEET基因家族的鉴定
从拟南芥信息资源(http://arabidopsis.org)中获得的17个AtSWEETs基因用作查询序列。依据已获得的拟南芥SWEET基因,对南瓜基因组数据库(http://cucurbitgenomics.org/)进行BLASTP分析,确定候选基因。去除冗余序列后,中国南瓜的所有候選SWEET蛋白序列都被提交到SMART数据库(http://smart.embl-hei-delberg.de)(Letunic et al., 2015)和Pfam数据库中(http://pfam.xfam.org)(Finn et al., 2016),消除不包含已知的MtN3/唾液/SWEET家族成员保守结构域和基序的基因。用同样的方法从南瓜基因组数据库中检测出印度南瓜SWEET基因家族成员。
运用ExPASy的“Compute pI/Mw”工具(http://web.expasy.org/compute_pi/)来预测南瓜SWEET蛋白的等电点和分子量;运用Plant-mPLoc server(http://www.csbio.sjtu.edu.cn/bioinf/plant-multi/#)预测每个SWEET基因的亚细胞定位;运用TMHMM Server v2.0(http://www.cbs.dtu.dk/services/TMHMM/)预测SWEET蛋白质跨膜(TM)螺旋数量。
1.2 中国南瓜SWEET基因的染色体分布
在南瓜数据库中获取SWEET基因的起始位置、终止位置和染色体长度,利用TBtools绘制中国南瓜全基因组CmSWEET基因家族成员分布图。
1.3 中国南瓜SWEET的结构分析和蛋白互作分析
在南瓜数据库中获取SWEET基因的cDNA及其相应基因的DNA序列,在此基础上利用在线软件GSDS(http://gsds.cbi.pku.edu.cn/) (Hu et al., 2015)绘制CmSWEETs的外显子-内含子结构图。利用在线软件MEME(http://meme.nbcr.net/meme/cgi-bin/meme.cgi)(Bailey & Elkan, 1995)对中国南瓜SWEET蛋白的保守基序进行预测和分析。MEME参数设置:motif数量为10,motif长度为5~50。以拟南芥SWEET蛋白为参考蛋白,利用在线STRING Version11.0平台进行分析。
1.4 中国南瓜SWEET启动子中的顺式作用元件分析
在数据库中获得SWEET基因的阅读框,利用TBtools获取起始密码子的前2 000 bp。在此基础上将所有基因的启动子序列使用PLant CARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)进行预测,对产生的数据进行统计分析。
1.5 多序列比对和系统发育树构建
通过ClustalW对所有南瓜SWEET蛋白进行多序列比对。中国南瓜SWEET蛋白与印度南瓜和拟南芥SWEET蛋白的系统发育关系由MEGA 5.10构建(Tamura et al., 2011)。使用成对删除空位的选项,采用bootstrap法对进化树进行评估,重复值设为1 000。
1.6 中国南瓜SWEET基因的共线性和基因复制
利用TBtools获取中国南瓜SWEET基因在印度南瓜和拟南芥中的共线性基因及这些基因在染色体上的位置。用Circos(Krzywinski et al., 2009)软件绘制SWEET基因的共线关系图。
使用TBtools程序对所有SWEET的CDS序列进行两两比对,并使用公式计算基因的比对覆盖率:基因比对覆盖率=(比对长度-错配)/较大基因的长度。依据先前在白菜物种的报道,当氨基酸一致性>80%、E期望值<1×10-10且基因比对覆盖率>0.75时,这些基因被认为是复制对(Yuan et al., 2019)。当两个基因间隔小于100 kb时被认为是串联复制基因(Wang et al., 2010)。使用KaKs calculator计算同义替换率(Ks),依据公式T=Ks / 2λ×10-6(MYA)(Emanuelsson et al., 2000)来计算SWEET基因的分化时间(T),式中λ为1.5×10-8。
2 结果与分析
2.1 南瓜SWEET基因家族成员的鉴定及其在染色体上的分布
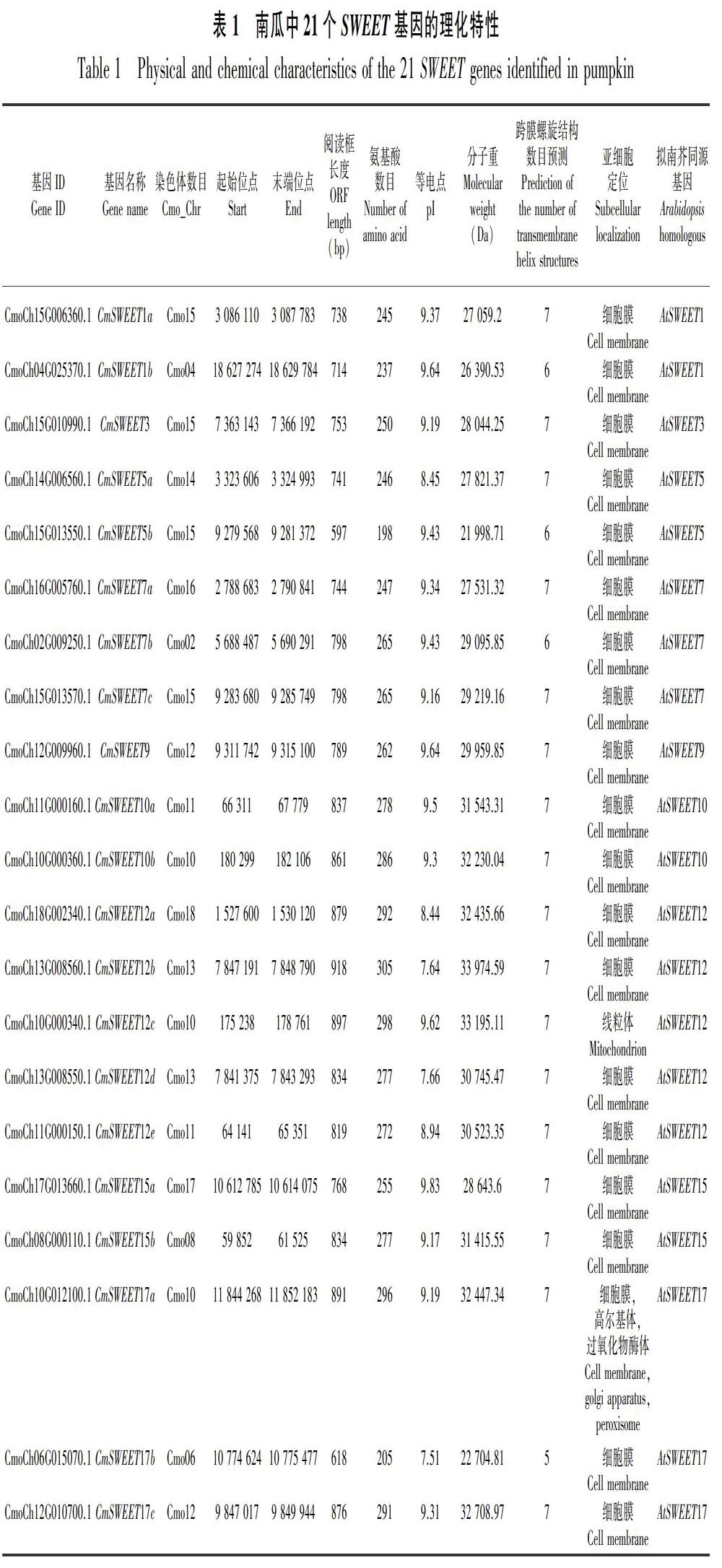
由表1可知,从中国南瓜基因组中共鉴定出21个具有保守结构域的CmSWEET基因。21个CmSWEET基因根据其在拟南芥中的同源基因命名。每个AtSWEET基因对应1~5个CmSWEET基因。CmSWEET基因的开放阅读框(ORF)的长度在597~918 bp之间,编码多肽的长度在198~305 aa之间。这些SWEET多肽的分子量(Da)和等电点(pI)分别为21 998.71~33 974.59 Da和7.51~9.83。此外,除了CmSWEET12c在线粒体中、CmSWEET17a在细胞膜/高尔基体/过氧化物酶体中,其他CmSWEETs都定位在细胞膜上。
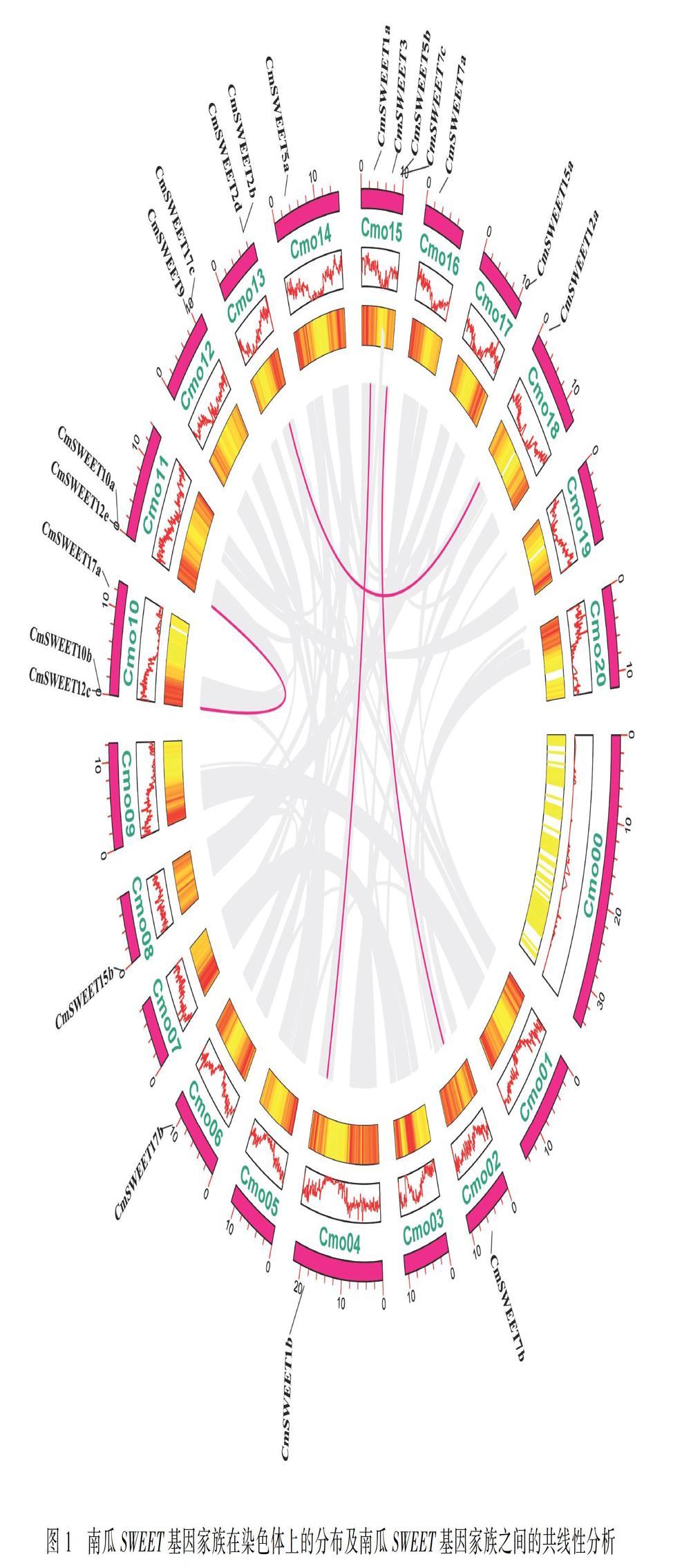
由图1可知,共有21个CmSWEET基因位于21条染色体中的13条染色体上。染色体Cmo15含有最多的CmSWEET基因(4个,19%),而染色体Cmo00、Cmo01、Cmo03、Cmo05、Cmo07、Cmo09、Cmo19和Cmo20上没有CmSWEET基因。染色体Cmo10上含有3个CmSWEET基因,染色体Cmo11、Cmo12和Cmo13上都含有2个CmSWEET基因,其余每一条染色体含有1个CmSWEET基因。由表2可知,共线性分析发现共有4对同源基因对,分别是CmSWEET1a_CmSWEET1b、CmSWEET12a_CmSWEET12d、CmSWEET10a_CmSWEET10b、CmSWEET7b_CmSWEET7c,其中依据氨基酸序列一致性>80%和基因比对覆盖率>0.75,本研究在中国南瓜染色体上共发现1对片段复制基因对(CmSWEET10b_CmSWEET10a),Ka/Ks<1.0表明这些复制对主要经历了纯化选择,分化时间为20.13 MYA。
2.2 中国南瓜SWEET蛋白的跨膜结构分析
使用默认值以FASTA格式提交给TMHMM Server v. 2.0推测了中國南瓜SWEET蛋白21个成员的氨基酸序列。图2结果显示,17个CmSWEET蛋白有7个TMs,1个CmSWEET蛋白有5个TMs,其余的3个CmSWEET蛋白(CmSWEET1b、CmSWEET5b和CmSWEET7b)有6个TMs。
2.3 中国南瓜SWEET蛋白的系统发育及结构分析
为更好地了解南瓜SWEET基因的进化起源和功能,利用Clustal Omega和MEGA 5.10软件,通过对21个CmSWEET蛋白序列和17个AtSWEET蛋白序列进行比对,构建了无根系统发育树。这些SWEET蛋白序列被分为4个亚族(图3)。图3比较了中国南瓜和拟南芥四个不同分支中的SWEET蛋白数量。每个分支都包含CmSWEET和AtSWEET。详细地说,第I亚族包含3个CmSWEETs (CsSWEET1a、CsSWEET1b和CsSWEET3)和3个AtSWEETs (AtSWEET1-3);第Ⅱ亚族包含5个CmSWEETs (CmSWEET5a-5b、CmSWEET7a-7c)和5个AtSWEETs (AtSWEET4-8);第Ⅲ亚族包含9个CmSWEETs (CsSWEET9、CsSWEET10a-10b、CsSWEET12a-12e和CsSWEET15a-15b)和7个AtSWEETs (AtSWEET9-15);第IV亚族包含3个CmSWEETs (CmSWEET17a-17c)和2个AtSWEETs (AtSWEET16-17)(图3)。
对基因结构的分析可以为CmSWEET基因家族的外显子-内含子和系统发育关系提供有价值的信息。在构建的系统发育树的基础上,利用GSDS工具分析了21个CmSWEET基因的外显子-内含子结构、内含子阶段和保守基序(图4)。基序分析发现基序1、2、3和5在所有的中国南瓜SWEET蛋白中都存在(图4: A, B;图5);基序4只在CmSWEET17b中不存在,基序7除在CmSWEET12a、CmSWEET17b蛋白中缺失,在其他蛋白中都存在;基序6只在第Ⅲ、第IV亚族中存在;基序8、9、和10只存在个别蛋白中(图4: B)。外显子-内含子分类模式与系统发育树一致(图4: C)。在中国南瓜SWEET基因家族中,所有CmSWEET基因的基因组序列都含有内含子。CmSWEET12a-12d、15a-15b、10a-10b、9、17a、17c、1a-1b、3和5a包含6个外显子,除CmSWEET15a外,它们的内含子阶段模式相似(图4: C)。
CmSWEET12e、7a-7c都包含5个外显子,除CmSWEET12e外,它们的内含子阶段模式相似。此外,CmSWEET17b和CmSWEET5b分别含有3个和4个外显子,且具有不同的内含子阶段模式(图4:C)。
2.4 中国南瓜SWEET基因启动子顺式作用调控元件的研究
为了解CmSWEETs基因的转录调控,我们从翻译起始位点(ATG)提取了21个基因的2.0 kb上游序列。在PlantCARE服务器上对21个基因上游序列中的顺式元件进行分析。图6:A为顺式作用元件在启动子上的位置。值得注意的是,在CmSWEETs的启动子区共鉴定出9种植物激素响应顺式元件,包括1种脱落酸响应元件(ABRE)、2种茉莉酸甲酯响应元件(CGTCA基序和TGACG基序)、3种赤霉素响应元件(GARE基序、P-box和TATC-box)、1个水杨酸响应元件(TCA元件)和2个生长素响应元件(TGA-box和AuxRR-core)(图6:B)。21个CmSWEET基因的启动子至少含有一个植物激素响应元件。CmSWEET7b含有最多(15个)植物激素响应元件(图6: B)。CmSWEET基因除了受植物激素响应元件的调控外,还可能对各种环境胁迫,如厌氧、高温、低温、干旱和昼夜节律(图6:B)。厌氧响应元件除在CmSWEET12d中不存在,存在所有的CsSWEET启动子中(图6),提示这些基因在厌氧诱导中的重要作用。然而,昼夜节律仅在CmSWEET12e、CmSWEET10a和CmSWEET17a中存在(图6: B)。这些数据表明CmSWEET可能通过一个复杂的机制参与环境胁迫,并且每个CmSWEET基因都可以被不同的环境胁迫所诱导。
2.5 中国南瓜SWEET蛋白相互作用关系网络分析
根据模式物种拟南芥中的同源序列,构建了中国南瓜SWEET蛋白相互作用关系网络。由图7可知,CmSWEET5a对应AtSWEET5(AtVEX1);CmSWEET9对应AtSWEET9;CmSWEET1a对应AtSWEET1;CmSWEET12a对应AtSWEET12; CmSWEET17a对应AtSWEET17;CmSWEET7a对应AtSWEET7;CmSWEET3对应AtSWEET3。图7结果显示,CmSWEET9分别与CmSWEET5a和CmSWEET1a;CmSWEET17a分别与CmSWEET12a、CmSWEET3和CmSWEET7a存在互作关系。
2.6 中国南瓜与印度南瓜SWEET蛋白系统发育分析
采用与中国南瓜相同的方法,从印度南瓜基因组中共鉴定到19个CmaSWEET基因(表3)。根据拟南芥的同源基因命名了19个CmaSWEET基因。无根进化树显示19个CmaSWEET被分为4个大分支,与前面中国南瓜SWEET的分类一致(图8)。其中,在CmaSWEET中,中国南瓜SWEET家族中第IV亚族成员又被分为3个小分支。每个大分支都包含中国南瓜SWEET蛋白和印度南瓜SWEET蛋白。从综合来看,两个栽培种SWEET蛋白系统进化相对保守。
2.7 中国南瓜与印度南瓜和拟南芥SWEET基因的共线性分析
利用TBtools工具获取CmSWEET基因在CmaSWEET和AtSWEET基因中的共线性基因,并对这些共线性关系进行了统计。图9结果表明,中国南瓜与印度南瓜SWEET的共线性基因对数目(18)大于中国南瓜与拟南芥的共线性基因对数目(12)。此外,统计中国南瓜SWEET基因在印度南瓜和拟南芥中的拷贝数的结果(图9,表4)发现,CmSWEET10a和CmSWEET10b对应印度南瓜1个SWEET基因(CmaSWEET10);AtSWEET1对应CmSWEET1a和CmSWEET1b;AtSWEET10对应CmSWEET10a和CmSWEET12c两个基因;AtSWEET5对应CmSWEET15b和CmSWEET17a两个基因。因此,我们认为中国南瓜SWEET基因与印度南瓜的进化关系相对拟南芥进化关系更加紧密,中国南瓜SWEET基因在葫芦科的多倍化事件中得到了扩张。
3 讨论与结论
SWEET基因家族在植物界广泛分布,并参与许多重要的生理和生化过程(Yuan & Wang, 2013;Chen, 2014;Chandran, 2015;Eom et al., 2015;Patil et al., 2015)。然而,仅在基因组水平上鉴定了SWEET基因家族的少数物種,如拟南芥、水稻、番茄和大豆(Chen et al., 2010;Yuan et al., 2014;Feng et al., 2015;Patil et al., 2015)。据我们所知,迄今为止尚未有关葫芦科南瓜属作物的SWEET基因家族的系统研究报告。本研究中,我们通过分析中国南瓜的系统发育、染色体分布、基因结构、保守基序、顺式作用调控元件等,探索了中国南瓜中的21个SWEET基因。
基因復制,包括片段复制、串联复制和全基因组复制,他们在生物的进化过程中起重要作用(Xu et al., 2012)。中国南瓜在经历了核心真双子叶植物共同的六倍体(core-eudicot common hexaploidization, ECH)事件(115~130百万年前) (Jaillon et al., 2007;Jiao et al., 2011)后,又经历了葫芦科共有的全基因组加倍(cucurbit-common tetraploidization, CCT)事件的证据(Wang et al., 2018),而后南瓜属共享一次最近的全基因组加倍事件CST[古多倍体事件(Cucurbita-specific tetraploidization, CST)](于继高等,2020)。在中国南瓜中发现4对SWEET共线基因对, 有一对复制基因(CmSWEET10b-CmSWEET10a),由于这两个基因不在同一条染色体上,因此我们认为是片段复制基因对。由Ka/Ks<1可知,中国南瓜SWEET基因正在进行纯化正向选择。
通过对中国南瓜和拟南芥SWEET蛋白进行系统发育树分析,发现他们被分配到4个亚族,这一分类与先前拟南芥的分类一致(Hu et al., 2017)。将中国南瓜和印度南瓜SWEET蛋白进行系统发育树分析,发现每一个大分支都含有中国南瓜和印度南瓜SWEET蛋白,这表明中国南瓜和印度南瓜在进化上可能来自同一个祖先。
通过SWEET基因结构分析,发现同一亚族中的大多数基因在或外显子数目或内含子数量方面具有相似的结构特征,这一现象与先前其他物种中SWEET家族结构特征相似(Yuan et al., 2014;Feng et al., 2015)。然而,同一个亚族中的CmSWEET17a和CmSWEET17b,与同一亚族中的其他CmSWEETs相比,它们含有不同数量的外显子,这说明中国南瓜SWEET基因家族的结构多样性。就特定的序列基序而言,在不同的亚族之间也检测到高度差异。然而,在一个共同的亚族中,大多数中国南瓜SWEET蛋白具有保守的基序,这表明同一亚族可能具有相似的功能。
本研究发现,21个CmSWEETs基因启动子序列中的植物激素顺式元件和胁迫响应相关。在所有的CmSWEETs基因启动子中,至少发现了2个与植物激素响应有关的顺式作用元件,这意味着相应的激素可能在其调节中起着重要作用。此外,所有CmSWEETs基因至少包含一个应激相关的顺式作用元件,表明这些基因在应激响应中也起着重要的调控作用。然而,还需要进一步地研究来确定这些顺式元件是否以及如何在中国南瓜中起作用。
在中国南瓜中发现21个CmSWEETs,在印度南瓜中发现19个CmaSWEETs。Hu et al.(2017)在黄瓜中发现17个CsSWEETs。中国南瓜、印度南瓜与黄瓜都是密切相关的,虽然黄瓜有7条染色体,中国南瓜和印度南瓜都有21条。详细的共线基因对和进化关系分析揭示了葫芦科三种重要作物染色体进化和重排的高度复杂性。因此,黄瓜、中国南瓜和印度南瓜中SWEET基因的相似性水平特别高,这些同源的SWEET基因可能具有相似的功能。
参考文献:
AYRE BG, 2011. Membrane-transport systems for sucrose in relation to whole-plant carbon partitioning [J]. Mol Plant, 4(3): 377-394.
BAILEY TL, ELKAN C, 1995. The value of prior knowledge in discovering motifs with MEME [J]. Proc Int Conf Intell Syst Mol Biol, 3: 21-29.
CHANDRAN D, 2015. Co-option of developmentally regulated plant SWEET transporters for pathogen nutrition and abiotic stress tolerance [J]. IUBMB Life, 67(7): 461-471.
CHEN HM, LI W, MA XF, et al., 2017. SWEET gene family in plants:research advances [J]. Chin Agric Sci Bull, 33(19): 34-39. [陈慧敏, 李威, 马雄风, 等, 2017. 植物SWEET基因家族的相关研究进展 [J]. 中国农学通报,33(19): 34-39.]
CHEN LQ, 2014. SWEET sugar transporters for phloem transport and pathogen nutrition [J]. New Phytol, 201(4): 1150-1155.
CHEN LQ, HOU BH, LALONDE S, et al., 2010. Sugar transporters for intercellular exchange and nutrition of pathogens [J]. Nature, 468(7323): 527-532.
CHONG J, PIRON MC, MEYER S, et al., 2014. The SWEET family of sugar transporters in grapevine: VvSWEET4 is involved in the interaction with Botrytis cinerea [J]. J Exp Bot, 65(22): 6589-6601.
COHN M, BART RS, SHYBUT M, et al., 2014. Xanthomonas axonopodis virulence is promoted by a transcription activator-like effector-mediated induction of a SWEET sugar transporter in cassava [J]. Mol Plant Microb Int, 27(11): 1186-1198.
EMANUELSSON O, NIELSEN H, BRUNAK S, et al., 2000. Predicting subcellular localization of proteins based on their N-terminal amino acid sequence [J]. J Mol Biol, 300(4): 1005-1016.
EOM JS, CHEN LQ, SOSSO D, et al., 2015. SWEETs, transporters for intracellular and intercellular sugar translocation [J]. Curr Opin Plant Biol, 25: 53-62.
FENG CY, HAN JX, HAN XX, et al., 2015. Genome-wide identification, phylogeny, and expression analysis of the SWEET gene family in tomato [J]. Gene, 573(2): 261-272.
FENG CY, 2018. Effect of SlSWEETs on sugar metabolism of tomato fruits and functional verification of four SlSWEETs genes [D]. Shenyang: Shenyang Agricultural University. [冯超阳, 2018. 番茄果实糖代谢中关键SlSWEETS基因的鉴定及其功能验证 [D]. 沈阳: 沈阳农业大学.]
FENG L, FROMMER WB, 2015.Structure and function of SemiSWEET and SWEET sugar transporters [J]. Trends Biochem Sci, 40(8): 480-486.
FINN RD, COGGILL P, EBERHARDT RY, et al., 2016. The Pfam protein families database: Towards a more sustainable future [J]. Nucl Acids Res, 44(1): 279-285.
HU B, JIN J, GUO AY, et al., 2015. GSDS 2.0: An upgraded gene feature visualization server [J]. Bioinformatics, 31(8): 1296-1297.
HU LP, ZHANG F, SONG SH, et al., 2017. Genome-wide identification, characterization, and expression analysis of the SWEET gene family in cucumber [J]. J Integr Agric, 16(7): 1486-1501.
HU LP, ZHANG F, XU H, et al., 2017. Research advances in the structure, function and regulation of SWEET gene family in plants [J]. Biotechnol Bull, 33(4): 27-37. [胡麗萍, 张峰, 徐惠, 等, 2017. 植物SWEET基因家族结构、功能及调控研究进展 [J]. 生物技术通报, 33(4): 27-37.]
HU Y, ZHANG JL, JIA HG, et al., 2014. Lateral organ boundaries 1 is a disease susceptibility gene for citrus bacterial canker disease [J]. Proc Natl Acad Sci USA, 111(4): 521-529.
JAILLON O, AURY JM, NOEL B, et al., 2007. The grapevine genome sequence suggests ancestral hexaploidization in major angiosperm phyla [J]. Nature, 449(7161): 463-467.
JIAO YN, WICKETT NJ, AYYAMPALAYAM S, et al., 2011. Ancestral polyploidy in seed plants and angiosperms [J]. Nature, 473(7345): 97-100.
KHN C, GROF CPL, 2010. Sucrose transporters of higher plants [J]. Curr Opin Plant Biol, 13(3): 288-298.
KRZYWINSKI M, SCHEIN JE, BIROL I, et al., 2009. Circos: an information aesthetic for comparative genomics [J]. Genome Res, 19(9): 1639-1645.
LE HR, SPINNER L, KLEMENS PA, et al., 2015. Disruption of the sugar transporters AtSWEET11 and AtSWEET12 affects vascular development and freezing tolerance in Arabidopsis [J]. Mol Plant, 8(11): 1687-1690.
LETUNIC I, DOERKS T, BORK P, 2015. SMART: Recent updates, new developments and status in 2015 [J]. Nucl Acids Res, 43: 257-260.
LI AQ, 2017. Expression characteristics and genetic transformation of SlSWEET7a in tomato fruit [D]. Shenyang: Shenyang Agricultural University. [李安琪, 2017. 番茄果實中SlSWEET7a基因的表达特性及遗传转化 [D]. 沈阳: 沈阳农业大学.]
LIU HT, LYU WY, TIAN SH, et al., 2019. The SWEET family genes in strawberry: Identification and expression profiling during fruit development [J]. S Afr J Bot, 125: 176-187.
MIAO HX, SUN PG, LIU Q,et al., 2017. Genome-wide analyses of SWEET family proteins reveal involvement in fruit development and abiotic/biotic stress responses in banana [J]. Sci Rep, 7(1): 35-36.
PATIL G, VALLIYODAN B, DESHMUKH R, et al., 2015. Soybean (Glycine max) SWEET gene family: insights through comparative genomics, transcriptome profiling and whole genome re-sequence analysis [J]. BMC Genomics, 16(1): 520.
RUAN YL,2014. Sucrose metabolism: Gateway to diverse carbon use and sugar signaling [J]. Ann Rev Plant Biol, 65: 33-67.
SEO PJ, PARK JM, KANG SK, et al., 2011. An Arabidopsis senescence-associated protein SAG29 regulates cell viability under high salinity [J]. Planta, 233(1): 189-200.
SLEWINSKI TL, 2011. Diverse functional roles of monosaccharide transporters and their homologs in vascular plants: a physiological perspective [J]. Mol Plant, 4(4): 641-662.
SOSSO D, LUO D, LI QB, et al., 2015. Seed filling in domesticated maize and rice depends on SWEET-mediated hexose transport [J]. Nat Genet, 47(12): 1489-1493.
SUN HH, WU S, ZHANG GY, et al., 2017. Karyotype stability and unbiased fractionation in the paleo-allotetraploid cucurbita genomes [J]. Mol Plant, 10(10): 1293-1306.
TAMURA K, PETERSON D, PETERSON N, et al., 2011. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods [J]. Mol Biol Evol, 28(10): 2731-2739.
WANG JP, SUN PC, LI YX, et al., 2018. An overlooked paleotetraploidization in Cucurbitaceae [J]. Mol Biol Evol, 35(1): 16-26.
WANG LQ, GUO K, LI Y, et al., 2010. Expression profiling and integrative analysis of the CESA/CSL superfamily in rice [J]. BMC Plant Biol, 10(1): 282-297.
WANG PP, 2019. Preliminary analysis on the function of 7 sugar transporter (SWEET) genes in jasmine [D]. Fuzhou: Fujian Agriculture and Forestry University. [王盼盼, 2019. 茉莉花7个糖转运蛋白(SWEET)基因的功能初步分析 [D]. 福州: 福建农林大学.]
XU GX, GUO CC, SHAN HY, et al., 2012. Divergence of duplicate genes in exon-intron structure [J]. Proc Natl Acad Sci USA, 109(4): 1187-1192.
XUAN YH, HU YB, CHEN LQ, et al., 2013. Functional role of oligomerization for bacterial and plant SWEET sugar transporter family [J]. Proc Natl Acad Sci USA, 110(39): 3685-3694.
YU JG, WANG JY, WANG JY, et al., 2020. Comparative genomic analysis of pumpkin, grape and wax gourd [J]. Mol Plant Breed.https://kns.cnki.net/kcms/detail/46.1068.S.20200501.1536.002.html. [于继高, 王金禹, 汪建宇, 等, 2020. 南瓜与葡萄和冬瓜的比较基因组学分析 [J]. 分子植物育种. https://kns.cnki.net/kcms/detail/46.1068.S.20200501.1536.002.html.]
YUAN JP, LIU TK, YU ZH,et al., 2019. Genome-wide analysis of Chinese cabbage IQD gene family and the response of BrIQD5 in drought resistance [J]. Plant Mol Biol, 99(6): 603-620.
YUAN M, WANG SP, 2013. Rice MtN3/saliva/SWEET family genes and their homologs in cellular organisms [J]. Mol Plant, 6(3): 665-674.
YUAN M, ZHAO JW, HUANG RY, et al., 2014. Rice MtN3/saliva/SWEET gene family: Evolution, expression profiling, and sugar transport [J]. J Integr Plant Biol, 56(6): 559-570.
ZHOU Y, LIU L, HUANG WF, et al., 2014. Overexpression of OsSWEET5 in rice causes growth retardation and precocious senescence [J]. PLoS ONE, 9(4): e94210.
(責任编辑 蒋巧媛)

