基于图卷积的蛋白质复合物筛选模型
高盼
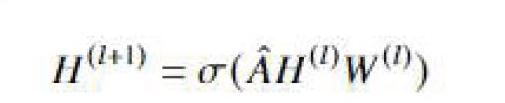


摘要:蛋白质复合物的预测对生物研究至关重要,现有的预测算法主要是基于蛋白质相互作用网络的局部结构发现算法,其存在一定的局限性,无法利用已知复合物作为先验知识,无法有效融合蛋白质生物关联性数据,因此其预测结果中会存在部分不符合复合物形成规律的样本。本文提出基于图卷积的复合物筛选模型,该模型充分考虑了蛋白质特征,在蛋白质复合物对应的局部图中将特征进行深度融合,从而有效地对蛋白质复合物进行评分,识别并剔除一般复合物预测算法结果中的低分复合物样本,提高其预测的准确性。
关键词:蛋白质复合物;生物信息学;图神经网络
中图分类号:TP183 文献标识码:A
文章编号:1009-3044(2021)07-0186-02
1 引言
蛋白质复合物是蛋白质相互结合完成某一项生物功能的集合。生物学上蛋白质复合物的识别与研究在生物检测、细胞分析、药物研发等领域发挥至关重要的作用,能有效帮助研究人员发掘生命活动的规律。传统的基于生物实验的方法可以识别蛋白质复合物,但其成本较高、周期较长,无法满足大规模数据时代的研究需求。现有的蛋白质复合物预测算法主要是基于计算的算法,将蛋白质之间广泛的相互作用抽象成图,蛋白质复合物抽象为图中的局部结构,此时蛋白质复合物预测问题转换为局部子图发现问题。但是基于计算的预测算法具有一定的局限性,其无法利用已知复合物作为先验知识,无法有效融合蛋白质生物关联性数据,因此其预测结果中会存在部分不符合复合物形成规律的样本。本文提出基于图卷积的复合物筛选模型,该模型充分考虑了蛋白质特征,在蛋白质复合物对应的局部图中将特征进行深度融合,从而有效地对蛋白质复合物进行评分,识别并剔除一般复合物预测算法结果中的低分复合物样本,提高其预测的准确性。
2 复合物特征子图数据集构建
已验证蛋白质复合物数据集中每一个样本为蛋白质集合,代表某一个复合物中蛋白质种类。但是集合数据不具有可学习性,因此本文提出了复合物特征子图的结构化数据生成方法,将集合数据转换为可学习的结构化数据。下面介绍具体的复合物特征子图数据集生成过程。
基于蛋白质相互作用数据集构建蛋白质相互作用网络(简称互作网络)。迄今为止,酿酒酵母(Saccharomyces cerevisiae)相关的蛋白质领域研究较为深入,其标准复合物数据包括MIPS数据集、CYC2008数据集等等,蛋白质相互作用数据包括DIP数据集、Biogrid数据集、Gavin数据集等,因此其数据基础是比较完备的。本文基于DIP[1]蛋白质相互作用数据集展开研究,其中包含着多对蛋白质相互作用,每一对蛋白质相互作用有相应的编号i和编号j,表示这两个蛋白质之间存在相互作用关系。本文以数据集中的互作关系作为邻边构建蛋白质相互作用网络,形成图结构(Graph)的形式G={V,E},其中V表示所有蛋白质结点,E表示所有蛋白质相互作用邻边。
在互作网络中嵌入结点特征,形成带特征的蛋白质相互作用网络(简称特征互作网络)。图结构是高维的复杂的数据,可能包含噪声和冗余信息,结点特征嵌入就是将网络数据从高维表示隐射维低维表示,其低维嵌入维度通常越小于网络的结点数量,从而达到网络去噪以及结点表示的目的。本文提出了两种结点特征嵌入方法,第一种是基于图自编码器嵌入(Graph Auto Encoder,GAE)[2],维度16维,第二种是基于深度随机游走嵌入(Deepwalk),维度64维。结合GAE和Deepwalk结点特征嵌入维度为80维。
从特征互作网络中提取特征子图。特征子图是由蛋白质复合物的集合数据作为结点集合在特征互作网络中提取子图构建。按照蛋白质复合物数据来源划分,特征子图分为正样本特征子图、中间样本特征子图、负样本特征子图和待筛选特征子图。训练数据集中正样本特征子图由CYC2008标准复合物数据集[3]提取,中间样本特征子图由COACH算法的预测结果提取,负样本特征子图由随机子图。在训练数据集中,子图不同类别具有不同标签,同时依据标准复合物邻居相似性评分指标,子图具有相应0~1的评分,评分越接近1表示该复合物越接近标准复合物。待筛选特征子图由多种基于密集子图的复合物预测算法构成,分别为Dpclus算法[4]、Clique算法和IPCA算法。
3 基于图卷积的复合物评价模型
卷积神经网络快速发展,其具有高效的特征提取能力,但其只能处理平移不变性的欧氏空间数据。特征子图的结构化数据是非欧式空间数据,每个结点的局部结构由于其邻边和对应的邻居结点是各不相同的,因此其是具有差异性的,图卷积神经网络[5](Graph Convolution Network,简称GCN)基于邻居特征聚集以及特征转换矩阵作为卷积核的思想巧妙地实现了图结构数据上的卷积过程。在数据集构建阶段本文已将复合物的集合数据转换为特征子图数据,特征子图为非欧式空间高维图结构,本文基于图卷积的算法本文对蛋白质复合物的高维图结构进行深度融合,从而提取复合物特征并对特征做进一步处理。
本文采用了如下的GCN的更新方法,在图结构中,输入为公式如下所示.:
其中H为n*m的矩阵,代表图结构中结点特征矩阵,n为图结构中结点数量,m为特征维度。其中(l)上标表示第l层特征,(l+1)上标表示第l+1层特征,上式即表示特征层级更新方法。A为n*n矩阵,表示图结构邻接矩阵,而A-hat标志表示经过拉普拉斯平滑的邻接矩阵。图卷积的过程本质是结点之间的信息汇总及更新过程,平滑处理使得信息流动会依据源点的度数和汇点的度数做相应的归一化处理,对度数过大的结点进行正则化惩罚。W为m*m的矩阵,表示图卷积的核参数,对应着特征汇聚之后的转换。σ表示激活函数。
基于圖卷积神经网络,本文提出了复合物评价模型,该模型将特征子图中的结点特征进行深度融合和动态更新,提取全局子图特征,并基于全局子图特征进行分类与评分,其具体过程如图1所示。
从图中可以得出,该模型的输入为特征子图的初始结点特征矩阵,其特征维度为80维,由GAE和Deepwalk嵌入拼接构成。采用了两层图卷积神经网络提取特征,经过第一层GCN层特征更新,80维初始结点特征转换为64维结点隐层特征,使得结点获取直径3的局部特征结构数据,经过第二层GCN层特征更新,64维结点隐层特征转换为更深层次的64维结点隐层特征,使得结点获取直径5内的局部特征结构数据。结点特征深度嵌入之后,将子图所有结点特征池化,以池化特征作为复合物子图整体特征表示,至此就完成了复合物的特征嵌入。具体的池化过程为将所有结点特征分别进行平均值池化和最大值池化,拼接起来维1*128维特征。在子图特征基础上,经过两层感知器模型以及Softmax激活函数,得到子图的分类结果,计算相应的分类损失;经过另外两层感知器模型以及Sigmoid激活函数,得到子图的评分结果,计算相应的评分损失。最终的损失函数为分类和损失与评分损失综合起来。
4 实验设计与结果
本文对比了基于随机特征的模型和基于图论拓扑特征的模型。基于随机特征的模型输入结点特征为随机数据,基于图论拓扑特征的模型采用度分布,结点数等统计特征直接作为子图特征。实验中在DIP网络中训练模型,在待筛选数据中保留分类结果与评分结果为达到阈值的样本,形成筛选后数据。评价阶段对比了筛选前后复合物数据的F1值指标,具体结果如图2所示。
对比结果表明基于图卷积的蛋白质复合物筛选模型有效地提高了F1评价指标,同时证明了结点特征的有效性以及图卷积特征融合方法的有效性。
5 结束语
本文提出了基于图卷积的蛋白质复合物筛选模型。提出了蛋白质复合物特征子图的构建方法,包括蛋白质互作网络构建,基于GAE和Deepwalk的结点特征嵌入和复合物特征子图提取。本文将图卷积神经网络应用于蛋白质复合物的特征提取中,经过多层图卷积获得结点深入嵌入特征,池化方法获取子图特征,基于子图特征获取复合物评分结果和分类结果。通过对复合物的分类与评分筛选一般性预测算法的有效复合物,经过多个对比实验表明,基于图卷积的筛选模型筛选之后的复合物样本F1评价指标在三种预测算法中均有大幅提升。
参考文献:
[1] Salwinski L , Miller C S , Smith A J , et al. The Database of Interacting Proteins: 2004 Update[J]. Nucl Acids Res,2004(32): D449-451.
[2] Kipf T N , Welling M . Variational Graph Auto-Encoders[J]. 2016.
[3] Shuye P , Jessica W , Brian T , et al. Up-to-date catalogues of yeast protein complexes[J]. Nucleic Acids Research, 2009, 37(3):825-831.
[4] Altaf-Ul-Amin M , Shinbo Y , Mihara K , et al. Development and implementation of an algorithm for detection of protein complexes in large interaction networks[J]. Bmc Bioinformatics, 2006,7(1):1-13.
[5] Kipf T N, Welling M. Semi-Supervised Classification with Graph Convolutional Networks[A]. 5th International Conference on Learning Representations[C].Open Review.net,2017.
【通聯编辑:光文玲】

