咽喉反流性疾病患者肠道菌群变化的初步研究△
崔小缓 蒋兴旺# 张延平 李丽娜 冉桃桃 毕欣欣佟明望 段国英 陈浩 陈援凯 刘倩 韩灵
咽喉反流性疾病(laryngopharyngeal reflux, LPRD)是指胃内容物反流至食管上括约肌以上部位,引起一系列症状和体征的总称[1],其发病机制不明,症状复杂多样,体征无特异性,缺乏诊断的金标准,给该病的诊断和治疗带来很多困惑,亟待进行深入的研究。研究发现LPRD患者普遍存在食管上括约肌低张力、食管体部运动能力减退等胃肠道动力异常现象[2],但原因不清楚。
咽喉部微生态失衡、致病菌占据优势时,可能引起感染和炎症,而胃内容物进入咽喉及口腔可进一步加重患者咽喉部菌群失调程度[3];有研究发现,炎症可以使喉-食管上括约肌和咽-食管括约肌感受器功能下降,有可能诱发LPRD[4],因此咽部微生物状态可能与LPRD的发生和发展密切相关,但由于口腔和咽部微生物受环境因素影响远大于基因背景的影响程度[5],给研究该部位微生态环境带来一定的困难。有研究发现胃液、口腔[6,7]、喉[8]、食管远端[9]、胃粘膜[8]和粪便[10]中放线菌、拟杆菌门、厚壁菌门、梭菌门和变形菌门均占主导地位,口腔微生物与肠道菌群间种群重复率可以达到45%[11],59%的口腔微生物会频繁向肠道转移和定植[12],提示肠道菌群与口腔和咽、喉菌群组成具有一致性,肠道菌群变化情况在一定程度上可以反映口腔及咽喉部微生物状态。本研究通过16S rDNA测序方法对LPRD患者与健康人肠道菌群的多样性和差异性进行研究,希望为探索LPRD发病机制、发现好的诊断和治疗方法提供参考。
1 资料与方法
1.1研究对象及分组 从2019年1~5月就诊于解放军总医院第八医学中心耳鼻喉科门诊疑似LPRD的患者中,以随机数字法随机选取30例为LPRD组,其中男21例,女9例,年龄8~60岁,平均37.17±12.15岁;以与病例组性别、年龄相匹配的健康志愿者30例为对照组,其中男女各15例,年龄10~64岁,平均34.63±9.81岁。
LPRD组纳入标准:符合LPRD诊断标准[1],有咽部不适症状,病史1个月以上,RSI>13;对照组纳入标准为无咽喉及胃部不适,无全身性疾病。排除标准:①结核病、红斑狼疮、麻风、茎突综合征、梅毒等可能引起咽部特异性炎症表现的全身性疾病;高血压、糖尿病、恶性肿瘤及相关手术史;②胃食管反流病(gastroesophageal reflux disease,GERD)〔既往诊断GERD或胃食管返流病自测表(GerdQ)量表评分≥8视为GERD〕;③1个月内服用抗生素或微生态制剂;④2周内服用质子泵抑制剂;⑤1个月内饮食结构和习惯有变化。
本研究经过解放军总医院第八医学中心医学伦理委员会批准(309201905171045),入组前征得患者本人同意,并签署知情同意书。
1.2肠道菌群检测标本采集 LPRD组、对照组均采用TinyGene粪便微生物DNA采集保存套装(CJ-01KA,上海微基生物技术有限公司)按照说明书留取粪便,-20 ℃冰箱存储待检测。
1.3肠道菌群检测方法
1.3.116S rDNA测序 由上海微基生物技术有限公司完成,DNA获取与质检:粪便样本采用化学裂解法裂解细胞膜及细胞核膜,提取DNA,以样本提取的DNA为模板,聚合酶链反应(PCR)扩增16S rDNA基因的V4-V5区域;全部PCR产物回收、定量,均一化混匀后完成文库构建,在IlluminaMiSeq 2×300 bp平台上完成测序。
1.3.2生物信息学分析 对测得的原始数据通过barcode分配样品reads,得到每个样本的有效序列,采用Trimmomatic软件,将测序结果末端低质量的序列去掉,根据PE reads 之间的overlap 关系,采用flash软件将成对的reads 拼接成一条序列,同时采用mothur软件对序列质量进行质控和过滤,将模糊碱基、单碱基高重复区、过长和过短的序列以及PCR过程中产生的一些嵌合体去除,从而得到优化序列,之后进行OTU(operational taxonomic unit)聚类(UPARSE software),OTU代表序列与silva 128数据库比对进行物种信息注释。基于分类学信息,在门、纲、目、科、属、种分类水平上进行群落结构的统计分析。在上述分析的基础上,进行一系列群落结构和系统发育等的统计学和可视化分析。利用Mothur(Version 1.33.3)进行Alpha多样性、Beta多样性分析,物种的差异分析采用LEfSe方法。
1.4统计学方法 在进行统计分析之前,进行Shapiro-Wilk test方法检验来评估所有定量变量的分布正态性。采用配对样本t检验或kruskal_wilcox非参数检验行统计学分析;除有特殊说明数据外,其余数据以均数±标准差表示;P<0.05为差异有统计学意义。
2 结果
2.1物种单元分类及OTU聚类 共获得2 364 658条有效序列,平均每个样本的序列数目为39 410.97±5 513.34条,序列长度主要集中在247~451 bp之间,平均长度为410.15±0.44 bp。对照组30例标本共1 205 013条有效序列,其平均序列数目为40 417.1±4 920.23条,进行OTU聚类后得到1 027 170条优化序列(平均序列数目为34 239±4 188.42条);LPRD组30例标本共1 159 645条有效序列,其平均序列数目为38 654.83±6 037.38条,进行OTU聚类后得到967 183条优化序列(平均序列数目为32 239.43±5 485.52条)。两组间有效序列数、优化序列数含量均无统计学差异(P=0.29,P=0.12)。
2.2肠道菌群稀释性曲线分析 稀释性曲线是从样本中随机抽取一定数量的个体,统计这些个体所代表的物种数目,并以个体数与物种数来构建曲线。它可以用来比较测序数据量不同的样本中物种的丰富度,也可以用来说明样本的测序数据量是否合理。从图1可以看出,随着抽样数量的增加,曲线趋向平坦,说明测序数据量合理,基本达到测序深度。

图1 LPRD患者和健康对照组肠道菌群的稀释性曲线
2.3肠道菌群的物种分布及多样性分析 60个样本中的1 994 353条优化序列归至细菌界的10个门、19个纲、29个目、47个科、116个属、130个种,99.99%的菌群可以划分到10个菌门中,其中厚壁菌门、拟杆菌门、变形菌门、放线菌门、软壁菌门占菌群的99.37%。
2.3.1α多样性分析 LPRD组患者肠道菌群Ace指数、Chaoz指数和香农指数略高于对照组,辛普森指数略低于对照组,但差异均无统计学意义(P>0.05),提示与健康人相比,LPRD患者肠道菌群丰度和多样性均无明显改变(图2)。
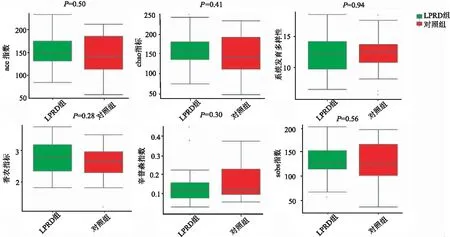
图2 LPRD组与健康对照组间alpha多样性分析 分别采用ace 指数(P=0.50)、chao指数(P=0.41)、系统发育多样性指数(P=0.94)、香农指数(P=0.28)、辛普森指数(P=0.30)、sobs指数(P=0.56)行两组间alpha多样性分析均无显著差异
2.3.2β多样性分析 主坐标分析(PCoA):横坐标表示一个主成分,纵坐标表示另一个主成分,百分比表示主成分对样品差异的贡献值;图3中的每个点表示一个样品,以TM和TF开头的为LPRD组,以CM和CF开头的为对照组。如果样品距离越接近,表示物种组成结构越相似,因此群落结构相似度高的样品倾向于聚集在一起,群落差异很大的样品则会远远分开。如图3所示,LPRD组和对照组样本较为聚集未出现明显分离倾向,说明两组菌群的物种组成无明显差异。
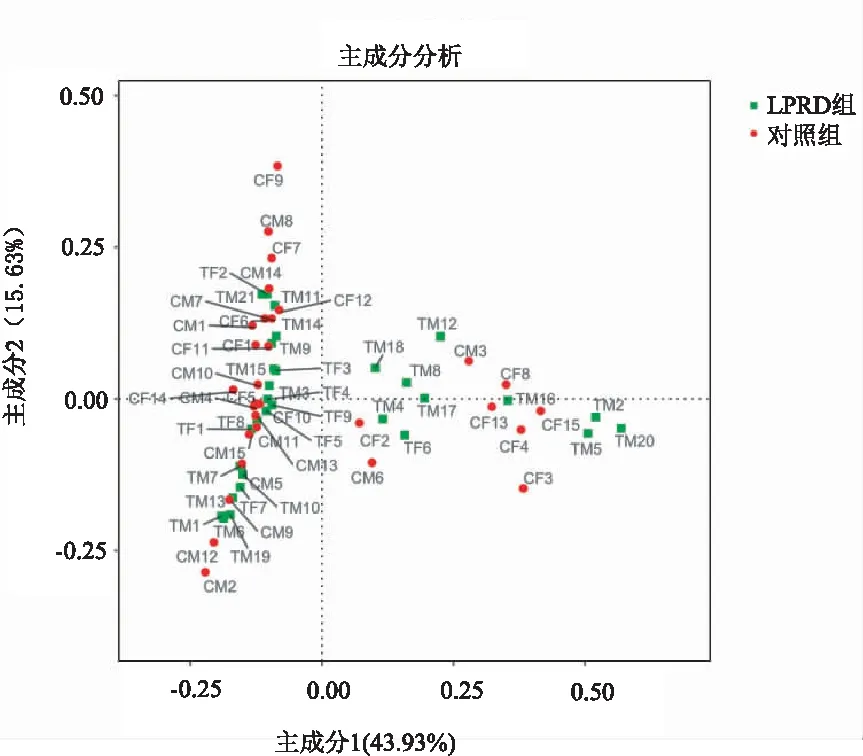
图3 LPRD组与健康对照组间Beta多样性分析
2.4LPRD组和对照组肠道菌群的结构差异分析
2.4.1门水平差异菌分析 在门分类水平上,厚壁菌门相对丰度在两组中均为最高,相对丰度最高的前5位一致,分别为厚壁菌门、拟杆菌门、变形菌门、放线菌门、梭杆菌门,与对照组相比,LPRD组放线菌门相对丰度显著高于对照组(P=0.03,图4)。
2.4.2纲水平差异菌分析 LPRD组放线菌门的红蝽菌纲相对丰度显著高于对照组(P=0.011,图4)。
2.4.3目水平差异菌分析 LPRD组红蝽菌目相对丰度显著高于对照组(P=0.011),假单胞菌目相对丰度显著低于对照组(P=0.018,图4)。
2.4.4科水平差异菌分析 LPRD组红蝽菌科、链球菌科相对丰度显著高于对照组(P=0.011,P=0.048),生丝微菌科、假单胞菌科相对丰度显著低于对照组(P=0.022,P=0.04,图4)。
2.4.5属水平差异菌分析 LPRD组瘤胃球菌属、柯林斯氏菌属相对丰度显著高于对照组(P=0.007 9,P=0.004 5),海洋杆菌属、假单胞菌属相对丰度显著低于对照组(P=0.022 2,P=0.040 1,图4)。
2.4.6种水平差异菌分析 LPRD组普雷沃氏菌种、梭状杆菌种、乳杆菌种相对丰度显著高于对照组(P=0.02,P=0.037,P=0.042,图4)。

图4 不同分类水平两组肠道菌群的结构差异
2.4.7LPRD组与对照组肠道菌群LEfSe差异分析 在LDA值≥2的条件下,两组中有显著性差异的物种有9种,其中有2个在对照组富集,即生丝菌科(P=0.022)和海洋杆菌属(P=0.022),另外7个在LPRD组富集,即放线菌门(P=0.030)、红蝽菌纲(P=0.010)、红蝽菌目(P=0.010)、红蝽菌科(P=0.010)、柯林斯氏菌属(P=0.004)、瘤胃球菌属(P=0.008)和普雷沃氏菌种(P=0.019)(图5),其中普雷沃氏菌在对照组样品中相对丰度为零,提示LPRD患者肠道菌群发生菌群失调,上述物种可能与LPRD有关(图6)。

图5 LEfSe差异分析LPRD组与健康对照组物种间差异 在LDA值≥2的条件下,两组中有显著性差异的物种有9种,生丝菌科(P=0.022)和海洋杆菌属(P=0.022)在对照组富集,放线菌门(P=0.030)、红蝽菌纲(P=0.010)、红蝽菌目(P=0.010)、红蝽菌科(P=0.010)、柯林斯氏菌属(P=0.004)、瘤胃球菌属(P=0.008)和普雷沃氏菌种(P=0.019)在LPRD组富集

图6 普雷沃氏菌种在LPRD组与健康对照组肠道相对丰度 两组间LEfSe差异分析普雷沃氏菌种相对丰度比较,对照组相对丰度趋向于零,提示了明显的差异
3 讨论
本研究采用16S rDNA测序的方法比较了LPRD患者与健康对照者肠道菌群的差异,对LPRD组和对照组肠道微生物菌群多样性进行了比较,结果显示代表肠道菌群丰度指数的Ace和Chao指数以及代表肠道菌群多样性的香农指数和辛普森指数均无明显差异,PCoA分析显示LPRD组和对照组样本较为聚集未出现明显分离倾向,提示LPRD患者较正常人肠道菌群丰度和多样性均无明显改变。对两组肠道微生物菌群结构比较发现显著差异菌,门、纲、目、科、属、种各水平分析显示放线菌门、红蝽菌纲、红蝽菌目、红蝽菌科、链球菌科、瘤胃球菌属、柯林斯氏菌属、普雷沃氏菌种、梭状杆菌种、乳杆菌种相对丰度LPRD组高于对照组,假单胞菌目、生丝微菌科、假单胞菌科、海洋杆菌属、假单胞菌属相对丰度对照组高于LPRD组。采用LEfSe组间群落差异分析两组肠道菌群,显示放线菌门、红蝽菌纲、红蝽菌目、红蝽菌科、柯林斯氏菌属、瘤胃球菌属、普雷沃氏菌种在LPRD组富集,生丝菌科、海洋杆菌属在健康对照组富集。放线菌门、红蝽菌纲、红蝽菌目、红蝽菌科、柯林斯氏菌属、瘤胃球菌属、普雷沃氏菌种可能为LPRD组的标志物,提示这些优势菌与LPRD可能具有相关性,可能在LPRD发生和发展中起作用。
胃肠道与支配其活动的神经中枢之间形成了一个双向的神经通路,称为脑-肠轴,而肠道菌群与脑肠轴之间还存在着密切的双向联系,构成了微生物-脑-肠轴。迷走神经含有80%的传入神经纤维,分布于胃肠道各个部位,通过感知其中的各种变化,向中枢神经系统传递信息,在微生物和大脑沟通中起重要的作用[13]。研究发现LPRD患者普遍存在迷走神经功能减低,以及交感神经功能的亢进[14],肠道菌群是否通过微生物-脑-肠轴影响自主神经功能、引起胃肠动力异常与LPRD的发病相关,目前还不清楚。Henke等[15]发现Crohn’s病患者肠道瘤胃球菌属合成活跃并分泌一种具有鼠李糖骨架链和葡萄糖侧链的复合葡聚糖多糖,这种葡聚糖多糖很有可能作用于TLR4,能有效诱导树突细胞分泌炎性细胞因子(TNFα),提示瘤胃球菌属在肠道炎症反应中可能起作用;Jie[16]的研究发现冠心病患者肠道内瘤胃球菌属增多,提示瘤胃球菌属与冠心病的发生有相关性。有研究发现TLR4信号传导通路是目前发现的重要的炎性通路之一,肾小管上皮细胞、心脏、呼吸道上皮细胞和肠上皮细胞也均表达TLR4[17]。Hoyles等发现肠道菌群的代谢产物可以通过TLR4信号通路对血脑屏障起调节作用[18],而迷走神经传入纤维上表达TLR4,这些纤维能感知细菌产物[19],本研究中瘤胃球菌属在LPRD组富集,为其优势菌,推测肠道瘤胃球菌属的代谢产物通过作用于迷走神经传入纤维的TLR4调节大脑,并通过迷走神经传出纤维调节食管、胃肠道,在LPRD发病中起作用。
放线菌门是目前公认的主要谱系中最大的分类学单位之一[20],GERD患者远端食管组织中存在放线菌的富集,可能在GERD发病中起作用[21]。神经系统通过胆碱能抗炎通路参与炎症的调节[22,23],也就是迷走神经将炎症信号传入中枢,中枢神经系统通过整合促使迷走神经释放乙酰胆碱,后者与炎症细胞表面的α7亚基N型胆碱能受体(α7 nicotinic acetylcholine receptors,α7nACHR)结合,调控炎症反应。研究发现放线菌中的肽聚糖相关脂蛋白具有诱导TLR2和TLR4表达增加的作用[24,25]。本研究结果显示LPRD组不仅放线菌门相对丰度显著高于健康对照组,而且同属于放线菌门的红蝽菌纲、红蝽菌目、红蝽菌科、柯林斯氏菌属均在LPRD组富集,为其优势菌。推测放线菌在LPRD患者肠道中富集,增加迷走神经传入纤维上的TLR表达,对肠道菌群的炎性代谢产物更加敏感,一方面通过传入中枢神经系统,刺激释放大量乙酰胆碱,使迷走神经传出活性增强,激活胆碱能抗炎通路试图抑制炎症反应,另一方面有些放线菌还可合成胆碱酯酶抑制剂,产生拟胆碱样作用,进一步激活迷走神经传出活性;大量的乙酰胆碱则激活了N受体[26],使自主神经节突触传递受阻,迷走神经功能下降,可能引起食管括约肌、食管体部甚至等胃肠道动力改变,从而与LPRD发生有关。
本研究还发现链球菌科、普雷沃氏菌种、梭状杆菌种在LPRD组富集,为LPRD的优势菌。有学者认为梭杆菌属、普雷沃菌属、消化链球菌属等为牙髓根尖周病的优势菌[27];一项单侧鼻窦炎的鼻窦菌群研究发现普雷沃氏菌属、梭杆菌属在上颌窦富集,为其感染菌[28]。这些研究提示梭杆菌属、普雷沃菌属、消化链球菌属在一定的环境下为致病菌,可能与上气道炎性疾病的发生相关。本研究通过LEfSe差异分析发现普雷沃氏菌种在健康对照组相对丰度为零,提示其可能在LPRD发病中起到更重要的作用。
本研究发现,LPRD患者肠道菌群与健康人相比虽然多样性无显著差异,但菌群结构存在不同,表现为一些特定优势菌的富集,提示LPRD患者可能存在肠道菌群失调,肠道菌群与LPRD具有相关性,优势菌可能在LPRD发病中起作用。推测LPRD患者肠道优势菌或其代谢产物可能通过TLR通路作用于迷走神经传入纤维,通过微生物-肠-脑轴的整合作用,将信号传递到迷走神经传出纤维而引起食管上、下括约肌松弛和胃肠功能变化,在LPRD发病中起作用。因咽喉菌群与肠道菌群有一致性的特点,LPRD患者这些优势菌或其代谢产物可能直接作用于咽喉部黏膜或通过迷走神经反射在LPRD发病中起作用。本研究只是初步对LPRD患者和健康人肠道菌群差异性进行了研究,推测肠道菌群与LPRD可能存在相关性,具体机制还需要进行深入的研究。

