基于DNA折纸术求解图的顶点着色问题的方法
麻晶晶 许 进
①(山西财经大学统计学院 太原 030000)
②(北京大学信息科学技术学院 北京 100871)
1 引言
分子计算的设想是由Feynman[1]在20世纪60年代初首先提出的,他指出单个分子或原子能够被用来构建计算机的组成元件。1994年,Adleman[2]第1次通过生物化学实验求解了一个7个顶点的哈密尔顿路问题,说明了DNA计算在解决复杂的数学问题方面的能力。随着生物学和纳米技术的快速发展,分子计算也得到了极大的发展。许多不同的方法已经证明生物分子能够作为开发更好的计算系统和提高计算能力的新工具。各种不同的DNA计算模型被设计出来,如粘贴模型[3]、剪接系统模型[4]、表面与芯片DNA计算模型[5]、发夹状DNA计算模型[6]、质粒DNA计算模型[7]、基于k-臂的DNA计算模型[8]、自组装DNA计算模型[9]、插入-删除系统模型[10]、试管型DNA计算模型[11]、图灵机DNA计算机模型[12]、布尔DNA计算机模型[13]等,以及将DNA计算的思想应用于基因分析与疾病诊断与治疗的模型等[14]。2016年,Xu[15]提出了一种探针机模型,该模型能够求解当今电子计算机无法处理的NP完全问题。近些年来的研究表明,DNA计算在理论、模型构建、实验方法和检测技术上都取得了很大的进展。
与此同时,DNA自组装技术的研究也取得了巨大的进展。1998年Winfree等人[16]在“简单自组装瓦片结构”的基础上,构建了具有足够刚性的5种DX元件。2003年,LaBean教授的研究组构造出一种十字形的支架结构(即四星元件)[17],利用这种模块组装成方形DNA自组装芯片。2006年,Rothemund等人[18]成功地应用了一条长为7000个碱基左右的M13噬菌体的单链,借助于200多条短的寡核苷酸链(钉书针链),通过碱基互补,在特定部位折叠成复杂的2 维D N A 自组装结构。依照Rothemund的方法,DNA分子可以构造人们想要的任何平面图形,因此,他的这一方法被形象地称为“DNA折纸术”。不满足于仅仅构造2维平面的简单图形,科学家们成功地在其它类型的DNA分子基础上设计出了新的DNA折纸方法,构造出了更为复杂高级3维DNA自组装结构。2011年,Han Dongran等人[19]利用DNA折纸的折叠技术,在3维空间中设计和构造了具有复杂的空间曲面的自组装DNA纳米结构,如球面、椭圆球面、瓶子等。之后,DNA折纸术的相关研究取得了很大的进展[20—30]。
图的顶点着色问题是著名的NP完全问题。2018年,Xu等人[31]提出了一种新颖的图顶点着色DNA计算模型,该模型以一个3-着色的61个顶点的图为例,设计了一种并行型聚合酶链反应(Polymerase Chain Reaction, PCR)操作技术,应用这种技术一次可以对图中多条边来删除非解,使得生物操作次数大大减少,极大地提高了运行速度。实验表明,99%的非可行解在构建初始解空间时就被删除,并利用DNA自组装和并行PCR方法,通过识别、拼接以及组装等技术得到解。该模型还通过如下3种方法来克服解空间指数爆炸问题:顶点颜色集的确定方法;子图分解方法以及子图中的顶点优化排序方法。本文提出了一种基于DNA折纸术求解图的顶点着色问题的方法,利用DNA折纸术可以构建出具有特定形状的DNA折纸结构。这些结构可以用来编码图的顶点和边,由于这些结构具有粘性末端,因此可以通过特异的分子杂交组装成代表不同的图的顶点着色方案的高级结构。利用DNA-纳米颗粒共聚体的属性和电泳等实验方法,可以筛选出正确的符合条件的图的顶点着色方案。这种方法是一种高度并行的方法,可以极大地降低求解图的顶点着色问题的复杂度。
2 图的顶点着色问题
本文所言之图皆指有限、无自环、无圈的简单无向图,通常用G表示图。本文用V(G), E(G)分别表示图G的顶点集和边集。一个图G的着色是指对G中的每个顶点分配一种颜色,使得相邻的顶点着不同的颜色。换言之,是指对图G中的顶点集V(G)的划分:V(G)=V1∪V2∪···Vk, Vi≠φ(空集),Vi∩Vj=φ, i =1,2, ···, k。满足图G正常着色的最小的颜色数称为色数,记为χ(G)。图G的一个k-正常顶点着色,简称为图的k-顶点着色,是指用k[k≥χ(G)]种颜色对图G进行着色。通常用Ck(G)表示图G的全体k着色构成的集合。由于本文仅考虑图的3-着色问题,故k=3,我们总是假定颜色集为C3(G)={r,b,y},其中,r表示红色,b表示蓝色,y表示黄色。所以,求解图的3-着色问题可以认为是寻找从图的顶点集V(G)到颜色集{r,b,y}的一种映射:f: V(G)→{r,b,y},使得对于∀uv ∈E(G),f(u)≠f(v)。图的3-着色问题是一个NP-完全问题。本文以一个具有6个顶点的简单图(图1)为例说明所设计的基于DNA折纸术求解图的顶点着色问题的算法。
3 基于DNA折纸术求解图的顶点着色问题的算法
DNA折纸术最先由Rothmund提出,这种新的DNA自组装策略是DNA纳米技术的巨大进步,依据这种自组装方法,不仅2维平面的不同图形,如正方形、圆形、笑脸等形状可以被构造出,而且3维立体的高级结构也可以被构造出。本文利用DNA折纸术构造的不同形状的结构对图G的每个顶点进行编码,通过DNA分子的杂交和一系列实验方法获得图G顶点着色的正确解,由于DNA折纸结构可以在原子力显微镜下被观测出,所以图G的可行的顶点着色方案可以通过观察原子力显微镜而得到。本文设计的基于DNA折纸术求解图的顶点着色问题的算法包含以下4个步骤:(1)用DNA折纸结构来编码图G的信息;(2)将DNA折纸结构进行退火反应,通过自组装过程生成问题的所有可能的解;(3)删除非解,并提取出正确的解;(4)识别结果。以下是具体方法的描述。
3.1 编码
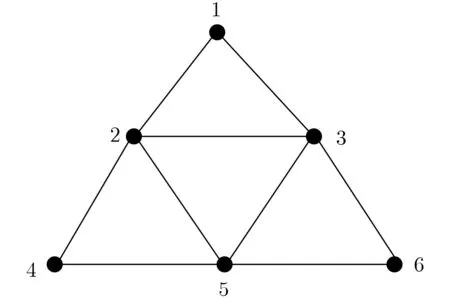
图1 一个简单无向图G
本文所使用的图如图1所示,它包括6个顶点、9条边。图2中以顶点1和顶点2为例展示了本文用DNA折纸结构编码图G的顶点和边的具体方法。由于本文求解的是图的3-着色问题,所以每个顶点都有可能染3种颜色,即红色、黄色和蓝色。为此以DNA折纸结构的不同形状来代表这3种颜色,即如图2所示,正方形的DNA折纸结构代表顶点被染成红色,圆形的折纸结构代表顶点被染成黄色,三角形的DNA折纸结构代表顶点被染成蓝色。每个DNA折纸结构上还具有突出的单链DNA,即粘性末端,这些粘性末端能依照图G中顶点的邻接关系相互杂交,粘性末端杂交的原理即DNA的两条单链在合适的实验室条件下依照碱基互补配对的规则(A与T 配对,G与C 配对)杂交形成双螺旋结构。
例如顶点1与顶点2、顶点3之间有边相连,所以顶点1有两条突出的粘性末端,分别能与顶点2、顶点3上的粘性末端杂交。按照这种方法,每个顶点都要设计正方形、圆形和三角形3种不同的DNA折纸结构,并且需要设计好需要的粘性末端序列,每个顶点所需要设计的粘性末端的数目恰好等于该顶点的度。同时,DNA折纸结构上还需要设计代表顶点序号的标记,以便在原子力显微镜下分辨顶点。
3.2 退火
设计好DNA折纸结构之后,就需要将所有代表顶点和边的信息的DNA折纸结构进行退火反应,这个过程由DNA粘性末端序列中碱基的互补配对完成,这是一个随机自由组合的反应,也是一个高度并行的过程,这种高度的并行性能在非常短的时间内形成问题的所有可能的解。图3中以顶点1和顶点3为例展示了DNA折纸结构的杂交方式。因为顶点1和顶点3有边相连,所以代表顶点1和顶点3的DNA折纸结构的其中两条突出的粘性末端可以互相配对杂交,杂交的方式如图中形成的双链所示。值得注意的是,由于顶点被赋予了颜色,所以两个顶点结构杂交之后可能是颜色相同的,也可能是颜色不同的,如图3(a)所示的杂交结构为顶点1和顶点3都是红色,如图3(b)所示的杂交结构是顶点1为黄色,顶点3为红色。有边相连的顶点被染成相同的颜色是不符合图的顶点染色问题的要求的,所以这种错误的解需要被删除掉。为了实现这个目的,本文在设计DNA折纸结构时预先给每一种颜色的DNA折纸结构设计了一段代表其颜色的特异序列,如图3中所示,在两个顶点结构的双链杂交部分的两侧即为本文预先设计的代表顶点颜色的特异序列,如图3(a)所示的顶点杂交结构中,顶点1和顶点3都被染成了红色,所以特异序列也是代表红色的序列,图中用红线表示;而如图3(b)所示的顶点杂交结构中,顶点1染成了黄色,顶点3为红色,所以特异序列也为相应的黄色和红色序列,图中分别用黄线和红线表示。可以想象,当顶点结构的粘性末端没有杂交形成双螺旋结构之前,这些特异序列(图3中的短的红色和黄色线段)是游离的而且靠近的几率很小,只有当顶点结构的粘性末端杂交形成双螺旋之后这些特异序列才会被拉近距离并且靠得很近无法分开。这样的设计将在后面删除非解的步骤中发挥作用。

图2 编码顶点和边的DNA折纸结构

图3 DNA折纸结构的退火方式
3.3 删除非解并提取正确的解
经过DNA折纸结构的退火之后,图G的顶点三着色问题的所有可能的解都已经形成,接下来的操作就是删除所有的非解,即删除掉退火结构中所有的顶点有边相连并且被染成相同颜色的染色方案,这个过程通过与DNA-纳米颗粒共聚体的杂交和琼脂糖凝胶电泳分离来实现。图4展示了用DNA-纳米颗粒共聚体与退火结构进行杂交的方法。如图4所示,金纳米颗粒上修饰了能与代表顶点颜色的两段特异序列杂交的互补序列,值得注意的是这段互补序列正好是特异序列的两倍,当且仅当两个杂交的顶点结构为染相同的颜色时,代表顶点颜色的两段特异序列才能被拉近距离,这样就能与DNA-纳米颗粒共聚体上的互补序列杂交。图4中以顶点1和顶点3为例展示了DNA-纳米颗粒共聚体与退火结构进行杂交的方法。顶点1和顶点3有边相连,它们有3种被染成相同颜色的情况,即同为红色、同为黄色和同为蓝色。如果退火结构中任意两个有边相连的点都染异色,那么DNA-纳米颗粒共聚体就不会与其杂交,这是因为本文设计的特异序列比较短,依照双链DNA分子的特性,在实验所需的温度条件下,单独一段长度较短的特异序列无法与互补序列杂交形成双链,只有两段特异序列组成的长序列才能与互补序列杂交形成双链。
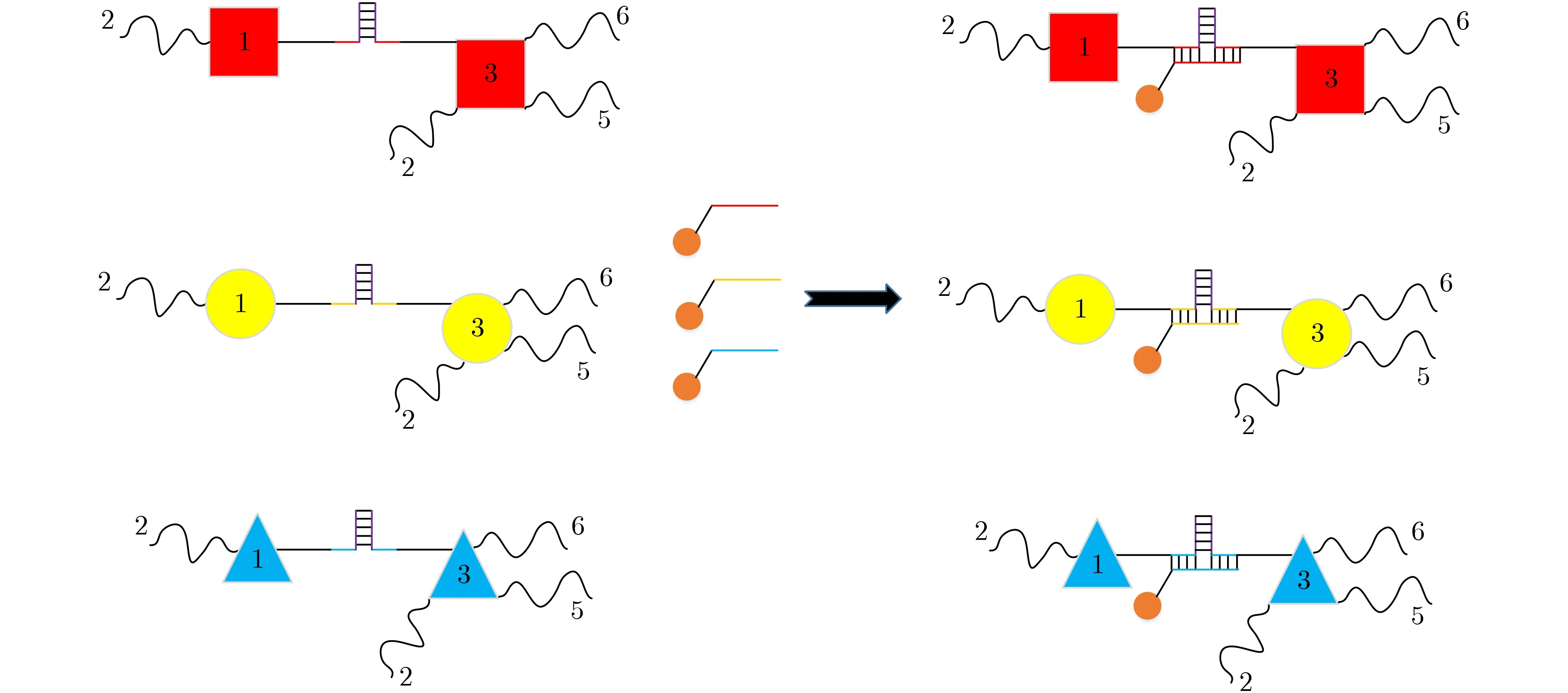
图4 DNA-纳米颗粒共聚体与退火结构的杂交
经过与DNA-纳米颗粒共聚体杂交之后,就要将产物进行琼脂糖凝胶电泳。在琼脂糖凝胶电泳中杂交了DNA-纳米颗粒共聚体的退火结构由于分子量较大在电泳的凝胶中处于落后的位置,没有杂交DNA-纳米颗粒共聚体的退火结构由于分子量较小处于领先的位置,就可以被回收回来,即为正确的解,将会用于下一步的识别和分析。
3.4 识别结果
由于DNA折纸结构可以通过原子力显微镜观察到,因此对于上一步回收的退火结构,可以用原子力显微镜进行观察,以确定最终的图G的顶点三着色方案。由于红色、黄色、蓝色分别由正方形、圆形和三角形DNA折纸结构表示,因此在原子力显微镜下就可以通过辨认折纸结构的形状和顶点的标记确认正确答案。图5显示了一种杂交结构,这是一种正确的图G的顶点三着色方案。

图5 图G的顶点三着色方案的正确解
4 结论
图的顶点着色问题有着非常广泛的理论与应用研究意义,各种DNA计算模型都将其作为研究的目标。DNA自组装技术为DNA计算提供了新的方法,各种DNA自组装计算模型也被用于解决各种不同的NP完全问题。本文中,我们利用DNA折纸结构编码图的信息,通过DNA折纸结构上粘性末端的自组装过程,构建了一种图的顶点着色问题的非确定性算法。通过构建数以亿计的参与计算的DNA折纸结构,该算法可以并行地测试每种可能的顶点着色方案。对于给定的n个顶点和m条边的图,顶点着色方案存在与否以及存在几种顶点着色方案都可以通过相应的DNA折纸结构的自组装过程和一系列实验方法计算出来,计算结果可以直接通过原子力显微镜观察到,这种方法具有高度的并行性,类似的基于DNA折纸术的方法在求解NP完全问题方面具有巨大的潜力。

