蓖麻高密度遗传图谱构建亲本SNP多态性分析
杨俊芳 曹越 王宙 王亚 张宏斌 赵宜婷 王宏伟

摘要:为了筛选最佳构建蓖麻高密度遗传图谱的亲本组合,以农艺性状差异较大的蓖麻两性系SL1为父本,镶嵌型雌性系HCH3和HCH1分别为母本,基于全基因组重测序(WGS)技术和生物信息学分析方法对2组亲本进行SNP标记多态性分析。结果表明,2组亲本间多态性标记均比较丰富,其中以HCH1为母本的组合2的亲本多态性更佳,SNP多态性标记总数为581 158个,可用aa×bb型SNP标记为181 791个。最佳亲本组合的确定为构建蓖麻的高密度遗传图谱、多种农艺性状定位和基因挖掘奠定了良好的基础。
关键词:蓖麻;全基因组重测序;亲本;SNP;多态性分析
中图分类号:S563.903.2 文献标志码: A 文章编号:1002-1302(2021)09-0053-05
蓖麻(Ricinus communis L.,2n=20),属于大戟科的单型蓖麻属[1],是雌雄同株异花植物,正常株蓖麻花序轴上方为红色雌花,下方为黄色雄花,圆锥花序,常异花授粉,多级分枝,无限花期。蓖麻是一种重要的能源油料作物,因其蓖麻油具有的特殊物理性质,被应用于航空、航天、军工、工程塑料、化工、纺织等数十个行业[2]。但是受困于蓖麻传统育种的局限性,在选择具有理想性状的植株,特别是多基因控制性状时,需要很长时间且缺乏精确性,蓖麻品种改良进程缓慢。分子标记辅助育种(MAS)方法可以有效地解决这些问题。遗传图谱和数量性状定位(QTL)已成为多种植物分子标记辅助育种的重要工具[3-8]。
在蓖麻上关于遗传图谱构建的研究起步较晚,研究者也较少。国内学者毕川等最早开始蓖麻图谱构建工作[9],2016年构建了第1张相对完整的遗传图谱[10],共331个标记(317个SSR、7个SRAP标记、3个SSRAP标记、3个形态学标记、1个ISSR标记)分布在10个连锁群上,覆盖 1 164.73 cM 基因组,平均标记间隔为3.63 cM。2019年又新构建了1张SSR标记遗传图谱并对蓖麻雌性复杂性状进行了定位[11]。2016年Tomar等通过遗传群体筛选出 141 个(RAPD、 ISSR、SSR)标记,构建了遗传连锁图谱,找到了关于蓖麻抗枯萎病的2个 QTLs[12]。2017年Tomar等筛选出 336 个(RAPD、ISSR、SSR) 标记,构建了遗传连锁图谱,并定位到了与蓖麻木炭腐病相关的新型QTLs[13]。2019年Yu等基于GBS测序技术对200个重组自交系(RIL)群体进行测序分析,构建了10个连锁群(LGs)的高分辨率遗传图谱包含8 896个高质量的SNPs,覆盖1 852.33 cM基因组,筛选到了16个控制种子大小和质量的QTLs[14]。
近年来,随着第2代测序技术迅速发展和测序成本的降低,高通量测序技术为植物基因型鉴定和遗传作图带来了方法上跨越式的突破[15]。以SNP 标记为代表的新一代分子标记大批量地用于植物高密度遗传图谱的构建[16-19]。传统的SSR、RAPD、AFLP、RFLP等分子标记上图量少,标记间遗传距离较大、基因组覆盖率低,而SNP标记的上图量远远超过传统分子标记,图谱分辨率高、定位精度高。蓖麻全基因组测序完成于2010年,形成了蓖麻的第1张基因草图,基因组大小约350 M,组装水平为Scaffold,Scaffolds有25 763条[20]。蓖麻测序工作的完成为开展蓖麻基因组水平的研究奠定了基础,但是由于蓖麻基因组组装水平并未到染色体,而且Scaffolds也比较多,组装水平还远远落后于很多作物。据此可以借助构建高密度遗传图谱的方法,对蓖麻的多种农艺性状进行QTL定位,挖掘相关基因,加快蓖麻分子标记辅助育种的进程。而且高密度的遗传图谱可以辅助基因组组装,提高全基因组精细图谱完整性。
本研究以农艺性状差异较大的蓖麻两性系SL1为父本,镶嵌型雌性系HCH3和HCH1为母本分别构建F1、F2、BC1群体。基于全基因组重测序(WGS)技术和生物信息学分析方法对2组亲本进行SNP标记多态性分析,以便筛选出最佳组合,为构建蓖麻高密度遗传图谱奠定良好的基础,进而对蓖麻多种农艺性状进行QTL定位和基因挖掘。
1 材料与方法
1.1 試验材料
以蓖麻两性系SL1为父本,镶嵌型雌性系HCH1和HCH3为母本,2017年分别杂交收获F1,2018年F1自交收获F2、与母本回交收获BC1。父本SL1为绿秆、密刺、中秆、早熟、花籽的正常两性株,且雄花比例极大,单株主穗甚至无雌花,仅在分枝穗上有少量雌花。母本1:HCH3为红秆、无刺、高秆、晚熟、小黑籽有极少量镶嵌雄花雌性系。母本2:HCH1为红秆、密刺、高秆、晚熟、小黑籽有少量镶嵌雄花的雌性系。2019年5月将3个亲本材料、F2及回交BC1群体种植于山西农业大学(山西省农业科学院)经济作物研究所,正常田间管理。
1.2 基因组DNA提取及测序
用剪刀取幼嫩蓖麻叶片0.1~0.5 g,用植物基因组DNA提取试剂盒(Solar Bio)提取每个样品的基因组DNA。利用Eppendorf AG 22331蛋白/核酸分析仪(Eppendorf公司,德国)检测DNA浓度及纯度,用1.0%琼脂糖凝胶电泳检测DNA质量。按照高通量测序的要求,每样品的DNA总量≥1 μg,浓度≥50 ng/μL;样品纯度D260 nm/280 nm为1.8~2.0,D260 nm/230 nm为1.9~2.4。电泳结果主带清晰,无降解,可用于高通量测序。检验合格的DNA样品寄送北京诺禾致源生物信息科技有限公司进行建库,并利用illumina HiSeqTM PE150进行测序。
1.3 信息分析方法
原始测序数据基于Raw data进行质量评估,去除掉包含接头信息、低质量碱基、未测出的碱基(以N表示)等干扰信息,最终得到有效数据,即Clean data 或 Clean reads。经质控过滤后的有效测序数据通过 BWA 软件(参数:mem-t4-k32-M)比对到蓖麻参考基因组(蓖麻全基因组数据库TIGR,http://castorbean.jcvi.org/downloads.php),比对结果经 SAMTOOLS 去除重复(参数:rmdup)。采用GATK等软件进行群体SNP的检测。
1.4 亲本间标记开发
基于蓖麻亲本基因型检测结果,进行亲本间多态性标记开发。过滤掉亲本信息缺失的位点,筛选父母本间具有多态性的位点,并从中筛选出符合F2群体作图标记类型的位点。用于F2群体作图的标记类型为aa×bb型,筛选父母本都为纯合且亲本间具有多态性的位点,即在某个SNP位点亲本1基因型为GG,亲本2基因型为AA,亲本基因型都为纯合,且亲本间基因型不相同。
2 结果与分析
2.1 测序数据统计及质量评估
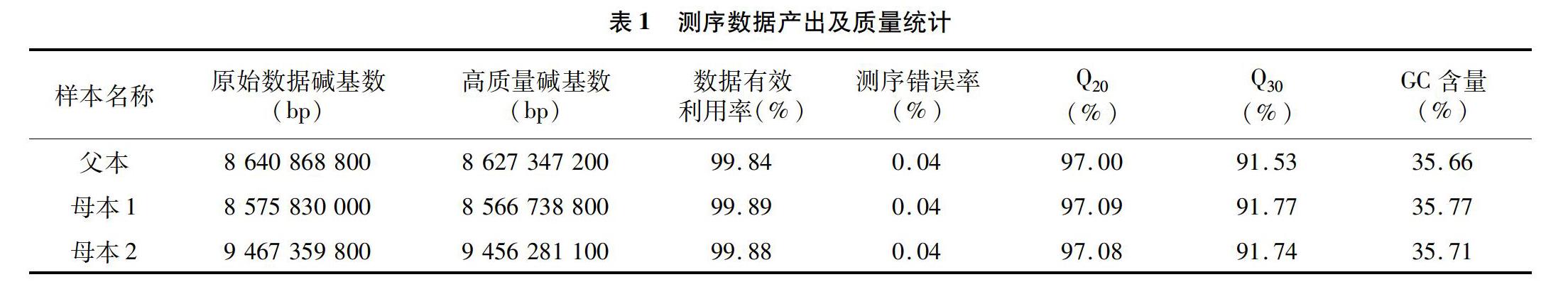
对经过严格质控过后的亲本测序数据进行统计(表1),可以看出母本2的碱基数最大,达到了947×109 bp。父本次之,为8.64×109 bp。母本1的碱基数最少,为8.58×109 bp。3个亲本的有效数据产量均达到了99.8%以上,测序错误率同为004%,Q20、Q30、GC含量也达到了测序要求。最终获得了高质量的Clean data,能够顺利进行下一步的比对工作。
2.2 Reads与参考基因组比对情况
蓖麻参考基因组大小为 336 968 299 bp。3个亲本的比对率在98%以上,对参考基因组(排除N区)的平均覆盖深度在20X以上,1X覆盖深度(至少有1个碱基覆盖)95.73%及以上,4X覆盖深度(至少有4个碱基覆盖)在92.35%及以上。亲本比对结果正常(表2),可用于后续的变异检测及多态性分析。
2.3 亲本SNP检测结果
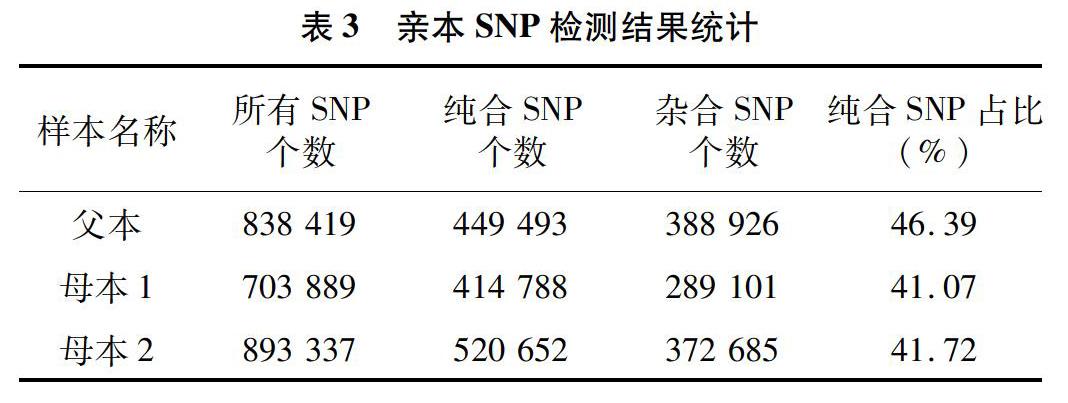
将比对到蓖麻基因组上的reads挑选出来,采用 GATK对数据进行群体SNP的检测,统计得到的SNP相关信息。父本和母本2的SNP标记总数和纯合标记数量均高于母本1。亲本SNP检测结果见表3。
2.4 标记多态性分析
以父本SL1和母本HCH3(母本1)为杂交组合1构建F2群体,多态性标记总数为542 928个,可用aa×bb型标记为171 829个(表4)。以父本SL1和母本HCH1(母本2)为杂交组合2构建F2群体,多态性标记总数为581 158个,可用aa×bb型标记为181 791个(表5)。通过2个杂交组合间不同类型标记数量比较,可见由父本SL1和母本HCH1(母本2)搭配的组合2的总标记数和可用型标记数都较优。这与田间观察到的表型性状分离情况也一致,组合2构建的F2群体、BC1群体分离的多样性比组合1更丰富。同时,通过表4和表5明显看出aa×bb型标记并不是最多的标记,hk×hk类型标记最多,超过25万个,在组合1中同样存在类似的问题。由于hkxhk与nnxnp、lmxll型标记均适用于由杂合亲本构建的F1作图群体。而本试验的亲本均为纯合,且构建的为F2作图群体,故同一组合间不同标记数量的比较没有太大的参考价值,本试验仅对不同组合间的同一aa×bb型标记进行比较分析。
3 讨论与结论
在构建遗传图谱前首先要确定亲本组合,若亲本材料选取不当, 会直接影响图谱的质量以及准确性[21]。传统遗传图谱构建亲本组合的确定,需经过分子标记多态性筛选[22-25],高密度遗传图谱构建亲本的选择同样也需要进行标记多态性分析。
2018年花生上基于WGS技术对2个亲本及91个RIL群体进行测序,构建了高密度遗传图谱,亲本间总的SNPs标记数为97 571个,可用多态性SNPs标记为18 252个,最终上图标记数为8 869个,覆盖3 120 cM基因组,平均遗传距离1.45 cM[26]。2018年大豆上基于SLAF测序技术对亲本和149株RILs个体进行测序,构建了高密度遗传图谱,亲本间总的SNPs标记数为391 476个,可用多态性SNPs标记为53 132个,最终上图标记数为5 111个,覆盖 2 909.46 cM 基因组,平均遗传距离0.57 cM[27]。2019年在胡萝卜上基于WGS技术对2个亲本及137个F2群体进行测序,构建了超高密度遗传图谱,亲本间可用多态性SNPs标记为411 891个,最终上图标记为378 738个,覆盖1 306.8 cM基因组,平均遗传距离0.46 cM[28]。2020年在烟草上基于WGS技术构建了高密度遗传图谱,亲本间可用多态性SNPs標记为1 626 811个,最终上图标记为 45 081 个,遗传距离为3 486.78 cM,平均遗传距离0.495 cM[29]。本研究中获得的组合1亲本间多态性标记总数为542 928个,可用多态性标记为 171 829 个;组合2亲本间多态性标记总数为 581 158 个,可用多态性标记为181 791个。最终的上图标记量还需要经过对F2群体多态性标记分析,之后再过滤掉一部分缺失的、偏分离的标记才能确定。但是目前来看,通过2组亲本间的可用多态性标记分析,进一步对2个组合子代F2群体进行重测序均是可行的。相比较而言,组合2优于组合1。
在群体构建亲本选择恰当的基础上,通过高通量测序获得的SNP标记构建的图谱质量一般要远高于传统标记图谱。然而通过选择不同的测序技术、测序群体种类和大小获得的图谱质量会有很大差异。测序方法上常用到的有全基因组测序和简化基因组测序,简化基因组测序技术根据文库构建的不同原理又分为RAD、GBS、2b-RAD、ddRAD、SLAF测序技术[30]。理论上同一群体利用全基因组测序比简化基因组测序获得的图谱质量更高。但全基因测序的方法在成本上比简化基因组测序要高很多,因此简化基因组测序要比全基因组测序应用得更多。近年来玉米[31]、大豆[32]、花生[33]、芝麻[34]、红豆[35]、向日葵[36]、甜瓜[37]等多种作物均有用简化测序技术的研究成果。蓖麻基因组相对于其他作物较小,在测序成本方面利用全基因组测序技术相比其他作物更具有优势。
本研究通过对3个亲本的全基因重测序及亲本间的标记多态性分析表明,2组亲本间多态性标记都比较丰富,均可用来作为构建高密度遗传图谱的亲本。组合1亲本多态性标记总数为542 928个,可用aa×bb型标记为171 829个。组合2亲本多态性标记总数为581 158个,可用aa×bb型标记181 791。经比较确定了以SL1为父本和HCH1为母本的组合2为最佳亲本组合。最佳亲本的确定为构建蓖麻的高密度遗传图谱及进行多种农艺性状定位和基因挖掘奠定了良好的基础。
参考文献:
[1]Alexandrov O S,Karlov G I. Molecular cytogenetic analysis and genomic organization of major DNA repeats in castor bean (Ricinus communis L.)[J]. Molecular Genetics and Genomics,2016,291(2):775-787.
[2]楊俊芳,王 亚,曹 越,等. 蓖麻性别决定基因研究进展[J]. 山西农业科学,2020,48(7):1164-1167.
[3]Wheeler N,Sederoff R. Role of genomics in the potential restoration of the American chestnut[J]. Tree Genetics & Genomes,2008,5(1):181.
[4]Wu J,Li L T,Li M,et al. High-density genetic linkage map construction and identification of fruit-related QTLs in pear using SNP and SSR markers[J]. Journal of Experimental Botany,2014,65(20):5771-5781.
[5]Chen J,Wang N,Fang L C,et al. Construction of a high-density genetic map and QTLs mapping for sugars and acids in grape berries[J]. BMC Plant Biology,2015,15:28.
[6]Ren X,Wang J,Liu L,et al. SNP-based high density genetic map and mapping of btwd1 dwarfing gene in barley[J]. Scientific Reports,2016,6:31741.
[7]Jiang N,Shi S,Shi H,et al. Mapping QTL for seed germinability under low temperature using a new high-density genetic map of rice[J]. Frontiers in Plant Science,2017,8:1223.
[8]Zhao Y,Su K,Wang G,et al. High-density genetic linkage map construction and quantitative trait locus mapping for hawthorn (Crataegus pinnatifida Bunge)[J]. Scientific Reports,2017,7(1):5492.
[9]毕 川,陆建农,殷学贵. 蓖麻遗传图谱构建初报[J]. 内蒙古民族大学学报(自然科学版),2013,28(5):532-535,564.
[10]Liu S,Yin X E,Lu J N,et al. The first genetic linkage map of Ricinus communis L. based on genome-SSR markers[J]. Industrial Crops and Products,2016,89:103-108.
[11]Lu J N,Shi Y Z,Yin X E,et al. The genetic mechanism of sex type,a complex quantitative trait,in Ricinus communis L.[J]. Industrial Crops and Products,2019,128:590-598.
[12]Tomar R S,Parakhia M V,Thakkar J R,et al. Development of linkage map and identification of QTLs responsible for fusarium wilt resistance in castor (Ricinus communis L.)[J]. Research Journal of Biotechnology,2016,11(5):67-73.
[13]Tomar R S,Parakhia M V,Rathod V M,et al. Molecular mapping and identification of QTLs responsible for charcoal rot resistance in castor (Ricinus communis L.)[J]. Industrial Crops and Products,2017,95:184-190.
[14]Yu A,Li F,Xu W,et al. Application of a high-resolution genetic map for chromosome-scale genome assembly and fine QTLs mapping of seed size and weight traits in castor bean[J]. Scientific Reports,2019,9(1):11950.
[15]赖国荣,张 静,刘 函,等. 基于GBS构建玉米高密度遗传图谱及营养品质性状QTL定位[J]. 农业生物技术学报,2017,25(9):1400-1410.
[16]唐立群,肖层林,王伟平. SNP分子标记的研究及其应用进展[J]. 中国农学通报,2012,28(12):154-158
[17]Li B,Lu X,Dou J,et al. Construction of a high-density genetic map and mapping of fruit traits in watermelon (Citrullus lanatus L.) based on whole-genome resequencing[J]. International Journal of Molecular Sciences,2018,19(10):3268.
[18]Jiang J,Fan X,Zhang Y,et al. Construction of a high-density genetic map and mapping of firmness in grapes (Vitis vinifera L.) based on whole-genome resequencing[J]. International Journal of Molecular Sciences,2020,21(3):797.
[19]Talukder Z,Underwood W,Ma G,et al. Genetic dissection of phomopsis stem canker resistance in cultivated sunflower using high density SNP linkage map[J]. International Journal of Molecular Sciences,2020,21(4):1497.
[20]Chan A P,Crabtree J,Zhao Q,et al. Draft genome sequence of the oilseed species Ricinus communis[J]. Nature Biotechnology,2010,28(9):951-956.
[21]鄭 剑,李兴星,苏华英,等. 粳稻资源热粳35遗传图谱构建与耐热QTL分析[J]. 核农学报,2017,31(5):844-851.
[22]单红丽,李文凤,黄应昆,等. 甘蔗抗褐锈病基因定位亲本间多态性SSR标记筛选[J]. 核农学报,2019,33(11):2119-2125.
[23]许梦琦,李双铃,任 艳,等. 花生作图亲本间分子标记的多态性分析[J]. 湖北农业科学,2015,54(11):2763-2766.
[24]黄焕焕,张桂华,韩毅科,等. 黄瓜作图亲本间分子标记的多态性分析[J]. 华北农学报,2007,22(2):47-49.
[25]贺 丹,吴芳芳,张佼蕊,等. 牡丹转录组SSR信息分析及其分子标记开发[J]. 江苏农业学报,2019,35(6):1428-1433.
[26]Agarwal G,Clevenger J,Pandey M K,et al. High-density genetic map using whole-genome resequencing for fine mapping and candidate gene discovery for disease resistance in peanut[J]. Plant Biotechnology Journal,2018,16(11):1954-1967.
[27]Zhang Y,Li W,Lin Y,et al. Construction of a high-density genetic map and mapping of QTLs for soybean (Glycine max) agronomic and seed quality traits by specific length amplified fragment sequencing[J]. BMC Genomics,2018,19(1):641.
[28]Luo X,Xu L,Wang Y,et al. An ultra-high-density genetic map provides insights into genome synteny,recombination landscape and taproot skin colour in radish (Raphanus sativus L.)[J]. Plant Biotechnology Journal,2020,18(1):274-286.
[29]Tong Z,Zhou J,Xiu Z,et al. Construction of a high-density genetic map with whole genome sequencing in Nicotiana tabacum L.[J]. Genomics,2020,112(2):2028-2033.
[30]魏庆镇. 黄瓜果实长度性状QTL定位及候选基因的筛选[D]. 南京:南京农业大学2016:19-20
[31]Wu X,Feng F,Zhu Y,et al. Construction of high-density genetic map and identification of QTLs associated with seed vigor after exposure to artificial aging conditions in sweet corn using SLAF-seq[J]. Genes,2019,11(1):37.
[32]Liu D L,Chen S W,Liu X C,et al. Genetic map construction and QTL analysis of leaf-related traits in soybean under monoculture and relay intercropping[J]. Scientific Reports,2019,9(1):2716.
[33]Zhang S,Hu X,Miao H,et al. QTL identification for seed weight and size based on a high-density SLAF-seq genetic map in peanut (Arachis hypogaea L.)[J]. BMC Plant Biology,2019,19(1):537.
[34]Du H,Zhang H,Wei L,et al. A high-density genetic map constructed using specific length amplified fragment (SLAF) sequencing and QTL mapping of seed-related traits in sesame (Sesamum indicum L.)[J]. BMC Plant Biology,2019,19(1):588.
[35]Li Y,Yang K,Yang W,et al. Identification of QTL and qualitative trait loci for agronomic traits using SNP markers in the adzuki bean[J]. Frontiers in Plant Science,2017,8:840.
[36]Zhou F,Liu Y,Liang C,et al. Construction of a high-density genetic linkage map and QTL mapping of oleic acid content and three agronomic traits in sunflower (Helianthus annuus L.) using specific-locus amplified fragment sequencing (SLAF-seq)[J]. Breeding Science,2018,68(5):596-605.
[37]Oren E,Tzuri G,Dafna A,et al. High-density NGS-based map construction and genetic dissection of fruit shape and rind netting in Cucumis melo[J]. Theoretical and Applied Genetics,2020,133(6):1927-1945.

