野生和栽培大理茶居群的遗传多样性与群体结构
毛娟,江鸿键,杨如兵,李崇兴,马成英,陈亮,马建强*
野生和栽培大理茶居群的遗传多样性与群体结构
毛娟1,2,江鸿键3,杨如兵4,李崇兴5,马成英6,陈亮1,马建强1*
1. 中国农业科学院茶叶研究所,浙江 杭州 310008;2. 西藏自治区农牧科学院,西藏 拉萨 850000;3. 云南省临沧市农业农村局,云南 临沧 677000;4. 云南省临沧市临翔区农业农村局,云南 临沧 677000;5. 云南省临沧市茶叶科学研究所,云南 临沧 677000;6. 广东省农业科学院茶叶研究所,广东 广州 510000
大理茶是栽培型茶树的野生近缘种,了解大理茶居群的遗传多样性和群体结构,对于大理茶资源的有效保护和开发利用至关重要。利用30对茶树核心SSR引物,对3个代表性的野生和栽培大理茶居群进行遗传分析。结果表明,30对SSR引物在所有大理茶样本中都能扩增出特异性产物,每个位点检测到的等位基因数为2~14个,范围为0.041~0.877,平均0.491。3个大理茶居群具有中等水平的遗传多样性,其中大雪山(DXS)野生居群的遗传多样性相对较低。香竹菁(XZQ)和白莺山(BYS)栽培居群的近交水平很高,近交系数()分别为0.728和0.913。居群配对分析显示,居群间遗传分化指数()均小于0.15,基因流()大于1。分子方差分析表明3个大理茶居群的遗传差异主要来源于居群内(94.1%)。聚类分析显示,野生和栽培大理茶单株的遗传距离相对较大。基于PCoA和Structure的群体结构分析显示,野生居群的遗传背景单一,栽培居群的遗传背景较复杂,其中7个BYS栽培居群单株可能是大理茶和阿萨姆茶通过渐渗杂交形成。
大理茶;SSR;遗传多样性;群体结构
大理茶[(W.W. Smith) Melchior]属于山茶属茶组[Sect.(L.) Dyer],是栽培型茶树[(L.) O. Kuntze]的野生近缘种。大理茶是茶组植物中较原始的种,相比栽培型茶树,其典型形态学特征为子房5(7)室、柱头5(7)浅裂或条、子房被茸毛、嫩枝和顶芽无茸毛等[1]。大理茶自然种群主要集中分布在怒江和澜沧江流域,扩散至云桂黔交界和中缅边境地区,是分布范围最广、数量最多的野生型茶树[2]。大理茶资源的农艺性状和生化成分存在较丰富的遗传变异[3],Ogino等[4]利用低咖啡碱大理茶特异资源,发掘了咖啡碱合成酶1(TCS1)的优异等位基因,开发了能够鉴别咖啡碱含量的功能标记。此外大理茶还可能参与了阿萨姆茶[var.(Masters) Kitamura]的驯化过程[5]。由此可见,大理茶具有重要的经济和科学研究价值。
深入了解种质资源的遗传多样性和遗传结构是有效保护和利用这些资源的前提。基于分子标记的遗传学分析,避免了形态学和生化指标易受环境和基因显隐性的影响,能更好地评估群体遗传参数。AFLP、SSR和SNP等分子标记已被普遍应用于茶树遗传学研究[6],但针对大理茶的相关研究较少。季鹏章等[7]采用AFLP标记对大理茶居群进行分析,发现居群间的遗传分化较低,遗传变异主要存在于居群内。Zhao等[8]利用SSR标记对野生、过渡型和栽培大理茶居群进行分析,发现大理茶整体上具有中等程度的遗传多样性,野生居群的遗传多样性相对较低,过渡型大理茶是由野生大理茶驯化而来。李苗苗等[5]利用SSR标记对野生大理茶、混栽的过渡型大理茶和阿萨姆茶居群进行分析,发现大理茶和阿萨姆茶之间遗传分化显著,在混栽居群中过渡型大理茶向阿萨姆茶有明显的种间杂交渐渗。
云南被认为是茶树的起源中心[2],茶树种质资源十分丰富。临沧市位于云南省西南部,地处澜沧江与怒江之间,是大理茶的主要分布区。双江大雪山大理茶居群是已发现的海拔最高、密度最大的野生大理茶居群[9],风庆香竹箐和云县白莺山大理茶居群是现存最古老的栽培大理茶居群之一。本研究评估了30对茶树核心SSR引物在大理茶中的通用性和多态性,并对3个最具代表性的大理茶居群进行遗传多样性分析,进一步认识其遗传组成和亲缘关系,为保护和利用这些资源提供科学理论依据。
1 材料与方法
1.1 材料
试验样品来源于云南省临沧市,包括3个大理茶居群,其中大雪山(DXS)为野生大理茶居群,分布于原始森林中,香竹箐(XZQ)和白莺山(BYS)为栽培大理茶居群,与阿萨姆茶居群混生。基于样本包含尽可能多的遗传变异原则[10],参考Zhao等[8]研究结果,并结合居群的实地情况,确定每个居群取样数量为20~30株(表1)。采样时先根据居群分布进行分区,在各区内随机取样,单株间距离10 m以上。采集的幼嫩枝条用脱脂棉包裹保湿带回实验室,取完整叶片用液氮快速冷冻,放置于–80℃冰箱保存备用。
1.2 SSR检测
DNA提取参照多糖多酚植物基因组DNA提取试剂盒(天根生化科技有限公司)方法进行,使用1%琼脂糖凝胶电泳和超微量分光光度计(NanoDrop 2000)检测DNA质量和浓度。检测合格的DNA稀释至20 ng·μL-1,放入–20℃冰箱保存备用。30对茶树核心SSR引物由本实验室前期开发[11],SSR荧光引物委托上海祥音生物科技有限公司合成,PCR反应体系与扩增程序参见毛娟等[12]方法进行。PCR产物使用3730xl DNA分析仪进行毛细管凝胶电泳检测。
1.3 数据分析处理
根据3730xl DNA分析仪检测结果,统计扩增片段大小,使用GenAlEx 6.5[13]将数据转换成不同分析软件所需格式。遗传多样性参数分析,分别使用PowerMarker 3.25[14]计算等位基因数()、基因型数()、多态信息含量();使用PopGene 1.32[15]计算期望杂合度()、观测杂合度()、居群配对Nei’s遗传相似性()和遗传距离(),以及Hardy-Weinberg平衡卡方检验。根据等位基因频率,使用BOTTLENECK 1.2.02[16]检测各居群的瓶颈效应;使用Arlequin 3.5[17]进行分子方差分析(Analysis of molecular variance,AMOVA),计算居群内近交系数()、以及居群配对遗传分化系数()和基因流()。
根据Nei’s遗传距离,使用PowerMarker 3.25利用邻接法(Neighbor-joining tree,NJ)构建系统进化树。主坐标分析(PCoA)在GenAlEx 6.5上完成。使用Structure 2.3.4[18]进行群体遗传结构分析,K值设置为2~7,Length of Burnin Period设置为10 000,Number of MCMC Reps after Burnin设置为100 000,Interation设置为10;使用Structure Harvest[19]计算ΔK,确定最佳K值;利用CLUMPP[20]对最佳K值运算结果进行重复抽样分析。
2 结果与分析
2.1 SSR多态性
30对核心SSR引物在所有样本中都能扩增出特异性产物,总共检测到176个等位基因和326个基因型(表2)。单个位点检测到的等位基因为2~14个,平均5.9个,基因型为2~32个,平均10.9个。值范围为0.041~0.877,平均0.491,其中高多态性位点有18个(≥0.5),中度多态性位点5个(0.25≤<0.5),低度多态性位点7个(<0.25)。观测杂合度范围为0.100~0.943,平均0.444,期望杂合度范围为0.042~0.895,平均0.532。大多数(27/30)位点的观察杂合度低于期望杂合度。卡方检验结果显示83.3%(25/30)位点显著偏离Hardy-Weinberg平衡(<0.01)。
2.2 遗传多样性
在3个居群中,BYS的多态性位点为100%,DXS和XZQ为80%(表3)。BYS居群检测到的等位基因(144)、基因型(191)和特异等位基因(45)均最多,平均每个位点4.8个等位基因和6.4个基因型;其次为DXS居群,共检测到100个等位基因(平均3.3)、169个基因型(平均5.6)和14个特异等位基因;XZQ居群最少,共检测到99个等位基因(平均3.3)、146个基因型(平均4.9)和8个特异等位基因。BYS居群的值相对较高(0.519),其次为XZQ居群(0.391)和DXS居群(0.381)。BYS和XZQ居群的观测杂合度低于期望杂合度,而DXS居群的观测杂合度略高于期望杂合度。

表1 大理茶居群采样信息

表2 30个SSR位点的遗传参数
注:*Hardy-Weinberg平衡卡方检验,<0.01,下同
Note: *<0.01 for Chi-square test of Hardy-Weinberg equilibrium (HWE), the same below

表3 3个大理茶居群的遗传多样性
3个大理茶居群的瓶颈效应检测结果如表4所示。在IAM模型的假设下,DXS和XZQ居群都表现出显著的杂合度过剩(<0.01),而在SMM模型下,杂合度过剩位点相对较多,但未达到显著水平。BYS居群在2种模型的假设下,都未检测到显著的杂合度过剩。因此推测DXS和XZQ居群可能在近期内经历过瓶颈效应的影响。
2.3 遗传分化
BYS和XZQ居群的近交系数分别为0.728和0.913,表明居群内近交水平很高,而DXS居群仅为0.028(表3)。居群配对基因流和遗传分化系数显示,各居群之间的基因流水平很高,遗传分化指数()较低(表5)。分子方差分析的结果表明,遗传变异主要来源于居群内(94.1%),而居群间的遗传差异仅为5.9%(表6)。
2.4 聚类分析
居群配对遗传相似性和遗传距离结果显示,两个栽培大理茶居群XZQ和BYS的遗传距离最小(0.148),遗传相似性最高(0.862);野生居群DXS和两个栽培居群的遗传距离较大,遗传相似性较低,表明野生和栽培大理茶居群之间的遗传差异较大,亲缘关系较远(表7)。基于Nei’s遗传距离的聚类分析结果显示,野生大理茶聚为一类,栽培大理茶聚为一类,XZQ和BYS居群的单株之间具有较紧密的亲缘关系(图1)。

表4 大理茶居群瓶颈效应检测
注:IAM:无限等位基因模型;SMM:逐步突变模型;He/Hd:杂合子过剩位点/杂合子不足位点
Note: IAM: Infinite Allele Model. SMM: Stepwise Mutation Model. He/Hd: Loci with heterozygosity excess/deficiency

表5 居群配对基因流和遗传分化系数
注:矩阵上方为基因流,下方为遗传分化指数;#<0.001
Note: Pairwise gene flow values were showed in upper triangular, and fixation index were in lower triangular. #<0.001

表6 大理茶居群的遗传多样性分子方差分析

表7 居群配对遗传相似性和遗传距离
注:矩阵上方为成对遗传相似性,下方为遗传距离
Note: Pairwise genetic identity values were showed in upper triangular, genetic distances were in lower triangular

注:DXS01—DXS30:大雪山居群单株;XZQ01—XZQ20:香竹菁居群单株;BYS01—BYS20:白莺山居群单株
2.5 群体结构
利用PCoA分析构建70个大理茶样本的空间排序图,结果表明,野生DXS居群与两个栽培居群可以完全区分,而栽培居群中,7个BYS单株与其他单株可以完全区分,其余单株之间则区分度不明显(图2-A)。利用Structure进行群体遗传结构分析,结果表明K=3时,ΔK获得最大值(图2-B),即试验样本的最适亚群数为3。当K=3时,DXS居群的全部单株属于同一亚群,BYS居群的7个单株属于1个亚群,其他单株与XZQ居群属于1个亚群(图2-C)。基于PCoA和Structure的群体结构聚类分析结果具有较好的一致性。
3 讨论
SSR标记被广泛用于茶树种质资源的遗传多样性分析。本研究选用的30对茶树SSR引物[11],在大理茶样本检测到较高的多态性,表明这些标记具有很好的通用性,可以作为核心引物进行茶组植物遗传多样性的比较分析。30个SSR位点在所有大理茶样本中共检测到176个等位基因,平均为0.491,低于利用同一套SSR标记对茶树育成品种(=0.510)[11]和地方品种(=0.565)[12]进行分析获得的结果,说明本研究中大理茶群体的遗传多样性整体上呈中等水平,但相比其他茶树种质资源较低。
3个大理茶居群中,BYS居群的遗传多样性相对较高,而DXS和XZQ居群可能在近期内经历过瓶颈效应的影响,导致遗传多样性相对较低。XZQ和BYS居群的近交系数很高,出现观测杂合度低于期望杂合度的现象(表3),其原因可能是两个居群均为栽培大理茶,居群内个体呈零星分布,由于生境片段化导致花粉传播受限,随机交配系统被打破,造成居群内极高的近交程度。遗传分化指数表明,居群间出现了一定程度的遗传分化,但基因流水平很高(>1),说明居群间存在较频繁的基因交流,能抑制由遗传漂变引起的遗传分化,AMOVA结果也表明遗传变异主要存在于居群内个体间(94.1%),这与李苗苗等[5]、季鹏章等[7]和Zhao等[8]的研究结果类似,也符合多年生异交物种的遗传变异规律。
聚类分析和遗传结构分析结果表明,野生和栽培居群具有不同的遗传背景。DXS野生大理茶居群的遗传背景较一致,聚为一个亚群,且与两个栽培居群的遗传距离较大。XZQ和BYS栽培大理茶居群的遗传背景较复杂,可能由3个祖先亚群杂交而来,其中BYS居群的7个单株聚为1个亚群(图2)。栽培大理茶通常来自大理茶的野生群体,通过移栽或采集开放授粉种子进行繁殖和驯化而成,其遗传背景与同域分布的茶树居群密切相关[7-8]。本研究中采集的BYS居群样本与阿萨姆茶混生,因此推测上述7个单株组成的亚群是由大理茶和阿萨姆茶通过渐渗杂交形成,在表型上接近大理茶,但遗传上与阿萨姆茶更近。
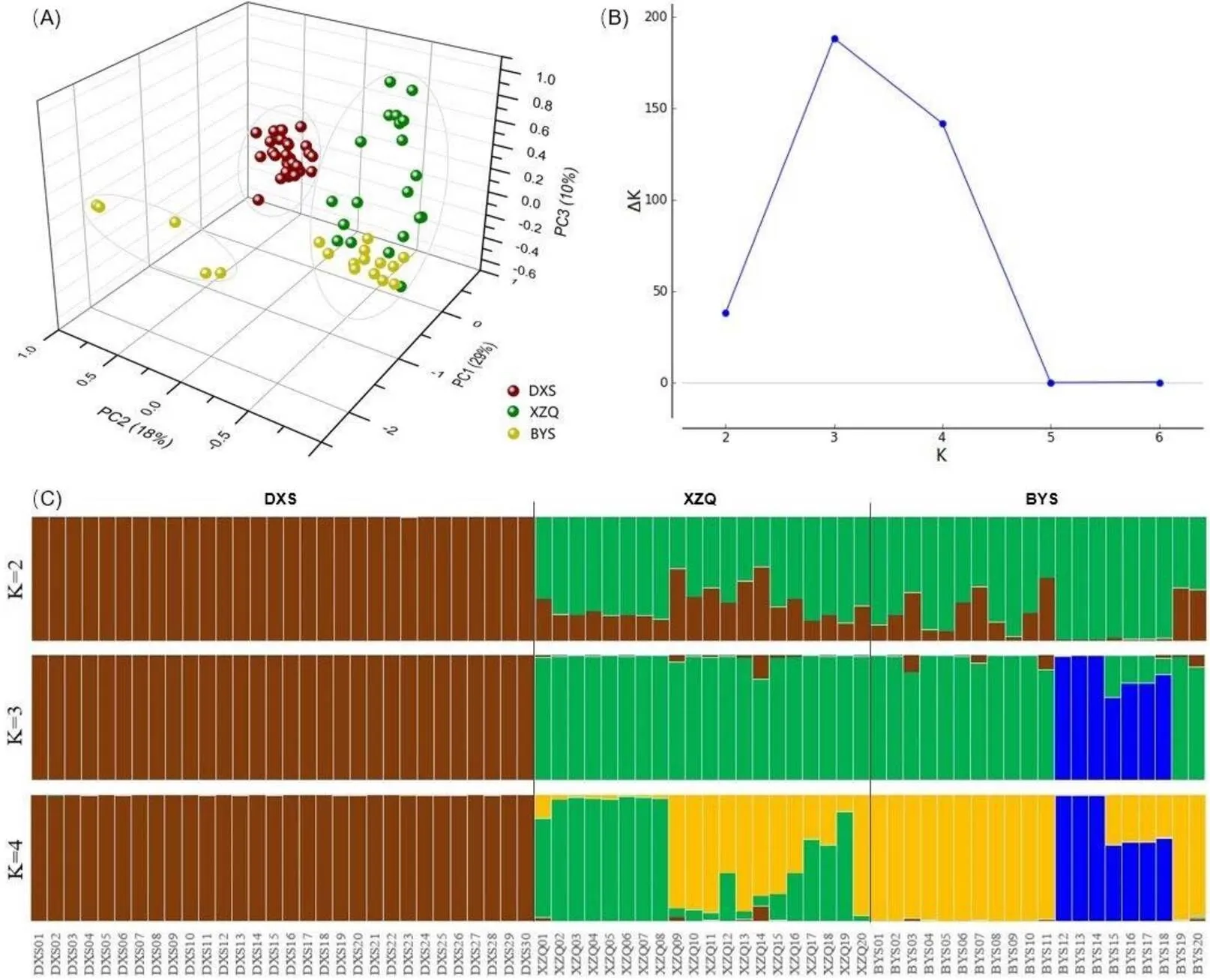
注:A:70个大理茶单株的主坐标分析;B:ΔK随亚群数K变化的关系图;C:3个大理茶居群K=2, K=3和K=4时的群体结构;DXS:大雪山居群;XZQ:香竹菁居群;BYS:白莺山居群
大理茶是研究茶树起源和栽培驯化的重要材料,也是茶树品种遗传改良的重要基因资源。近年来因野生茶和古树茶的市场炒作,对大理茶的非法采伐和过渡采摘时有发生,其遗传多样性受到严重威胁。本研究发现3个临沧市代表性大理茶居群表现出中等水平的遗传多样性,低于茶树育成品种和地方品种[11-12],因此应及时加强这些资源的保护;同时在制定保护策略时,因遗传变异主要存在于居群内,应首先考虑原地保护,并结合迁地保护对具有突出性状的优异资源进行扩繁,保证这些珍稀资源能够合理利用和有效保护。
[1] 陈亮, 虞富莲, 童启庆. 关于茶组植物分类与演化的讨论[J]. 茶叶科学, 2000, 20(2): 89-94.
Chen L, Yu F L, Tong Q Q. Discussions on phylogenetic classification and evolution of Sect[J]. Journal of Tea Science, 2000, 20(2): 89-94.
[2] 陈亮, 虞富莲, 杨亚军. 茶树种质资源与遗传改良[M]. 北京: 中国农业科学技术出版社, 2006: 11-45.
Chen L, Yu F L, Yang Y J. Germplasm and genetic improvement of tea plant [M]. Beijing: China Agricultural Science and Technology Press, 2006: 11-45.
[3] 段志芬, 杨盛美, 唐一春, 等. 云南大理茶遗传多样性分析[J]. 山西农业科学, 2019, 47(12): 2068-2072.
Duan Z F, Yang S M, Tang Y C, et al. Genetic diversity analysis offrom Yunnan province [J]. Journal of Shanxi Agricultural Sciences, 2019, 47(12): 2068-2072.
[4] Ogino A, Taniguchi F, Yoshida K, et al. A new DNA marker cafless-TCS1 for selection of caffeine-less tea plants [J]. Breeding Science, 2019, 69(3): 393-400.
[5] 李苗苗, Meegahakumbura M K, 严丽君, 等. 核基因组微卫星标记揭示大理茶参与了阿萨姆茶的驯化过程[J]. 植物分类与资源学报, 2015, 37(1): 29-37.
Li M M, Meegahakumbura M K, Yan L J, et al. Genetic involvement ofin the domestication ofvar.(Assimica Tea) revealed by nuclear microsatellite markers [J]. Plant Diversity and Resources, 2015, 37(1): 29-37.
[6] Sharma V S, Kumudini Gunasekare M T. Global tea science[M]. Cambridge: Burleigh Dodds Science Publishing Limited, 2016: 3-19.
[7] 季鹏章, 汪云刚, 蒋会兵, 等. 云南大理茶资源遗传多样性的AFLP分析[J]. 茶叶科学, 2009, 29(5): 329-335.
Ji P Z, Wang Y G, Jiang H B, et al. Genetic diversity offrom Yunnan province of China revealed by AFLP analysis [J]. Journal of Tea Science, 2009, 29(5): 329-335.
[8] Zhao D W, Yang J B, Yang S X, et al. Genetic diversity and domestication origin of tea plant(Theaceae) as revealed by microsatellite markers [J]. BMC Plant Biology, 2014, 14: 14.
[9] 宋永全, 苏祝成. 云南古茶树资源现状与保护对策[J]. 林业调查规划, 2005, 1(5): 108-111.
Song Y Q, Su Z C. Status quo of ancient tea tree resources of Yunnan and measures for protection [J]. Forest Inventory and Planning, 2005, 1(5): 108-111.
[10] 金燕, 卢宝荣. 遗传多样性的取样策略[J]. 生物多样性, 2003, 11(2): 155-161.
Jin Y, Lu B R. Sampling strategy for genetic diversity [J]. Chinese Biodiversity, 2003, 11(2): 155-161.
[11] 黄丹娟, 马建强, 陈亮. 茶树PVP申请品种的SSR分子标记鉴定和系谱关系分析[J]. 茶叶科学, 2016, 36(1): 68-76.
Huang D J, Ma J Q, Chen L. SSR identification and pedigree analysis of PVP application cultivars in tea plant [J]. Journal of Tea Science, 2016, 36(1): 68-76.
[12] 毛娟, 江鸿键, 李崇兴, 等. 云南白莺山地区茶树遗传多样性研究[J]. 茶叶科学, 2018, 38(1): 69-77.
Mao J. Jiang H J, Li C X, et al. Genetic diversity analysis of tea plant in Baiyingshan mountain of Yunnan [J]. Journal of Tea Science, 2018, 38(1): 69-77.
[13] Peakall R O D, Smouse P E. GENALEX 6: genetic analysis in Excel. Population genetic software for teaching and research [J]. Molecular Ecology Resources, 2006, 6(1): 288-295.
[14] Liu K, Muse S V. PowerMarker: an integrated analysis environment for genetic marker analysis [J]. Bioinformatics, 2005, 21(9): 2128-2129.
[15] Yeh F C. Population genetic analysis of co-dominant and dominant markers and quantitative traits [J]. Belgian Journal of Botany, 1997, 129: 157.
[16] Luikart G, Cornuet J M. Empirical evaluation of a test for identifying recently bottlenecked populations from allele frequency data [J]. Conservation Biology, 1998, 12(1): 228-237.
[17] Excoffier L, Lischer H E L. Arlequin suite ver 3.5: A new series of programs to perform population genetics analyses under Linux and Windows [J]. Molecular Ecology Resources, 2010, 10: 564-567.
[18] Hubisz M J, Falush D, Stephens M, et al. Inferring weak population structure with the assistance of sample group information [J]. Molecular Ecology Resources, 2009, 9(5): 1322-1332.
[19] Earl D A. STRUCTURE HARVESTER: a website and program for visualizing STRUCTURE output and implementing the Evanno method [J]. Conservation Genetics Resources, 2012, 4(2): 359-361.
[20] Jakobsson M, Rosenberg N A. CLUMPP: a cluster matching and permutation program for dealing with label switching and multimodality in analysis of population structure [J]. Bioinformatics, 2007, 23(14): 1801-1806.

Genetic Diversity and Population Structure of Wild and CultivatedPopulations
MAO Juan1,2, JIANG Hongjian3, YANG Rubing4, LI Chongxing5, MA Chengying6, CHEN Liang1, MA Jianqiang1*
1. Tea Research Institute, Chinese Academy of Agricultural Sciences, Hangzhou 310008, China; 2. Tibet Academy of Agricultural and Animal Husbandry Sciences, Lhasa 850000, China; 3. Lincang Municipal Bureau of Agriculture and Rural Affairs, Lincang 677000, China; 4. Linxiang District Bureau of Agriculture and Rural Affairs, Lincang 677000, China; 5. Tea Research Institute of Lincang, Lincang 677000, China; 6. Tea Research Institute, Guandong Academy of Agricultural Sciences, Guangzhou 510000, China
is a wild relative of tea plants (). Understanding of the genetic diversity and population structure ofis important and helpful for protecting and utilizing these germplasm resources. In this study, a set of 30 core SSR markers derived from tea plants were used for genetic analysis of three representative wild and cultivatedpopulations. The results show that all SSR markers successfully yielded specific amplification, with a range of polymorphic alleles from 2 to 14. Thevalue was between 0.041 and 0.877, with an average of 0.491. The threepopulations showed moderate levels of genetic diversity, and it was relatively lower for the Daxueshan (DXS) wild population. The inbreeding coefficient () of Xiangzhuqing (XZQ) and Baiyingshan (BYS) cultivated populations were 0.728 and 0.913 respectively, which meant high levels of inbreeding. Pairwise comparisons of the genetic differentiation index of three populations were low (<0.15), while the rates of gene flow were high (>1). The results of AMOVA exhibite that 94.1% of the genetic variation was within population. The genetic distances of wild and cultivatedindividuals were relatively higher. The genetic background of wild population was similar, while it was complex for cultivated populations. Seven individuals of BYS population were possibly originated from hybridization and introgression betweenandvar..
, SSR, genetic diversity, population structure
S571.1;Q949
A
1000-369X(2021)04-454-09
2020-12-28
2021-02-04
中国农业科学院科技创新工程(CAAS-ASTIP-2017-TRICAAS)、财政部和农业农村部:国家现代农业产业技术体系(CARS-19)、浙江省农业新品种选育重大科技专项(2016C02053)、广东省农业科学院院长专项(201838)
毛娟,女,硕士研究生,主要从事茶树种质资源与遗传育种研究。*通信作者:majianqiang@tricaas.com
(责任编辑:赵锋)

