基于生成对抗网络的肝脏CT图像分割
邓鸿 邓雅心 丁廷波 严中红 王富平 陈忠敏
0 引言
医学图像分割的目的是从医学图像中分离出目标区域,例如脏器、组织,再通过数字图像处理的手段实现可视化、图形引导手术、虚拟内径等[1]。在腹部成像,肝脏分割具有直接潜在临床应用:(1)自动肝脏体积测量,肝脏体积是肝外科手术一个重要的预后指标[2],也是确定肝肿瘤放射栓塞的放射剂量的重要参数[3];(2) 肝脏3D重建,肝脏3D重建能帮助外科手术制定手术计划[4]。同时肝脏分割也能应用到许多新兴的应用,如多回波磁共振(magnetic resonance ,MR)检查质子密度脂肪级(quantification of proton density fat fraction,PDFF)的量化,以协助诊断非酒精性脂肪肝病(nonalcoholic fatty liver disease,NAFLD)和肝形态学评估的管理,以帮助检测肝硬化[5]。
目前,肝脏图像的分割方法可以分为人工分割、半自动分割和自动分割。人工分割需要具有经验的影像科医生依据经验对肝脏CT图像进行手工划分,这种方法不仅耗时而且会因为医师的主观意愿而导致分割结果的差异性较大。半自动分割方法主要是根据CT图像中的灰度和梯度信息对肝脏进行分割,包括水平集法[6]、阈值插入法[7]、区域生长法[8]等,这些方法都需要人手工地设置参数,参数设置的不同会导致分割效果不同,同时这些方法如果是处理肝脏灰度值比较均匀的图像可能会出现欠分割或过分割现象[6-8]。自动分割方法主要指依赖于神经网络的一些方法,比如FCN[9]和Unet[10]网络;Shelhamer等提出的FCN网络因为其使用卷积层来替代了传统神经网络中最后一层的全连接层,因此FCN网络可以接收任意大小图片的输入从而实现图像分割端到端的训练。同时它使用增大了数据尺寸的反卷积层,能够输出精细的结构;结合不同深度层结果的跳级结构可以提高图像分割的鲁棒性和精确性。Unet网络基于FCN网络的基础上提出,融合了所有池化层的特征,使得该网络可以使用较少的训练图像就可以得到比较精准的分割结果。FCN网络和UNET网络已经可以分割出肝脏的大体位置,但是对于肝脏边缘的分割结果较差。
Vorontsov等[11]提出了对肝脏和肝脏病变区域进行级联分割的模型,该模型使用两个级联的全卷积神经网络,一个网络负责对肝脏区域进行分割,另一个网络负责对病变区域进行分割。该网络对于肝脏的分割效果欠佳,是因为没有考虑到相邻层的信息。Li 等[12]提出了一种混合紧密连接型Unet,该模型由两部分组成,分别为2D和3D的Unet,2D部分负责提取切片内的特征,3D部分负责对切片的Z轴的信息特征进行分层整合,该网络可以用于肝脏和肝脏肿瘤的分割。但是因为该模型运用了3D卷积网络,使得其变得不容易训练而且对硬件的需求较高。可以看到在对肝脏肿瘤进行分割时,研究者们都会先对肝脏组织进行分割,然后将只含有肝脏组织的图像送入到肿瘤分割网络进行肿瘤的分割。因此对于肝脏区域分割的准确与否也会影响到后续的肿瘤分割的精确度[11-12]。
生成式对抗网络[13]是2014年由Good-fellow等提出。生成对抗网络主要包括两个模块:生成器模块(generative model)和判别器模块(discriminative model),在训练过程中,生成网络的目标就是尽量生成真实的图片去欺骗判别网络。而判别网络的目标是把生成网络生成的图片和真实的图片区分开。这样,生成网络和判别网络构成了一个动态的“博弈过程”。通过在Unet的训练中引入生成式对抗网络,可以使得Unet网络预测的分割结果更加接近于真实标注的图像。
基于上述内容,本文将生成对抗网络和Unet结合为Gan-Unet网络对肝脏CT序列图像进行分割训练,从而使得肝脏自动分割预测的结果更加清晰、分割准确度进一步提高。同时,在进行对抗训练时探索了不同的距离约束函数对于分割结果的影响。
1 方法
1.1 分割网络
本文参考条件生成对抗网络(conditional generative adversarial nets,CGAN)[14]搭建肝脏图像分割网络,其框架示意图如图1所示,整个网络由一对互相对抗的生成器网络和判别器网络组成。
生成器采用Unet网络对肝脏CT图像进行图像分割预测,具体结构如图2所示。该网络包含收缩路径和扩张路径两部分,两部分结构是对称的,收缩路径主要通过卷积和池化操作对输入的CT图像进行不同尺度上的特征提取。收缩路径部分每一层都使用2次3×3的卷积核对图像进行卷积,再通过池化层将图像大小缩小一半继续进行相同的卷积操作,共进行4次下采样操作。在扩张路径部分,通过上采样操作从最底层开始对图像进行恢复。通过Concat操作对采样恢复的图像和收缩路径对应层的图像进行融合,将融合后的图像通过2个3×3卷积后继续重复上述操作最终将图像还原到输出大小,最后通过一个1×1的卷积层得到最后输出的分割结果预测图。整个网络在特征提取的时候卷积核大小设置为3×3是为了在可以最小化神经网络的复杂性的同时又能保证网络的预测分割准确率[15]。
判别器采用由5个卷积层组成的神经网络,如图3所示。输入层通过Concat操作把真实的图像标签和预测的图像标签结合在一起输入卷积网络,再通过4个步长为2的4×4卷积层卷积,将得到的结果最后经步长为1的4×4的卷积层进行卷积得到输出结果。
1.2 分割网络训练过程
整个分割网络的训练过程如下,首先通过生成器预测生成一张肝脏区域图片,然后将生成器预测的肝脏分割图和真实肝脏标签图片一起送入判别器中进行判别。如果生成的肝脏分割图和实际的分割图一样,那么判别器就会输出一个接近于1的数,否则输出一个接近于0的数。生成器再根据判别器的输出继续去生成预测分割图像,最终判别器不能区分出送进这个网络的到底是来自于生成器的图片还是真实的肝脏分割图片,这个网络就达到了一种纳什均衡的状态,也就完成了分割网络的训练。
1.3 分割网络损失函数
生成对抗网络的目的是让生成器产生的图像可以去欺骗判别器,让判别器不能够区分送进网络图片的真实性。故整个网络的优化目标:
Σln[1-D(label|G(x))]
(1)
式中:G和D分别代表生成器和判别器模型;D(label|x)代表肝脏分割标签图送入判别器网络计算后的输出;G(x)代表的是通过生成器生成的预测分割图;D(label|G(x))代表生成器网络生成的预测分割图送入到判别器网络计算后的输出。
整个网络的对抗训练目标是通过最小化生成器的损失函数Gloss[见式(2)],从而最大化判别器的输出。最终当网络达到纳什均衡以后整个网络就会表现出比较好的结果。
Gloss=μ1∑lnD(label|G(x))+
μ2∑Distance(G(x)-label)
(2)
式中:Distance()表示距离约束函数;μ1,μ2分别表示生成器损失函数和距离约束函数所占的权重。
2 实验设置
2.1 数据集的获取与处理
本次实验训练集采用的肝脏CT图像序列来自于LiTS(Liver Tumor Segmentation Challenge)[16]竞赛中公开的肝脏肿瘤数据集,该数据集总共采集了131例病患的腹部CT扫描图像,并且由具有经验的放射科医师对肝脏和肿瘤区域进行标注。原始的CT数据和标注数据都以nii格式保存。通过ITK-SNAP软件可以打开此文件进行查看(图4)。可以看到原始的CT序列文件可以在横断面、矢状面以及冠状面进行重建成像,在重建后,在横断面图像的大小为固定的512×512,在矢状面和冠状面的图像大小都会根据扫描的情况不同而变化,同时切片的数量根据每位患者的情况是不定的,一般从几百到上千张不等。故本次实验选取横断面的CT图像作为训练数据。
由于本文使用的是2D神经网络,故输入网络的图像也应该为2D图像。本文通过Python中的nibabel库将原始的nii图像切分为2D图像,同时将CT图像的窗宽窗位设置为[400,60],得到横断面的切片图像并保存。得到的训练集CT图像如图5所示。训练集中的标注图像对肝脏及肝脏肿瘤进行了标注,在图像上用不同的灰度值分别表示肝脏和肿瘤[图6(a)],本次实验因为只对肝脏进行分割,通过阈值化将肝脏肿瘤部分的灰度值设置为与肝脏灰度值相同[图6(b)]。经处理后,共有58 638张CT切片图像;其中超过一半以上的图片都是未含有肝脏的CT图像,如果把这些不含肝脏的图片全部送入网络训练,因为其标注图像的像素值全为0,在训练时如果选取的这个batch图像均为不含肝脏的图像,就会导致loss为零或者过大从而导致训练的不稳定,同时若含有太多的负样本可能对神经网络的训练导致过拟合或欠拟合现象。故本次实验从LiTS数据转换的切片中按照正负样本比例9∶1选取了18 962张含有肝脏的CT图像和2 033张不含肝脏的图像作为训练数据集。
图5 调节窗宽窗位至[400,60]的CT训练数据Figure 5 CT training data after adjusting window width and window level to [400,60]

图6 处理后的标注图像Figure 6 Annotated image after processing
本次实验采用的测试集来自于CHAOS[17]公开的肝脏CT数据集。CHAOS数据集中CT图像包含带有标注的20例训练集和不带标注的20例测试集数据,本次实验使用其中的20例带有标注的训练集数据作为分割网络的测试集。CHAOS数据其原始格式是dicom文件。通过Python的Pydiocm库对原始文件读取后调整窗宽窗位为[400,60],最后得到2 874张带有标注的CT图像。
2.2 肝脏图像分割评价指标
本文采用Dice、IoU、PA、RVD以及RSSD对肝脏分割网络进行评价。
2.2.1 Dice系数
(3)
式中:Ground是专家手动标注的金标准图像;Predict为分割网络预测出来的图像;Dice的取值范围为[0,1],Dice越接近1,分割网络预测的结果与金标准就愈加重合。
2.2.2 交并比
(4)
式中:Ground 是专家手动标注的金标准图像;Predict为预测图像。IoU分数和Dice一样评价预测图像和真实图像之间的重合度。
2.2.3 像素精确度
(5)
根据标注图像和预测图像之间的关系,式中TP(true positive)代表预测值为1,真实值为1的部分;TN(true negative)代表预测值为0,真实值为0的部分;FP(false positive)代表预测值为1,真实值为0的部分;FN(false negative)表预测值为0,真实值为1的部分。PA计算的是预测分割途中预测正确的像素占图像总像素的比例,该比值越接近1,说明网络预测的准确性越高。
2.2.4 相对体积误差
(6)
式中:Vground代表通过真实标注三维重建后的肝脏体积;Vpredict代表网络预测的分割图通过三维重建后的体积。可以看出该式的值越接近于0,表示分割网络预测的结果更加接近于真实的肝脏。肝脏体积对于肝脏精准外科手术和精准放疗是一个很重要的参数[18]。这个参数影响着治疗计划的制定以及患者术后的预后。所以使用相对体积误差对分割网络进行评价具有重大意义。
2.2.5 相对表面积误差
(7)
式中:Sground代表通过真实标注三维重建后的肝脏表面积;Spredict代表网络预测的分割图通过三维重建后的表面积。通过表面积的差异,可以清楚地了解到三维重建后肝脏表面的粗糙程度。同样,该式越接近于0,代表预测结果和真实的标注结果愈加接近。
3 实验结果与分析
3.1 实验环境及超参数设计
本实验采用的硬件配置:(1) CPU Intel(R) Core(TM) i5-7600K CPU @ 3.80 GHz;(2) NVIDIA RTX 2080Ti;(3) 内存16 GB ;(4) 操作系统:Windows10。
本实验主要探究不同网络结构对肝脏分割效果的影响,故将训练时的一些超参数如优化方法、训练周期数、Dropout率等根据经验以及实际的硬件配置设置为相同,如表1。

表1 超参数设置Table 1 Hyperparameter settings
3.2 GanUnet与Unet的训练与结果分析
为了对比GanUnet与传统Unet的分割效果,在分割网络训练时,GanUnet网络的Gloss中选取生成器自身部分的loss,即在式(2)中将μ2设置为0,μ1为1;Unet网络的loss函数采用交叉熵(Cross Entropy)作为损失函数。在训练时,每训练一个周期将训练模型的结果保存。在训练结束后,将测试集的CT图像序列输入到每个周期训练的模型中得到预测图像,统计测试集在每个训练周期上的Dice系数、IoU分数和PA分数的平均值。并在20组中选取结果最好的一组肝脏预测图片。将所得的预测图片在原始CT图像上提取出对应的部分后得到每一位患者肝脏分割图。通过3D Slicer软件对得到的肝脏分割图进行三维重建后计算RVD和RSSD分数。得到的结果如下(统计结果去掉最大值和最小值以防止误差过大)。
从图7可以看出,提出的网络在Dice和IoU上某些周期是高于Unet的,且提出的网络在PA指标上明显高于Unet。这证明本文搭建的网络在表现性能上是优于Unet的。GanUnet 3个指标出现的波动情况是由于生成对抗网络训练的特性导致的,同时受限于计算机性能,不能够保存每一步训练的结果,所以每个周期最后一步保存的模型可能是性能比较差的,以至于结果出现了波动。综合3个指标来看,本文提出网络的性能是优于传统的Unet网络。
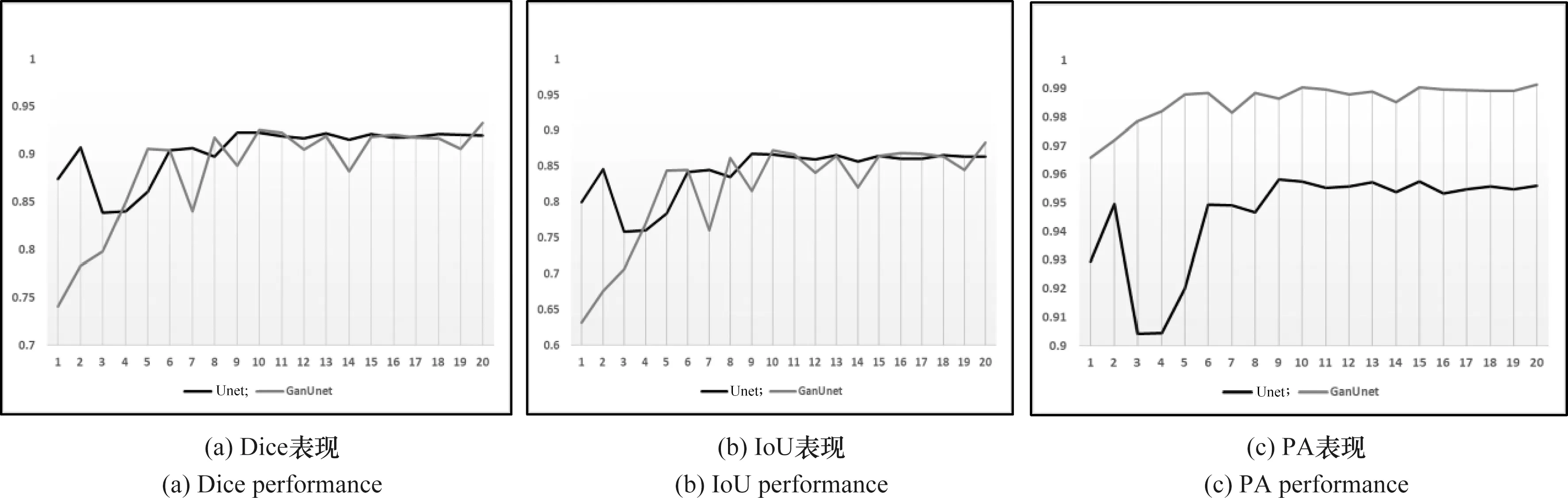
图7 GanUnet与Unet在测试集上Dice、IoU和PA的表现Figure 7 Performance of the test set Dice,IoU and PA in GanUnet and Unet
从表2可以看出,本文搭建的GanUnet网络,在4个评价指标上均优于传统的Unet分割方法,Dice系数相较于Unet提升了1.1%;IoU系数提升了1.8%;PA提升了3.5%;RVD降低了48.3%,RSSD降低了23.6%。使用的方法在Dice、IoU和PA上的提升不是很大,但是在RVD和RSSD上看来,都有较高的提升。故从评价指标上来看,本文使用的方法均优于传统的Unet方法。从图8可以看出,Unet网络能识别出肝脏所在区域的大体位置并进行分割,但是Unet网络对于肝脏边界的识别能力不足,导致预测分割图的边缘表现十分粗糙。本文使用的网络,很好地解决了Unet对于肝脏边缘识别不准确而导致的边缘分割粗糙的问题,使得预测图像更加接近标注图像。

表2 GanUnet与Unet指标定量分析Table 2 GanUnet and Unet quantitative analysis
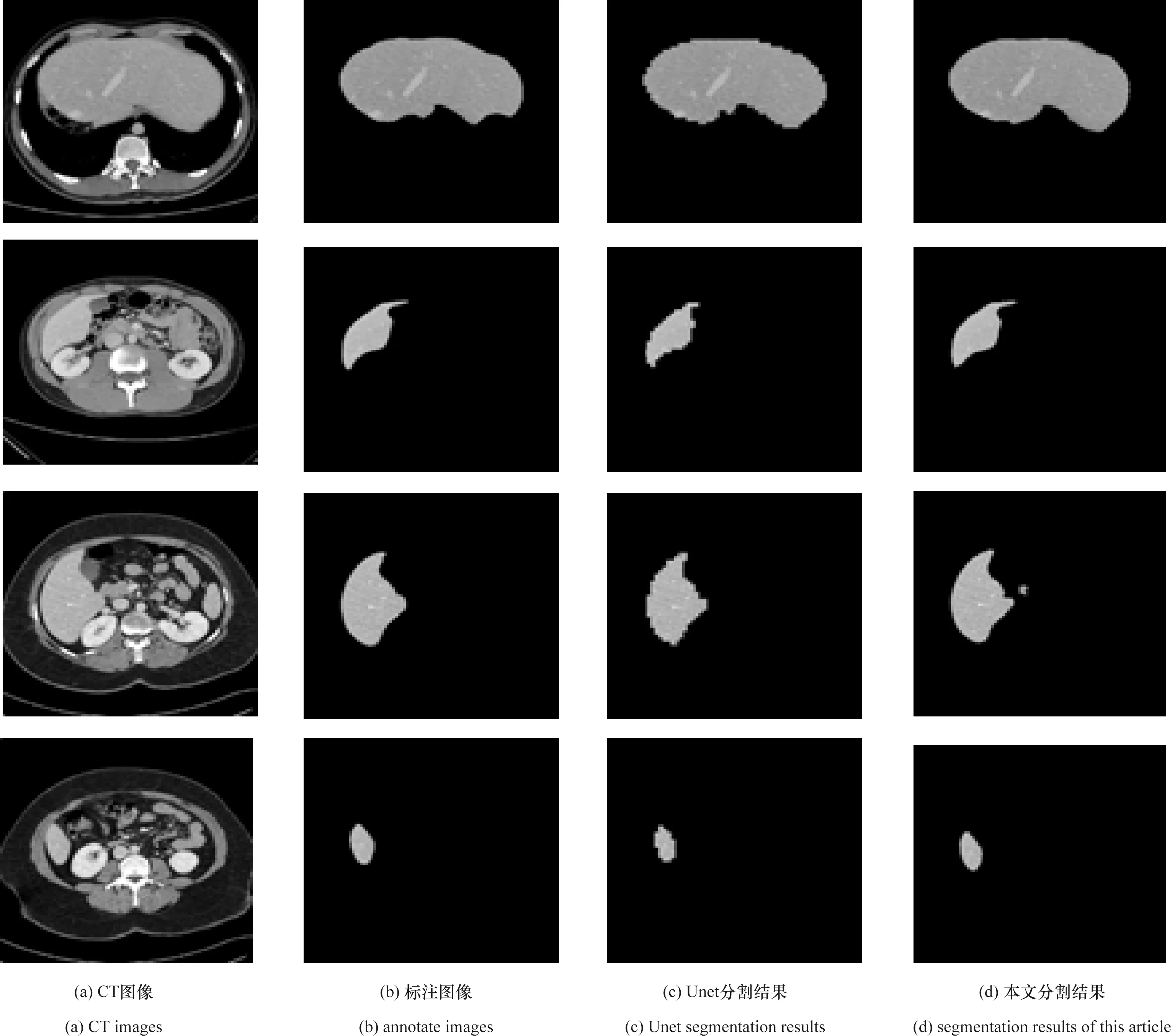
图8 使用不同方法的分割效果Figure 8 Segmentation results with different methods
3.3 加入距离约束函数对GanUnet的影响
为了进一步的提高GanUnet的肝脏分割效果,在GanUnet网络的Gloss中加入距离约束函数L1、L2、SmoothL1对肝脏分割效果图进行约束。即在式(2)中μ1=μ2=1,将Distance函数分别替换为L1、L2范数和SmoothL1距离约束函数得到如下的损失函数:
Gloss_l1=ΣlnD(label|G(x))+Σ|G(x)-label|(8-1)

Gloss_smoothL1=ΣlnD(label|G(x))+ΣSmoothL1(x)
(8-3-1)
(8-3-2)
同样的对L1、L2以及SmoothL1都训练20个周期,统计测试集在每个训练周期上的Dice系数、IoU分数和PA分数。并在20组中选取结果最好的一组肝脏预测图片。将所得的预测图片在原始CT图像上提取出对应的部分后得到每位患者的肝脏分割图,将其进行三维重建后计算出RVD和RSSD。
从图9可以看出,L1、L2以及SmoothL1约束后的GanLoss的表现性能都要优于单独的Gloss,同时在引入了约束函数后,网络的训练会变得更加的稳定,波动没有那么大。其中加入L2约束函数后的性能是在这几种方法中表现最好的。从表3可以看出,在训练时将Gloss通过L1、L2以及SmoothL1距离函数约束后,5个评价指标都有一定程度的提高,说明网络的分割性能有进一步的提升。其中Gloss+L2的分割效果要优于其他3种方法,可能的原因是L2距离函数计算的是两幅图像距离平方的差距,对于分割网络来说,标注的图像很大一部分区域都是为0的,只有含有肝脏区域的部分为1,用L2范数计算标注图像和预测图像之间差异的方法会体现出更大的差异;而L1和 SmoothL1函数在这个任务中大部分计算的是标注图像和预测图像之间的L1范数,差异较小。在Dice系数、IoU、PA、RVD和RSSD上,Gloss+L2相较于单一的Gloss分别提高了1.7%,3.4%,0.2%,38.1%,58.7%。可以看到加入L2约束后,一些二维评价指标的上升幅度不是很大,但是三维的评价指标RVD和RSSD都有明显的提高,说明加入L2距离函数对于分割性能有一定的提升。
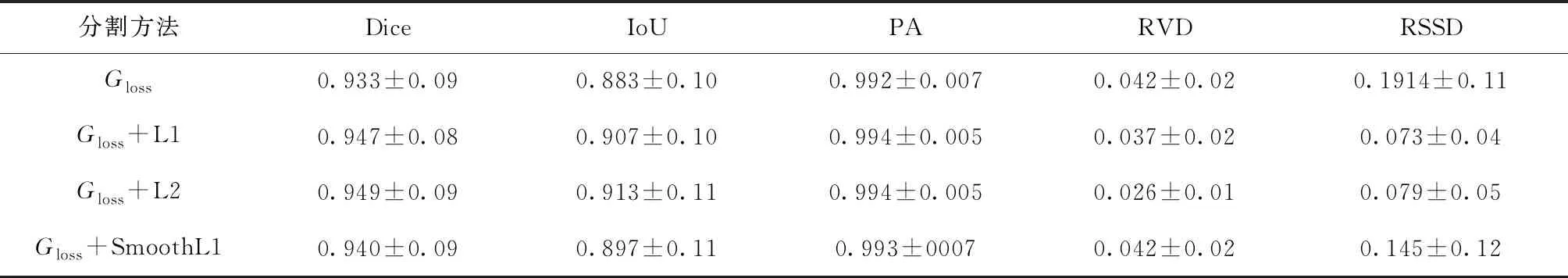
表3 不同方法的评价指标定量分析Table 3 Quantitative analysis of evaluation indexes of different methods

图9 引入距离约束函数后测试集的表现Figure 9 The performance of the test set after introducing the distance constraint function
4 讨论与结论
肝脏CT图像分割现在依然是一个值得研究的问题,一般的肝脏分割方法包括手动分割、半自动分割和自动分割。自从Unet网络被提出来后,研究人员基于Unet提出了一系列的改进方法。这些改动大多都是基于网络本身的改动,例如将简单的卷积层换为深度残差网络[19]或者空洞卷积[20],融合不同的损失函数[21]。这些方法虽然对分割效果有一定的提高,但是仍不理想。也有研究人员为了取得更好的分割效果基于Unet改造出了3D的Unet,但是3D-Unet网络对于计算机硬件的要求高,并且不容易训练。故本文从训练方式上改进Unet,使用基于生成对抗网络的Unet。GanUnet使得肝脏边缘的分割变得更加平滑和清晰。该网络在Dice系数、IoU系数、PA、RVD和RSSD这些评价指标上都优于传统的Unet网络,特别是在三维评价指标RVD和RSSD上GanUnet都明显优于Unet网络,RVD和RSSD分别提高了48.3%和23.6%。同时,为了进一步提高GanUnet的分割效果,引入了L1、L2和SmoothL1 距离约束函数对Gloss进行改造。结果表明L2距离约束函数可以进一步提高分割网络的效果,Dice、IoU和PA分别达到了94.9%、91.3%、99.4%,相比于Unet的Dice、IoU和PA为92.3%、86.7%、95.8%有一定的提升;在三维指标中,相对体积误差、相对表面积误差为0.026、0.079,相比于Unet的0.042、0.191有明显的下降。该网络预测结果的三维重建效果明显优于传统的Unet网络,肝脏体积这个参数对于肝脏的精准外科手术是非常重要的。
本文使用的网络分割结果在三维重建后更接近于标准肝脏体积,因此可以运用到肝脏的三维打印[22]和肝脏精准手术的术前规划[4]等。本文从训练方式改进Unet取得了一定效果,可以看到对抗训练方式不仅可以提高分割的精度,同时也能够提高图像分割的细节,这个对于医学辅助诊断治疗是非常重要的。在对抗训练中引入L1、L2和SmoothL1约束函数对Gloss进行约束后还可以进一步提高分割精度,可以看出不同的约束函数可能会适应于不同的任务。本文存在的不足:实验突出了GanUnet相较于传统Unet的优越性,但没有进一步训练调参找出最优的超参数。后续研究方向:可以针对GanUnet通过数据集增强、超参数调优以及引入其他约束函数对Gloss进行改造来进一步提高肝脏分割效果。

