叠氮溴化丙锭筛选环境活性微生物的原理及其应用
胡晓婧,白 鑫,曲娟娟,刘俊杰,孔令辉,王光华
(1.中国科学院 东北地理与农业生态研究所 黑土区农业生态重点实验室,黑龙江 哈尔滨 150081; 2.东北农业大学 资源与环境学院,黑龙江 哈尔滨 150030)
0 引 言
环境微生物是自然生态系统的重要组成部分,是驱动地球各圈层物质循环与能量交换的引擎。微生物介导的特定生态功能过程主要通过活性微生物来执行[1]。活性微生物是指具有或潜在具有生长代谢特性和适应改变既定环境能力的微生物[2],也是微生物生态学中微生物分离培养或功能鉴定的主要目标。尽管高通量测序已成为研究环境微生物多样性及其结构功能的主流技术[3],但环境样品中DNA的直接扩增测序无法反映出真实的活性微生物群,很可能高估了环境中活性微生物的种类和数量[4]。这是因为,环境微生物总DNA主要包括:(1)活性细胞DNA,具有代谢活性且可培养的完整细胞DNA[5];(2)休眠细胞DNA,低代谢活性且不可培养的活细胞和抗生素持留菌细胞,但在一定条件下或可恢复其代谢活性和可培养性的完整细胞DNA[6];(3)死细胞DNA,包括细胞膜受损且正在裂解的和已死亡的非完整细胞DNA[7];(4)胞外DNA,包括游离DNA和附着于其他物质上的DNA[8]。尽管细胞死亡后,其所含DNA可能被环境中的脱氧核糖核酸酶(Deoxyribonuclease,DNase)快速降解,但这种降解是不充分的,仍有大量死细胞DNA和胞外DNA长期存留于环境中[9]。因此,如何准确区分并量化环境活性微生物群,是当前环境微生物生态学研究中亟待解决的关键问题。
目前,国际上对活性微生物的判定主要有三个标准:(1)具有完整且有功能的细胞膜;(2)存在细胞代谢和能量流动过程;(3)具有能够自我复制的DNA并能转录成RNA,进而翻译成蛋白[10]。在此基础上,多种活性微生物筛选区分方法被逐步开发出来(图1),它们的相对优缺点见表1,主要分为两大类:可培养法和不可培养法[2]。可培养法已远不能满足探究环境微生物多样性的需求。而ATP生物发光技术、微生物呼吸法、酶活性检测法以及基于等温微热量法的热流动技术等都受到通量的限制,对环境活性微生物的检测没有普适性[11-13]。在满足高通量检测的基础上,基于RNA转录组学或蛋白组学技术、稳定性同位素示踪技术(DNA-SIP)已被广泛用于检测具有代谢活性的微生物类群,并适用于多种环境样品[14-16]。然而,这些方法的实验步骤较为繁琐,实验成本较高,且实验过程中也存在很多限制因素,如RNA极易降解从而增加了实验操作难度[17];稳定性同位素示踪技术通常针对某些特定的功能微生物,且存在培养周期长、同位素耗材昂贵等问题[18]。因此,急需建立更加普适性且操作简便的环境活性微生物的研究方法。
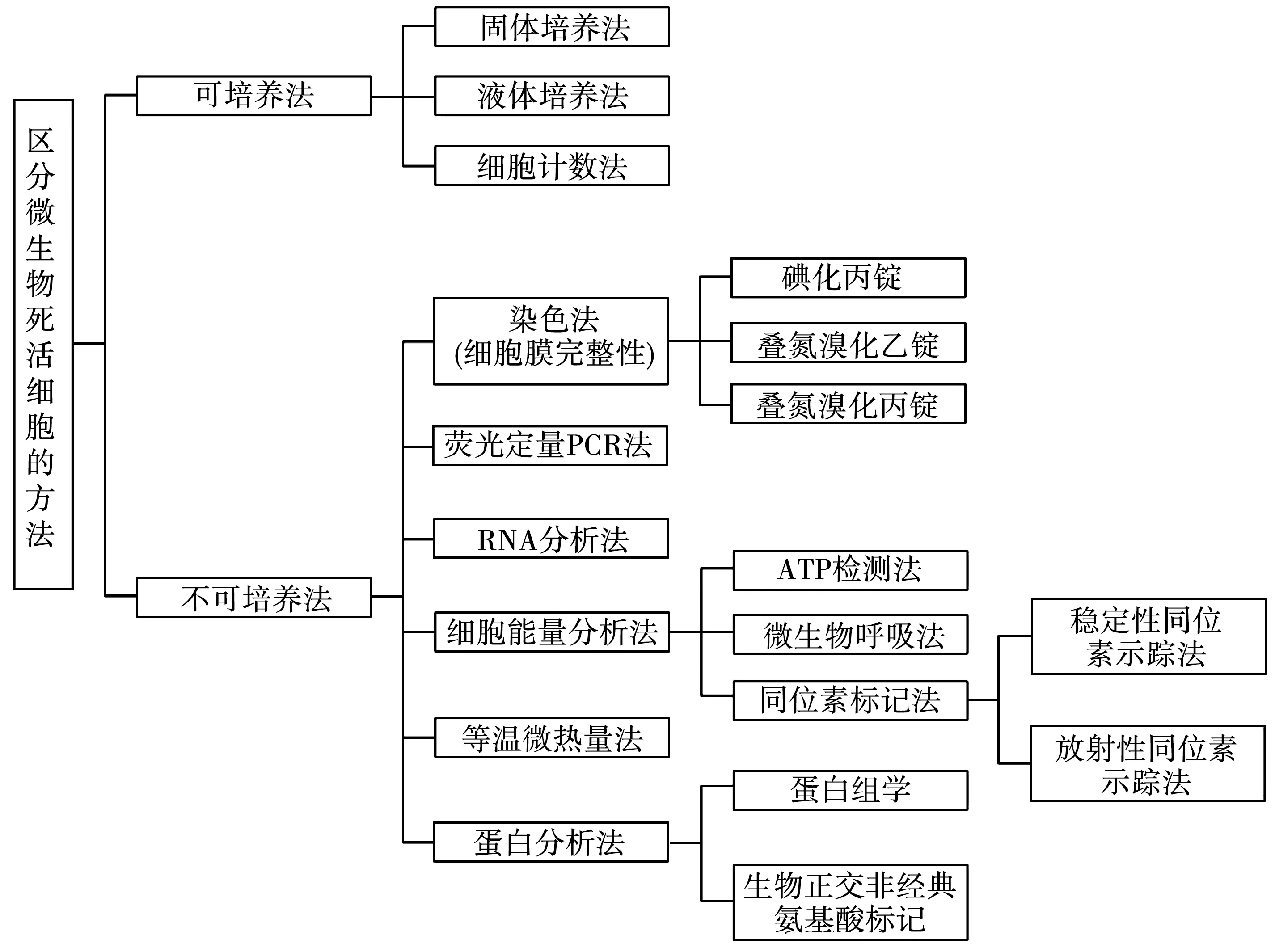
图1 活性微生物筛选方法概括[2]Fig.1 Highlight of techniques to distinguish live from dead microbes

表1 活性微生物检测的常用技术手段Table 1 Comparison of commonly used techniques to examine viable microbes
维持细胞膜的完整性是保持微生物活性的必要条件[19],因此,细胞膜也可以被当作活性微生物细胞的分子标记物。针对细胞膜完整性的检测,一系列核酸荧光染料应运而生,也为基于DNA的活性微生物检测开辟了新的思路。其中,碘化丙锭(Propidium iodide,PI)、叠氮溴化乙锭(Ethidium monoazide,EMA)和叠氮溴化丙锭(Propidium monoazide,PMA)是常用的光敏性核酸染料[20-22]。这类染料自身的荧光非常微弱,但与核酸结合后荧光信号增强,它们通常不能穿透活性细胞完整的细胞膜,但能够进入膜损伤后的死细胞,修饰钝化其DNA[22]。PI染料法只能在荧光显微镜、激光共聚焦扫描显微镜或流式细胞仪等仪器辅助下进行活性细胞的检测,无法实现样品的高通量测定,而PMA和EMA可与荧光定量PCR及高通量测序等技术相结合,更适合于复杂环境样品中活性微生物组的检测[23]。
1 PMA筛选活性微生物的基本原理
PMA染料法是一种简便、快速的筛选区分活性微生物的方法。它对DNA具有高度亲和性,在暗培养过程中可高效穿透死细胞的细胞膜,与暴露出来的DNA产生共价交联。在可见光(最大吸收波长460 nm左右)的作用下,PMA所携带的光敏性叠氮基团会转化为高反应性的氮宾自由基,并与其结合位点附近的任意碳氢化合物反应形成稳定的共价碳氮键,致使DNA分子被永久修饰,最后阻断DNA分子后续的PCR扩增等反应(图2)。同时那些残留在溶液中没有与DNA分子发生交联的PMA,在强光照射下可与水分子反应生成没有活性的羟胺[22,24-25]。以上过程均在环境样品DNA提取之前完成,由于PMA不能穿透活性细胞完整的细胞膜,故不会影响后续活性微生物的DNA提取和PCR扩增,最终使relic DNA在核酸纯化中被选择性剔除。PMA筛选后的活性微生物样品可用于后续的高通量测序等多组学研究[26-27]。

图2 PMA染料法选择性检测活性微生物细胞的流程Fig.2 Procedure of PMA dye in selectively detecting viable microbial cells
需要说明的是,EMA与PMA的工作原理基本类似,但EMA在随后的实验中被发现可穿透一些活性微生物的细胞膜,如EscherichiacoliO157:H7、Streptococcussobrinus、Micrococcusluteus、Staphylococcusaureus和Mycobacteriumavium等都能够吸收EMA[22],致使这些活性细胞被当作死亡的细胞而造成了假阴性结果。此外,由于EMA比PMA多携带一个正电荷,更易穿透活细胞的细胞膜,与带负电荷的DNA结合[27],因此,EMA被发现较PMA具有更高的细胞毒性,可能会杀死一些本来具有完整细胞膜的活性微生物[28-29],如EMA对Listeriamonocytogenes和Legionellapneumophila的细胞毒性就要比PMA强[24-25]。相比而言,PMA因其较高的活性细胞选择特异性,更适用于复杂环境样品中活性微生物的研究。
目前,基于光敏性核酸染料筛选区分活性微生物的方法大多应用于临床诊断、食品安全和动植物病原菌检验检疫等方面[26,29-30],并初步应用在复杂环境样品中活性微生物群的研究中,但仍处于起步阶段。本文结合最新国内外研究进展,从底泥、土壤和水体等环境样品方面综述了目前PMA染料法在微生物生态学中的应用,并提出PMA技术现存的一些问题及改进对策,为切实有效地开展环境活性微生物多样性及功能研究提供参考。
2 PMA在底泥研究中的应用
微生物是底泥生态系统的重要组成部分,驱动着底泥中养分循环与转化,是底泥生物活性的源泉[31]。Nocker等[32]首次将PMA应用于海洋和水库的底泥样品研究,结合PCR-DGGE技术研究发现,与未处理样品相比,PMA能够有效地抑制底泥样品中relic DNA的PCR扩增,导致微生物群落的DGGE指纹图谱和条带密度产生改变,从而筛选区分出底泥样品中活性微生物。Ramirez等[33]采集了北冰洋和太平洋区域多个地点的海洋底泥,用PMA去除样品中relic DNA,发现大多数样品处理前后的细菌16S rRNA基因拷贝数没有显著差异;基于细菌16S rRNA基因的测序结果显示,尽管有个别地点的relic DNA含量相对较高,但它们对原核微生物的群落组成并未造成显著影响;物种水平分析表明,原位的relic DNA可能来源于不同时期海洋底泥中的微生物,显示了PMA染料法也有助于海洋底泥微生物起源与演化的研究。Wagner等[34]采用PMA染料法筛选发酵罐底泥中活性微生物,发现与处理前样品相比,PMA处理降低了底泥微生物的DNA产量,且底泥中活性微生物数量与传统平板培养法得到的结果存在很大差异;底泥质地的复杂性可能对微生物DNA或PMA分子产生吸附作用,从而影响DNA与PMA的结合,导致结果出现假阳性;而发酵底泥暗黑色的表面也可能会阻碍可见光的透射性,进而使DNA与PMA无法发生有效的交联反应。因此,应用PMA染料法筛选区分环境底泥中活性微生物群具有可行性,但现有实验方法还需进一步优化改进。
3 PMA在土壤研究中的应用
微生物在陆地生物地球化学循环和维持土壤肥力方面扮演着重要角色[35]。土壤微生物总DNA中除活性微生物DNA外,还包括大量relic DNA[36],它们能够与土壤矿物质和腐殖质结合并可长期存留于土壤中[8,37]。Carini等[38]首次利用PMA对采自美国地区大尺度下不同生态类型的31份土壤样品进行活性微生物的研究,他们对去除relic DNA的土壤样品提取微生物DNA,以细菌16S rRNA和真菌ITS基因为靶标,进行荧光定量PCR测定和高通量测序,结果发现,与未经PMA处理的土壤相比,去除relic DNA后的土壤微生物丰度平均降低达40%,微生物群落多样性降低最高达到55%,表明土壤中relic DNA的存在明显干扰了样品处理对微生物群落结构影响的解读,以及空间尺度上微生物群落分布与环境条件之间关系的建立。需要说明的是,尽管该研究发现,relic DNA对微生物物种相对丰度影响的偏差最高能达到25%,但该研究也显示绝大多数土壤优势微生物物种在去除relic DNA后仍然可被检测到,并且不同的土壤类型对微生物群落的影响要明显大于relic DNA的影响,表明relic DNA的存在对土壤微生物的群落组成并未产生实质性的改变。Heise等[39]将PMA应用于土壤活性产甲烷古菌的研究中,发现PMA筛选活性产甲烷古菌的有效性与土壤质地紧密相关,黏土和粉土中的relic DNA不能被PMA有效地去除,而PMA可以成功地抑制砂土中relic DNA扩增。Gustave等[40]针对装配有微生物燃料电池的稻田土壤,经PMA处理后发现,尽管微生物燃料电池运行过程中增加了土壤中relic DNA的含量,且影响了一些物种(如Geobacter和CandidatusMethanoperedens)的相对丰度,但relic DNA对活性微生物的群落组成和结构并没有产生显著影响,这可能是由于土壤中微生物活性细胞和非活性残体的周转速率始终保持着动态平衡。尽管PMA在筛选土壤活性微生物群及评估relic DNA残留对土壤微生物群落结构的影响程度方面已有一定报道,但PMA对不同类型土壤和微生物类群的适用性及其最佳实验条件仍需大量数据的积累。例如,某些微生物细胞死亡后可能仍保持有完整的细胞膜,而浓度尚不充足的PMA或许无法有效地穿透这些完整的细胞[41]。因此,PMA在复杂土壤样品的应用中仍存在许多问题亟待解决。
4 PMA在水体研究中的应用
相比底泥和土壤样品,水体中的relic DNA可能会更快速地被脱氧核糖核酸酶降解,因而其含量相对较少,如Dell′Anno和Danovaro[42]报道过水体中relic DNA的降解比其相应的底泥中的所需时间少7~100倍之多。目前,针对水体样品,PMA染料法大多应用于选择性检测饮用水等水体中活性病原菌的研究。仝铁铮等[43]以大肠杆菌(Escherichiacoli)作为模式菌,应用PMA-qPCR技术研究了氯和一氯胺消毒对饮用水中病原菌的灭活效果,结果表明,PMA能够钝化99.94%和99.99%的分别来自非活性大肠杆菌和沙门氏菌(Salmonella)的DNA,说明PMA-qPCR技术可有效区分活性菌与非活性菌,并有助于更准确地评价氯和一氯胺消毒对饮用水病原菌的灭菌效果。Singh等[44]采用PMA-qPCR技术对印度北部昌迪加尔市的饮用水及其源头水中活性沙门氏菌进行检测,发现该地区饮用水及其源头水中活性沙门氏菌含量分别为2~7 160 CFU·100 mL-1和2.1×104~2.6×106CFU·100 mL-1,而净化处理后的饮用水中未检出活性沙门氏菌,表明该技术能够有效地用于城市饮用水污染的监管。Yuan等[45]采集了美国密苏里地区城市管道饮用水和多处河流水样品,并对这些水样接种动物粪便大肠杆菌,研究发现添加5 μmol的PMA并曝光处理10 min,即可抑制1×105CFU·mL-1大肠杆菌死细胞DNA的扩增。该研究还显示,PMA的添加浓度和曝光时间根据采样季节的不同而有所差异,表明需确定合适的PMA前处理方法才能较为准确地量化水样中的活性病原菌,从而有效地评估水样质量。
5 PMA在其他环境样品研究中的应用
除底泥、土壤和水体等常见环境样品外,PMA染料法在筛选区分其他环境样品活性微生物的应用仅有少量报道。Vaishampayan等[46]采用PMA结合高通量测序技术对国际空间站宇航员居住舱内的活性微生物进行检测,研究发现,太空舱内总微生物和活性微生物的群落组成差异很大,这可能是由于舱内属极度洁净区域,仅有的微生物大多属于稀有物种,而PMA会完全结合某些稀有物种的全部DNA(它们可能只以relic DNA形式存在),如加入PMA后,无法再检测到一些具有抗性生理特性的细菌。Seidel等[47]等利用PMA对人和多种动物粪便中活性拟杆菌(Bacteroides)进行检测,研究发现添加0.5%的二甲基亚砜(DMSO)能够显著增强PMA对拟杆菌死细胞PCR扩增信号的抑制效果;同时,增加目的片段的扩增长度,可提高PMA去除拟杆菌死细胞DNA的效率。由此可见,除PMA添加浓度、暗培养和曝光时间等应用条件需进一步优化外,还应对后续的定量PCR和高通量测序等结合技术进行相应调整和改进,以期进一步提高PMA筛选区分环境样品活性微生物的有效性和准确性。
6 存在的问题及展望
PMA染料法用于环境活性微生物的筛选研究尚处于起步阶段,由于环境样品的复杂性,该方法的广泛应用仍受到很多限制,还需大量研究加以改进和完善:
(1)针对不同类型的环境样品,需首先确定最佳的PMA添加浓度、暗培养和曝光时间。在暗培养和曝光过程中,需轻微震荡离心管以避免局部染料过多破坏细胞的活性,同时使PMA与relic DNA充分结合并产生共价交联,以及多余的PMA与溶液中水分子发生结合形成羟胺,避免影响后续活菌裂解及其DNA的扩增反应。也可通过多循环PMA处理加以改进,即样品添加PMA后曝光之后再次添加PMA并曝光。
(2)可见光类型和光照强度也是影响PMA处理效果的因素之一。目前绝大多数基于PMA染料法的研究,在其曝光步骤中,都是采用500~650 W卤素灯对样品进行照射10~20 min,同时将样品放置距离卤素灯15~20 cm左右的冰面上,以避免温度过高对活性微生物造成损伤。然而,卤素灯可变的光强度和未知的光谱特性,可能会造成实验结果难以解释或产生偏差,加之卤素灯的温度非常高,当样品受热不均时可能会造成完整细胞膜的破损或是塑料管子的熔化,存在一定实验风险。目前可应用专业的PMA LED光解仪来代替卤素灯,其自动的冷却循环系统和适当的PMA光解波长有助于提高实验的准确性。
(3)环境样品的浊度、pH和盐分含量等也会影响PMA筛选活性微生物的有效性。如土壤样品,可通过稀释土样使土壤颗粒充分分散于缓冲液中,从而提高PMA与relic DNA的结合效率。与此同时,稀释后的土壤悬液可能存在微生物浓度较低、定量不准确等问题,或可采用数字PCR对样品中微生物的绝对丰度进行准确定量。
(4)适当增加目的基因的片段扩增长度和PCR循环次数也可提高PMA去除环境样品中relic DNA的准确性。扩增较长的目的基因片段有助于减少实验中relic DNA的假阳性结果。此外,也可采用两步扩增的巢式PCR法,以达到准确检测活性微生物数量的目的。
(5)添加适当的PMA促进剂也可提高其处理效果,如脱氧胆酸盐可促进PMA对革兰氏阴性死细菌的结合效率,同时不会对活性细胞产生影响[48]。二甲基亚砜(DMSO)和乙二胺四乙酸(EDTA)可增强PMA对细胞膜的穿透性[49]。尽管如此,这些促进剂的使用需在保证不损伤活性细胞的前提下进行。
(6)尽管PMA染料法的广泛应用尚存在一些问题,但它具有快速、准确、灵敏度高和低成本等优点,不仅能增强人们对复杂环境样品中活性微生物类群的认识,还可与高通量测序等多组学技术相结合,在环境微生物的种质资源挖掘与功能开发利用等方面具有较大的应用前景。

