盐胁迫下秋茄根系转录组的De novo 测序及分析
邢建宏,陈 伟
(1.三明学院资源与化工学院/ 福建省资源环境监测与可持续经营利用重点实验室,福建 三明 365004;2.福建农林大学生命科学学院,福建 福州 350012)
近年来土壤盐碱化问题日益突出,对农业可持续发展造成重大威胁[1].因此,深入研究植物的耐盐机理,挖掘耐盐基因资源对改变日趋严重的土壤盐碱化问题有重要意义.红树植物生长在潮间海岸带,受海水的周期性浸渍,形成了一套有别于淡水植物或陆生植物的耐盐机制[2],值得深入研究,并挖掘其中有价值的基因资源.Ilumina 高通量测序技术的出现,为从基因组和转录组层面深入解析植物耐盐分子机理提供了有力手段.近年来研究者利用高通量测序技术对红树(Rhizophora apiculata)[3]和木榄(Bruguiera gymnorrhiza)[4]进行转录组分析,结果表明抗逆基因的表达可能是提高其抗极端环境能力的关键因素.对泌盐红树植物海榄雌(Avicennia officinalis)盐胁迫下根系的RNA-seq 分析表明,ABA 和乙烯介导的植物激素信号通路在其响应盐胁迫中发挥重要作用[5].这些研究为从转录组层面揭示红树植物耐盐机理提供了前期探索,但对非泌盐植物根系的耐盐分子调控机制还未见系统和深入的研究.秋茄(Kandelia candel)是红树植物中典型的非泌盐植物[6],其耐盐能力通过自身基因表达调控实现,蕴藏着丰富的耐盐基因资源,因此从分子水平解析秋茄的耐盐机理可为揭示木本植物耐盐机制提供新的证据.本研究利用Illumina HiSeqTM2000 平台对不同浓度NaCl 处理下秋茄幼苗根系转录组进行测序,并建立转录组数据库,为今后从分子层面深入解析其耐盐机制提供基础数据.
1 材料与方法
1.1 材料
供试秋茄胚轴采自福建省漳州市漳江口红树林国家自然保护区(23°55′N,117°26′E).挑选大小与成熟度相近、无机械损伤和病虫害的胚轴种植于45 cm×35 cm×25 cm 塑料盆的干净河沙中,每盆20 株,每盆浇灌1L Hoagland 营养液,每天傍晚用自来水补充散失的水分.
1.2 方法
1.2.1 NaCl 处理 待幼苗长至4 叶后(60 d),用含不同盐浓度(0、200、400、600 mmol·L-1NaCl)的Hoagland 营养液浇灌幼苗根部,为避免原有水分对处理浓度的影响,处理前2 d 先放干培养容器中的溶液,停止水分补充.为避免高浓度盐处理引起盐激效应,400、600 mmol·L-1NaCl 处理均从200 mmol·L-1开始增加,每隔1 d 施加1 次盐处理,并在同一时间达到相应处理浓度,3 d 后取样.每个处理选取20 株幼苗,剪取所有植株的根,迅速放入液氮中速冻20 min 后,置于-80 ℃冰箱保存备用.每个处理2 次生物学重复.
1.2.2 总RNA 的提取及文库构建 采用十六烷基三甲基溴化铵(CTAB)-LiCl 沉淀法提取秋茄根系中总RNA[7].将4 种不同NaCl 浓度处理的秋茄幼苗根系单独提取RNA,每个处理2 个生物学重复,准确测定所提取RNA 浓度,将所有样品RNA 等量混合,构建含有不同处理浓度的RNA 样品池进行测序.将检测合格(浓度、纯度、完整性和片段大小符合建库要求)的RNA 按照Illumina 使用说明书构建cDNA 文库.首先将RNA 通过片段缓冲液(Ambion,Austin,TX,USA)进行孵育并破碎;接着应用随机六聚体引物hexamer-primers (Illumina,San Diego,CA,USA) 和逆转录酶(Invitrogen,Carlsbad,CA,USA) 合成第一链cDNA;然后应用DNA 聚合酶I(Invitrogen,Grand Island,NY,USA)合成第二链cDNA;将双链cDNA 片段应用QIAquick PCR extraction kit 进行纯化处理,并连接Illumina 转录适配器(Illumina,San Diego,CA,USA),随后选择合适的片段进行PCR 扩增.
1.2.3 Illumina 测序、组装及注释分析 转录组文库质量检测合格后,应用Illumina HiSeqTM2000(Illumina,San Diego,CA,USA)仪器按照Illumina 公司的标准操作步骤进行转录组测序.对原始reads(双端序列)进行评估,剔除低质量的reads(more than 20% of the bases showed a Q-value≤10)和含有超过5%未知序列“[N]”的reads,获得clean reads.随后采用Trinity(v2012-10-05)软件对去杂后的clean reads进行转录本De novo 组装,获得Unigene 库(Unigene Library),利用Nonredundant protein (Nr)、Kyoto Encyclopedia of Genes and Genomes (KEGG)、Clusters of Orthologous Groups(COG)、Gene Ontology (GO)、Swiss-Prot 等数据库对每条Unigene 进行BLAST 比对,获得每个数据库的比对结果及功能注释.
1.2.4 耐盐相关基因的qRT-PCR 验证 为证明本研究所得数据库包含了秋茄耐盐相关基因[8],选取前人发现的6 个红树植物盐胁迫相关基因,以本研究获得的序列信息设计引物,对不同NaCl 浓度处理下的基因表达量进行qRT-PCR 验证.所验证基因信息及引物见表1,方法见参考文献[8].
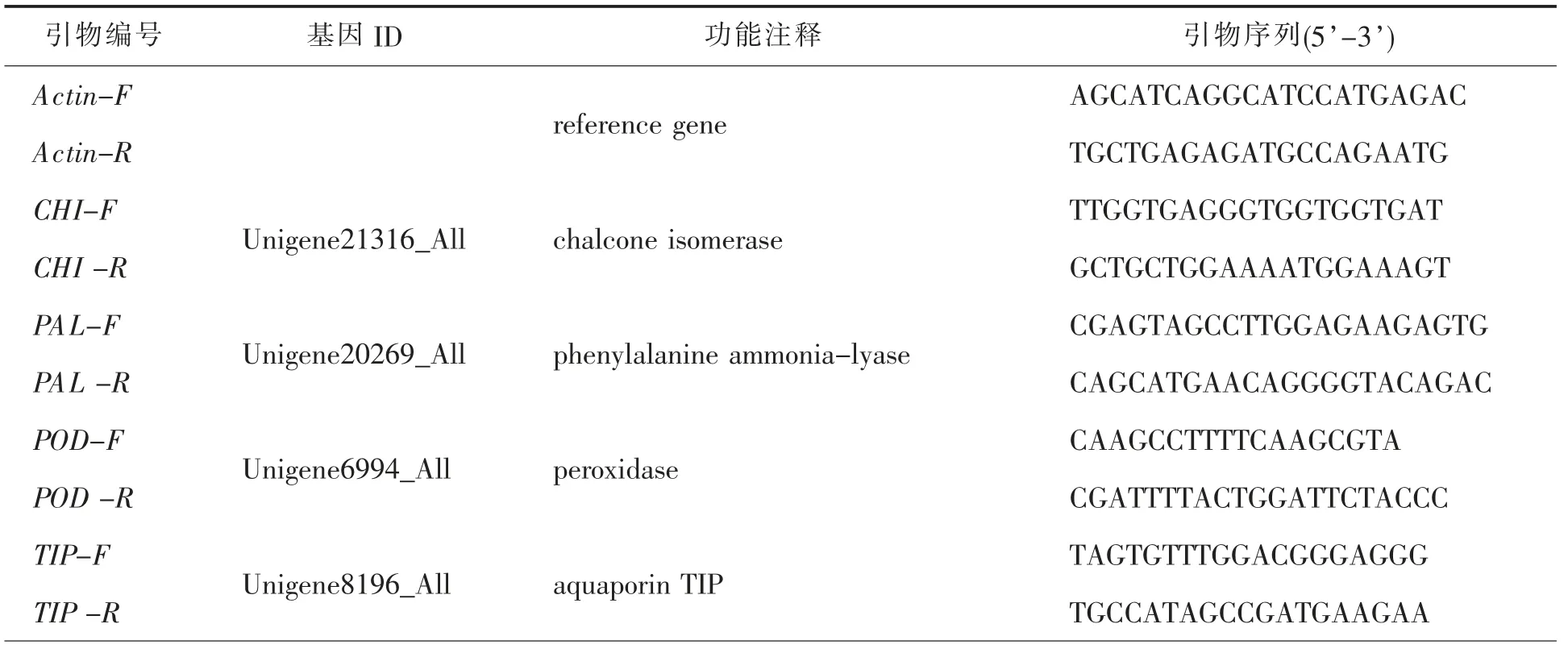
表1 不同浓度NaCl 处理下差异表达基因qRT-PCR 验证引物

续表 1
2 结果与分析
2.1 总RNA 提取与质量检测
分别对4 种NaCl 浓度处理下的8 个样品的总RNA 进行琼脂糖凝胶电泳检测,结果(图1)显示,电泳条带明亮、清晰,无拖尾现象,28S 的亮度在18S 的两倍以上,表明所提取的RNA 质量较高,符合建库要求.

图1 琼脂糖电泳初步检测总RNA 质量
2.2 测序结果评估与序列组装
由表2 可知,对不同NaCl 浓度处理下秋茄根系样品进行转录组测序总共得到了65.79 M 的clean reads,每个样品的clean reads 均超过7.50 M;测序总碱基数为6.64 Gb,每个样品的总碱基数均高于760 Mb;各样品碱基Q30 均大于86%;各样品的GC 含量大约45%,与一般物种中的GC 含量相近.上述结果表明,秋茄根系的转录组测序质量较好.应用Trinity 组装平台,共获得112 243 条Transcripts 和61 970条Unigenes,而且Transcripts 与Unigenes 的N50 分别为2 361 和1 510 bp,这表明秋茄根系转录组片段组装完整性较高.

表2 组装结果统计

续表2
采用Bowtie 软件将测序得到的reads 与Unigene 库进行比对,总比对效率大于85%,并且多个位置比对read 所占百分比在41%左右,证明测序数据的总体比对效果较好,可以用于各转录本表达量的确定.以比对到参考基因序列上的reads 在基因上的分布情况进行测序随机性评估表明,本研究中样品reads 在参考基因上对应的分布曲线比较平缓(图2),表明样品的测序随机性良好.依据测序碱基数量和发现基因数进行测序饱和度评价,结果显示(图3),本研究中总RNA 在当前的测序量条件下已达到饱和,表明测序结果可以用于全转录组分析.

图2 cDNA 片段的随机性检验图

图3 测序饱和度分析图
2.3 Unigenes 基因功能注释
经详细比对分析,本研究最终成功获得26 804 个秋茄根系转录组Unigenes 序列的注释信息.基因注释的统计结果见表3.注释结果显示组装成功的61 970 条Unigenes 序列有26 668 条可以在Nr 数据库中获得注释,占总数的43.03%,17 761 条Unigenes 序列与Swissprot 数据库中获得注释,占28.66%;22 143 条Unigenes 序列在GO 库中获得注释,占35.73%;8 324 条在COG 数据库中得到注释,占13.43%;6 258 条可以注释到KEGG 库中,占10.10%.获得成功注释的基因为深入研究秋茄对盐胁迫的应答提供了参考依据,尚有35 166 条Unigenes 序列无法从现有数据库中获得注释,获得注释的基因为将来进一步挖掘秋茄中的新基因提供了有价值的信息.

表3 Unigenes 序列注释结果统计表
2.4 Unigenes 基因的COG 分类
为了进一步分析秋茄根系NaCl 处理下转录组序列的功能,本研究将COG 注释到的8 324 条Unigenes 基因进行了功能分类,共获得11 719 个功能注释信息,可划分为25 个功能类群,结果见图4.

图4 秋茄根系NaCl 处理下Unigenes 基因的COG 功能注释分布图
COG分类结果显示,包含基因数量排在前10 位的类别分别为,一般功能预测基因(ceneral function prediction),达2 194 个,占26.36%;复制、重组与修复基因(replication,recombination and repair)1 022个,占12.28%;与转录(transcription)相关的功能基因1 008 个,占12.11%;与翻译、核糖体结构与生物合成(translation,ribosomal structure and biogenesis)相关的基因948 个,占11.39%;与信号转导机制(signal transduction mechanisms)相关基因887 个,占10.66%;翻译后修饰、蛋白转运和分子伴侣(posttranslational modification,protein turnover,chaperones)基因872 个,占10.48%;碳水化合物运输与代谢(carbohydrate transport and metabolism)基因654 个,占7.86%;氨基酸转运和代谢(amino acid transport and metabolism)相关基因588 个,占7.06%;能量产生和转换(energy production and conversion)相关基因467 个,占5.61%;无机离子转运和代谢(inorganic ion transport and metabolism)相关基因406 个,占4.88%.从COG 分类结果来看,排在前10 位的基因类群大多数和植物应答生物与非生物胁迫相关,这表明本研究所建立的秋茄NaCl 处理下的转录组库包含了秋茄抵御逆境的相关基因.
2.5 Unigenes 基因GO 分类
对GO 注释的2 2143 个Unigenes 基因进行功能显著性富集分析,结果(图5)表明,本研究获得的秋茄根系Unigenes 基因涉及细胞组分(cellular component,CC)、分子功能(molecular function,MF)和生物学过程(biological process,BP)3 个大类中56 个小类.3 个大类中生物学过程获得的注释最多,占51.53%,包含:细胞过程(cellular process)、代谢过程(metabolic process)、刺激响应(response to stimulus)、生物学调节(biological regulation)、发育过程(developmental process)和细胞组分组织与生物合成(cellular component organization or biogenesis)等24 个小类.细胞组分类获得注释占35.59%,包含:细胞部分(cell part)、细 胞(cell)、细 胞 器(organelle)、膜(membrane)、细 胞 器 部 分(organelle part)、大 分 子 复 合 体(macromolecular complex membrane part)等16 个小类.分子功能类获得注释较少,占12.88%,包含:核酸结合转录因子活性(nucleic acid binding transcription factor activity)、结合(binding)、催化活性(catalytic activity)、转运体活性(transporter activity)、结构分子活性(structural molecule activity)、分子传感器活性(molecular transducer activity)和酶调节活性(enzyme regulator activity)等16 个小类.上述结果显示,秋茄盐胁迫下转录组中涉及生物学过程基因较多,表明秋茄应答盐胁迫的分子调控与细胞生物学过程关系密切.

图5 秋茄根系NaCl 处理下Unigenes 基因的GO 分类图
2.6 Unigenes 基因的代谢通路分析
对基因的KEGG pathway 分析可以帮助我们深入了解基因的生物功能及相互作用关系.对不同浓度NaCl 胁迫下秋茄转录组库中Unigenes 基因的KEGG pathway 富集分析显示,获得注释的6 258 条Unigenes 基因参与了116 条代谢途径,按照获得注释的基因数多少,排在前10 位的通路如表4 所示.

表4 Unigenes 基因的KEGG pathway 富集结果统计
Unigenes 基因通路富集结果显示核糖体、内质网蛋白加工、植物信号转导、氧化磷酸化、RNA 转运剪接、核酸代谢以及糖代谢等途径为包含较多基因的通路,而这些通路已被证明与植物逆境应答密切相关.这表明,本研究建立的秋茄转录组库包含了多个与抗逆相关的代谢途径,可以作为后续秋茄耐盐相关基因筛选的参考数据库.
2.7 Unigenes 基因开放阅读框预测
对Unigenes 基因开放阅读框(ORF)进行预测,共获得61 690 条序列,平均长度393.63 bp,其中长度大于2 000 bp 的有1 703 条,N50 为984 bp,与其他物种的预测结果基本一致.由于秋茄缺乏全基因组测序信息,转录组库中尚有大量序列无法从现有数据库中得到注释,而这些序列可能为新的蛋白编码序列.
2.8 耐盐相关基因的qRT-PCR 分析
依据本研究获得的相关基因数据库,对前人在红树植物中发现的盐胁迫相关的6 个基因在不同NaCl 浓度下的表达差异进行了qRT-PCR 分析,结果(表5)表明,上述6 个基因在不同NaCl 浓度处理下差异表达,表明上述基因可能参与了秋茄对高盐环境的响应,也表明本研究构建的数据库序列可以用于秋茄耐盐基因差异表达研究.

表5 本研究检测到的部分与植物耐盐相关的基因qRT-PCR 分析结果
3 结论与讨论
测序深度是保证转录组库质量的关键因素.当测序量达到一定标准时,检测到的表达基因数量就会逐渐趋近饱和.本研究共获得33 246 108 条的reads,总碱基数达到了6.71 G,达到饱和测序量的200倍,表明测序深度很高,利用此数据构建的转录组库结果可靠,能保证低丰度基因的检测.
选择合适的组装软件是获得高质量转录组序列的关键.常见的几种组装软件中Trinity 是进行无参考基因组非模式生物转录组从头组装的最佳软件[9].本研究利用Trinity 组装共得到112 243 条Transcripts 和61 970 条Unigenes,Transcripts 与Unigenes 的N50 分别为2 361 bp 和1 510 bp,有13 642 条Unigenes序列的长度大于1 000 bp,平均长度为790.75 bp,这高于前人获得的同为红树植物且无参考基因组的海桑(Sonneratia caseolaris)(589 bp)[10],表明本研究获得秋茄转录组序列总数、平均长度、以及片段组装完整性都比较好.
对测序结果的准确注释是开展基因功能及代谢通路分析的前提.本研究借助Nr、Swissprot、GO、COG和KEGG 数据库对秋茄根系Unigenes 基因进行了注释,共获得了43.25%的注释信息,尚有56.75%的序列没有获得注释信息,这主要原因可能是因为组装出的短序列难以达到具有统计意义的匹配阀值,无法进行同源性比对,也可能是因为未比对上的序列可能为秋茄特有基因或非编码序列,这需要后续工作进一步挖掘.
GO 富集分析、COG 功能分类和KEGG pathway 三者相互结合成为从新物种中挖掘功能基因的重要手段[11].本研究通过对Unigenes 基因的GO 富集、COG 功能分类和KEGG 通路分析发现,秋茄在盐胁迫下启动了大量与逆境胁迫相关的基因表达,这些基因参与了多种代谢调控途径,这预示着秋茄响应盐胁迫的过程相当复杂,涉及到多条代谢通路交叉调控,需要多个基因组成调控网络来调控.
综上所述,通过对不同NaCl 浓度处理下秋茄根系转录组库测序,共得到112 243 条Transcripts 和61 970 条Unigenes,Transcripts 与Unigenes 的N50 分别为2 361 bp 和1 510 bp,表明本研究建立的转录组库片段的组装完整性.利用Nr、SwissProt、GO、COG 和KEGG 数据库对Unigenes 基因进行注释,有26 804 个Unigenes 基因获得注释信息.对Unigenes 基因的生物信息分析表明,这些抗逆相关基因是秋茄能够适应滨海潮间带高盐环境的分子基础,对这些基因功能及所参与的代谢路径的深入研究将为木本植物耐盐分子机理的解析及未来作物抗盐基因工程育种奠定理论基础.
[责任编辑 杨玉玲]

