芳樟叶片转录组测序及萜类合成调控相关基因表达分析
曹瑞兰 胡冬南 周增亮 刘 爽 陈尚钘 刘 娟
(1. 江西农业大学林学院江西省森林培育重点实验室,江西 南昌 330045;2. 郑州工业应用技术学院,河南 郑州 451150)
芳樟(Cinnamomum camphora),又名香樟,是樟科(Lauraceae)樟属的一种常绿阔叶树种,主要分布在热带和亚热带地区[1]。樟树在建筑、香料、园林造景、生态环境和生态文化建设等方面具有广泛的用途。根据樟树精油中主要化学成分不同,将樟树分为5种类型,包括芳樟(芳樟醇含量58%~92%)、龙脑樟(右旋龙脑含量67%~82%)、脑樟(樟脑含量54%~97%)、油樟(桉叶油素含量32%~52%)和异樟(异-橙花叔醇含量16%~57%)等[2]。其中,从芳樟枝和叶中提取的天然芳樟醇是生产使用最多且需求量最大的一类广谱性香料,对其特性及其生物合成途径的研究已成为研究热点。芳樟醇是芳香类植物香气的主要挥发性物质,是一种重要的单萜类化合物[3]。芳樟醇合成酶(LIS)是催化单一底物基质牻牛儿基焦磷酸(GPP)转化成单一产物芳樟醇的关键酶。目前,已有研究在不同的芳香植物中开展芳樟醇合成酶基因的表达和克隆等相关研究,但是对芳樟中含量较高芳樟醇合成酶基因差异表达的报道有限。
转录组学是从RNA水平研究物种的基因表达,广泛应用于对特定生物学过程的分子机理研究[4]。因其具有成本低、通量高、时间短、可重复性高等优点,许多植物已经完成了转录组测序分析。樟树转录组研究也取得了重要进展。如江香梅等对芳樟醇型、龙脑樟型、樟脑型、油樟型和异樟型等5种樟树化学类型进行叶转录组分析,发现9条Unigene可能参与编码了芳樟醇合成酶基因[5]。Chen等对芳樟和龙脑樟叶片开展转录组测序分析,解析了芳樟醇型和龙脑樟型萜类合成途径中表达差异的基因,并通过比较分析鉴定了龙脑樟中有关单萜合成酶的3个关键基因[6]。已有的文章主要关注樟树精油中多种萜类化合物合成相关的差异基因,但是他们对芳樟醇含量较高的芳樟中芳樟醇合成酶关键基因的挖掘十分有限,需要针对芳樟展开更深入研究。
本研究基于已选育的芳樟优良无性系,并以化学成分较为复杂且芳樟醇含量较低的杂樟为对照组,利用Illumina Hi-seqTM2000高通量测序平台进行2种化学类型樟树叶片转录组测序。基于获得的基因序列,进行GO、KOG、KEGG以及Pathway功能注释,进行差异基因的表达分析,同时重点关注与芳樟醇合成酶合成调控相关的差异表达基因,并开发SSR分子标记。本研究将对推动芳樟分子育种及其樟科植物分子生物学研究和发展具有极大一定的促进作用。
1 材料与方法
1.1 研究材料
本研究材料来源于江西省抚州市金溪县陆坊乡石岗村吉源芳樟基地(116°47 E,28°01′ N)的芳樟人工原料林种质资源圃内,选择长势良好且无病虫害的优良芳樟无性系和杂樟实生苗。2017年6月,在优良芳樟无性系3个个体上,分别采集其新叶;在杂樟实生苗的3个个体上采集新叶作为对照。样品采集后,立即放入液氮中粉碎样品,-80 ℃低温保存。芳樟样品不同个体分别装样,实生苗杂樟的叶片则将不同个体混合后匀成3份混样,进行转录组测序。
1.2 方法
1.2.1 RNA提取与文库构建
采用RNeasy Plant Mini Kit获得芳樟和杂樟叶(对照)共6个样品的总RNA,琼脂糖凝胶电泳检测其完整性,Nanodrop检验总其质量和纯度。合格后,磁珠富集和片段化后,用随机引物(random hexamers)合 成cDNA,AMPure XP beads纯化双链cDNA。双链cDNA经末端修复、加测序接头、加poly(A)等过程,AMPure XP beads筛选片段长度。进行PCR扩增后,经AMPure XP beads纯化,最后得到小片段测序文库,使用Illumina Hi-seqTM 2000进行高通量转录组测序及分析。
1.2.2 转录组测序与组装
经CASAVA碱基识别(base calling)分析,使原始测序序列(sequenced reads)被转化为高通量转录测序序列。对所得原始序列(raw reads)进行过滤,去除重复的(N>5%)、测序质量低的(Q≤20碱基数占50%以上)和带接头的reads,得到过滤后序列(clean reads)。采用短reads组装软件Trinity[7]将这些短reads连在一起,获得无法在两端延伸的序列,称为Unigene。
1.2.3 Unigene功能注释
采用BLAST将Unigene与NT(NCBI non-redundant nucleotide sequences)、NR(NCBI non-redundant protein sequences)、Pfam(Protein family)、Swiss-Prot(an annotated protein sequence database)、GO(gene ontology)、COG/KOG(Clusters of orthologous groups/EuKaryotic orthologous groups)、KEGG(Kyoto encyclopedia of genes and genomes)七大数据库进行对比,获得Unigene注释信息。最后以CcEF1α基因作为内参基因,选取潜在的差异基因进行q-PCR,验证其在不同化学类型中的表达差异。
1.2.5SSRs(simple sequence repeats)检索与分析
采用MISA对样品转录本数据进行SSR搜索,参数设置分别为:1-10,2-6,3-5,4-5,5-5,6-5(如:1-10表示如果使用单个核苷酸作为重复单元,其重复数≥10才可被检测到),并且对不同SSR类型在转录本的密度分布进行统计。
1.2.4基因表达水平及表达差异量分析
采用RSEM软件得到比对到转录本上的clean reads表达量[8],对其进行FPKM(Expected number of fragments per kilobase of transcript sequence per millions base pairs sequenced)转换,并分析基因的表达水平。将所得的转录本进行基因差异表达(DEGs)分析。选用的差异基因筛选标准为校准后的P<0.05且|log2FoldChange|>1,并对差异表达基因进行GO注释和KEGG pathway富集分析。
2 结果与分析
2.1 芳樟和杂樟叶精油成分测定
从江西省金溪县芳樟人工原料林的种质资源圃内采集樟树叶片样品,芳樟叶(L-leaf)和杂樟叶(B-leaf)采用GC-MS分析了鲜叶挥发油的化学成分(表1)。在2种化学型的提取物中,芳樟叶的芳樟醇含量达到88.68%,远高于杂樟叶的芳樟醇含量。
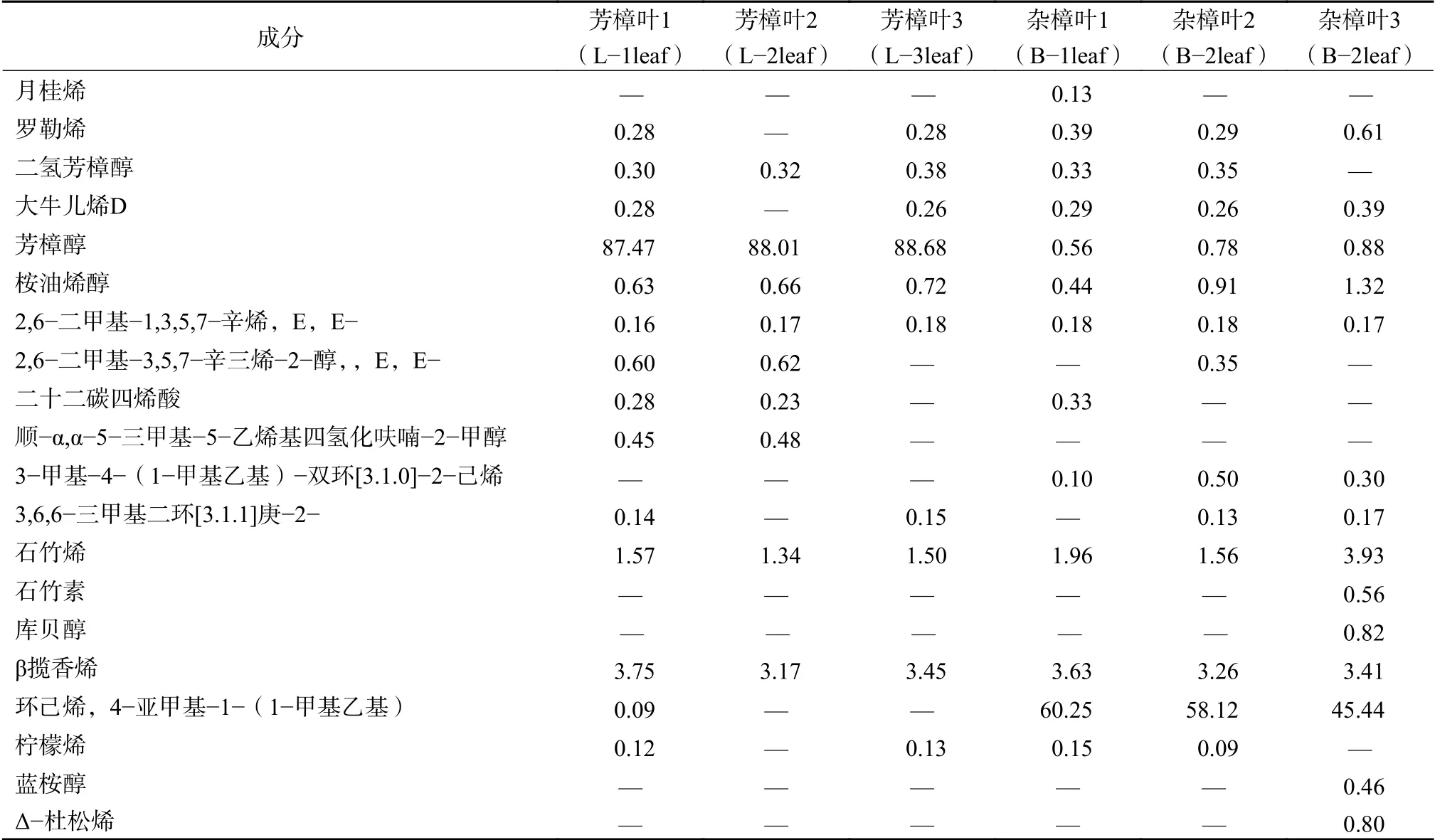
表1 樟树叶提取物的组成Table 1 Composition of leaf extracts of C. camphora %
2.2 芳香樟和杂樟转录组测序与组装
芳樟叶(L-1 leaf,L-2 leaf,L-3 leaf)和杂樟叶(B-1 leaf,B-2 leaf,B-3 leaf)转录组测序后所得raw reads,去除杂质获得clean reads(表2)。结果表明,质量参数Q20最低为96.17%(芳樟叶),最高达96.91%(杂樟叶);过滤后不确定的碱基比例N值小于0.01%;其中GC含量为45.72%~46.43%。共生成34.45 GB clean reads数据。
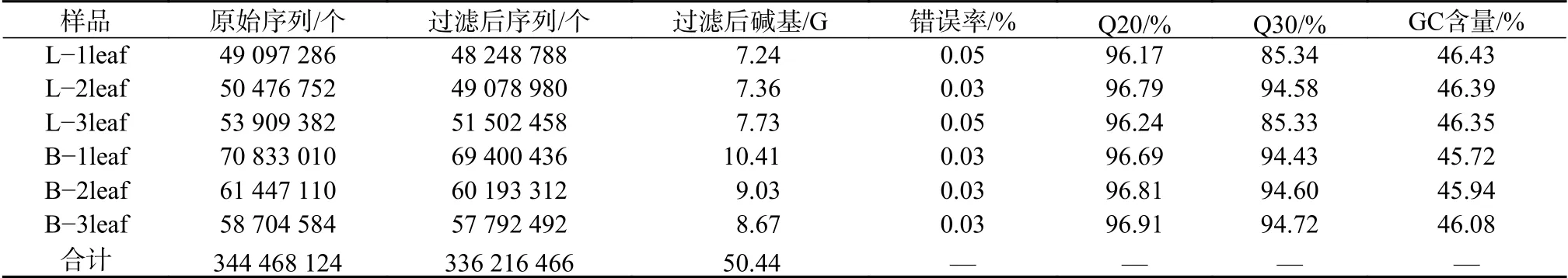
表2 芳樟和杂樟叶测序产量统计Table 2 The statistics sequencing yield of 2 different types of C. camphora
2.3 Unigene注释与功能分类
利用BLAST获得的Unigene,分别在Nr、Nt、GO、KOG、Swiss-Prot、Pfam和KEGG数据库中进行比对,对所注释的Unigene进行统计,并通过BLASTN将其比对到Nt(E值<0.000 01),获得其功能注释信息和具有最高相似性的蛋白(表3)。结果显示,312 457条Unigene在NR数据库中存在98 579条同源匹配到信息,Nt 67 148条,Ko 40 723条,SwissProt 80 310条,Pfam 85 151条,GO 87 053条,KOG 33 177条。

表3 Unigenes注释到不同数据库统计Table 3 The statistics of Unigenes annotation to different databases
2.4 Unigene的GO功能注释和分类
基于Nr的注释信息,在GO数据库对芳樟和杂樟的叶转录组Unigene进行生物学特征功能分类。结果显示,GO数据库注释Unigene可分为生物过程(biological process)、分子功能(molecular function)和 细 胞 组 分(cellular component)3个GO类别,56个小组。在生物过程中,共含有207 529个GO条目,25个功能组,其中“代谢过程”45 038个,“细胞进程”47 138个,“单一生物体”过程35 746 个。在细胞组分中,共涉及132 394个GO条目,21个功能组,其中“细胞”26 278个,“细胞要素”26 259个,“细胞器”17 788个。在分子功能中,共涉及95 197个GO条目,分为10个功能组,其中“催化活性”有36 459个,“结合活性”41 049个,“细胞聚集”2个,“细胞外基质组分”7个。
2.5 差异表达基因的表达分析
为了分析芳樟(L-leaf)和杂樟(B-leaf)2种不同化学类型叶片的差异基因表达,参考edgeR进行差异基因分析,筛选阈值选择为false discovery rate(FDR)≤0.05,|log2FoldChange|≥1,共鉴定出11 685个单基因,其中芳樟相对于杂樟有6 481个上调基因和5 204个下调基因,芳樟特有差异基因794个,杂樟特有842个差异基因,10 049个基因在2种化学类型中均有表达(图1)。

图1 样品间差异表达基因的数量Fig. 1 Number of differentially expressed genes between 2 samples
2.5.1 差异基因GO功能注释
将芳樟和杂樟叶所获得的转录本用DEGseq软件进行比较分析,获得差异表达基因11 685个,将差异基因进行GO分类统计(表4),参与生物学过程差异基因分为20类,其中磷代谢过程(727个)、含磷化合物代谢过程(725个)和大分子修饰(640个)中所占比例最高。参与分子功能中包含了20个功能小类,差异基因主要分布在催化活性(2 810个)、转移酶的活性(1 180个)和核苷酸绑定(821个)中。在细胞组分这一类中,外部封装结构(98个)和细胞壁(79个)中差异基因较高。

表4 差异基因在GO中的功能注释Table 4 Functional annotations of different genes in GO
2.5.2 差异基因KEGG富集分析
将获得的11 685个DEG映射到KEGG代谢途径数据库,共有2 200条DEG获得功能注释,占DEG总数的18.83%,其中归入KEGG的代谢途径有116条。将富集的上调和下调差异基因最多的前20条KEGG代谢途径列出(表5),主要包含植物-病原物互相作、糖代谢、植物激素信号转导、内质网蛋白加工、苯丙烷生物合成和吞噬体等,其中,上调差异基因中涉及倍半萜和三萜生物合成的Unigene有12个,下调差异基因中发现单萜类生物合成的Unigene有5个。
2.5.3 与萜类生物合成调控相关差异表达基因
为了探索不同萜类化合物积累模式的调控机制,对萜类生物合成相关基因的表达进行深入分析。共鉴定39个编码萜类合成酶的Unigenes,其中单萜生物合成有6个,二萜生物合成有7个,倍半萜和三萜生物合成有12个,萜类骨架合成14个。表5是萜类生物合成调控的差异基因,在杂樟叶中有5个单萜合成酶(TPS14)基因、3个二萜合成酶基因、2个萜类骨架合成酶基因显示上调,而在芳樟叶中有1个单帖生物合成基因、4个二萜合成酶基因12个Unigene参与倍半萜和三萜类合成酶基因和12个萜类骨架合成酶在表达上调(表6)。
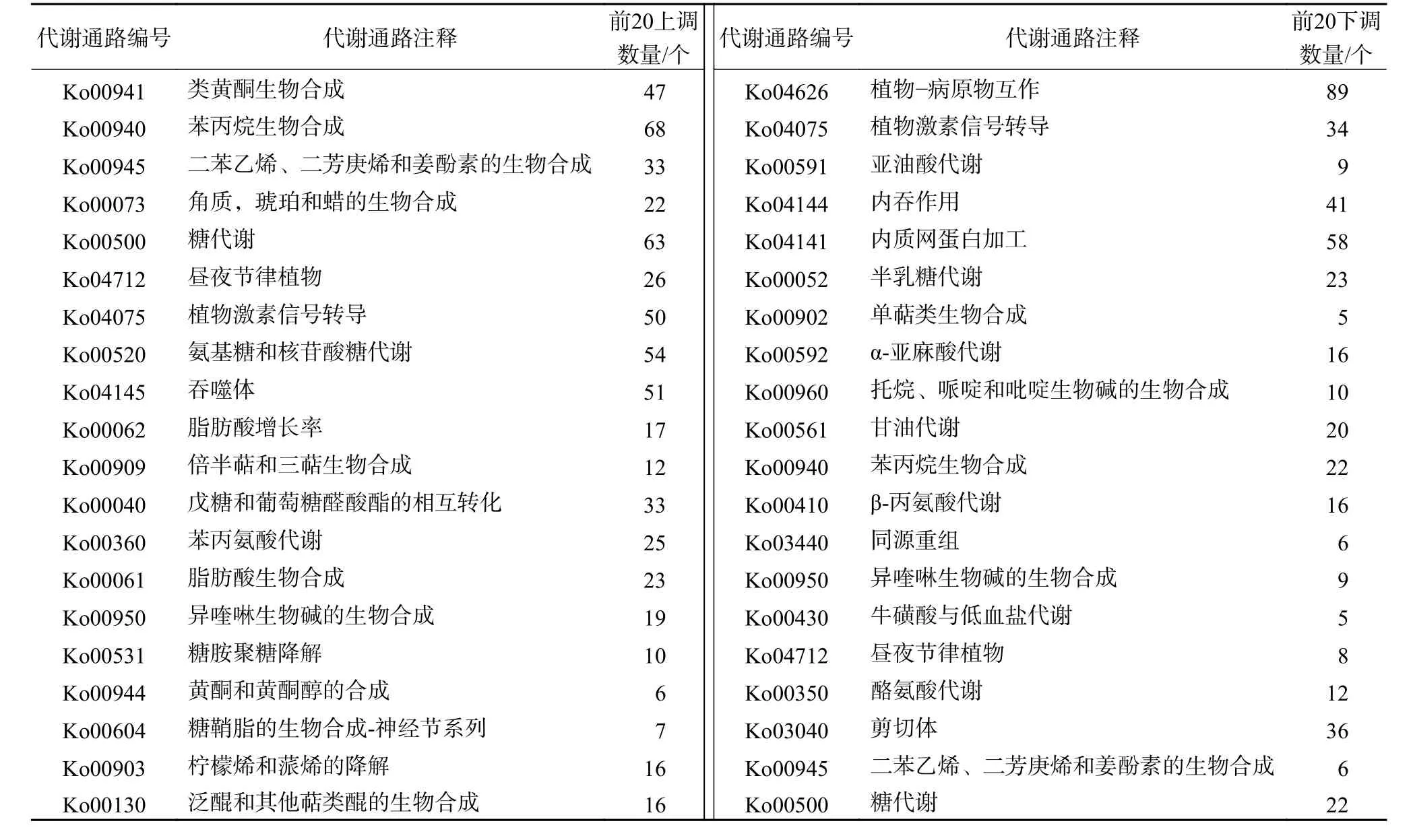
表5 前20个上调和下调的差异基因KEGG代谢通路显著性富集列表Table 5 The top 20 up-regulated and down-regulated differential genes KEGG metabolic pathways with significant sexual enrichment
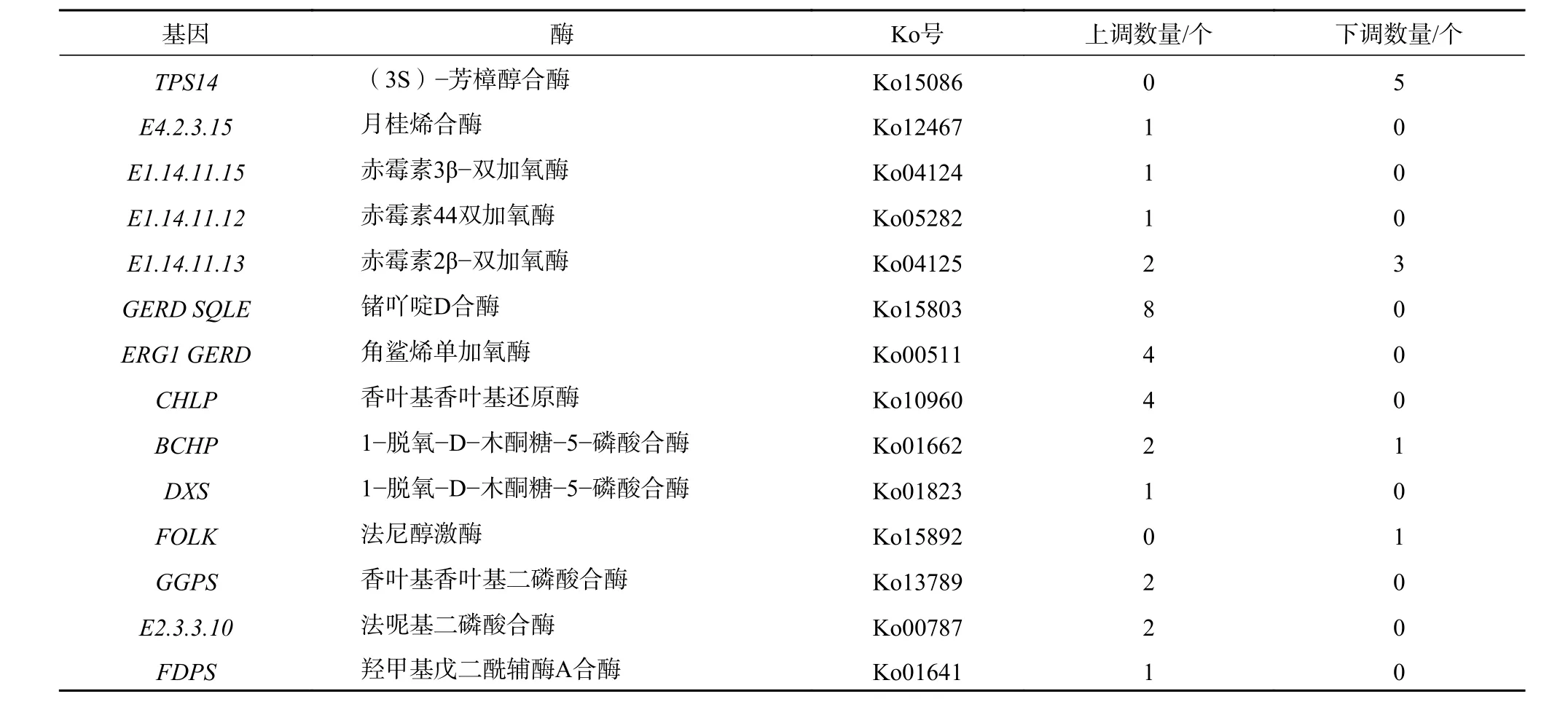
表6 与萜类生物合成调控相关差异表达基因Table 6 The list of differentially expressed genes related to the regulation of terpenoid biosynthesis
2.5.4 芳樟醇合酶(TPS14)基因的表达量分析
为验证RNA-Seq分析获得的萜类生物合成基因的表达模式,本研究采用q-PCR方法检测了芳樟(L-1leaf、L-2leaf和L-3leaf)和杂樟(B-1leaf、B-2leaf和B-3leaf)2种化学类型中3个单帖合成酶(TPS14)基因的表达水平(图2a)。结果显示,3个TPS14基因在2个化学类型中的q-PCR表达模式与RNA-Seq(图2b)的FPKM的结果基本一致,均表现为3个TPS14基因在芳樟中的表达量比杂樟中的较低。

图2 TPS14基因在两种化学型中叶表达模式分析Fig. 2 Analysis of the expression patterns of TPS14 genes in 2 chemical types of leaves
2.6 SSR分析情况
利用MISA对转录组测序获得的312 457条Unigene进行检索,共找到符合条件的SSR位点有134 851个,分布于88 016条Unigene中。其中含单个SSR位点的Unigene有103 478条,含2个及2个以上SSR位点的Unigene有31 373条。复合型SSR数目为20 277个(表7)。

表7 转录组中SSR信息分析Table 7 SSR information analysis in transcriptome
3 结论与讨论
芳樟的枝叶经减压精馏分离而来的高纯度芳樟醇,在香精、香料和医疗保健等方面具有广泛的应用价值[9]。而杂樟,通常是指野生樟树中精油主成分不明显的一类,因在自然演化历史中,经反复杂交和环境因素的影响,其化学成分和含量十分复杂,生产利用途径有限[10]。目前关于樟树的研究主要集中在生长发育[11-12]、栽培管理[13-14]、油脂组成及提取工艺[15-16]等方面,有关其分子生物方面研究有限,尤其是其主要成分合成途径及其分子机理尚不清楚。本研究通过Illumina HiSeqTM 2000高通量基因测序技术对芳樟和杂樟的叶转录组进行比较分析,转录组测序共生成34.45 GB clean数据,组装成312 457个Unigene,鉴定出11 685个差异表达Unigenes。相对杂樟,芳樟叶中有6 481个上调的Unigenes和5 204个下调Unigenes,其中有2 200条差异基因在KEGG代谢途径数据库中获得功能注释,占DEG总数的18.83%,被归入116条KEGG的代谢途径中。
KEGG pathways分析结果表明,注释到次生代谢生物合成途径中的Unigene有1 184条,占2.91%。樟科的各个家族中萜类化合物含量丰富[17-18],目前已在山苍子(Litsea cubeba)[19]、细毛樟(Cinnamomum tenuipilum)[20]、土肉桂(Cinnamomum osmophloeum)[21]、月桂(Laurus nobilis)[22]等类群中成功鉴定参与萜类生物合成的关键基因。针对樟树的不同化学类型,研究者也展开了萜类合成酶关键基因的探索。如江香梅等通过樟树5种化学型的转录组分析,鉴定发现了参与编码芳樟醇的9个关键基因,且这些基因在芳樟中表达水平远高于油樟等其他化学类型[5]。这与本研究的结果相似,本研究发现的39个与萜类生物合成调控相关基因,涉及单帖合成途径的的Unigene有6个,二萜合成通径的Unigene有7个,倍半萜合成通径的Unigene有12个,萜类骨架合成通径的Unigene有14个。其中,5个单萜合成酶(TPS14)基因、3个二萜合成酶(E1.14.11.13)基因、2个萜类骨架合成酶(BCHP和FOLK)基因在芳樟叶中表达水平较低,而在杂樟叶中表达水平较高。
江香梅等对芳樟和龙脑樟的转录组分析中发现它们存在67条与萜类合成相关的差异表达基因,且多个基因被注释为同一种酶[5]。本研究基于芳樟和杂樟叶转录组数据,鉴定了39个萜类合成相关的差异表达基因。造成结果的差异,可能是由于与所选用材料的萜类物质含量不同。同时也发现5个TPS14基因均被注释为芳樟醇合酶。Chen等的结果显示,TPS14在芳樟中表达量较低,且在樟树的5种化学型叶片荧光定量表达分析发现相同的规律,即TPS14在芳樟醇含量较高的芳樟中反而表达最低[6]。本研究的TPS14基因转录组FPKM数据和q-PCR表达量也证实了该结论,潜在的3个TPS14基因在杂樟的表达量要高于芳樟。然而,在拟南芥中,TPS14被认为是花部器官中调控芳樟醇合成的关键酶[23]。已有报道发现,TPS少数氨基酸的变化就会导致其特定酶合成萜类谱发生剧烈变化[24]。Liu等通过研究油茶的萜类合成代谢途径,发现选择性剪切可改变关键酶的转录,进而影响芳樟醇合成[25]。TPS14基因在芳樟中的转录过程及其功能需要进一步探索和证实。
目前,在樟树精油产业中,种质资源混乱和缺少优良无性系是制约其发展的主要因素之一。由于不同化学类型的精油含量及其品质很难依靠樟树外部形态进行精准划分,而精油提取和化学成分分析的方式存在时效慢、受季节和生长发育阶段的影响等缺点,因此急需新的鉴定手段。SSR分子标记法,普遍认为具有共显性、重复性好、多态性丰富、操作简便等优点[26],目前已成为开展植物品种鉴定、遗传多样性分析、遗传图谱构建和分子辅助育种[27]的主要方式之一。张震等以油茶转录组测序数据为基础,利用MISA软件对SSR进行搜索,发现104 515个SSR,分布在80 724条Unigene中[28]。本研究基于芳樟和杂樟叶的转录组数据SSR位点分析,有88 016条Unigene鉴定到SSR位点。SSR特征分析的结果,为芳樟和杂樟及其同属物种的目标基因的标定、遗传多样性分析、遗传图谱构建和基因组差异分析提供了数据依据。

