通过生物信息学分析预测肺鳞癌病人预后的免疫信号
樊雨乔 林玫 徐文华


[摘要] 目的 基于免疫相關基因(IRGs)构建肺鳞癌(LUSC)病人预后风险模型。
方法 从癌症基因组图谱(TCGA)数据库下载551例LUSC病人的基因表达谱,并从ImmPort数据库中获得IRGs,再利用R软件筛选差异表达的免疫相关基因(DEIRGs);构建转录因子调控网络,探讨其异常表达的机制。应用单变量Cox回归确定DEIRGs的预后价值,应用Lasso回归和多变量Cox比例风险回归构建预后风险模型,并对模型的预测性能进行验证。评估这些危险因素与各种临床变量和免疫浸润细胞之间的关系。
结果 由PLAU、JUN、RNASE7、FOS、IGGD3-22、IGKV1-6、SEMA4C、APLN、FGFR4和TRAV39共10个IRGs构成的风险模型显示出良好的预测性能,有望成为预测LUSC病人预后的独立因素,且该模型还能对不同预后的病人进行分层,风险评分高的病人更容易出现免疫细胞浸润。
结论 IRGs可用于评估LUSC病人的预后和肿瘤免疫微环境的状态。
[关键词] 肺肿瘤;转录因子;计算生物学;ROC曲线;预后
[中图分类号] R734.2
[文献标志码] A
[文章编号] 2096-5532(2021)05-0679-06
doi:10.11712/jms.2096-5532.2021.57.169
[开放科学(资源服务)标识码(OSID)]
[网络出版] https://kns.cnki.net/kcms/detail/37.1517.R.20210909.1511.001.html;2021-09-09 17:35:37
A BIOINFORMATICS ANALYSIS OF IMMUNE SIGNALS FOR PREDICTING THE PROGNOSIS OF PATIENTS WITH LUNG SQUAMOUS CELL CARCINOMA
FAN Yuqiao, LIN Mei, XU Wenhua
(Qingdao University Medical College, Medical Inspection Department, Qingdao 266071, China)
[ABSTRACT] Objective To establish a prognostic risk model for patients with lung squamous cell carcinoma (LUSC) based on immune-related genes (IRGs).
Methods The gene expression profiles of 551 LUSC patients were downloaded from The Cancer Genome Atlas (TCGA) database, and IRGs were obtained from the ImmPort database. R software was used to screen out differentially expressed IRGs (DEIRGs), and a transcription factor regulatory network was constructed to investigate the mechanism of abnormal expression. A univariate Cox regression analysis was used to investigate the prognostic value of DEIRGs; Lasso regression analysis and multivariate Cox proportional-hazards regression model were used to construct a prognostic risk model, and the predictive performance of this model was verified. In addition, the association of these risk factors with various clinical variables and immune infiltrating cells was evaluated.
Results The risk model based on the 10 IRGs of PLAU, JUN, RNASE7, FOS, IGGD3-22, IGKV1-6, SEMA4C, APLN, FGFR4, and TRAV39 showed good predictive performance and was expected to be used as an independent factor for predicting the prognosis of LUSC patients. The model was able to stratify patients based on prognosis, and patients with high risk scores were more likely to develop immune cell infiltration.
Conclusion IRGs can be used to evaluate the prognosis of patients with LUSC and the state of tumor immune microenvironment.
[KEY WORDS] lung neoplasms; transcription factors; computational biology; ROC curve; prognosis
肺鳞癌(LUSC)是肺癌常见的亚型[1-2],病人病死率高[3]。目前,LUSC治疗策略主要包括手术、化疗、靶向治疗和免疫治疗。早期病人多采用手术切除治疗,晚期则以化疗为主[4],但后者预后不佳[5-6]。致癌基因的发现对肺癌的治疗策略选择产生了重大的影响,但LUSC病人很少发生表皮生长因子受体(EGFR)基因突变和间变性淋巴瘤激酶(ALK)易位,这限制了其靶向分子治疗的选择[7-10]。目前,免疫治疗成为提高LUSC病人生存率的重要方法[11],阻断免疫检查点通路是抗肿瘤治疗的热点[12-14]。然而,一些病人对免疫检查点抑制剂不敏感[15]。有研究结果表明,免疫相关基因(IRGs)不仅与病人的预后有关,还通过影响肿瘤免疫微环境影响病人对免疫治疗的敏感性[16]。本研究旨在开发一种基于多个IRGs的预后信号,评估LUSC病人的预后及对免疫治疗的敏感性。
1 资料和方法
1.1 数据采集
数据资源下载于TCGA数据库(http://portal.gdc.cancer.gov/)。①登录TCGA数据下载官网(https://portal.gdc.cancer.gov/),点击Repository,进入数据存储地;②点击Case,选择肿瘤原发部位、项目、疾病类型;③点击Files,选择基因表达的测序数据HTSeq-FPKM进行下载(https://gdc-hub.s3.us-east-1.amazonaws.com/latest/TCGA-LUSC.htseq_fpkm.tsv.gz)。其中包括所有病人的基因表达谱和临床统计数据,总共获得了502例LUSC组织和49例正常肺组织。临床信息包括病人的年龄、性别、TNM分期、T分期、N分期、M分期、生存時间和生存状态。从ImmPort数据库(http://www.immport.org)下载了2 483个IRGs[17]。
IRGs根据功能不同,分为细胞因子、肿瘤坏死因子家族受体、B细胞受体信号通路和白细胞介素等17个免疫类别。从AnimalTFDB(http://bioinfo.life.hust.edu.cn/AnimalTFDB/)下载了1 665个转录因子。
1.2 差异表达免疫相关基因(DEIRGs)的鉴定
从TCGA数据库下载LUSC及正常肺组织样本,用R软件(3.6.1版)进行分析,鉴定肿瘤组织和正常肺组织之间差异表达的基因。使用火山图和热图对筛选出的差异表达基因进行可视化处理,分别通过R软件的ggplot2包和pheatmap包进行构建。然后,从中筛选IRGs,获得DEIRGs。
1.3 转录因子调控网络
构建转录因子调控的网络,通过R软件分析LUSC和正常肺组织间差异表达的转录因子,并构建火山图和热图;然后建立转录因子调控网络,探讨其与DEIRGs的关联性。
1.4 模型建立与验证
从TCGA数据库中选取有完善预后信息的LUSC病人215例,随机分为训练集(n=108)和测试集(n=107)。在训练集中建立风险回归模型,并在测试集中进行验证。通过单变量Cox回归分析,确定与预后相关的风险基因。通过Lasso回归分析,去除彼此之间高度相关的风险基因;通过多变量Cox回归分析建立与预后相关的风险回归模型。
1.5 生存分析
根据预后模型计算每个LUSC病人风险评分。以中位风险评分作为临界值,将LUSC病人分为低风险组和高风险组,用R软件中的survival包绘制Kaplan-Meier生存曲线,散点图示病人随访时间。
1.6 肿瘤免疫微环境分析
使用肿瘤免疫微环境估计资源(TIMER,http://cistrome.dfci.harvard.edu/TIMER/)算法[18],分析预后模型的风险评分与肿瘤浸润免疫细胞之间的相关性。
1.7 统计分析
应用R软件包limma进行差异表达基因分析,采用Benjamini-Hochberg法进行校正,以|log2 fold change(FC)|>1和FDR<0.05作为筛选差异基因的标准。应用R软件中survival包分析受试者工作特征(ROC)曲线,ROC曲线下面积(AUC)>0.60被认为是一个可接受的预测模型,而AUC>0.70被认为具有显著的预测价值[19-20]。比较高风险组和低风险组的临床参数,连续变量比较采用t检验,分类变量比较采用χ2检验、对数秩检验和Cox比例风险回归模型。以P<0.05为差异有显著性。
2 结 果
2.1 DEIRGs的鉴定
根据TCGA数据库,共鉴定出8 478个差异表达的基因。其中,与正常肺组织相比较,LUSC组织中有5 893个基因表达上调,2 585个基因表达下调。从获得的差异表达基因中进一步筛选出593个DEIRGs,在这17个当中,307个DEIRGs表达上调,286个DEIRGs表达下调。
2.2 转录因子调控网络
共发现了70种在正常肺组织和LUSC组织之间显著差异表达的转录因子,其中17个与DEIRGs异常表达显著相关(r>0.4,P<0.05)。在这17个当中,3个转录因子负调控IRGs的表达,14个转录因子正调控IRGs的表达。
2.3 与预后相关的DEIRGs的鉴别
单变量Cox回归分析表明,共有24个DEIRGs与LUSC病人的预后显著相关(P<0.01)(图1)。
2.4 风险模型中与预后相关的DEIRGs的鉴定
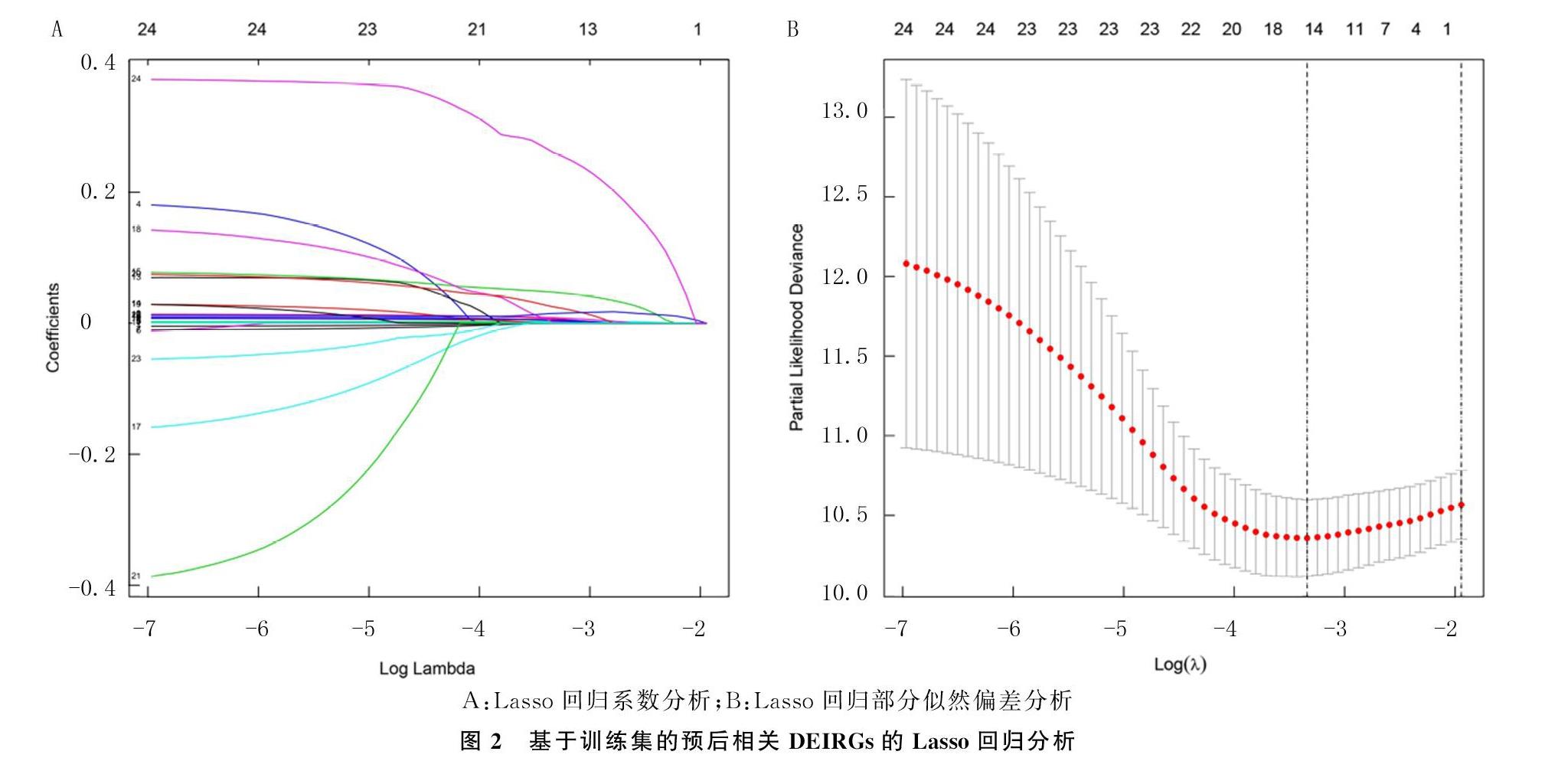
基于预后指标对LUSC病人总体生存率的影响,进一步筛选预后指标,构建基于训练集数据的Cox回归风险模型。Lasso回归分析获得了14个候选基因(图2),经多变量Cox比例风险回归分析最终获得了10个高危基因,分别为PLAU、JUN、RNASE7、FOS、IGGD3-22、IGKV1-6、SEMA4C、APLN、FGFR4和TRAV39。这10个基因均与LUSC病人预后不良有关。
2.5 基于训练集的预后风险模型
为了探讨风险基因在评估LUSC病人预后中的意义,使用以下公式计算每个病人的风险评分:风险评分=(0.002 071 216×PLAU的表达量)+(0.005 001 859×JUN的表达量)+(0.011 714 662×RNASE7的表达量)+(0.001 904 47×FOS的表达量)+(0.008 147 631×IGGD3-22的表达量)+
(0.000 379 557×IGKV1-6的表达量)+(0.013 048 762×SEMA4C的表达量)+(0.061 256 145×APLN的表达量)+(0.059 219 257×FGFR4的表达量)+(0.397 970 139×TRAV39的表达量)。以中位风险评分作为临界值,将训练集中的病人分为高风险组和低风险组。Kaplan-Meier曲线分析显示,高风险组和低风险组在训练集中的存活时间差异有显著性(χ2=11.4,P<0.05)(图3A);时间相关的ROC曲线分析结果显示,建立的预后模型可靠,ROC曲线AUC在3年和5年分别为0.721和0.715(图3B、C),低风险评分病人的生存状况优于高风险评分病人(图3D、E)。热图分析结果显示,与低风险组相比,高风险组10种风险基因的表达水平高于低风险组(图3F)。
2.6 预后模型性能的验证
分别在测试集和整个TCGA集中验证建立的预后风险模型的预测性能。根据每个病人的风险评分,将测试集和整个TCGA集的病人分别按照风险评分中位数分为高风险组和低风险组,进一步绘制Kaplan-Meier曲线和时间相关的ROC曲线,比较高风险组和低风险组的3年生存率和5年生存率。结果表明,无论是在测试集还是整个TCGA集中,低风险组的生存率都高于高风险组(图4A、B)。在测试集和整个TCGA集中,3年生存率AUC分别为0.631和0.679(图4C、D),5年生存率AUC分别为0.634和0.692(图4E、F)。与低风险组相比,高风险组病人生存状况更差,风险基因表达水平更高。表明建立的预后风险模型具有良好的预测性能。
2.7 风险评分在整个TCGA集中的独立预后价值
单变量Cox回归分析显示,病理分期(P=0.005)、T分期(P=0.005)和风险评分(P<0.001)与LUSC病人的生存结果显著相关。多变量Cox回归分析結果表明,风险评分可作为预测LUSC病人预后的独立危险因素(P<0.001)。时间相关的ROC曲线分析显示,3年时病理分期、T分期和风险评分的AUC分别为0.587、0.597和0.691,5年时分别为0.536、0.526和0.694。
2.8 预后模型的临床有效性
基于整个TCGA集分析模型变量(风险基因和风险评分)和临床变量(年龄、性别、病理分期和TNM分期)之间的关系显示,随着PLAU表达的增加,LUSC的T分期进展迅速(P=0.021);不同年龄的病人某些危险基因的表达也不同,APLN
(P=0.038)、JUN(P=0.019)和PLAU(P=0.008)
在65岁以上LUSC病人中的表达水平明显高于年轻病人;FGFR4在男性LUSC病人中的表达水平显著高于女性病人(P=0.020)。风险评分与B细胞、CD8+T细胞、树突状细胞、巨噬细胞和中性粒细胞等浸润程度呈正相关(r=0.106~0.171,P<0.05)。表明风险评分能够评估LUSC病人肿瘤免疫微环境的状态。
3 讨 论
肺癌是一种病死率较高的恶性肿瘤,虽然近年来在诊疗方面取得了很大进展,但生存率仍然不容乐观。免疫治疗的兴起及临床应用为肺癌的诊疗手段提供了新的思路。越来越多的证据表明,肿瘤免疫微环境可以影响肿瘤的恶性表型[21-23]。在多种恶性肿瘤中,免疫浸润与临床结果密切相关[24-25],尤其是在肺癌中[26-27]。本文研究基于TCGA数据库,确定了与预后相关的DEIRGs,并构建了一个预后风险模型来评估LUSC病人的生存结果及肿瘤免疫微环境的状态。
本研究分析了LUSC组织和正常肺组织之间DEIRGs,并构建了转录因子调控网络,得到17个转录因子与IRGs异常表达相关。表明肿瘤转录因子可能通过调节IRGs的表达来影响LUSC病人的预后。本研究还探讨了DEIRGs与LUSC病人预后之间的相关性,并评估了与LUSC预后相关的DEIRGs在预测病人预后中的效率。通过Lasso回归分析,最终获得10个风险基因,纳入预后风险模型,并在整个TCGA数据集中进行验证。本文研究结果表明,本文建立的预后模型能够有效区分不同风险的病人,风险评分可作为预测LUSC病人预后的独立因素,且优于其他临床参数。因此,风险模型可用于筛查高危病人,以便通过早期治疗改善预后。本文研究评估了危险因素和一些临床变量之间的关系,结果显示,一些风险基因的表达与LUSC进程呈正相关,表明所构建的风险模型能够有效预测LUSC进程。
免疫疗法在肺癌的治疗中显示出不同的临床效果,这部分取决于肿瘤浸润淋巴细胞的数量和特征[28-29]。有研究认为,肺恶性肿瘤中浸润的免疫细胞可能对病人的免疫治疗反应和预后产生重要影响[22]。本文分析了风险评分与肿瘤浸润免疫细胞之间的关系,结果表明,病人风险评分越高,免疫细胞浸润程度越高,说明风险模型在评估LUSC生存结果中可信度较高。
基于IRGs的風险模型被广泛用于评估多种恶性肿瘤病人的预后。本文研究具有以下优势:①分析了IRGs在LUSC中的表达模式;②进行Lasso回归,删除彼此高度相关的基因,增加了结果的可信度;③所建立的模型不仅可用于预测LUSC病人的预后,还可用于评估病人肿瘤免疫微环境的状态,从而辅助制定个性化的治疗方案。
综上所述,本文通过生物信息学分析,得到了基于IRGs的预后信号,该信号可用于评估LUSC病人的预后和肿瘤免疫微环境的状态,从而帮助制定个性化的治疗方案,提高病人的生存率。
[参考文献]
[1]SORBER L, ZWAENEPOEL K, DESCHOOLMEESTER V, et al. Circulating cell-free nucleic acids and platelets as a liquid biopsy in the provision of personalized therapy for lung cancer patients[J]. Lung Cancer (Amsterdam, Netherlands), 2017,107:100-107.
[2]CHEN W J, GAN T Q, QIN H, et al. Implication of downregulation and prospective pathway signaling of microRNA-375 in lung squamous cell carcinoma[J]. Pathology, Research and Practice, 2017,213(4):364-372.
[3]XU F, ZHANG H P, CHEN J X, et al. Immune signature of T follicular helper cells predicts clinical prognostic and therapeutic impact in lung squamous cell carcinoma[J]. Internatio-
nal Immunopharmacology, 2020,81:105932.
[4]KURIBAYASHI K, FUNAGUCHI N, NAKANO T. Chemotherapy for advanced non-small cell lung cancer with a focus on squamous cell carcinoma[J]. Journal of Cancer Research and Therapeutics, 2016,12(2):528.
[5]THATCHER N, HIRSCH F R, LUFT A V, et al. Necitumumab plus gemcitabine and cisplatin versus gemcitabine and cisplatin alone as first-line therapy in patients with stage Ⅳ squamous non-small-cell lung cancer (SQUIRE): an open-label, randomised, controlled phase 3 trial[J]. The Lancet Oncology, 2015,16(7):763-774.
[6]SCAGLIOTTI G V, PARIKH P, VON PAWEL J, et al. Phase Ⅲ study comparing cisplatin plus gemcitabine with cis-platin plus pemetrexed in chemotherapy-naive patients with advanced-stage non-small-cell lung cancer[J]. Journal of Cli-nical Oncology: Official Journal of the American Society of Clinical Oncology, 2008,26(21):3543-3551.
[7]PEREZ-MORENO P, BRAMBILLA E, THOMAS R, et al. Squamous cell carcinoma of the lung: molecular subtypes and therapeutic opportunities[J]. Clinical Cancer Research, 2012,18(9):2443-2451.
[8]GERBER D E, GANDHI L, COSTA D B. Management and future directions in non-small cell lung cancer with known activating mutations[J]. American Society of Clinical Oncology Educational Book American Society of Clinical Oncology Annual Meeting, 2014:e353-e365.
[9]ROSE-JAMES A, TT S. Molecular markers with predictive and prognostic relevance in lung cancer[J]. Lung Cancer International, 2012, 2012:729532.
[10]REKHTMAN N, PAIK P K, ARCILA M E, et al. Clarifying the spectrum of driver oncogene mutations in biomarker-verified squamous carcinoma of lung: lack of EGFR/KRAS and presence of PIK3CA/AKT1 mutations[J]. Clinical Cancer Research: an Official Journal of the American Association for Cancer Research, 2012,18(4):1167-1176.
[11]SENOO S, NINOMIYA K, HOTTA K, et al. Recent treatment strategy for advanced squamous cell carcinoma of the lung in Japan[J]. International Journal of Clinical Oncology, 2019,24(5):461-467.
[12]LIM J S J, SOO R A. Nivolumab in the treatment of metasta-
tic squamous non-small cell lung cancer: a review of the evidence[J]. Therapeutic Advances in Respiratory Disease, 2016,10(5):444-454.
[13]GANDHI L, RODRGUEZ-ABREU D, GADGEEL S, et al. Pembrolizumab plus chemotherapy in metastatic non-small-cell lung cancer[J]. The New England Journal of Medicine, 2018,378(22):2078-2092.
[14]SOCINSKI M A, OBASAJU C, GANDARA D, et al. Current and emergent therapy options for advanced squamous cell lung cancer[J]. Journal of Thoracic Oncology, 2018,13(2):165-183.
[15]BODOR J N, BOUMBER Y, BORGHAEI H. Biomarkers for immune checkpoint inhibition in non-small cell lung cancer (NSCLC)[J]. Cancer, 2020,126(2):260-270.
[16]QU Y, CHENG B, SHAO N, et al. Prognostic value of immune-related genes in the tumor microenvironment of lung
adenocarcinoma and lung squamous cell carcinoma[J]. Aging, 2020,12(6):4757-4777.
[17]BHATTACHARYA S, ANDORF S, GOMES L, et al. ImmPort: disseminating data to the public for the future of immunology[J]. Immunologic Research, 2014,58(2/3):234-239.
[18]LI B, SEVERSON E, PIGNON J C, et al. Comprehensive analyses of tumor immunity: implications for cancer immunotherapy[J]. Genome Biology, 2016,17(1):174.
[19]CHO S H, PAK K, JEONG D C, et al. The AP2M1 gene expression is a promising biomarker for predicting survival of patients with hepatocellular carcinoma[J]. Journal of Cellular Biochemistry, 2019,120(3):4140-4146.
[20]HAN M E, KIM J Y, KIM G H, et al. SAC3D1: a novel prognostic marker in hepatocellular carcinoma[J]. Scientific Reports, 2018,8(1):15608.
[21]MONY J T, SCHUCHERT M J. Prognostic implications of heterogeneity in intra-tumoral immune composition for recurrence in early stage lung cancer[J]. Frontiers in Immunology, 2018,9:2298.
[22]LIU X Y, WU S C, YANG Y H, et al. The prognostic landscape of tumor-infiltrating immune cell and immunomodulators in lung cancer[J]. Biomedecine & Pharmacotherapie, 2017,95:55-61.
[23]XIONG Y F, WANG K, ZHOU H, et al. Profiles of immune infiltration in colorectal cancer and their clinical significant: a gene expression-based study[J]. Cancer Medicine, 2018,7(9):4496-4508.
[24]FRIDMAN W H, PAGS F, SAUTS-FRIDMAN C, et al. The immune contexture in human tumours: impact on clinical outcome[J]. Nature Reviews Cancer, 2012,12(4):298-306.
[25]FRIDMAN W H, GALON J, DIEU-NOSJEAN M C, et al. Immune infiltration in human cancer: prognostic significance and disease control[J]. Current Topics in Microbiology and Immunology, 2011,344:1-24.
[26]BREMNES R M, BUSUND L T, KILVR T L, et al. The role of tumor-infiltrating lymphocytes in development, progression, and prognosis of non-small cell lung cancer[J]. Journal of Thoracic Oncology, 2016,11(6):789-800.
[27]KUREBAYASHI Y, EMOTO K, HAYASHI Y, et al. Comprehensive immune profiling of lung adenocarcinomas reveals four immunosubtypes with plasma cell subtype a negative indicator[J]. Cancer Immunology Research, 2016,4(3):234-247.
[28]RIZVI N A, HELLMANN M D, SNYDER A, et al. Cancer immunology. Mutational landscape determines sensitivity to PD-1 blockade in non-small cell lung cancer[J]. Science (New York, N Y), 2015,348(6230):124-128.
[29]TUMEH P C, HARVIEW C L, YEARLEY J H, et al. PD-1 blockade induces responses by inhibiting adaptive immune resistance[J]. Nature, 2014,515(7528):568-571.
(本文編辑 黄建乡)

