海南桑树青枯病病原菌鉴定及其分子鉴定
娄德钊 武华周 卢芙萍 耿涛 涂娜娜 王树昌







摘 要:桑树青枯病是由青枯雷尔氏菌引起的细菌性病害,热带、亚热带地区发病严重,严重影响蚕桑产业的可持续发展。雷尔氏菌不同种间致病力和宿主各不相同,其防治策略也相应不同,准确地分离鉴定病原菌是青枯病有效防控的先决条件。本研究采集、分离了海南省琼中县桑青枯病发病桑园(‘桂桑优62’)桑树根部、茎部病原菌,并通过致病性、生理小种、生化变种测定,结合16S rDNA、特异性引物、复合PCR检测体系、序列变种等分子鉴定方法初步确定了病原菌的种类和分类地位。结果表明,引发海南省琼中县桑青枯病的病原菌属于青枯雷尔氏菌(Ralstonia solanacearum)、生理小种5(race 5)、生化变种Ⅴ(biovar Ⅴ),病原菌遗传进化分析结果显示病原菌属演化型Ⅰ(phylotype Ⅰ)即亚洲分支菌株,序列变种12(sequevar 12)。这些结果将为海南桑青枯病的有效防控奠定基础。
关键词:桑树青枯病;病原鉴定;雷尔氏菌;分子鉴定;系统发育
中图分类号:S888.71 文献标识码:A
Abstract: Bacterial wilt of mulberry is a bacterial disease caused by Ralstonia solanacearum. It is serious in tropical and subtropical areas, which restricts the development of sericulture industry. The pathogenicity and host of R. solanacearum are different from each other, and the control strategy is also different. Accurate isolation and identification of the pathogen is the prerequisite for the effective control of bacterial wilt. In this study, we collected and isolated the pathogenic bacteria of mulberry (‘Guisangyou 62’) in Qiongzhong County, Hainan Province. The strains were selected and tested for pathogenicity, race and biovar, combined with 16S rDNA molecular identification, specific primers, multiplex PCR detection system and sequence variation, and with phylogenetic analysis. All the three strains were R. solanacearum. According to the traditional classification the pathogen belongs to physiological race 5 and biovar V. The genetic evolution of the pathogen was further analyzed by molecular biology methods. The pathogen belonged to phylotype I (the Asiaticum division), sequevar 12. The results would lay a foundation for the effective control of mulberry bacterial wilt in Hainan.
Keywords: Morus alba L. bacterial wilt; pathogen identification; Ralstonia solanacearum; molecular identification; phylogeny
DOI: 10.3969/j.issn.1000-2561.2021.11.027
桑樹(Morus alba L.)是一种重要的经济作物,桑叶、桑葚、桑根、桑枝均可入药,桑叶不仅是家蚕饲料,而且可以制成桑茶、桑叶菜、桑叶粉及桑叶饮料等,被列入“既是食品又是药品的物品”名单。桑树种植面积广,朝鲜、日本、蒙古、中亚各国、欧洲、东南亚等地以及非洲均有栽培。桑树栽培起源于中国,长期以来种植于我国长江、黄河中下游地区;随着“东桑西移”“北桑南移”,我国桑蚕重心逐渐向亚热带和热带地区发生转移[1]。海南岛地处108°37′~ 118°03′ E,18°10′~20°10′ N,气候属热带季风季候,年平均气温22 ℃~26 ℃,年均降水量2000~2400 mm,桑树全年生长,与其他地区相比,海南的蚕桑产业拥有得天独厚的地理优势[1],近年来种植面积不断扩大[2]。
青枯雷尔氏菌(Ralstonia.solanacearum)寄主范围广泛,能够引起植物的青枯症状,主要表现为,初期上部叶片迅速卷曲、萎蔫,而后整株植株叶片卷曲、萎蔫,萎蔫叶片仍为青色,最终导致整株植株枯死,在多种作物上造成巨大的经济损失[3]。青枯菌的鉴定主要依据菌落形态鉴定(TTC平板划线),然后根据青枯菌生理生化性质划分为生理小种(race)与生化变种(biovar);Fegan等[4]提出了演化型分类框架,将青枯菌划分为种(species)、演化型(phylotype)、序列变种(sequevar)、克隆(clone)4个不同水平的分类单元,种(species)根据特异性引物759/760 PCR扩增结果确定[5];演化型(phylotype)以ITS区设计的演化型复合PCR检测体系确定[6];根据青枯菌种部分内源葡聚糖酶基因(endoglucanase, egl)同源性序列确定序列变种(sequevar)[6];用基因组指纹方法(AFLP、re-PCR)确定克隆(clone)[4]。
国内外对桑树青枯病的研究主要在病原菌生理小种、生化变种鉴定、病原菌检测等方面,分子水平鉴定及种内划分报道较少,而有关海南省热区桑树青枯病病原菌未见报道。准确区分病原菌地理起源、种内分类,有利于病害的精准防控,因此本研究通过致病性、生理小种、生化变种测定,结合16S rDNA、特异性引物、复合PCR检测体系、序列变种等分子鉴定方法,对海南省桑青枯病病原进行分离鉴定,并确定病原菌种内分类及演化型,为桑青枯病的有效防控提供前提条件。
1 材料与方法
1.1 病害调查、样品采集和病原菌分离
2019年4月在海南省琼中县营根镇(19º02′ N, 109º76′ E)调查并采集典型桑青枯病发病植株(‘桂桑优62’),观察发病症状、坏死植株根茎部木质部形态等,记录发病情况,计算发病率,并将病样带回实验室分离鉴定病原菌。
参照Buddenhagen等[7]、方中达[8]的方法,将采集的根部组织冲洗干净,剥去表皮,用无菌水冲洗后,75%酒精消毒1 min,最后用无菌水冲洗3次,放入无菌管中,加入1 mL无菌水浸泡10 min。用TTC培养基[每升TTC培养基包含酸水解酪蛋白1.0 g、蛋白胨10.0 g、葡萄糖5.0 g、琼脂17.0 g,pH=7.0,121 ℃灭菌20 min,待冷却至55 ℃后,添加1% 2,3,5-三苯基氯化四氮唑溶液(TTC)5 mL]、M-SMSA培养基(每升S-SMSA培养基含酸水解酪蛋白1.0 g、甘油5.0 mL、蛋白胨10.0 g、琼脂17.0 g,pH=7.0,121 ℃灭菌20 min,待冷却至55 ℃后,加入1%结晶紫5 mL、1%多粘菌素β硫酸盐10 mL、1%杆菌肽2.5 mL、1%氯霉素0.5 mL、1%青霉素0.5 mL、1%放线菌酮10 mL和1%TTC 5 mL),对致病菌进行分离、纯化。纯化获得的菌株分别加入终浓度为20%的甘油中于–80 ℃保存。另取一部分纯化菌株,加入无菌水于常温保存。实验共分离到6个菌株,选取3株代表性菌株(MRS1、MRS2、MRS3)进行后续实验。
1.2 桑青枯菌致病性测定
将常温保存的纯化菌株在TTC培养基上划线活化,室温下培养48 h后,挑取单一菌落,放入LB液体培养基,置于28 ℃、150 r/min摇床中;24 h后采用稀释平板法,与紫外分光光度计法,根据OD600吸光光度值绘制标准曲线,将菌悬液浓度稀释至1×108 CFU/mL。对照菌株为R. solanacearum 448,由中國热带农业科学院环境与植物保护研究所提供。
接种选用1年生5~7叶桑种子苗(‘桂桑优62’)。桑种子苗是将桑树种子用清水洗净,放入75%酒精中消毒2 min后,用无菌水清洗3次,用30 ℃无菌水清洗3次,用无菌土进行培育所得。选取长势均匀、健康桑种子苗,减掉根部4~5根侧根,以每次5 mL浓度为1×108 CFU/mL的菌悬液浇灌至根部,连续浇灌3 d,每天1次。以接种空白的LB营养液为对照,每个处理接种15株桑苗。接种后观察记录发病情况,收集发病植株根部组织,按1.1方法再次对致病菌分离纯化并保存。
1.3 生理小种的测定
参照Buddenhagen等[7, 9-10]的研究方法。采用注射接种法,将浓度为1×108 CFU/mL的代表菌株菌液分别接种桑(‘桂桑优62’)、茄子(紫红长茄)、番茄(珍珠番茄)、辣椒(‘丰椒18’)、烟草(本生烟)、姜(海南本地肉姜)、香蕉(巴西蕉),测定桑青枯菌对这些植物的致病性。桑、茄子、番茄、辣椒分别接种30株,烟草、番茄、香蕉、姜分别接种15株,每株接种30 µL。
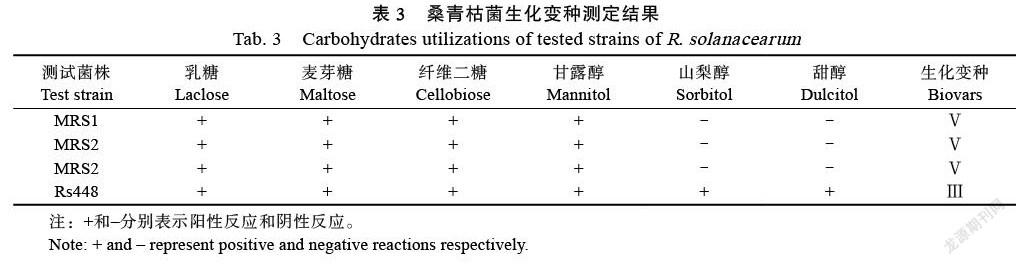
1.4 生化变种的鉴定
参照方中达[8]、Hayward[11]的方法,配制3种双糖(乳糖、麦芽糖、纤维二糖)、3种己醇(甘露醇、山梨醇、甜醇)的10%母液,经灭菌后添加至基础培养基至终浓度1%,调节pH至7.0后添加溴百里酚蓝指示剂。菌株活化后接种于此培养基,28 ℃恒温培养10 d,记录观察结果,确定生化变种分类。
1.5 病原菌的分子鉴定及分子特征性分析
1.5.1 青枯菌基因组DNA的提取 用细菌基因组DNA提取试剂盒提取青枯菌基因组DNA,试剂盒由QIAGN公司提供的QIAamp DNA Mini Kit。提取的DNA OD260/280比值在1.82.0之间,提取后的DNA放于–20 ℃冰箱中保存。
1.5.2 青枯菌16S rDNA、特异性引物PCR、演化型鉴定 通过细菌通用引物和青枯雷尔氏菌特异性引物(表1),应用TIANGEN的Taq DNA polymerase试剂盒进行PCR扩增,反应体系50 μL,反应程序参照表1,其中模板DNA 6 pmol,引物各12 pmol。PCR仪型号Bio-RAD T100。扩增产物在1%琼脂糖凝胶电泳后,通过Biorad凝胶成像仪观察结果。扩增后16S rDNA呈单一条带的PCR产物送生工生物工程(上海)股份有限公司测序。同时参照Fegan和Prior方法采用复合引物PCR扩增,方法参照表1文献,反应体系为50 μL。取7 μL反应产物进行琼脂糖凝胶电泳,凝胶浓度1%,根据条带确定青枯菌演化型。
1.5.3 青枯菌序列变种鉴定 应用Endo-F、Endo-R扩增代表性菌株egl基因,扩增产物在1%琼脂糖凝胶电泳后,呈单一条带的PCR产物送生工生物工程(上海)股份有限公司测序。根据Xu等[15]报道的序列变种菌株与测试菌株进行序列变种分析。数据分析应用软件MEGA7.0,采用邻接法(neighbor-joining)Bootstrap 1000次进行聚类分析,构建进化树,确定测试菌株序列变种的分类。
2 结果与分析
2.1 病原菌的分离和培养
田间观察表明:植株上部叶片首先发病,快速失水萎蔫,继而全株叶片萎蔫,植株枯死,呈青枯状(图1A)。病株根部横截面观察表明:维管束组织变褐、坏死,显微镜观察到有大量细菌呈雾状喷出,呈现典型的感染青枯病植株后的“溢菌”现象(图1B)。从发病植株和根际土壤中分类获得6个独立细菌株系,其中3个株系在TTC平板上的菌落形态不规则,中间隆起;颜色呈粉红或淡红色,边缘白色;菌落表面光滑、湿润具有流动性,具备青枯雷尔氏菌的典型特征(图1C)。在N-SMSA培养基上进一步观察发现,这3个株系菌落形态同样不规则,颜色呈紫红色,表面光滑湿润且具有流动性。革兰氏染色表明该菌为革兰氏阴性菌;电子显微镜观察表明该菌呈杆状、着生鞭毛,菌体大小为(0.46~0.87)µm × (1.02~2.13)µm(图1D),据此初步判定,这3个株系为青枯雷尔氏菌。
2.2 致病性测定
将分离到的3个独立菌株(MRS1、MRS2、MRS3)分别回接生长至5~7叶的桑种子苗。结果表明,接种7 d后植株开始出现萎蔫症状(图2A);10 d后部分叶片完全坏死并逐渐脱落;14 d后病株枯萎死亡。统计结果表明:MRS1的发病死亡率为85%,MRS2的发病死亡率为80%,MRS3的发病死亡率为93%,相应的空白对照发病死亡率为0。进一步观察表型发现,接种植株与田间桑树发病症状表现一致,同时均能够在接种植株中分离到接种菌株。由此认为,分离获得的病原细菌是桑青枯病的致病菌,且具有较强致病力。
2.3 生理小种鉴定
将分离到的3个代表性菌株(MRS1、MRS2、MRS3)室内注射接种7种寄主植物,结果表明,3株菌株能够侵染桑树且具有强致病力,3株菌株侵染番茄、茄子,但其致病力较弱,且并不致死;除MRS2菌株外均可侵染辣椒,但致病力较弱;3
株菌株均不侵染烟草、姜、香蕉。对照菌株Rs448分离自番茄,可侵染番茄、茄子、辣椒,对桑树、烟草、姜、香蕉无致病力(表2)。根据茄科雷尔氏菌生理小种分类标准[7, 9-10],从桑树分离到的3株测试菌株属于生理小种5(race 5),对照菌株Rs448属于生理小种1(race 1)。
2.4 生化变种鉴定
3种双糖、3种己醇利用实验结果表明:3个菌株可以有效利用麦芽糖、纤维二糖、乳糖和甘露醇,但不可以有效利用山梨醇和甜醇;且青枯菌利用糖、醇效率不同,其中乳糖在接种7 d后才发生颜色变化(表3),根据Hayward提出的生化变种划分方法[16],初步鉴定本研究分离到的桑青枯病菌属于生化变种Ⅴ。
2.5 病原菌的分子生物學鉴定
以细菌通用引物扩增3个代表性菌株及对照菌株,均能扩增出1500 bp左右的特异性片段(图3A),序列拼接后全长为1430 bp,经BLAST序列比对结果显示,序列与标准菌株同源率为99%。应用R. solanacearum的特异性引物对3个代表性菌株PCR扩增结果显示,引物759/760能够扩增出280 bp左右的单一条带(图3B),fliC-F/R能够扩增出400 bp左右的单一条带(图3C),结果证明测试菌株属于R. solanacearum复合种。
2.6 病原菌序列变种分析
根据Fegan等[4]提出的茄科雷尔氏菌演化型分类方案,采用复合PCR检测体系,3个菌株均可同时扩增得到144 bp和280 bp 2条特异片段(图4B),其中280 bp片段为茄科雷尔氏菌特异性扩增条带,144 bp片段为演化型Ⅰ(亚洲型)的特异性扩增条带。
用引物对Endo-F、Endo-R扩增病原菌内切葡聚糖酶基因(egl),菌株MRS1、MRS2、MRS3均能扩增出840 bp左右的条带(图4A),测序后与从GenBank中下载的青枯菌菌株的egl基因序列进行聚类分析。结果显示(图5)菌株MRS1(MW294008)、MRS2(MW294009)、MRS3(MW294010)属于亚洲型(phylotypeⅠ),序列变种12(sequevar 12)。
3 讨论
青枯病的发生严重制约热带地区蚕桑产业的发展,桑园发病率最高可达80%[17],导致经济损失严重。本研究从海南省琼中县发生青枯病的桑树植株茎和根中分离致病菌,经鉴定确定为变形菌门(Proteobacteria)、伯克氏菌科(Burk¬holderiales)、雷尔氏菌属(Ralstonia)。
根据青枯菌对不同寄主侵染能力的差异,提出了青枯菌生理小种(race)的划分[7, 9-10],小种1能够侵染烟草、茄科等多种作物;小种2对香蕉、海里康等植物具有较强的侵染力;小种3主要侵染马铃薯及其他茄科植物;亚洲姜上分离到的青枯菌被划分为小种4;1983年何礼远等[17]发现了对桑树具有强致病力的青枯菌菌株,并将其划分为小种5。国内研究发现侵染桑树的生理小种属于1和5,He[10]发现对桑树具有强致病力菌株,并将其划分为生理小种5,曹梦琪等[18-19]通过qPCR、PMA-qPCR等技术建立了青枯菌生理小种5的快速检测及活体检测方法。而赖文姜等[20]在广东分离到的菌株对茄科作物致病力强,但不侵染辣椒,属于生理小种1,这与吕志强等[21]在浙江分离到的菌株研究结果一致。本研究通过注射接种法从海南省琼中县分离到的3株青枯雷尔氏菌菌株均属于生理小种5。
Hayword[16]根据青枯菌对3种双糖和3种己醇的氧化能力,将青枯菌分为5个生化变种(biovar)。本研究通过3种双糖(麦芽糖、纤维二糖、乳糖)和3种己醇(甘露醇、山梨醇和甜醇)氧化产酸能力的不同,证明引起海南省桑树青枯菌的菌株为茄科雷尔氏菌生化变种Ⅴ。曾宪铭等[22]认为桑树生化变种属于Ⅰ、Ⅲ、Ⅴ,而吕志强等[21]在不同桑树组织分离到的青枯菌大多属于生化变种Ⅴ,部分属于生化变种Ⅲ,并且发现相同组织分离到菌株生化变种可能不一致;华静月等[23]同样发现桑青枯菌生化变种大部分属于V,只有M6菌株属于生化变种Ⅲ;但苗雪等[24]在广东省不同县市分离到的3株菌株均属于生化变种Ⅲ。表明在某些情况下桑青枯菌生化性状可能极易变异,导致同一样地、同一植株、同一组织内青枯菌生化变种可能不同。
传统的分类框架不能体现青枯菌地理起源与遗传进化[25]。Fegan等[4]提出了演化型分类框架,确定了种、演化型、序列变种及克隆4个分类单元。青枯菌的egl基因主要编码内切葡聚糖酶[26],同时内切葡聚糖酶作为一种胞外多糖,能够在植物细胞内分解纤维素,对于病原菌致病性有着重要作用。苗雪等[24]在广东感病桑园分离到的3株菌株属于演化型Ⅰ,这与本研究结果一致。Xu等[15]認为国内桑青枯菌属于序列变种12、48;但序列变种48菌株同时也能够引起马铃薯、番茄和姜[27]等植物的青枯病。本研究通过egl部分基因序列与多系统发育分析,结果表明,3个菌株均与R292、M4聚类于同一分支,同时与egl基因序列同源性达到99%,推断海南省琼中县桑青枯菌属于序列变种12;同时与苗雪等[24]分离到的菌株G12-9聚类于同一分支,属于同一序列变种;但海南省桑青枯病菌是否存在序列变种48,仍需进一步验证。
本研究系统地鉴定了桑树青枯病病原菌,鉴定结果为生理小种5(rice 5)、生化变种Ⅴ(biovarⅤ)、演化型Ⅰ(phylotypeⅠ)、序列变种12(sequevar 12)。
参考文献
[1] 刘吉平, 黄正恩, 朱方容, 等. 海南省发展蚕桑产业的SWOT分析[J]. 中国蚕业, 2015, 36(2): 49-54.
[2] 王树昌, 耿 涛, 黄华平, 等. 海南蚕桑[M]. 海口: 南海出版公司, 2017: 50-53.
[3] Denny T. Plant pathogenic Ralstonia species[M] Plant-Associated Bacteria. Springer Netherlands, 2007.
[4] Fegan M, Prior P. How complex is the Ralstonia solanacea-rum species complex[M]//Bacterial Wilt Disease and the Ralstonia solanacearum Species Complex. USA: The American Phytopathological Society, 2005: 449.
[5] Opina N. A novel method for development of species and strain-specific DNA probes and PCR primers for identifying Burkholderia solanacearum (formerly Pseudomonas solanacearum)[J]. Asia-Pacific Journal of Molecular Biology and Biotechnology, 1997, 5(1): 19-30.
[6] Fegan M, Taghavi M, Sly L I, et al. Phylogeny, diversity and molecular diagnostics of Ralstonia solanacearum[M]//Prior P, Allen C, Elphinstone J. Bacterial wilt disease: molecular and ecological aspects. Berlin, Springer, 1998.
[7] Buddenhagen I, Sequeira L, Kelamn A. Designation of races in Pseudomonas solanacearum[J]. Phytopathlolgy, 1964, 52: 726.
[8] 方中达. 植病研究方法[M]. 3版. 北京: 中国农业出版社, 1998
[9] Pegg K G, Moffett M L. Host range of the ginger strain of Pseudomonas solanacearum in Queensland[J]. Australian Journal of Experimental Agriculture, 1971, 19(53): 696-698.
[10] He L Y. Characteristics of strains of Pseudomonas solana-cearum from China[J/OL]. Plant Disease, 1983, 67(12): DOI:10.1094/PD-67-1357..
[11] Hayward A C. Characteristics of Pseudomonas solanacea-rum[J]. Journal of Applied Microbiology, 2010, 27(2): 265-277.
[12] Weisburg W G, Barns S M, Pelletier D A, et al. 16S ribo-somal DNA amplification for phylogenetic study[J]. Journal of Bacteriology, 1991, 173(3): 697-703.
[13] Ito S, Ushuima Y, Fujii T, et al. Detection of viable cells of Ralstonia solanacearum in soil using a semiselective me-dium and a PCR technique[J]. Journal of Phytopathology, 1998, 146(8/9): 379-384.
[14] Schönfeld J, Heuer H, Van F J D, et al. Specific and sensi-tive detection of Ralstonia solanacearum in soil on the basis of PCR amplification of fliC fragments[J]. Applied & Environmental Microbiology, 2003, 69(12): 7248-7256.
[15] Xu J, Pan Z C, Prior P, et al. Genetic diversity of Ralstonia solanacearum strains from China[J]. European Journal of Plant Pathology, 2009, 125(4): 641-653.
[16] Hayward A C. Biology and epidemiology of bacterial wilt caused by Pseudomonas solanacearum[J]. Annual review of phytopathology, 1991, 29(1): 2965-2987.
[17] 何礼远, 华静月, 张长龄, 等. 我国细菌性青枯病的发生及防治[J]. 植物保护, 1983, 9(3):8-10.
[18] 曹梦琪, 包 奇, 杨采风, 等. 基于qPCR技术快速检测青枯劳尔氏菌5号生理小种的方法[J]. 蚕业科学, 2015, 41(5): 807-814.
[19] 曹梦琪, 王旭东, 王 俊, 等. 基于PMA-qPCR检测青枯菌5号生理小种活菌的方法[J]. 蚕业科学, 2015, 41(6): 1004-1010.
[20] 赖文姜, 曾宪铭, 谭炳安, 等. 桑青枯病病原细菌的鉴定[J]. 华南农学院学报, 1982, 3(1): 66-73.
[21] 吕志强, 王汉荣, 周 勤, 等. 浙江省桑树青枯病菌生理小种及生化型的测定[J]. 浙江农业学报, 2007, 19(4): 306-309.
[22] 曾宪铭, 董 春. 广东农作物青枯病菌的生化型[J]. 华南农业大学学报, 1995, 16(1): 50-53.
[23] 华静月, 张长龄, 何礼远. 我国植物青枯菌的生化型和其他生理差异[J]. 植物保护学报, 1984(1): 43-50.
[24] 苗 雪, 唐翠明, 李 治, 等. 来自桑树的3株青枯劳尔氏菌分离株的亚分类研究[J]. 蚕业科学, 2017, 43(5): 737-743.
[25] 潘哲超. 植物青枯菌遺传多样性及致病力分化研究[D]. 北京: 中国农业科学院, 2010.
[26] Schell M A, Denny T P, Huang J. Extracellular virulence factors of Pseudomonas solanacearum: role in disease and regulation of expression[M]//Molecular Mechanisms of Bacterial Virulence, Springer Netherlands, 1994.
[27] Thano P, Akarapisan A. Phylotype and sequevar of Ralstonia solanacearum which causes bacterial wilt in Curcuma alismatifolia Gagnep[J]. Letters in Applied Microbiology, 2018, 66(5): 384-393.
责任编辑:谢龙莲

