U-Net及其在肝脏和肝脏肿瘤分割中的应用综述
张 欢,刘 静,冯毅博,仇大伟
山东中医药大学 智能与信息工程学院,济南 250355
近年来,医学临床数据呈现爆发式增长。据统计,我国的医学数据增长速率约为30%。海量的医学影像数据极大地增加了医生诊断的负担,但也推动了人工智能在医疗领域的快速发展与应用,“智能医疗”“辅助诊断”等新兴词汇逐渐走向大众视野[1-2]。传统的机器学习通过手工设计特征提取算法进行疾病分类,这种方法泛化性能较差,且准确率不高。深度学习作为机器学习的分支,由于其能自动从大样本数据中学习获得优良的特征表达,有效提升各种机器学习任务的性能,近年来得到了迅速发展[3]。其中,卷积神经网络是深度学习中的经典网络结构,具有很强的提取图像特征的能力,且网络参数量小,泛化能力强[4]。U-Net[5]网络作为卷积神经网络的一种,于2015年提出。其采用编解码器的思想,实现了图像端到端的自动分割,特别是针对少量的医学数据,仍具有较好的分割性能,故在医学图像分割领域得到了广泛应用。针对U-Net做出的各种改进工作也对医学图像处理技术的提升做出了很大的贡献。
肝癌是发生在人体肝脏部位的恶性肿瘤,对肝癌的早期诊断能够有效降低患者死亡率。而实现肝脏区域的精准分割以及对肝脏肿瘤数量和良恶性的判断能够辅助医生对肝脏疾病进行诊断和治疗[6]。传统分割方法需要耗费大量的人力和时间进行数据的手工标注,且肝脏与周围器官灰度相近,肝脏和肝脏肿瘤的形状和位置也因病人差异而不同,肝脏和肝脏肿瘤的分割面临很大的挑战[7]。故许多研究者针对肝脏肿瘤分割的相关工作进行了归纳综述,旨在分析肝脏研究工作发展现状,促进肝脏肿瘤分割领域的发展。如乐美琰等人[8]从非机器学习方法和机器学习方法两个角度对肝癌病灶自动分割的进展予以综述。马金林等人[9]则总结了全卷积网络、U-Net网络和生成对抗网络这三种深度学习分割方法在肝脏肿瘤CT图像分割工作中的应用。郭雯等人[10]对包括肝脏在内的多个器官自动分割研究进展工作进行了论述。
U-Net作为医学图像分割中的经典网络,虽然在很多医学图像的分割中表现良好,但原始的U-Net网络只针对二维图像进行分割,且在特征提取、分割精度、网络性能等诸多方面仍存在不足。对U-Net网络进行改进,从而实现肝脏和肝脏肿瘤的自动精准分割对临床诊断有着重要意义。因此,不同于上述综述,本文仅针对于U-Net结构改进及其在肝脏和肝脏肿瘤分割上的应用进行深入研究。本文介绍了肝脏及肝脏肿瘤分割常用数据集及评价指标,并归纳梳理了U-Net网络结构的相关改进工作,然后从单网络结构和多网络结构两个方面对U-Net网络在肝脏及肝脏肿瘤分割领域的应用进行了综述,最后讨论了U-Net及其相关改进网络应用于肝脏及肝脏肿瘤分割面临的挑战和未来的发展趋势。
1 常用数据集及评价指标
1.1 常用数据集
(1)LiTS数据集
LiTS数据集是肝脏肿瘤分割挑战赛(Liver Tumor Segmentation Challenge)所使用的公共数据集,也是目前肝脏及肝脏肿瘤分割研究中最为常用的数据集,由CodaLab组织提供。该数据集包括含有130例CT扫描的训练集和含有70例CT扫描的测试集。每次扫描包括的CT切片数量为42到1 026不等,轴向平面分辨率均为512×512像素,切片层间距为0.45 mm至6.0 mm之间。其中,训练数据集由世界各地的6个临床站点的4名放射科医生进行了手动标注,而测试集未进行标记。由于数据来源不唯一,使得数据集的重建层厚,同时切片的厚度、扫描图像的存储方向、图像质量和空间分辨率等存在显著差异,这在一定程度上增加了使用该数据集进行肝脏病灶分割的难度。但由于该数据集数量相对较多,图像质量相对较高,其仍然是目前最为广泛使用的肝脏和肝脏肿瘤分割数据集。
(2)3D-IRCADb数据集
用于算法数据库比较的三维图像重建数据集(3D image reconstruction for comparison of algorithm database,3D-IRCADb)作为公开数据集,在相关研究中也得到了广泛使用。该数据集提供了更为复杂的肝脏及其病变数据,包含匿名患者的医学图像和临床专家对感兴趣区域进行手动分割后的图像。其中,3D-IRCADb-01由10名女性和10名男性的增强CT扫描组成,其中75%的患者患有肝脏肿瘤;3D-IRCADb-02则包括2个匿名的胸腹增强3D CT扫描,如图1所示。该数据集的分辨率同样为512×512像素,但是数据集中有部分肝脏和肝脏肿瘤的对比度偏低,肝脏和肝脏肿瘤区域几乎重叠,这在一定程度上会影响模型训练,进而对分割结果产生影响。在使用时,需适当进行预处理。
图1 3D-IRCADb提供的模型Fig.1 Models offered by 3D IRCADb
(3)ISICDM 2019数据集
2019年图像计算与数字医学国际研讨会(the 2019 International Symposium on Image Computing and Digital Medicine,ISICDM2019)临床数据分析挑战赛提供的肝脏分割数据集为60例肝脏CT图像,数据集图像大小不同,但分辨率固定为1 mm×1 mm×5 mm,数据来源为江苏省人民医院。该数据集均为肝脏门静脉期CT增强图像,所有病例均人工勾画出了肝脏轮廓及病灶轮廓,以实现肝脏轮廓分割和肝脏病灶分割的目的。
(4)SLiver07数据集
The Segmentation of the Liver Competition 2007(SLIVER07)数据集来自MICCAI 2007的肝脏肿瘤分割挑战赛。该数据包括20例训练集数据和10例测试集数据。其中,训练集数据由专家进行了人工标注。该数据集轴向切片数量为64到502不等,切片层间距为0.7 mm至5.0 mm之间,切片分辨率为512×512像素。由于该数据集是2007年的公开数据集,近几年使用较少,在实际研究中,常将其与LiTS数据集合并,作为最常用的肝脏和肝脏肿瘤分割数据集。
(5)TCGA-LIHC数据集
肝癌基因组图谱(the cancer genome Atlas liver hepatocellular carcinoma,TCGA-LIHC)数据集是由美国国家癌症和肿瘤研究所以及国家人类基因研究所联合开展收集的,旨在通过提供与癌症基因组图谱(TCGA)中的受试者匹配的临床图像,将癌症表型与基因型联系起来。该数据集包括97名受试者的临床数据。数据类型包括CT图像、MR图像和正电子放射断层扫描成像(positron emission tomography,PT)。但由于该数据集是从世界各地许多地点进行收集的,故图像数据因为扫描仪模式、采集协议等存在异构。同时,由于该数据集采集目的更倾向于临床数据与基因组序列联系的研究,故未对肝脏和肝脏肿瘤边界进行标记,实际应用中需根据分割需求进行人工标记处理。该数据集相比于其他公开数据集在分割研究中应用较少。
(6)其他数据集
除了上述公开数据集外,还有一些研究者使用了一些内部数据或临床数据集,用于肝脏和肝脏肿瘤的分割研究。如Xu等人[11]在对肝脏肿瘤分割进行模型评估时,采用的是来自上海癌症中心的40名患者的数据集,肿瘤类型包括原发性和继发性两种。数据集包括术前T1 MR图像和术中未使用造影剂的CT图像。其中,MR图像的尺寸主要有320×260像素和256×256像素两种;CT图像尺寸大小均为512×512像素,厚度为3 mm,间距为0.732 mm×0.732 mm到0.920 mm×0.920 mm不等。Schlemper等人[12]则使用了CT-150数据集,该数据集由150名胃癌患者的腹部三维CT扫描图像组成,所有图像中胰腺、肝脏和脾脏的边界均由3名专业的研究人员进行了人工标注,并由一名临床医生手工验证,以确保数据集质量。各数据集基本情况,如表1所示。
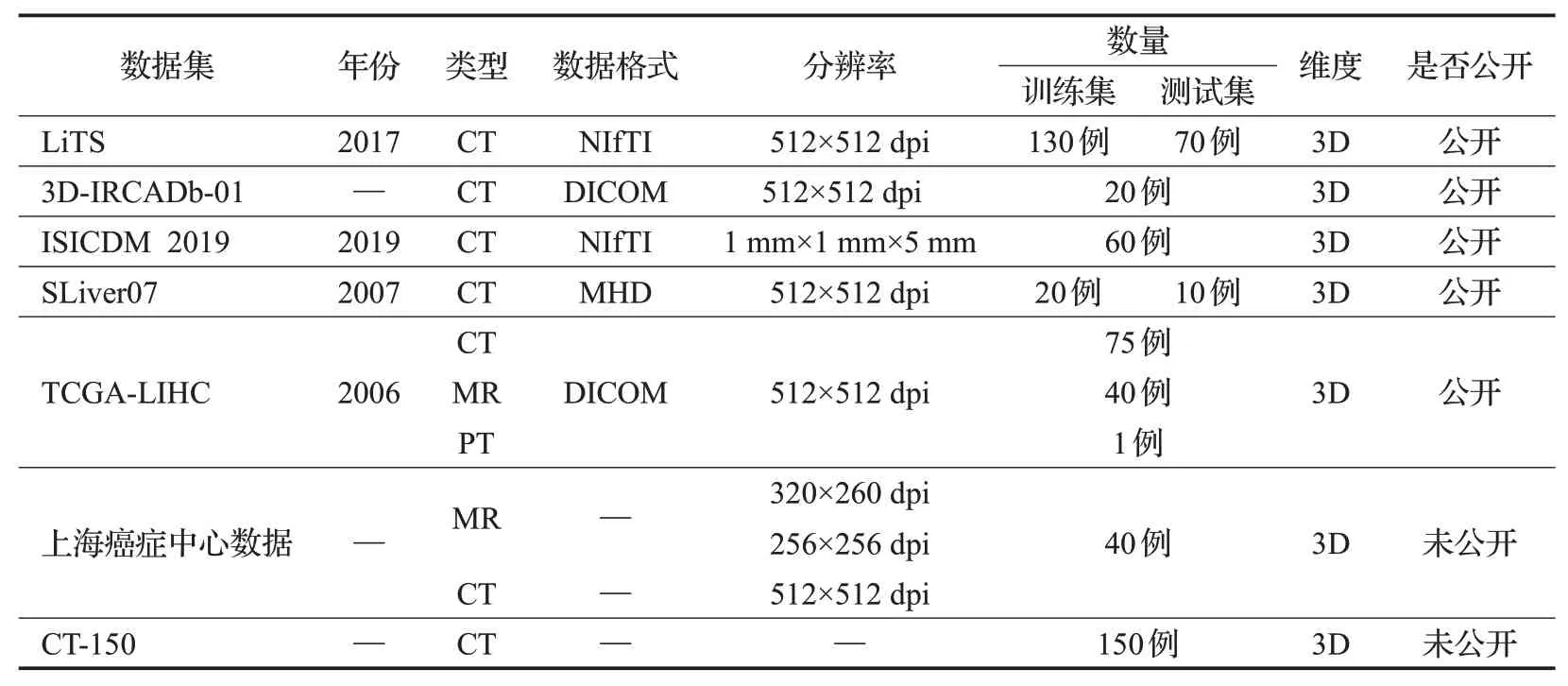
表1 肝脏及肝脏肿瘤分割常用数据集Table 1 Datasets used for segmentation of liver and liver tumors
1.2 评价指标
肝脏及肝脏肿瘤分割常用评价指标包括Dice系数、Jaccard相似系数、体积重叠误差(volume overlap error,VOE)、相对体积差异(relative volume difference,RVD)、平均对称表面距离(average symmetric surface distance,ASSD)、最大对称表面距离(maximum symmetric surface distance,MSSD)等[13]。若用A表示理论结果,用B表示实际分割结果,相关评价指标如下。
1.2.1 Dice系数
Dice系数是图像分割最常用的评价指标,它表示分割结果与标记之间的重叠相似度。取值范围在0~1之间,且越接近1,代表分割效果越好。具体计算公式为:

1.2.2 Jaccard相似系数
Jaccard相似系数同Dice系数类似,用于衡量分割结果与标记之间的相似性,取值范围在0~1之间。其值越大,代表样本的分割结果与标记之间的差异越小,分割效果越好。

1.2.3 VOE
VOE与Dice系数类似,是分割结果和标记的体积重叠误差,表示分割的错误率大小,取值范围在0~1之间。其值越大,代表分割效果越差。具体计算公式如下:

1.2.4 RVD
RVD用来表示分割结果与标记之间的体积相对差异,取值可正可负。具体计算公式如下:

1.2.5 ASSD
ASSD,又称ASD,它用于表示分割结果和标记对称位置的平均距离,以mm为单位。其值越接近0,说明分割效果越接近完美。具体公式如下:
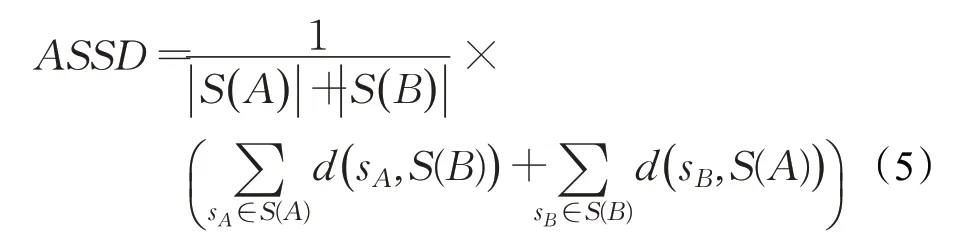
其中,S(A)表示A的表面体素集合,任意体素到S(A)的最短距离表示为:
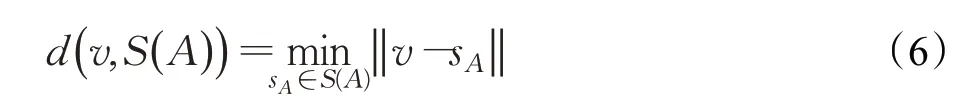
1.2.6 MSSD
MSSD,又称MSD,它用于表示分割结果和标记对称位置距离的最大值,同样以mm为单位。与ASSD类似,其值越接近0越表示分割效果接近完美。具体公式如下:
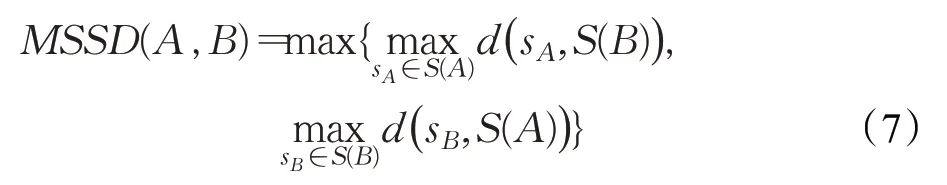
上述各项指标可大致归为两类。一类是基于体积差异进行衡量的指标,如Dice系数、Jaccard相似系数、VOE和RVD。其中,Dice系数和VOE相似,能够直观反映分割结果与标记之间的相似度,是最为常用的评价指标。而RVD所反映的差异仅仅是体积量的差异。RVD为0,并不意味着分割结果与标记完全重叠,故不单独用于分割效果的衡量。但是RVD与其他评价指标结合使用,不仅能反映体积信息,也能够体现出是否存在过分割或欠分割,这对于肝脏及肝脏肿瘤区域分割效果的判定十分重要。另一类是基于表面距离进行判断的指标,如ASSD和MSSD。ASSD也是最为常用的评价指标之一。由于MSSD对离群值很敏感,且能够反映出真实的最大值误差,在某些分割效果判定时,MSSD甚至比ASSD更重要。
针对于肝脏及肝脏肿瘤区域分割效果的评定而言,对体积误差的评估可能比距离测量更可取。但是,不同的评价指标强调了分割质量的不同方面。使用多种指标综合度量,能够传达更多的信息,更好地估计整体分割质量。故研究者常选择上述评价指标中的几种或全部,进行综合评价。
2 U-Net结构改进
U-Net网络模型于2015年由Ronneberger等人[5]提出,其网络最开始用于细胞壁的分割。由于其在医学图像分割领域,特别是针对小样本数据集的良好表现,逐渐被广泛应用于视网膜血管分割、脑肿瘤分割、肝脏肿瘤分割等各类医学图像分割中。U-Net网络模型与同时期提出的FCN网络[14]模型相比有诸多相似和不同之处。二者均使用了编码器和解码器的思想,但是U-Net相比于FCN而言,其网络结构更加完善。通过编码器进行特征提取后,再由解码器进行上采样,逐步恢复原始图像大小。同时,U-Net采用跳跃连接的方式,将底层特征与深层特征通过逐点相加的方式进行融合,充分利用上下文语义信息,提高网络分割性能。
随着医学图像处理领域的任务要求的提高,研究者们基于U-Net网络模型进行了诸多改进工作。通过结构的改进与完善,显著提升了网络的分割性能,加快了网络分割效率。根据U-Net模型的网络结构,对相关改进工作归纳如下。
2.1 编解码器改进
U-Net网络模型采用了编解码器的思想。编解码器均包含四个子模块,每个子模块内含有两个卷积层,并通过ReLU进行激活。下采样使用最大池化层,使得特征尺寸减半,通道数加倍,进行特征的提取;上采样通过反卷积操作,逐步恢复图像大小。原始U-Net的编解码器设计相对简单,故无法充分提取特征,进而影响了分割精度的提升。研究者在编解码器的基础上,进行了各种改进工作。针对编解码器部分的改进主要是引入残差模块、Dense模块和注意力模块等各种经典模块,或对子模块内部的普通卷积进行替换,使用可变形卷积、扩张卷积、循环卷积等,以充分提取特征,进而提高网络模型的分割性能。
2.1.1 引入新模块
(1)残差模块。早期神经网模型,如AlexNet、VGG、GoogLeNet等,主要是通过对网络层数的加深和参数优化来提高网络对特征的提取能力,进而提升网络性能。但是简单的层数加深操作除了会增加网络训练时间和参数量外,还易导致梯度消失和梯度爆炸。而ResNet网络中提出残差连接的概念,即将底层的输出通过残差路径输入到网络深层,由残差块进行残差的学习和拟合,促进训练过程中梯度的反向传播,从而有效解决了网络层数过深导致性能退化的问题,这种思想在后续网络改进中得到了很大应用。Milletari等人[15]提出的V-Net网络,首次将ResNet的短路连接思想应用于U-Net网络结构中。通过在编解码器的每个子模块内使用残差连接,从而确保短时间内收敛。Ibtehaz等人[16]提出的MultiResUNet网络中,为了缩小编码器和解码器之间的特征差异,则引入了1×1残差卷积模块,使得编码器和解码器中的两幅特征图更加同构。虽然残差连接的思想能够使得网络向更深层次发展,有效提高网络准确率,但是也会在一定程度上增加网络的训练时间。
(2)Dense模块。密集连接的思想来源于DenseNet,其将某层网络作为之后若干层网络的输入进行通道上的连接,实现了特征重用,在减少参数量和保证分割精度的同时,提高了模型效率;另外,相比于残差模块,Dense模块能够将多个层次的特征图进行连接,有助于充分结合上下文信息,进而能够处理更为复杂的图像数据。密集连接可以说是对残差连接思想的进一步升华,也为U-Net的相关改进提供了一些思路。如Zhang等人[17]在U-Net的基础上,提出了三种不同的多尺度密集连接,形成新的网络架构MDU-Net。该网络直接将相邻上下两层的不同大小的特征图进行融合,增强了特征在该层的传播;三种不同的密集连接的结合也减少了单一密集连接造成的过拟合。Guan等人[18]则将U-Net子模块替换为全密集连接模块(Dense Block),提出了Fully Dense UNet(FD-UNet)用于去除图像中的伪影。Dolz等人[19]提出的Dense Multi-path U-Net网络则应用Dense思想将不同模态所有卷积层进行密集连接,使得网络在任何层次下可以自由学习任何模式,从而提高了模型的学习能力。
虽然密集连接模块在保证分割精度的前提下降低了参数量和计算成本,并通过对特征的重用提高了模型效率,但这种特点也增加了网络的冗余度,在进行网络训练时会导致显存占用较高,在应用时需适当考虑显存优化问题。
(3)Inception模 块。Inception模 块 是 在 经 典 的GoogLeNet网络中首次提出的概念,通过3×3、5×5等不同尺寸大小的卷积核并行处理,能够进行多尺度特征提取,提高了网络对尺度的适应性;而借助1×1卷积能够有效减少特征通道数,进而在提取不同尺寸特征的同时,大大降低参数量。可以说,Inception模块的提出是将网络加宽的有效尝试。但是该模块也引入了一些无关信息,且并行多个卷积块会导致内存需求增加。因此,Ibtehaz等人[16]提出MultiResUNet网络,在U-Net网络中引入Inception模块,并加以改进。该网络使用了更小更轻量的3×3卷积块序列代替原有Inception模块中的5×5和7×7卷积,在保证多尺度特征提取的同时,又避免了并行多个卷积模块造成的较大内存需求的问题。
(4)Attention模块。Attention机制模仿了生物观察行为的过程,即通过快速扫描全局图像,获得需要重点关注的目标区域,进而对该区域投入更多注意力。Attention模块中的注意门控能够为特征图的元素赋予权重,使神经网络在进行学习时,有重点地进行关注,突出有用的显著性的特征,抑制无关特征,有效提升了网络分割效率,同时减少网络参数量。Oktay等人[20]提出的Attention U-Net是Attention机制在U-Net网络应用的典型。通过在编码器和解码器特征进行拼接之前插入Attention模块,使网络消除掉了跳跃连接过程中不相关信息和噪声,只合并相关内容。Li等人[21]则在具有密集跳跃连接的网络结构的基础上,提出了基于注意力的嵌套分割网络ANU-Net。该网络在嵌套的卷积块之间引入了注意力机制,从而在进行不同层次特征提取的时候增加目标区域的权值,同时抑制与分割任务无关的区域,使得网络具有更好的分割性能。
由此可以看出,Attention机制能够灵活捕捉全局信息和局部信息之间的联系,提高模型训练效率,减少训练时间。但是这种有侧重点的学习方式有可能破坏深层网络的特征信息,影响模型学习效果[22]。
(5)多模块结合。由于不同模块具有其自身的特点,在实际应用中,许多研究者往往综合考虑各模块优缺点,适当选择一种或几种模块,对编解码器进行合理改进,以实现最佳的网络性能。如Song等人[23]在U-Net的基础上提出了BSU-Net,该网络在编码器中同时引入了Dense模块、Inception模块和扩张卷积,用于避免梯度消失,并扩大感受野,提高分割精度。Zhang等人[24]提出的DENSE-Inception U-Net同样将残差模块、Inception模块和密集连接三者的思想进行了结合。通过Inception-Res模块替换标准卷积层来增加网络的宽度,设计Density-Inception模块来提取特征,在不增加其他参数的情况下,使网络能够更深层。实验结果也进一步证实了各模块的有效性。
2.1.2 引入新卷积
(1)可变形卷积。传统卷积块是固定不可变的,对未知变化适应性差,这在一定程度上导致网络泛化能力不强。而可变形卷积则在卷积核的每一个元素上额外增加了一个方向参数,使得卷积能够根据当前识别的图像内容自适应地学习并调整感受野范围,从而适应不同物体在形状、大小等方面的几何形变。受可变形卷积的启发,Jin等人[25]提出了名为Deformable U-Net(DU-Net)的网络,在编码器和解码器中使用可变形卷积块代替原有的卷积块用于捕获几何变化,从而使得网络能够根据血管的尺度和形状变化自适应地调整感受野大小,进而捕获各种形状和尺度的视网膜血管。但是由于可变形卷积引入了方向参数,需要对不连续的位置变量进行求导,这会在一定程度上增加网络模型的计算量。
(2)扩张卷积。原始FCN、U-Net等编解码器结构一般是在编码器部分通过先卷积再池化的操作来扩大感受野,以捕获更多特征信息。但是下采样的过程降低了图像分辨率,这会导致一些信息的丢失,影响分割结果。为了解决这个问题,扩张卷积被提出。扩张卷积又称为空洞卷积或膨胀卷积,是指在标准卷积核参数保持不变的同时,在相邻元素间注入空洞,扩大卷积核的尺寸,从而在不损失特征图尺寸的情况下增加了感受野范围。Chen等人[26]提出的三维扩张型多纤维网络(DMFNet)中,借鉴了扩张卷积的思想。其利用softmax约束并行扩张型卷积分支的权值,用于获得多尺度图像表示,从而提高了脑肿瘤的分割精度。根据扩张卷积特点可以看出,当网络需要较大感受野,而计算资源有限难以提高卷积核数量和大小时,能够考虑使用。但是扩张卷积的膨胀率设置不合理,也会导致信息丢失,影响分割精度,在应用时需注意合理设置膨胀率参数。
(3)循环卷积。循环卷积是周期卷积的一种,能够对具有相关性的序列数据进行处理,在UNet结构改进中也得到了应用。受残差模块、RCNN、U-Net的启发,Alom等人[27]在U-Net的基础上提出了循环卷积网络(RU-Net)和循环残差卷积结构(R2U-Net)。循环卷积和残差操作使得网络在不增加参数量的同时,提升了分割性能。虽然循环卷积能够很好地捕捉序列数据的语义信息,增强了网络模型对特征长期依赖的捕获能力,但是这种思想更多是在基于RNN的医学影像算法中使用,且对于单一切片的分割任务优势并不明显。
(4)协调引导卷积。经典卷积层之间的信息传递通常局限在某层的接受域内,这限制了经典卷积层表示全局位置信息的能力。而协调引导卷积是对传统卷积的扩展,通过添加额外的坐标通道来集成位置信息,弥补传统卷积的不足。Wang等人[28]在对肺部区域进行自动分割时,在解码器的最后一个转换中对传统卷积分别添加了三个额外通道表示输入的3D图像的x、y、z坐标,进而扩展为协调引导卷积,用于生成肺叶位置信息的附加特征图,有效减少了不同肺叶的误分类,提高了分割精度。但由于协调引导卷积增加了额外的参数量,会适当影响模型训练效率。
针对编解码器改进方法总结如表2所示。
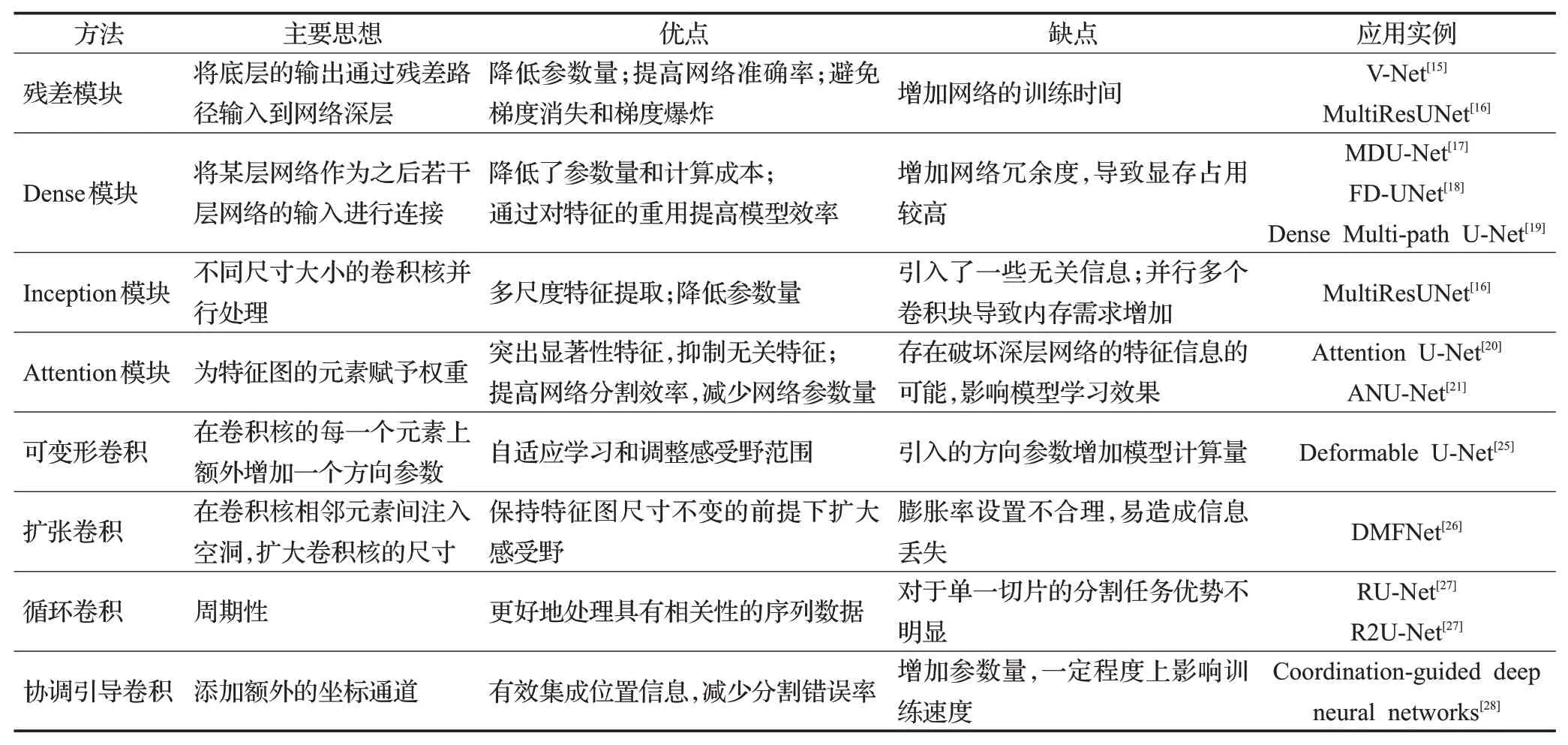
表2 U-Net编解码器改进方法小结Table 2 Summary of improved methods for U-Net encoding-decoding
2.2 跳跃连接改进
U-Net模型中,经过反卷积之后的输出结果将与下采样中对应子模块的输出结果进行逐点相加,然后再送入上采样子模块进行卷积。这种跳跃连接的方式,将浅层特征与深层特征进行了融合。但是由于特征层级不同,相关信息特征在融合时容易丢失。针对该问题,许多研究者尝试对U-Net结构中的跳跃连接结构进行了改进,以促进特征融合,尽量保留信息。针对跳连部分的改进主要有加法融合、密集跳跃连接、全尺度跳跃连接、加入attention机制等。
(1)加法融合。原始的U-Net网络中,通过跳跃连接将编码器和解码器的特征进行拼接融合,而在之后的网络改进中,有些研究者尝试将其以加法的形式进行跳连,也取得了很好的效果。如在Chen等人[29]提出的Bridged U-Net、Zhuang等人[30]提出的LadderNet网络中,均采用加法跳连,将两个分支的特征直接相加,从而有利于解决参数量过多的问题。
(2)密集跳跃连接。原始U-Net是将编码器中的浅层输出结果通过跳跃连接直接与解码器中的深层网络输出进行连接,这种将不同语义特征直接、单一进行融合的方式,会在一定程度上降低分割效果。为了满足医学图像处理对精度的高要求,Zhou等人[31]提出了UNet++网络结构。该网络主要在跳连部分增加了一系列嵌套的密集跳跃连接,用于增加梯度流,缩小语义差异,有效提高了分割精度。但是由于UNet++没有从全尺度充分挖掘信息,不能非常明确地标明器官位置和边界,为了弥补不足,Huang等人[32]进一步提出了UNet3+网络。在该网络中使用全尺度密集跳跃连接,使得网络能够充分利用多尺度特征,同时含有较少的参数。
(3)加入Attention机制。注意力机制除了在编解码器部分使用外,还可用于跳跃连接部分。基础的Attention可以对特征图元素赋予权重,以突出显著性特征。而Fu等人[33]则在此基础上,提出了包含位置注意模块(PA)和通道注意模块(CA)的双注意网络用于场景分割。位置注意模块能够根据所有位置特征的加权和有选择性地聚合每个位置的特征,因此无论距离多远,相似的特征都能够被关联起来。而通道注意模块则从通道角度对关联特征进行整合,从而选择性地强调相互依赖的特征图。两种注意模块的结合,能够有效提升分割精度。受双注意机制的启发,谷鹏辉等人[34]提出AtGBU-Net用于视网膜血管分割。为了使上采样获取的特征图包含更多的语义信息,该网络在进行反卷积操作后加入了PA;同时在进行跳跃连接前加入了CA,使得对应编码器的特征图包含更多的空间信息。
根据上述跳跃连接的改进工作能够明确,密集跳跃连接能够将不同层级的特征进行充分的融合,缩小了语义差异,能够在一定程度上提高分割性能,但是要以计算成本为代价。而加法融合的思想相比于原始U-Net的拼接融合,大大减少了参数量,但由于这种改进方式过于单一,其对网络性能提升所发挥的作用有限。而Attention机制能够通过对信息的选择性关注,在提高网络分割性能的同时,能够提高网络效率,减小计算量,在网络结构改进中发挥了重要作用。另外,除了基本的Attention机制外,研究者还可从通道注意、位置注意、多注意模块结合等角度对网络结构进一步优化,以实现更佳的分割效果。
2.3 整体结构改进
还有一些研究者在U-Net网络结构的基础上,保留编解码器的思想,根据需要进行了U-Net结构的堆叠或整体结构的变形。
(1)堆叠。堆叠即对U-Net的重复利用,使用两个甚至多个U-Net网络进行串联,分步完成由粗到细的分割,能够有效提升分割效果。如Chen等人[29]提出的Bridged U-Net采用桥接的方式将两个Unet结构进行连接,使得网络能够在多个层次上充分利用不同的特征,从而降低训练成本,加快网络收敛。Jha等人[35]提出的DoubleU-Net结构,在两个U-Net结构堆叠的基础上,还引入了空间金字塔池(ASPP)用于捕获网络内的上下文信息,以及SE模块用于减少冗余信息。虽然U-Net的堆叠改进能够有效提升分割精度,但这种操作实质上存在特征重复训练的弊端,从而影响了分割效率。因此,相关改进工作还需考虑如何在保证分割精度的同时,减少网络冗余。
(2)变形。U型结构主要包括编码和解码的过程,但有时候无法满足实际应用需求,故有些研究者对整体结构进行修改,如Valloli等人[36]在U-Net编解码器的基础上设计了形似W的网络结构W-Net。该网络通过编码器进行多尺度特征提取后,经过由密度地图(DME)分支和增强分支组成的解码器进行解码输出,生成密度地图,用于人群计数。Fu等人[37]在U-Net网络结构的基础上,增加了多尺度输入层(multi-scale input layer)和侧边输出层(side-output layer),构成M-Net模型架构,能够很好地在单阶段多标签系统中同时解决视盘和视杯的分割问题。变形操作能够根据应用需求有针对性地优化网络结构,但是这种改进是对整体结构的设计,存在一定难度。
2.4 U-Net结构改进小结
基于U-Net结构进行的改进和研究总结如表3所示。可以看出,围绕U-Net进行的结构改进在视网膜分割、脑肿瘤分割、皮肤癌病灶区域分割等各类医学图像领域中都得到了应用。通过引入经典模块和经典卷积,或对跳跃连接和整体结构进行完善,能够加强网络特征提取能力,大大提高网络性能。相关研究工作对U-Net在肝脏和肝脏肿瘤分割领域的改进和应用起到了一定的启发和促进作用。
3 U-Net在肝脏和肝脏肿瘤分割中的应用
实现肝脏及肝脏肿瘤区域的自动分割具有重要的临床价值,也是进行肝肿瘤计算机辅助诊断的基础,但是目前肝脏及肝脏肿瘤的自动精准分割仍面临很大的挑战。首先,在CT影像中肝脏与邻近脏器的灰度值相似性很高,甚至存在较大连接区域,难以区分。其次,肝脏肿瘤区域形状易受外界影响,由于患者不同、设备参数不同等原因,肿瘤区域在形状、大小、数量等方面存在诸多差异。相比于肝脏分割,肝脏肿瘤区域的自动分割更具挑战性。另外,目前肝脏和肝脏肿瘤分割数据集过少,且以3D数据居多,一定程度上会影响分割精度,增加计算成本。因此,将肝脏区域与周围器官更好地区分,对肿瘤区域边缘细化,实现精准分割,同时提高分割效率,控制计算成本等是目前肝脏及肝脏肿瘤区域分割任务中的主要需求。
传统的分割方法如区域生长、强度阈值处理等依赖于人工设计手动提取特征,特征表示能力有限且泛化性能较差,难以满足目前的分割要求。基于深度学习的全卷积神经网络FCN虽然在语义分割领域得到广泛关注和应用,但其更适用于样本较多的数据。而U-Net网络作为医学图像分割中的经典网络,在针对少量样本的医学数据集时,也能够具有良好的表现。因此,很多研究者选择在该网络及其改进网络的基础上继续改进,用于实现肝脏和肝脏肿瘤的分割。
针对上述应用需求,研究者在对U-Net网络改进时,主要从以下几个方面进行探索。首先是考虑加强网络对特征的提取和信息的利用以提高肝脏和肝脏肿瘤区域的分割精度,通过引入上述经典网络模块、卷积等对网络结构改进能够有效实现。其次,为了实现肝脏肿瘤区域的精准分割,研究者尝试粗细结合,分步骤完成分割任务,这种思路的实现能够通过采取级联思想或多网络结合的方式完成。能够充分利用三维信息而又控制计算成本,是研究者针对肝脏和肝脏肿瘤区域分割的重点任务。在实际改进工作中,可通过引入注意力机制、2.5D网络或轻量级网络等方式来减轻参数量,降低计算负担,进而在保证分割精度的前提下提高网络性能和效率,解决3D分割带来的计算成本增加问题。根据研究者们基于U-Net所提出的新的网络结构,本文从单网络结构和多网络结构两大方面来对相关工作进行总结。
3.1 单网络结构改进
单网络结构改进是指在单一U-Net网络模型的基础上进行的改进,这是一种相对简单和常用的改进方式。针对肝脏和肝脏肿瘤区域的分割工作,很大一部分改进工作是在U-Net结构的基础上,引入了常用的Dense模块、残差模块等,以提高网络分割性能。如Song等人[23]提出的bottleneck supervised(BS)U-Net用于肝脏和肿瘤的分割。该网络引入了常见的Dense模块、Inception模块和扩张卷积构成BaseU-Net,并设计了encoding U-Net和segmentation U-Net两个部分共同组成BSU-Net以实现自动编码。与传统U-Net相比,该网络结构能够有效减少假阳性和假阴性,控制形状失真。Xu等人[11]则在UNet++的基础上进行了改进,通过在卷积模块中增加残差结构,解决梯度消失问题;同时,使用二值交叉熵和Dice系数的组合作为损失函数,稳定收敛。实验结果表明,该模型优于U-Net和U-Net++,对快速、准确地进行配准以协助医师在CT影像指导下进行多模式肝恶性肿瘤消融手术有重要的意义。
大部分医学图像都是3D图像,如果仅使用二维卷积进行分割,无法充分利用三维空间信息,这对分割精度必然会产生一定的影响。特别是对于肝脏和肝脏肿瘤区域来说,分割的目标区域与其他区域差异较小,分割难度较大,更加需要特征的充分提取和信息的利用。故肝脏和肝脏肿瘤的分割任务,大部分是利用三维卷积进行3D分割。V-Net[15]和3D U-Net[38]的提出,使得U-Net在3D分割任务上的有效性得到了验证,也为后续进一步改进奠定了基础。如孙明建等人[39]为了解决肝脏区域精准分割的问题,提出了一种新型全卷积网络3D Unet-C2-CRF,如图2所示。该网络使用三维卷积,充分利用了肝脏CT图像的空间信息,通过级联的方式,将浅层特征与深层特征结合,同时增加网络层数以提取更深层次的特征。在初步分割后,使用三维条件随机场3D CRF解决了肝脏边界分割精准度低的问题,并有效降低肝脏分割的假阳性率。与V-Net和3D U-Net模型相比,该网络具有更好的特征表达能力和更强的泛化性能。
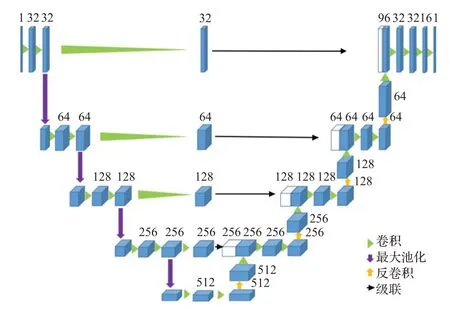
图2 3DUnet-C2-CRF网络体系结构示意图Fig.2 Schematic representation of 3DUnet-C2-CRF architecture
虽然三维卷积能够结合层间信息,但是也带来了很高的计算成本,对计算机性能提出了更高的要求。为了解决该问题,2.5D网络[40]的概念被提出。2.5D网络是二维卷积与三维卷积的结合,故2.5D既能够充分利用层内信息和层间信息,又避免了较高的计算成本问题。Han[41]受V-Net模型改进的启发,结合了U-Net模型远程跳跃连接和ResNet中短距离残差连接的思想,提出了DCNN。与V-Net在3D分割不同的是,该网络设计应用在了2.5D上。该网络既能够促进浅层高分辨率信息与深层语义信息的融合,又能够加速网络收敛,进而提高网络性能。Han等人[42]提出的垂直2.5D U-Net,将3个2.5D Res-UNets进行融合用于肝脏和肝脏肿瘤的分割,如图3所示。实验结果表明,该网络比2D U-Net的分割精度更高,同时又比3D U-Net模型尺寸更小,GPU内存需求更少,能够更广泛地部署在低配置的设备上。
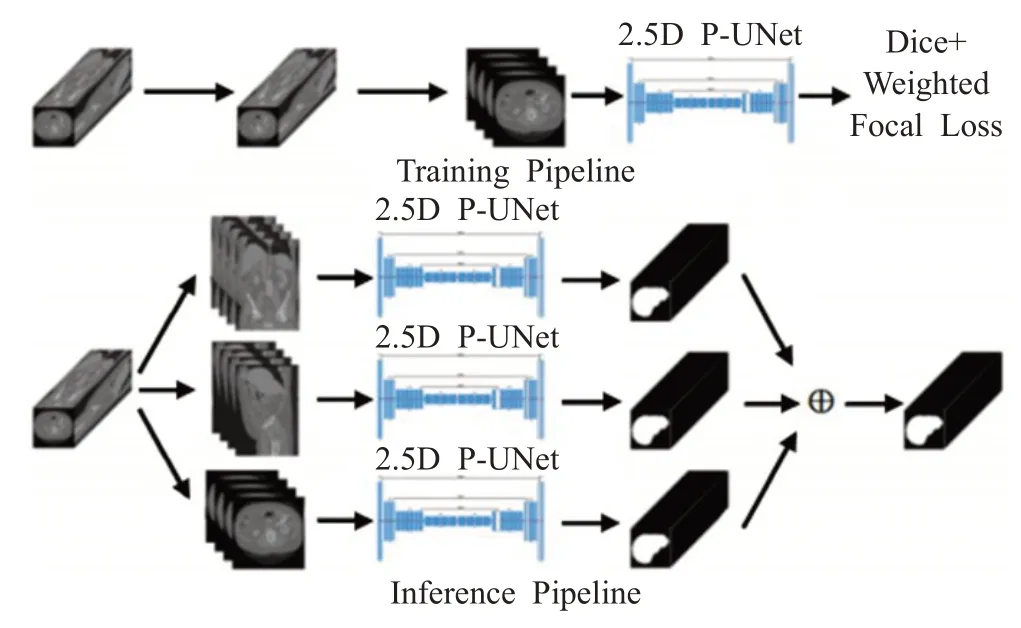
图3 2.5D U-Net结构示意图Fig.3 Schematic representation of 2.5D U-Net
针对3D分割带来的计算成本问题,除了使用2.5D分割外,还可以通过引入注意力机制,从而加强对特征的有效提取,减少无效特征带来的计算负担。Jin等人[43]提出的名为RA-UNet的三维混合残差注意力感知分割网络,是首次使用注意力残差机制(attention residual mechanism)来处理医学体积图像的网络。该网络在跳连部分增加了attention模块使得网络能够将注意力放在感兴趣的位置;同时为了避免单纯叠加注意力模块造成网络性能下降,引入残差机制,形成注意力残差模块。该网络在肝脏区域分割和肝脏肿瘤提取方面均具有良好的表现。为了进一步提高特征提取的有效性,Schlemper等人[12]提出了attention gated networks,网络结构如图4所示。该AG模型能够自动学习不同大小的目标区域,同时能够抑制无关特征,突出有效特征,从而在保证模型训练效率的基础上,减小计算开销,提高分割精度。同时,AG能够较为容易地集成到U-Net网络等标准CNN结构中,该网络模型能够很好地实现对胰腺、肝脏和脾脏区域的分割。
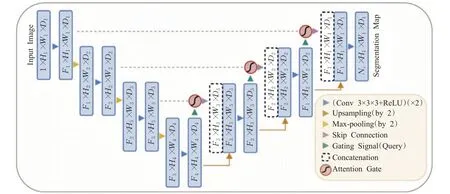
图4 attention gated networks结构示意图Fig.4 Schematic representation of attention gated networks
无论是2.5D网络还是3D网络,网络结构都具有较大的参数量,这在一定程度上使得计算成本和GPU内存过高。随着深度学习的发展,轻量级网络逐渐被应用。Lei等人[44]在V-Net的基础上提出了轻量级V-Net(LV-Net)用于肝脏的分割。该模型通过引入一个反向剩余瓶颈块(IRB块)和一个三维平均池化模块,显著减少了参数量。同时在训练阶段采用了3D深度监督来改进最终的损失函数。实验结果表明,LV-Net的可训练参数仅为普通V-Net的2.55%,但其仍具备更强的区分肝区和非肝区的能力。
3.2 多网络结构改进
对单一的U-Net网络结构改进是现有分割算法普遍使用的思路,通过加入经典网络模块等操作能够很好地实现对肝脏和肝脏肿瘤的分割。但是对于一些分割精度要求很高的任务,比如对肝脏肿瘤边缘区域的精细分割来说,由于肝脏肿瘤体积较小,形态差异较大,且边缘模糊,仅通过单阶段的U-Net网络结构无法对边界进行精准分割[45]。故研究者们尝试使用多网络结构进行改进。较为常见的方法是级联网络或将U-Net网络与其他网络结合,分步骤完成分割任务。
级联网络的基本思想是将前一个网络的输出作为后一个网络的输入,实现对特征的进一步提取和利用,以实现精准分割。全卷积网络FCN与U-Net类似,均采用编码器和解码器的结构。针对于肝脏和肝脏肿瘤区域的精准分割,有部分研究者选择使用FCN网络进行级联。如Christ等人[46]通过第一个FCN分割出肝脏,作为感兴趣区域(ROI),然后将结果输入第二个FCN分割出肝脏病变区域,最后使用3D条件随机场(conditional random fields,CRFs)进一步细化分割结果。Vorontsov等人[47]同样将两个全卷积网络串联,实现了端到端的训练。
虽然级联FCN能够在一定程度上实现精准分割,但是医学图像数据的显著特点是数据量小,而U-Net能够很好地在少量数据集下进行分割。因此,很多研究者选择级联U-Net网络。比如Isensee等人[48]提出的nnU-Net中使用到了一个2D U-Net、一个3D U-Net以及级联了两个3D U-Net的网络模型。nnU-Net打破了以往对U-Net网络模型的各种复杂改进,充分考虑非结构因素对网络性能的影响,使得网络能够自适应任何给定的医学图像数据集。刘云鹏等人[49]则将深度学习与医学影像组学结合,采用级联的U-Net网络结构对肝脏和肝脏肿瘤实现同时分割,并在U-Net结构中引入了注意力模块、密集连接和子像素卷积,使得网络能够更好地获取全局信息和局部特征,提升对微小肿瘤区域的分割精度。Xu等人[50]提出的多尺度DC-CUNets是一种基于双通道的级联U-Net网络结构,通过双通道分别提取肝脏和肝脏肿瘤动脉期和静脉期的特征,然后通过级联的方式进行特征融合,从而提高了网络的分割精度。
除了级联U-Net网络的方式,还有研究者选择将U-Net网络或其改进网络与其他经典网络结合,以充分发挥不同网络结构的优势。黄泳嘉等人[51]针对肝部区域图像处理存在的诸多不足,采用初始模块和优化模块的方式进行分割。初始模块为类似于U-Net的编解码器结构,其在上采样中增加池化层复制对应下采样层的特征图,以减小信息损失。优化模块则引入残差连接来学习初始分割图和专家标注图像之间的残差,优化分割边界。同时,该模型采用组归一化方法减小信息损失,并使用混合损失函数提高分割边界的清晰度和分割精度。该网络结构证明了混合损失函数和残差模块结合对U-Net网络性能提升的有效性。
肝脏及肝脏肿瘤分割所面临的2D分割无法利用三维信息,而3D分割计算成本过高的问题,也可以通过2D与3D网络组合的方式加以解决。常见的组合为2D网络粗略分割,3D网络聚合信息,完成精细分割。如Li等人[52]提出了一种混合密集的网络结构,即H-DenseUNet。该网络先通过二维DenseUNet充分提取特征信息,然后利用一个3D网络分层聚合体积上下文,从而实现三维卷积的效果。该方法在LiTS2017数据集和3D-IRCADb数据集上均表现出了这种新模式的优越性。Zhang等人[53]在对肝脏及肝脏肿瘤分割时,先使用2D的U-Net网络对肝脏区域粗略定位,减少非肝脏区域的干扰;然后通过3D的FCN网络对肝脏区域进行精细分割,并对肝脏肿瘤进行粗略的定位,以提高分割精度;最后通过提出的新的水平集方法完成对肝脏肿瘤区域的精细分割,网络结构如图5所示。2D U-Net与3D FCN的结合实现了肝脏和肝脏肿瘤区域由粗到细的逐步分割,既保证了分割效率,也提高了分割精度。

图5 用于肝脏和肝肿瘤分割的粗-细通道示意图Fig.5 Coarse-to-fine pipeline for liver and liver tumor segmentation
3.3 小结
由上述内容可知,由于医学图像大部分为3D图像,故单网络结构与多网络结构的很多改进工作都在围绕如何充分利用三维空间信息,而又适当减少3D分割带来的高昂的计算成本问题进行。单网络结构下,可以通过引入2.5D卷积,减少3D卷积的使用,或使用注意力机制,提高特征提取的有效性,减少无效特征对计算量的负担加以解决。多网络结构下,则可以通过2D网络粗分割与3D网络细分割相结合的方式,分步提取特征。各方法在肝脏及肝脏肿瘤区域分割的评价指标结果如表4、表5所示。
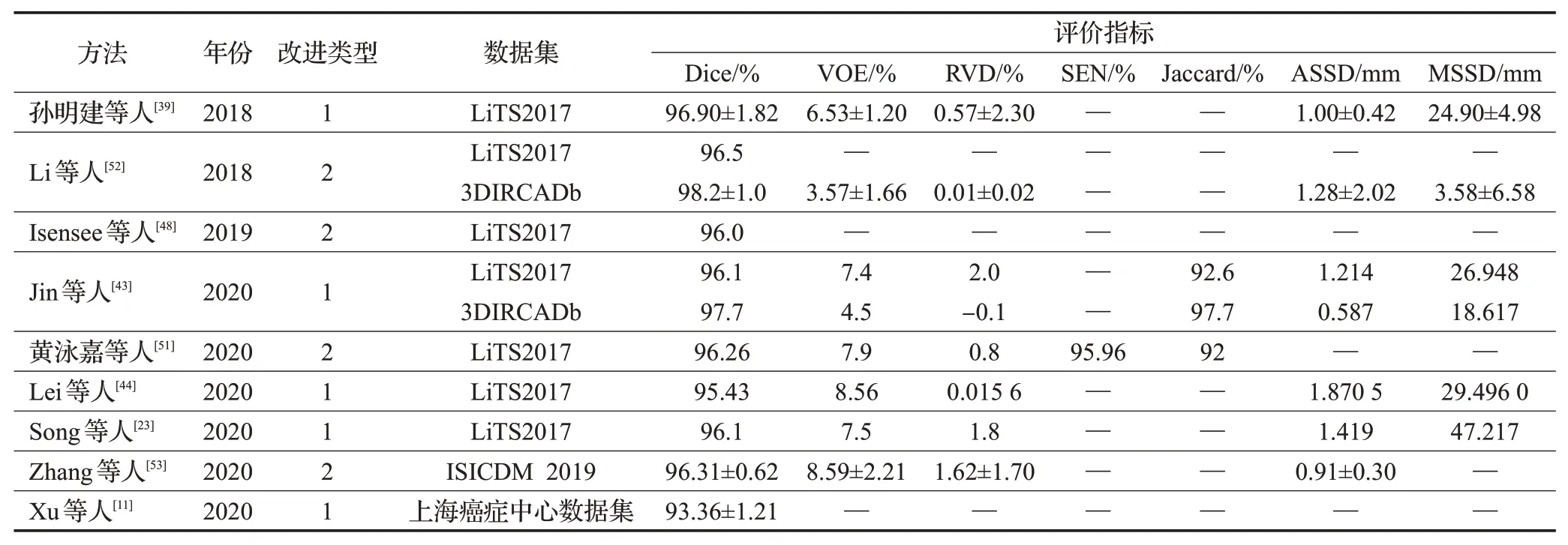
表4 肝脏分割的评价指标结果Table 4 Evaluation index results of liver segmentation
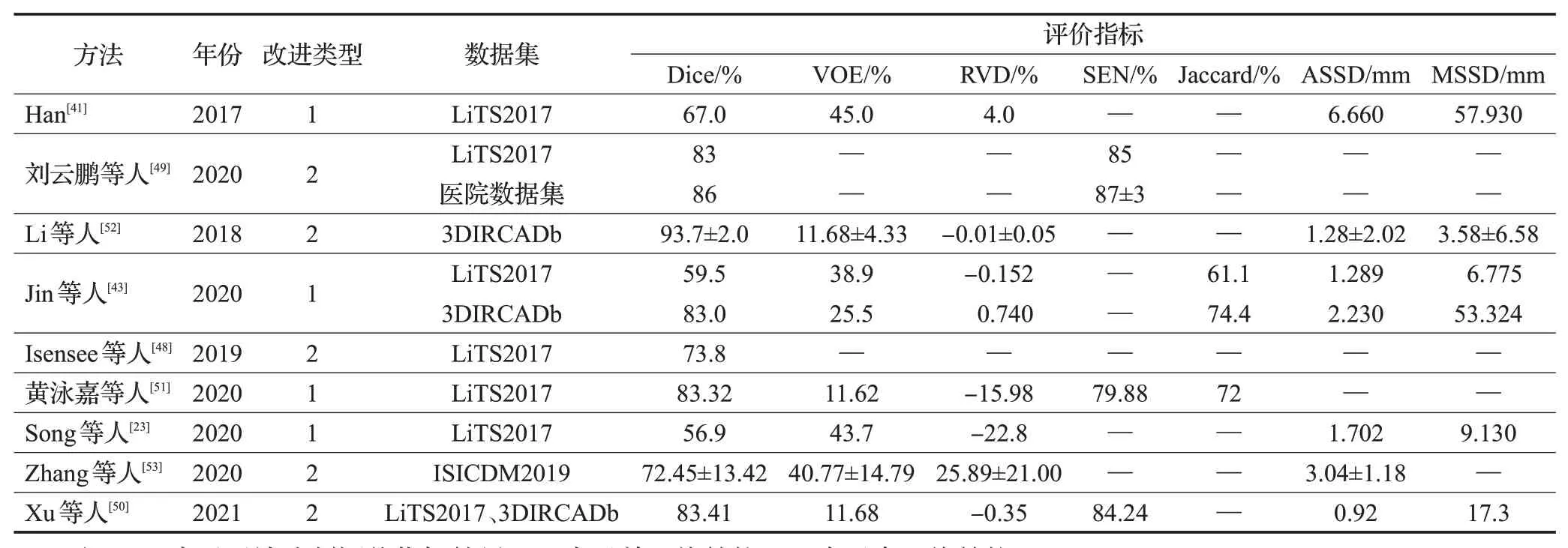
表5 肝脏肿瘤分割的评价指标结果Table 5 Evaluation index results of liver tumor segmentation
实验结果表明了各种改进方法的有效性。单网络结构通过引入残差模块、密集连接模块等经典模块提高网络的特征提取能力,进而提高分割精度。对单一U-Net网络结构的改进方式在对肝脏区域和较大的肝脏肿瘤区域分割时,效果改善明显,但是对细小肝脏肿瘤分割及肿瘤边缘区域的细化时,易造成信息的丢失,导致分割效果不佳。相比于单网络结构,多网络结构对小目标分割更具优势。通过级联能够将分割任务细化,充分利用特征信息,由粗到细地完成分割。而与其他经典网络结合,则可以结合不同网络模型的优势,提升分割效果。但是多网络结构增加了计算成本和存储开销,网络设计难度也较大,相关改进还需在网络性能和效率等方面加以探索。另外,从整体来看,大部分肝脏肿瘤区域分割的各项平均指标低于肝脏区域分割结果,这也从侧面印证了肝脏肿瘤区域分割的难度。因此,如何进一步提高分割效果,特别是实现肝脏肿瘤区域的精准分割,仍需要进一步研究。相信随着网络结构的不断完善,肝脏和肝脏肿瘤区域的分割能够更加精准高效,将为临床辅助诊断肝脏病变做出更多的贡献。
4 总结与展望
本文对U-Net结构改进及其在肝脏和肝脏肿瘤分割的应用进行了研究总结。通过对相关工作的分析可知,U-Net常见的结构改进方式为引入残差模块、注意力模块等经典模块或循环卷积、扩张卷积等。通过对跳跃连接的改进或整体结构的改进也能够有效提升网络性能,这些方法在对肝脏和肝脏肿瘤分割时得到了应用。由于肝脏和肝脏肿瘤图像的特殊性,在进行相关研究时,主要以3D分割为主。通过引入2.5D卷积或注意力机制,能够有效减少网络计算成本,提高分割效率。为了实现精细分割,研究者们常通过级联U-Net,或使用2D网络与3D网络结合的方式,逐步完成粗分割和细分割,细化分割边缘,提高分割精确度。
虽然相关改进工作在一定程度上提升了肝脏和肝脏肿瘤区域的分割效果,但仍存在一些不足和难点,具体如下:
(1)缺乏对非结构内容的改进和思考。网络结构的改进能够最直观地改善网络性能,也是最为常用的改进方式。但是在模型训练的过程中,选择合适的数据预处理方法,改进损失函数,或对分割结果进行适当的后处理等,均能在一定程度上对网络分割效果产生影响。如增加BN层可以减轻网络的过拟合现象,提高网络泛化能力而又不影响准确率;若增加Dropout层,则可以降低参数量和模型复杂度以缓解过拟合,使模型更具鲁棒性等[54]。因此,对非结构的改进工作不容忽视。
(2)目前,数据量少仍然是医学图像处理所面临的一大难点。针对于肝脏及肝脏肿瘤区域而言,其形状、大小不一,且与周围器官相近,往往需要专业医生进行标注。同时,由于设备参数不同,其CT图像灰度值也存在差异,进一步影响了数据集的质量和规模。若医学图像数据过少,而网络模型参数过多,易造成过拟合,影响网络性能。虽然U-Net网络针对小样本数据集具有良好的表现,但是对于肝脏肿瘤的精细分割以及较复杂结构的分割而言,网络性能会一定程度下降。因此,少样本训练问题亟待解决。
(3)分割精度与网络计算成本的平衡关系仍需探索。由上述改进工作可知,引入残差模块、Dense模块、可变形卷积等操作,虽能有效提升网络性能,但是也会在一定程度上增加计算量。另外,为了充分利用特征信息,肝脏及肝脏肿瘤区域往往需要3D分割,这也大大增加了计算成本。因此,如何在提高分割精度的同时,尽可能减少计算量的增加,值得继续探索。
根据对现有工作的归纳梳理,以及现阶段存在问题的探讨,本文认为未来肝脏及肝脏肿瘤区域的分割工作可以围绕以下几点展开:
(1)充分考虑结构与非结构因素对模型分割性能的影响,既要追求网络结构的创新改善,又要考虑非结构因素的改进。比如在对数据预处理时,进行数据扩增,避免样本过少导致的过拟合现象。常用的数据增强方法包括翻转、裁减等几何变换方法或灰度值增强等颜色变换方法。随机翻转通过将图像镜像翻转可直接将数据量扩大一倍,随机裁减可将数据量更大幅度提升。在对方向不敏感的任务比如分类任务中随机裁剪最为常用[55],经典的深度学习网络LeNet、AlexNet以及VGG等在训练中均有所涉及。针对曝光过度或不足的X光图像可以使用直方图均衡化的方法重新分布图像像素值,以增加局部对比度;针对对比度偏低而整体亮度偏高的情况,可选择伽马校正的方法以增强高灰度或低灰度细节,进而增加对比度。在实际应用中应考虑多种数据增强方法的结合,以实现最佳的数据增强效果,进而提高分割精度。
非结构因素还包括训练时使用的损失函数和后处理方法等。通过对损失函数进行合理改进,或采取一定的策略将多种经典损失函数组合,能够发挥不同损失函数的优势。比如将常用损失函数Dice和Cross-entropy结合,既能够保证背景被正确分割,提高分割精度,又能够使模型加快收敛[56];在后处理阶段可考虑使用CRF条件随机场等方法,进一步优化分割边缘,提高分割精度。
(2)针对数据量过小的问题,一方面,要医工结合,加强研究者与专业医生的合作交流,进一步搜集相关数据,提高数据集的质量。针对现有数据集,考虑使用翻转、裁减等数据增强方法扩大数据量。同时,完善网络结构,提高网络模型对小规模数据集的训练效果。另一方面,积极探索半监督学习、无监督学习与传统监督学习方式的结合。比如通过无监督学习中的生成对抗网络GAN进行训练,生成高质量的肝脏肿瘤图像,从而避免训练样本不平衡的问题,提高分割效果。
(3)加强对轻量级网络、2.5D网络的探索。为减小计算成本,同时能够有效改善网络性能,可加强对轻量级网络的研究,以减小网络参数量。同时,2.5D卷积概念的提出以及2.5D网络的应用也给以启示。针对三维肝脏及肝脏肿瘤图像分割工作,将2D分割与3D分割结合,能够有效改善分割效果,而又避免参数量的大幅增加。未来可在轻量级网络和2.5网络的应用上进一步探索。
(4)可以加强对多任务学习的研究。除了进一步加强对肝脏和肝脏肿瘤区域自动精准分割外,未来还可以将多种深度神经网络结合,在完成肝脏及肝脏肿瘤分割的同时,能够对肝脏肿瘤良恶性作出定性判断或实现对肝脏肿瘤区域的检测任务,以更好地辅助临床诊断工作。
5 结束语
U-Net及其改进网络在肝脏及肝脏肿瘤区域的分割中已经具有了良好的表现。相信随着研究工作的开展,肝脏及肝脏肿瘤的自动精准分割能够得到更加充分的发展,更好地为临床肝脏病变的早期发现和治疗提供辅助。

