三阴性乳腺癌相关miRNA筛选及其靶基因的生物信息学分析
胡鑫,麻慧慈,韩明盛,原晓红,杨鸣宇,马艳琴
山西农业大学生命科学学院,山西晋中030801
乳腺癌是女性最常见的恶性肿瘤之一,在女性癌症相关死亡中居第2 位[1]。三阴性乳腺癌(triple-negative breast cancer,TNBC)作为乳腺癌的1 个亚型,具有高度异质性、发病年龄偏小、分化程度低、侵袭性高、复发率高等特点[2]。TNBC由于雌激素受体(estrogen receptor,ER)、孕激素受体(progesterone receptor,PR)及人类上皮细胞生长因子受体2(human epithelial growth factor receptor 2,HER2)表达均为阴性,采用内分泌治疗及抗HER2 治疗后预后较差[2]。因此,探究与TNBC 发生、浸润及转移相关的分子机制对早期诊断、精准治疗及预后评估具有重要临床意义[3]。
微小RNA(miRNA)是一类小的非编码RNA,长度一般为18~25 个核苷酸,可通过抑制mRNA翻译或促进mRNA降解而对基因表达起负调控作用[4]。miRNA 通过调节癌基因和抑癌基因的表达来调控肿瘤细胞的存活、增殖、侵袭、转移、凋亡及对药物的反应性等,对肿瘤早期诊断、预后判断和治疗靶标价值较高[5-6]。本研究利用基因表达数据库(gene expression omnibus,GEO)中TNBC相关的miRNA 表达谱芯片数据集,筛选差异表达miRNA并预测其靶基因,进而对靶基因进行相关生物信息学分析,从而获得TNBC 发生发展中的关键miRNA 及其关键靶基因,旨在为TNBC 的研究提供潜在的分子靶点。
1 材料与方法
1.1 数据获取
从GEO 数据库中获取miRNA 表达芯片数据集GSE38167(agilent-029297 human miRNA microarray,GPL14943平台),包括31例原发性TNBC 样本和23例正常乳腺组织。
1.2 差异miRNA筛选
利用GEO2R 在线分析软件(https://www.ncbi.nlm.nih.gov/geo/geo2r/)分析正常乳腺组织和TNBC 组织的差异表达miRNA,筛选条件为adj.P<0.05,|log2FC|≥2。
1.3 差异miRNA靶基因预测
对差异表达倍数最大的5 个上调和5 个下调miRNA 进行靶基因预测。为降低软件预测结果的假阳性率,拟定至少在miRDB(http://mirdb.org/)、miRWalk(http://mirwalk.umm.uni-heidelberg.de/)和TargetScanHuman7.2(http://www.targetscan.org/vert_72/)这3 个预测软件中有记录的基因才可以作为靶基因。
1.4 靶基因功能富集分析
通过DAVID(https://david.ncifcrf.gov/)在线分析软件对靶基因进行基因本体(gene ontology,GO)功能注释和京都基因与基因组百科全书(kyoto encyclopedia of genes and genomes,KEGG)通路富集分析,筛选条件为P<0.05。
1.5 靶基因PPI网络及miRNA-靶基因网络构建
将上调和下调miRNA 的靶基因分别映射到STRING 数据库(https://string-db.org/)构建蛋白互作网络(protein-protein interaction network,PPI),中置信度(medium confidence)值为0.400。结合Cytoscope 软件的CytoHubba 插件筛选degree 值为Top10 的基因,并建立miRNA-靶基因调控网络,筛选关键miRNA和关键靶基因。
1.6 生存分析
利用GEPIA2数据库(http://gepia2.cancerpku.cn/)分析关键靶基因表达与TNBC 患者总体生存率的相关性。
2 结果与分析
2.1 差异miRNA的筛选
使用GEO2R 分析GSE38167数据集并绘制差异miRNA 的火山图(图1)。共筛选出486 个差异miRNA,其中上调的298 个,下调的188 个。上调倍数最大的5 个miRNA 分别是miR-452*、miR-611、miR-587、miR-615-5p、miR-1915*;下调倍数最大的5 个miRNA 分别是miR-339-5p、miR-511、miR-1205、miR-147b、miR-580(表1)。

表1 TNBC组织和正常乳腺组织中差异表达倍数最大的miRNATable 1 The largest differential expression of miRNA in TNBC tissue and normal breast tissue

图1 差异表达miRNA火山图Fig.1 Volcano map analysis of differentially expressed miRNA
2.2 差异miRNA靶基因的预测
通过miRWalk、TargetScanHuman7.2和miRDB分别对差异miRNA 进行靶基因预测并取交集,结果如图2~3 所示,上调miRNA 中miR-1915-5p、miR-615-5p、miR-587、miR-611、miR-452-3p 的靶基因分别有211、81、287、33、47 个;下调miRNA中miR-339-5p、miR-511-3p、miR-1205、miR-147b-3p、miR-580-3p 的靶基因分别有3、29、161、4、79个。

图2 表达上调倍数最大的5个miRNA的靶基因预测Fig.2 Target gene prediction of five miRNA with the largest up-regulated expression

图3 表达下调倍数最大的5个miRNA的靶基因预测Fig.3 Target gene prediction of five MiRNA with the largest down-regulated expression
2.3 靶基因的功能富集分析
GO 分析结果表明,上调miRNA 的靶基因主要参与的生物学过程(biological process,BP)包括转录、转录调控、轴突导向、细胞周期阻滞等;细胞成分(cellular component,CC)主要集中于细胞核、细胞质、细胞溶质等;分子功能(molecular function,MF)主要为转录因子活性、DNA 结合、RNA聚合酶Ⅱ核心启动子近端区域序列特异性DNA结合等(表2)。下调miRNA 的靶基因主要参与的BP 为突触传递的调节、成纤维细胞凋亡的正调控、伴侣蛋白介导的蛋白质折叠辅因子等;CC 主要定位于细胞质、晚期胞内体、细胞溶质;MF主要与RNA 结合等(表3)。KEGG 通路分析结果表明,上调miRNA 的靶基因主要参与ErbB 信号通路、癌症中的转录失调、催乳素信号通路等;下调miRNA 的靶基因主要参与cGMP-PKG 信号通路(表4)。
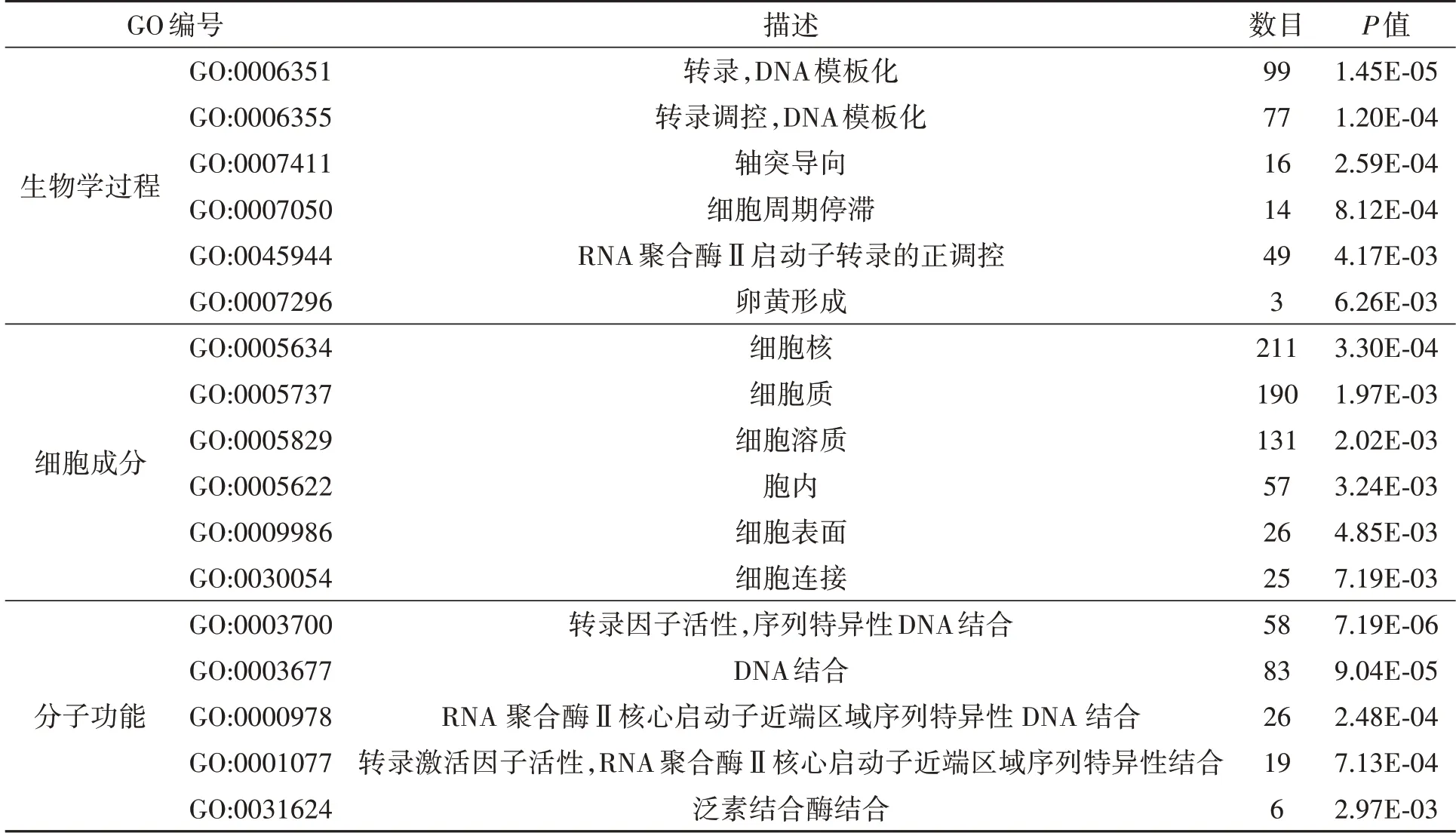
表2 部分表达上调的miRNA靶基因的GO功能注释Table 2 Partial results of GO function annotations of miRNA target genes with up-regulated expression

表3 部分表达下调的miRNA靶基因的GO功能注释Table 3 Partial results of GO function annotation of down-regulated miRNA target genes

表4 miRNA靶基因的KEGG通路富集分析(部分结果)Table 4 KEGG pathway enrichment analysis of miRNA target genes(partial results)
2.4 靶基因PPI 网络的构建和关键靶基因的筛选
利用STRING 在线数据库构建PPI网络,上调miRNA 的靶基因PPI 网络由608 个节点和1 228条边连接组成(图4A);下调miRNA 的靶基因PPI网络由271个节点和269条边连接而成(图4B)。

图4 差异miRNA靶基因的PPI网络图Fig.4 PPI network diagram of different miRNA target genes
利用Cytohubba 插件分析显著性最高的分子模块,进而获取关键靶基因。结果如图5所示,上调miRNA的关键靶基因为CDC27、UBE2D2、UBR1、ASB1、SPSB1、HERC2、RLIM、MKRN1、UBE3B、RNF138。下调miRNA 的关键靶基因为FBXL3、WSB1、KBTBD6、FBXL8、CBLB、UBE2W、PTPN11、ARF6、DNAJC6、COPS2。

图5 关键靶基因互作网络图Fig.5 Key target gene interaction network diagram
利用Cytoscape 构建miRNA-靶基因调控网络,如图6 所示,miR-611 可能调控6 个关键靶基因,分别为CDC27、UBE2D2、UBR1、SPSB1、HERC2、RLIM。miR-1205 可能调控7 个关键靶基因,分别为WSB1、FBXL8、UBE2W、PTPN11、ARF6、DNAJC6、COPS2,提示miR-611和miR-1205为TNBC的2个关键潜在调控miRNA。

图6 miRNA-靶基因调控网络图Fig.6 miRNA-target gene regulatory network diagram
2.5 靶基因的生存曲线分析
利用GEPIA2 数据库进一步分析关键靶基因表达与TNBC 患者生存率的关系,其中UBR1(P=0.007 2)和PTPN11(P=0.029)的高表达可显著降低TNBC患者的整体生存率(图7)。
3 讨论
TNBC不表达或低表达ER、PR以及HER2,其恶性程度高、侵袭性强、预后差,临床治疗难度较大[7]。近年来,人们逐渐认识到miRNA 可通过靶向调控mRNA的表达而促进肿瘤的生长、侵袭、迁移、血管新生及免疫逃逸[8]。本研究筛选出486个差异miRNA,包括298 个上调miRNA 和188 个下调miRNA。对差异倍数最大的5 个上调和5 个下调的miRNA 进行靶基因预测。miRDB、miRWalk和TargetScanHuman7.2 是3 个重要的miRNA 靶点预测工具,可进行miRNA 与mRNA 的启动子、5'非翻译区、编码区及3'非翻译区结合位点的计算和预测[9]。富集分析显示,靶基因主要参与ErbB信号通路、癌症的转录失调和cGMP-PKG 信号通路等。进一步分析miRNA-靶基因调控网络,获得与TNBC发生发展相关的关键miRNA及关键靶基因,可用于探究miRNA 对TNBC 的调控作用及发现新的分子靶点。本研究发现,多个mRNA(CDC27、UBE2D2、UBR1、SPSB1、HERC2和RLIM)共同受到miR-611 的调控,mRNAWSB1、FBXL8、UBE2W、PTPN11、ARF6、DNAJC6和COPS2共同受到miR-1205的调控。结合PPI网络分析关键模块,以上蛋白在整个蛋白调控网络中处于核心地位,对TNBC 的发生发展有重要的作用。生存分析进一步表明,靶基因UBR1和PTPN11的高表达与TNBC 患者整体生存率降低显著相关。研究表明,miR-611 可促进舌鳞状细胞癌细胞增殖、迁移、侵袭及上皮间质转化,其表达上调与TNM 分期呈正相关,与患者整体生存率呈负相关[10]。miR-1205 在喉鳞状细胞癌(laryngeal squamous cell carcinoma,LSCC)组织中表达下调,miR-1205 过表达可抑制LSCC 细胞的增殖、迁移和侵袭。miR-1205 高表达则预示LSCC 患者预后良好[11]。同时,在非小细胞肺癌和胃癌中也证实了miR-1205作为肿瘤抑制基因的作用[12-13]。本研究中,miR-611 在TNBC 组织中表达显著上调(log2FC=7.97,adj.P<0.05),miR-1205 表达显著下调(log2FC=-7.87,adj.P<0.05),提示其可作为TNBC 潜在的诊断标志物,并为深入研究TNBC 的发病机制奠定了理论基础。
靶基因UBR1能识别含不稳定氨基末端的蛋白质,导致底物蛋白质的最终降解[14]。Hsp90 抑制剂通过泛素蛋白酶体途径从而促进致癌蛋白激酶和转录因子的降解,可作为有效的化疗药物。UBR1可提高癌细胞对Hsp90抑制剂的敏感性[15]。但本研究结果显示,UBR1高表达显著降低了TNBC 患者整体生存率,但UBR1在TNBC 发生发展中的作用及作为治疗靶点的应用价值还需要进一步的研究。研究表明靶基因PTPN11编码SHP-2分子[16]。SHP-2 已证实与多种肿瘤的发生相关,包括胃癌、乳腺癌、肺癌、肝癌、血癌等[17]。童雅兰[18]研究发现,SHP-2通过调控TNBC干细胞而影响TNBC 的复发、转移和化疗耐药。Wu 等[19]通过生物信息学分析得出PTPN11是乳腺癌差异表达基因的中心基因之一,进一步通过实时荧光定量实验进行验证,结果发现在0.1 μmol·L-1Cd 处理下PTPN11表达显著增加,表明PTPN11基因可能与Cd 诱导的乳腺癌进展相关。本研究中靶基因PTPN11高表达可显著降低TNBC 患者整体生存率,提示其有望作为潜在的预后标志物。
综上所述,经筛选获得的关键miRNA 及其关键靶基因可作为潜在分子标记物用于TNBC 的早期诊断、治疗靶点选择和预后判断,并为未来更深入的研究提供理论参考依据。

