基于图卷积神经网络的小分子虚拟筛选
张凯睿 黄钢


摘 要:新药研发存在研发周期长、成本高和成功率低等问题。为了解决这一系列问题,提高早期药物研发效率,提出一种基于图卷积神经网络的虚拟筛选方法,并利用模型对EGFR(Epidermal Growth Factor Receptor, 表皮生长因子受体)靶点进行虚拟筛选。首先获取EGFR靶点的相关数据,对其进行数据处理后用于模型训练;随后应用模型筛选大量化合物,筛選出小分子后,将其与药物分子进行化合物相似性搜索,验证其是否与已知的EGFR药物存在相似性;同时,将图卷积神经网络(Graph Convolutional Networks, GCN)模型与其他传统机器学习模型进行比较,证明本研究模型在各项指标中均优于其他模型。实验结果表明,本研究提出的方法具有较好的预测性和准确性,为发现潜在药物提供了助力。
关键词:图卷积神经网络;虚拟筛选;EGFR;化合物相似性搜索;机器学习
中图分类号:TP391 文献标识码:A
Virtual Screening of Small Molecules based on Graph Convolutional Neural Network
ZHANG Kairui1,2, HUANG Gang1,2
(1. School of Health Science and Engineering, University of Shanghai for Science and Technology, Shanghai 200093, China;
2. Shanghai Key Laboratory of Molecular Imaging, Shanghai University of Medicine and Health Sciences, Shanghai 201318, China)
zhangkarry0328@163.com; huanggang@sumhs.cn
Abstract: New drug research and development has the problems of long research and development cycle, high cost and low success rate. In order to solve these problems and improve the efficiency of early drug research and development, this paper proposes a virtual screening method based on graph convolution neural network, and uses the model to perform virtual screening of the EGFR (Epidermal Growth Factor Receptor) targets. Firstly, the relevant data of EGFR targets are obtained and used for model training after data processing. After that, the model is used to screen a large number of compounds, and after small molecules are screened out, they are searched for compound similarity with drug molecules to verify whether they are similar to known EGFR drugs. At the same time, the graph convolution neural network model is also compared with other traditional machine learning models, and the proposed model is superior to other models in all indicators. Experimental results show that the proposed method has good predictability and accuracy, which facilitates the discovery of potential drugs.
Keywords: graph convolutional neural network; virtual screening; EGFR; compound similarity search; machine
learning
1 引言(Introduction)
随着疾病多样性以及药物耐药性等问题的出现,对新药的需求日益增加,但是新药研发存在研发周期长、研发成本高以及成功率低等问题。药物开发是一个昂贵且耗时的过程,通常来讲,一个新的药物从开始研发到最终上市,需要花费数十亿美元和10—15 年的时间[1]。在人力、物力投入高和耗时长的同时,小分子药物最终上市的成功率却只有6.2%,研发失败的风险比较高[2]。计算机辅助药物设计能够大幅度地缩短新药研发的时间,提高新药研发的成功率。传统的药物筛选方法有分子对接、药效团匹配[3]和相似性搜索[4]等。随着近年来计算机算力的提升和大数据时代的到来,以机器学习和深度学习为代表的算法进一步促进了药物研发的进程。
虚拟筛选是一种药物筛选方法,其使用计算机算法和模型来发现新的生物活性小分子药物。与高通量筛选(HTS)相比,虚拟筛选具有高效、低成本的特点。虚拟筛选的方法通常分为两类:基于结构的虚拟筛选和基于配体的虚拟筛选。
在基于结构的虚拟筛选中,算法通过模拟靶点蛋白质与小分子之间的物理相互作用,计算它们之间的亲和度[5-6]。根据与结合能相关的亲和度打分函数,对蛋白质和小分子化合物的结合能力进行评价,最终从大量化合物分子中筛选出结合方式合理、预测分数较高的化合物,用于后续的生物活性测试。
在基于配体的虚拟筛选中,通常不需要靶点的信息和结构,而是收集一系列作用于这个靶点的已知小分子化合物,从这些已知小分子开始,去发掘这部分小分子的内在结构规律。根据化合物相似性或者药效团模型在化合物数据库中搜索能与之匹配的化学分子结构,最后对所筛选出来的化合物进行实验筛选研究。
近年来,随着计算机计算能力的发展,深度学习[7]被广泛应用于计算机视觉、自然语言处理、语音识别等领域。由于各类组学以及生物学数据的积累,深度学习模型已在药物研发的各个领域崭露头角[8],并且在一部分领域展现出优于传统的机器学习模型的优势。
本文从DUD-E中收集了EGFR靶点的活性化合物和诱饵化合物数据,并对这些化合物数据进行数据处理,将其分割成训练集和测试集。利用收集好的化合物数据训练图卷积神经网络[9-10]模型,随后筛选了ZINC数据库中的大量数据,将筛选出的10 个小分子与DrugBank数据库中收集到的药物数据做相似性搜索,发现其中4 个小分子与已知EGFR药物分子存在较高相似性。同时利用相同数据,训练6 个传统机器学习模型,结果表明GCN模型要优于机器学习模型。实验结果证明了GCN模型在药物筛选方面具有比较好的预测性和准确性。
2 材料与方法(Materials and methods)
基于图卷积神经网络的药物筛选模型构建流程及方法主要包括从化合物数据库收集相关数据,对药物数据进行数据处理和筛选,训练图卷积模型,随后进行药物筛选及对筛选结果的相似性进行搜索,如图1所示。
2.1 数据来源
本研究使用的数据来源于多个公共化合物数据库,包括DUD-E[11]、ZINC[12]、DrugBank[13]。DUD-E(A Database of Useful Decoys: Enhanced)是由美国加州大学旧金山分校药物化学系的Shoichet实验室提供的数据库(http://dude.docking.org),在Target中可以选择想要选择的靶点,下载actives_final.ism和decoys_final.ism文件,获取对应靶點的活性化合物和诱饵化合物数据,用于训练模型。ZINC数据库是目前最大的有机小分子化合物库之一,不少类药分子的前期虚拟筛选都是基于这个数据库的,数据库中包含9.8亿多个小分子,根据后续的研究条件和需要设定过滤标准,将小分子数量锁定在10万个后下载数据文件,用于后续的虚拟筛选。DrugBank数据库是一个综合的、可自由访问的在线数据库,包含有关药物和药物目标的信息,在利用模型筛选完ZINC数据库中下载的小分子后,将筛选出的分子与DrugBank中的药物分子做相似性搜索,从而验证模型的准确性。
2.2 数据预处理
为了更好地应用来源于DUD-E数据库中的数据集从而构建一个有效的模型,需要确保活性化合物与诱饵化合物的分子性质相似。从DUD-E数据库中收集到的化合物数据都有各个化合物对应的SMILES(Simplified Molecular-Input Line-Entry System)号。SMILES是一种用文本字符串定义分子的常用方法,SMILES字符串以既简洁又直观的方式描述了分子的原子和键。在本研究中,可以利用化学信息学软件包RDkit,结合化合物的SMILES号,计算出化合物的分子量、LogP以及形式电荷,通过化合物的这些属性可以比较活性集和诱饵集的分布。根据活性集和诱饵集的分布,对其进行平衡,为后续的模型训练做好准备。
2.3 图卷积网络模型
本文使用图卷积神经网络模型来预测分子抑制EGFR的能力。图卷积神经网络是一类采用卷积操作的图神经网络,属于图神经网络(Graph Neural Network)[14]的一种。
对于图,为节点的集合,为边的集合。对于每个点,均有其特征,可以用表示。一个中有3 个比较重要的矩阵:邻接矩阵、度矩阵和特征矩阵。邻接矩阵用来表示节点间的连接关系。度矩阵是一个对角矩阵,每个节点的度指的是其连接的节点数,其中对角线元素。特征矩阵用于表示节点的特征,,其中是特征的维度。
深度学习中最重要的是学习特征:随着网络层数的增加,特征愈发抽象,然后用于最终的任务。对于图任务,深度模型从最开始的特征出发学习到更抽象的特征。任何一个图卷积层都可以写成如下非线性函数:
(1)
为第一层的输入,其中,为图的节点个数,为每个节点特征向量的维度,为邻接矩阵。这里指的是网络层数,就是网络第层的特征。不同模型的差异点在于函数的实现不同。
在图学习中,每个节点的新特征就是对该节点的邻域节点特征进行变换然后求和。其公式为:
(2)
其中,是学习权重,维度是,是激活函数,这是神经网络的基本单元。不难看出,乘以邻接矩阵就相当于对每个节点都加上了其相应邻域节点的特征。这里存在两个问题,一是计算节点的新特征时并没有考虑自身的特征;二是矩阵没有正则化,这可能导致网络训练过程中发生梯度爆炸或者是梯度消失的问题。对于第一个问题,解决方法是对每一个节点加上自环,即为。对于第二个问题,则是对矩阵进行正则化,使其每一行的和都为1,例如。
图卷积神经网络的最终形式为:
(3)
其中,第层网络的输入为(初始输入为);;为待训练的参数;为相应的激活函数。
2.4 评估标准
本研究使用马修斯相关系数(Matthews Correlation Coefficient, MCC)、准确率(Accuracy)、召回率(Recall)和F1分数(F1-Score)作为评价指标。MCC、Accuracy、Recall及F1-Score的公式如下:
其中,为真正例,表示正类正确预测为正类数;为真负例,表示负类正确预测为负类数;为假正例,表示负类错误预测为正类数;为假负例,表示正类错误预测为负类数。
2.5 Morgan指纹
化合物相似性搜索在化学信息学和新药研发中有着悠久的历史,许多算法都使用相似性搜索来验证正在研究的新化合物。
本研究通过计算化合物的Morgan[15]指纹(Morgan Fingerprints, 摩根分子指纹)来进行化合物相似性搜索。Morgan指纹是一种圆形指纹,也属于拓扑型指纹,是通过对标准的Morgan算法进行改造后得到的。Morgan指纹具有如下优点:计算速度快,没有经过预定义,可以包含手性信息,指纹中的每个元素代表一种特定子结构,可以方便地进行分析和解释,可以根据不同的需要进行相应的修改。Morgan指纹设计的最初目的是用于搜索与活性相关的分子特征,也可以用于相似性搜索、聚类、虚拟筛选等方向。
3 实验结果及分析(Experimental results and analysis)
3.1 虚拟筛选结果
将DUD-E中获取的EGFR的活性化合物和诱饵化合物数据进行数据处理、整合之后得到542 个活性小分子和35,050 个非活性小分子,将整合好的数据分割为训练集和验证集,然后用于GCN模型的训练。
训练好模型后,用模型筛选ZINC数据库中收集的化合物,将得分最高的10 个小分子取出。筛选出的小分子信息如表1所示,化学结构如图2所示。
3.2 相似性搜索
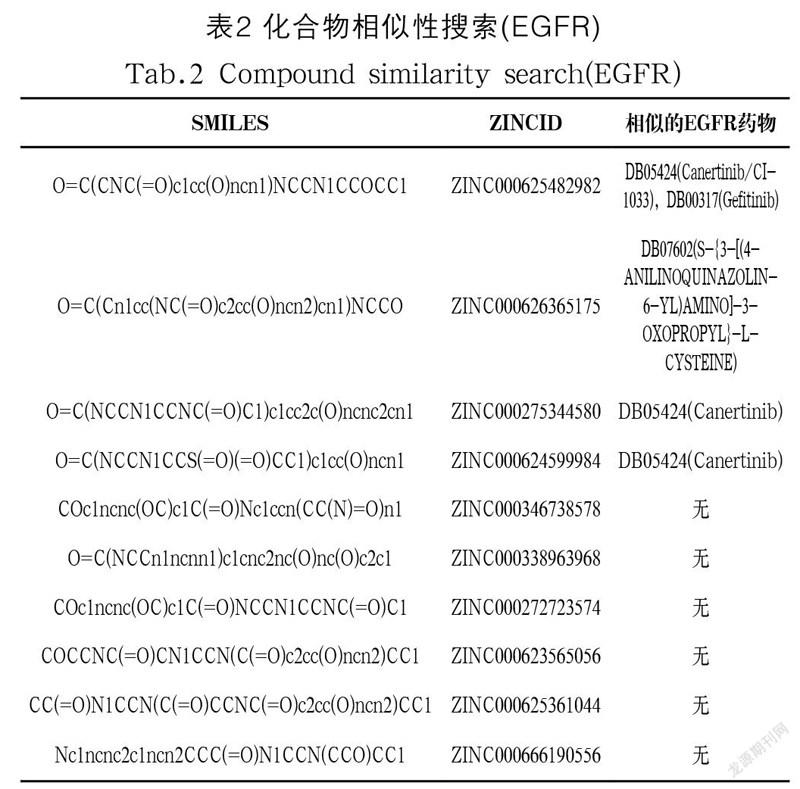
本研究通过计算分子的Morgan指纹将筛选结果中的小分子与DrugBank数据库中下载的药物小分子数据进行化合物相似性搜索,验证筛选结果是否与EGFR药物分子存在相似性。首先读入DrugBank数据库中的数据和需要查询相似性的小分子的SMILES号,然后计算查询分子与数据库分子的分子指纹,计算相似度并排序,输出相似度最高的前20 个药物分子,随后在DrugBank数据库中根据相似度最高的20 个药物分子的DRUGBANK_ID查詢其相关信息,看其是否为EGFR药物分子。
对筛选结果中的10 个小分子依次进行上述操作,结果显示4 个小分子与已知的EGFR药物分子存在相似性。相似性搜索的结果如表2所示。
3.3 模型比较
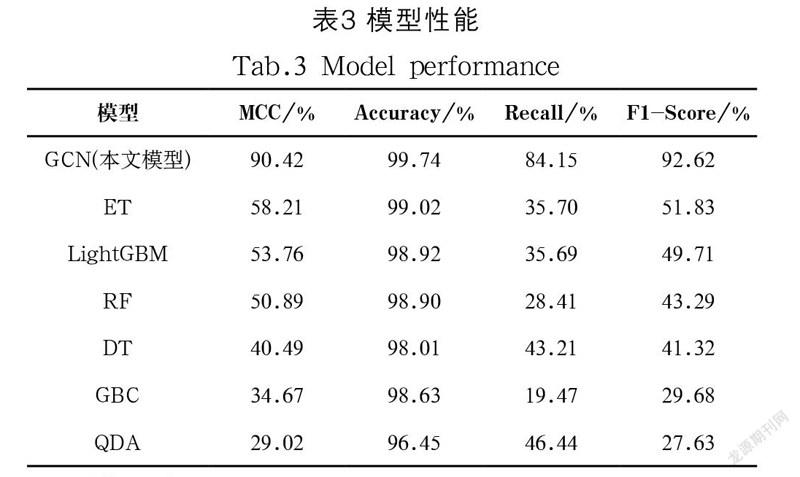
将数据处理后得到的EGFR活性化合物和诱饵化合物数据应用于六种机器学习模型进行训练,然后与GCN模型进行比较。七种模型的MCC、Accuracy、Recall、F1-Score结果如表3所示。结果显示,在四项指标中GCN模型均取得了最优结果。本数据集存在类别不均衡的情况,在这种情况下,MCC、F1-Score指标更具有说服力,在这两项指标中,GCN模型远远优于其他几种模型。
3.4 模型验证
为了防止模型仅对EGFR靶点产生较好的筛选效果,需针对其他靶点重复实验流程,从而进一步验证模型性能。
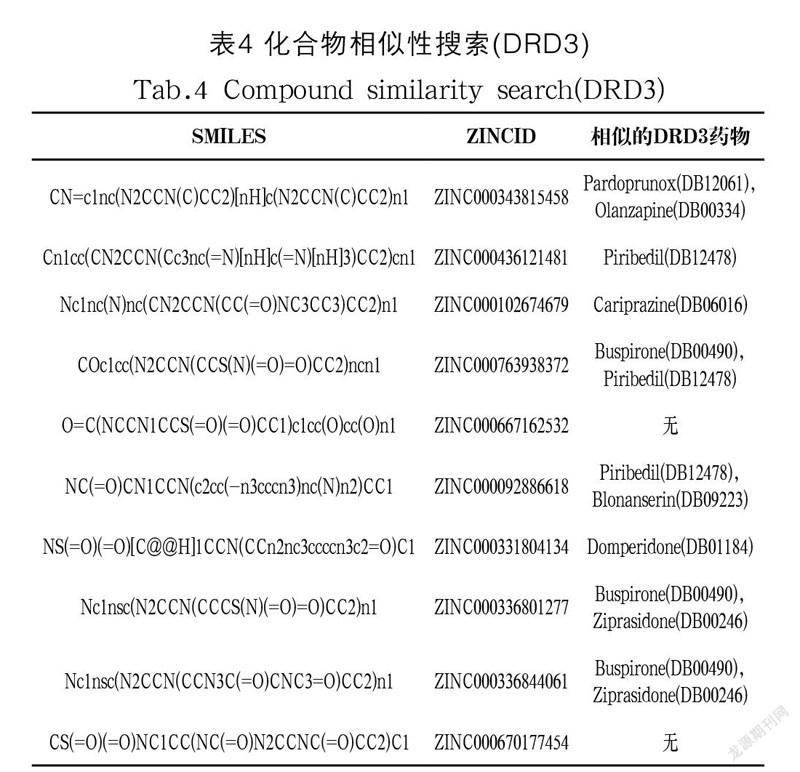
这里选择使用DRD3(Dopamine D3 Receptor, 多巴胺受体D3抗体)靶点进行模型验证。对DRD3靶点进行虚拟筛选和相似性搜索后得到的结果如表4所示,结果显示筛选出的10 个小分子中有8 个小分子与已知的DRD3药物分子存在相似性,证明模型对其他靶点也能产生较好的筛选效果。
4 结论(Conclusion)
本研究从DUD-E、ZINC、DrugBank数据库收集得到所需相关数据,基于图卷积神经网络方法进行虚拟筛选工作,并后续进行化合物相似性搜索,对模型性能进行验证,结果显示模型具有较好的预测性和准确性。同时,将本文模型与传统机器学习模型进行比较,通过十折交叉验证,使用MCC、Accuracy、Recall及F1-Score指标评估了模型,结果表明GCN模型优于其他传统机器学习模型,证明图卷积神经网络结合大数据在药物筛选方面的优越性。基于图卷积神经网络的虚拟筛选方法提升了早期药物研发工作的效率,为后续的生物实验及最终发现潜在药物提供了助力。
同时,在研究中也注意到图卷积神经网络在虚拟筛选中还存在的一些问题。当从DUD-E数据库中下载ACE靶点的活性化合物和诱饵化合物数据并用于模型训练时,会发现模型出现过拟合现象,这可能是因为ACE靶点的相关数据量远小于EGFR靶点的相关数据量,数据量过少导致了过拟合现象的产生。因此应当注意到,深度学习模型需要一定量的样本用来训练才能有效避免模型过拟合。为了解决这一问题,在后续的研究中需要考虑对模型进行改进,以应对小样本学习任务。
参考文献(References)
[1] DIMASI J A, GRABOWSKI H G, HANSEN R W. Innovation in the pharmaceutical industry: New estimates of R&D costs[J]. Journal of Health Economics, 2016, 47:20-33.
[2] WONG C H, SIAH K W, LO A W. Estimation of clinical trial success rates and related parameters[J]. Biostatistics, 2019, 20(2):273-286.
[3] WOLBER G, LANGER T. LigandScout: 3-D pharmacophores derived from protein-bound ligands and their use as virtual screening filters[J]. Journal of Chemical Information and Modeling, 2005, 45(1):160-169.
[4] WILLETT P, BARNARD J M, DOWNS G M. Chemical similarity searching[J]. Journal of Chemical Information and Computer Sciences, 1998, 38(6):983-996.
[5] KITCHEN D B, DECORNEZ H, FURR J R, et al. Docking and scoring in virtual screening for drug discovery: Methods and applications[J]. Nature Reviews Drug Discovery, 2004, 3(11):935-949.
[6] MENG X Y, ZHANG H X, MEZEI M, et al. Molecular docking: A powerful approach for structure-based drug discovery[J]. Current Computer-Aided Drug Design, 2011, 7(2):146-157.
[7] LECUN Y, BENGIO Y, HINTON G. Deep learning[J]. Nature, 2015, 521(7553):436-444.
[8] CHEN H, ENGKVIST O, WANG Y, et al. The rise of deep learning in drug discovery[J]. Drug Discovery Today, 2018, 23(6):1241-1250.
[9] KIPF T N, WELLING M. Semi-supervised classification with graph convolutional networks[Z/OL]. (2017-02-22) [2022-06-08]. https://arxiv.org/abs/1609.02907.
[10] 徐冰冰,岑科廷,黄俊杰,等.图卷积神经网络综述[J].计算机学报,2020,43(05):755-780.
[11] MYSINGER M M, CARCHIA M, IRWIN J J, et al. Directory of useful decoys, enhanced (DUD-E): Better ligands and decoys for better benchmarking[J]. Journal of Medicinal Chemistry, 2012, 55(14):6582-6594.
[12] STERLING T, IRWIN J J. ZINC 15-ligand discovery for everyone[J]. Journal of Chemical Information and Modeling, 2015, 55(11):2324-2337.
[13] WISHART D S, FEUNANG Y D, GUO A C, et al.
DrugBank 5.0: A major update to the DrugBank database for 2018[J]. Nucleic Acids Research, 2018, 46(D1):D1074-D1082.
[14] SCARSELLI F, GORI M, TSOI A C, et al. The graph neural network model[J]. IEEE Transactions on Neural Networks, 2008, 20(1):61-80.
[15] MORGAN H L. The generation of a unique machine description for chemical structures—a technique developed at chemical abstracts service[J]. Journal of Chemical Documentation, 1965, 5(2):107-113.
作者簡介:
张凯睿(1996-),男,硕士生.研究领域:生物医学工程.
黄 钢(1961-),男,博士,教授.研究领域:核医学分子影像.本文通信作者.

