利用加权基因共表达网络分析筛选天农麻鸡胴体性状候选基因
赵 迪,康慧敏,谭晓冬,刘冉冉,张正芬,李 华,,赵桂苹*
(1. 佛山科学技术学院生命科学与工程学院 广东省动物分子设计与精准育种重点实验室,佛山 528225;2. 中国农业科学院北京畜牧兽医研究所,北京 100193;3.广东天农食品集团股份有限公司,清远 511827)
中国是全球第二大鸡肉生产国和消费国,鸡肉产量约占全球鸡肉产量的14.7%,在中国,鸡肉是第二大肉类产品,占肉类总产量的20.5%。随着生活水平的提高,人们对鸡肉的需求正在从“数量”型向“质量”型转变。黄羽肉鸡肉质细腻,风味鲜美,深受我国消费者喜爱。清远麻鸡是慢速型黄羽肉鸡的代表性品种,也是广东最著名、最具有代表性的优质地方鸡种。天农麻鸡是以清远麻鸡为主要素材培育的优质肉鸡配套系,商品代抗病力强,肉质松软、风味好、鸡味浓、口感佳,体型外貌符合饲养地区消费市场对特优型地方鸡种的要求。无论是父母代种鸡还是商品代仔鸡,它们在同类产品中性能均处于领先水平,具有较强的市场竞争力。
胴体性状是肉鸡的重要经济性状,与肌肉、骨骼等多组织发育紧密相关,提高黄羽肉鸡的生产效率,增加产肉量一直是遗传育种工作的核心内容。随着高通量测序技术的快速发展,转录组测序(RNA-Seq)已被广泛应用于挖掘畜禽重要经济性状的候选基因。李国辉利用金茅黑鸡胸肌组织进行RNA-Seq,通过关联分析鉴定到107基因是影响体重和屠体重的候选基因,通过调节核质间的物质运输,参与生物的多种生长发育过程。Zhang等对白羽肉鸡、大恒肉鸡和商品代罗曼蛋鸡的胸肌进行RNA-Seq,共获得8 398个差异表达基因,发现与肌肉生长相关的基因15、2、3、2、-2、、等在不同品种中的表达量差异显著。
以RNA-Seq数据为基础,开发的加权基因共表达网络分析(weighted gene co-expression network analysis, WGCNA)是一种高效、准确的生物信息学和生物数据挖掘的方法。它可以系统地筛选目标性状关联的共表达基因,以便于进一步了解基因相互作用的机制。同时,利用WGCNA能够发现与目标性状高度相关的模块,快速识别目标性状的关键候选基因,进一步通过不同的筛选标准鉴定枢纽基因。WGCNA已在动物遗传学等生物领域广泛应用。在黄羽肉鸡的研究中,本课题前期曾报道,通过对京星黄鸡进行WGCNA,鉴定到转录因子3MBTL1和转录因子辅助因子1、1和6与高肌内脂肪和低腹脂重相关。采用WGCNA探究京星黄鸡肝中基因表达量与腹脂重的相关性,其中8、1、2和2基因被鉴定为腹脂重正相关模块中的枢纽基因,表明这些基因在肝脂质代谢中发挥着重要作用。
已有的关于慢速型黄羽肉鸡胴体性状遗传基础的研究相对较少,其候选基因仍有待进一步挖掘。本研究基于104只上市日龄天农麻鸡胸肌转录组数据进行WGCNA,筛选与胴体性状显著相关的共表达模块及其枢纽基因,并对模块内基因进行富集分析,挖掘影响胴体性状的作用通路,为解析黄羽肉鸡胴体性状的调控机制提供参考。
1 材料与方法
1.1 试验动物
本试验群体来源于广东天农食品集团股份有限公司提供的商品代天农麻鸡,该群体是慢速型优质黄羽肉鸡,1~125日龄于农户家中散养,饲养期间自由饮水和采食,所有个体健康状况良好。
1.2 样品采集及表型测定
天农麻鸡饲养至126日龄,宰前禁食12 h后测定活重,并随机选择104只母鸡进行颈动脉放血致死。统一采集右侧胸肌相同部位样品,置于液氮中迅速冷冻,-80 ℃保存备用。参照《家禽生产性能名词术语和度量统计方法》(NY/T823—2020)测定屠体重、全净膛重和胸肌重,并计算屠体率、全净膛率和胸肌率:屠体率(%)=屠体重/活重×100;全净膛率(%)=全净膛重/活重×100;胸肌率(%)=两侧胸肌重/全净膛重×100。
1.3 胸肌组织RNA提取
剪取黄豆大小的胸肌样本,使用TRIzol试剂(Invitrogen,美国)提取总RNA。使用K5500分光光度计(凯奥,中国)检测RNA纯度。同时使用生物分析仪2100系统的RNA Nano 6000检测试剂盒(安捷伦科技,美国)评估RNA的完整性和浓度。并将A260/A280比值在1.8~2.0之间,RNA完整性大于7.5的样品用于后续试验。
1.4 RNA-Seq数据分析
首先利用FastQC软件对下机数据进行质量评估并利用BBMap软件去掉read接头,通过Fastp过滤不合格read,获得clean data。利用HISAT2建立基因组(GRCg6A)索引,并进行比对,通过SAMtools软件将比对后的sam文件转换为bam文件,并进行排序、质控、索引,使用FeatureCount软件对基因进行定量,采用DESeq2软件对基因表达量进行标准化。
1.5 加权基因共表达网络分析
使用R语言WGCNA包的hclust函数对样本进行聚类,剔除离群样本。随后使用pick-SoftThreshold(sft)函数选择最佳软阈值,以无标度拓扑拟合指数()大于0.85为条件确定最佳软阈值(sft=7)。随后采用adjacency函数构建拓扑重叠矩阵(topological overlap matrix, TOM),并根据TOM矩阵的相异度,使用hclust函数对基因进行聚类。采用主成分分析法计算每个模块的特征值(module eigengene, ME),根据ME进行聚类,对相似度大于0.7的模块进行合并。
将合并过的模块重新计算ME,与表型矩阵进行关联分析,选择与表型最相关的模块为目标模块。随后计算目标模块内的Module membership(MM)与Gene significance(GS),以|MM| > 0.8、|GS| > 0.2为标准筛选到的基因为枢纽基因(hub gene),采用DAVID数据库(http://david.abcc.ncifcrf.gov/)对目标模块内的基因进行KEGG富集分析,以<0.05为标准筛选显著富集通路。
1.6 枢纽基因蛋白互作网络构建及可视化
采用String在线网络(https://string-db.org/)对每一个及全部枢纽基因分别进行蛋白质互作网络(protein-protein interaction network, PPI network)的构建,选取为参考物种。并使用Cytoscape软件中的cytoHubba插件,根据PPI网络节点中的Degree进行筛选,对排名前20的基因进行可视化。
1.7 统计分析
计算104个个体7种胴体表型的平均值及标准差,均以“平均值±3倍标准差”为标准剔除离群个体,利用SPSS软件计算58个枢纽基因的表达量与7种胴体表型的Spearman相关性,使用GraphPad Prism 8软件作图。
2 结 果
2.1 表型数据统计
对104只126日龄天农麻鸡的活重、屠体重等7种胴体表型进行基本的描述性统计(表1),126日龄母鸡平均活重为1 541.3 g,屠体重均值为1 370.5 g,全净膛重均值为1 040.0 g,胸肌重均值为168.9 g,屠体率均值为89.0%,全净膛率均值为67.7%,屠宰性能良好。

表1 胴体性状描述性统计Table 1 Descriptive statistics for carcass traits
2.2 基因共表达模块的构建
对转录组数据进行分析质控后,共得到15 092个基因用于后续分析。对104个样本进行聚类,剔除5个离群样本。以> 0.85为标准确定最佳软阈值sft=7(图1a),共得到24个模块。根据ME进行模块聚类,合并相似度大于0.7的模块(图1b),最后得到19个模块(图1c)。将共表达趋势不明显的基因统一归入Grey模块。

a.左图中纵轴代表对应的网络中log(k)与log(p(k))相关系数的平方;右图中纵轴代表相应的基因模块中基因邻接函数的均值;b.模块聚类图,红色线表示相似度达0.7;c.基因动态剪切聚类树,每个颜色代表一个模块,第一行为首次聚类的结果,第二行为模块合并后的结果a. The vertical axis of the left figure represents the square of the correlation coefficient between log(k) and log(p(k)) in the corresponding network; the vertical axis of the right figure represents the mean value of the gene adjacency function in the corresponding gene module; b. Module clustering graph, the height of the red line is 0.3, and the modules below the line are the modules that have greater similarity and need to be merged; c. Gene dynamic shear clustering tree, each color represents a module, the color in the first row is the result of the first clustering, the color in the second row is the result of the merging of the modules图1 加权基因共表达网络分析Fig.1 Weighted gene co-expression network analysis
2.3 目标模块的选择与分析
模块的 ME与表型矩阵进行相关性分析,得到模块与胴体性状间的相关系数及值(图2),选择与目标性状相关性较强(>0.39)且<0.05的模块为目标模块。最终确定4个目标模块,分别是“Magenta”模块,该模块对LW、DW、EW和BMW均呈显著负相关(LW:=-0.55,=4×10; DW:=-0.51,=9×10; EW:=-0.54,=1×10; BMW:=-0.50,=1×10)、“Tan”模块与DP呈显著正相关(DP:=0.47,=9×10)、“Green”模块与DP呈显著负相关(DP:=-0.58,=2×10)和“Purple” 模块与EP呈正相关(EP:=0.39,=6×10),这4个模块包含的基因数分别为:504、439、753、474个(表2)。根据|MM| > 0.8且|GS| > 0.2的标准确定模块的枢纽基因展开描述。

模块与胴体性状的相关性分析,红色代表正相关,蓝色代表负相关,颜色越深相关性越强The correlation analysis between the module and the carcass traits, red represents positive correlation, blue represents negative correlation, the darker the color, the stronger the correlation图2 模块性状相关图Fig.2 Module-trait relationship diagram
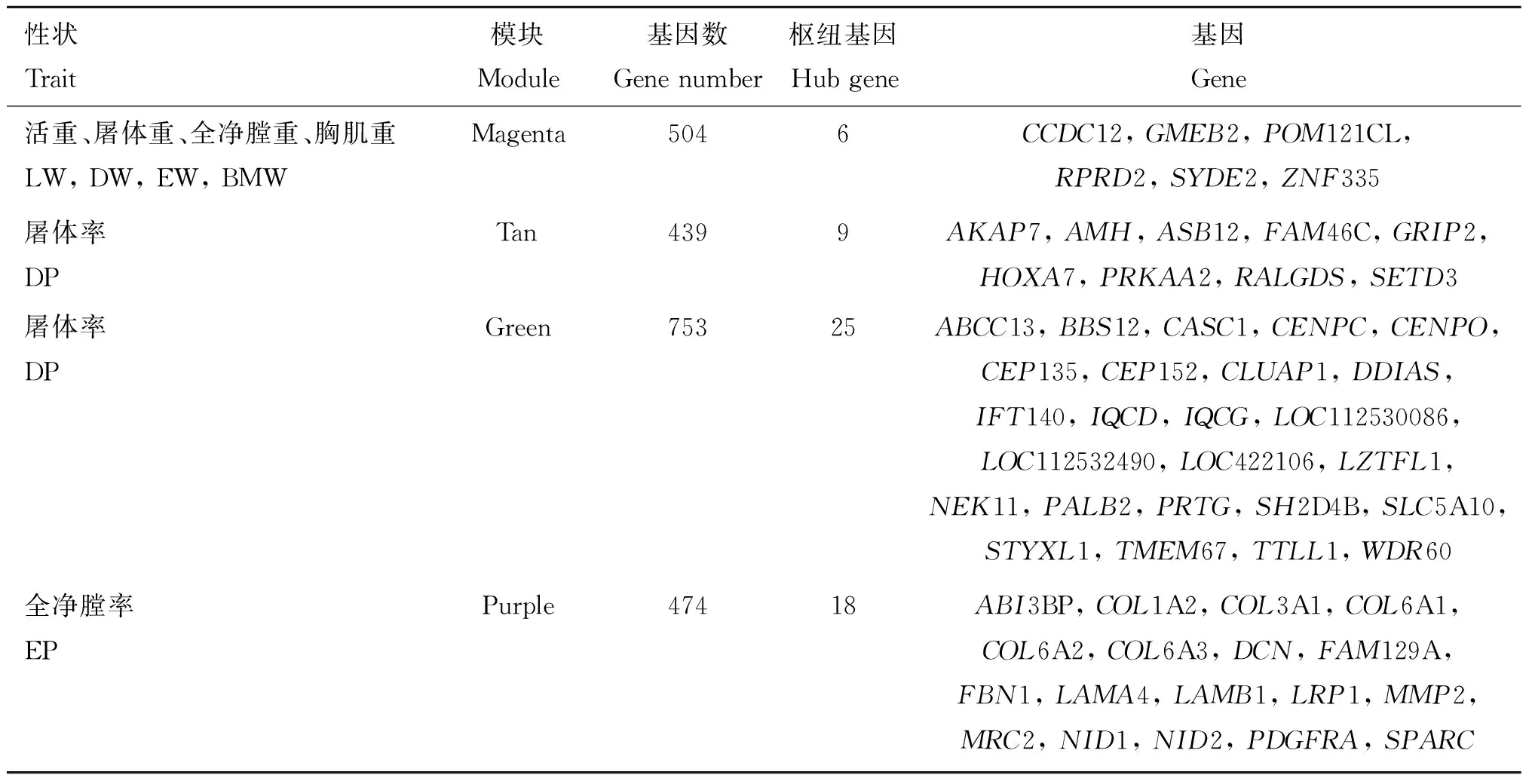
表2 目标模块及枢纽基因Table 2 Target modules and hub genes
2.4 目标模块中基因的KEGG通路富集
KEGG富集分析表明,目标模块共富集到18条 通路(图3)。Magenta模块内基因显著富集于mRNA 监测、Notch信号、黑色素生成、Wnt信号通路和TGF-β信号通路;Tan模块内基因显著富集于脂肪细胞因子信号通路、肌动蛋白细胞骨架的调节和FoxO信号通路;Green模块显著富集的KEGG通路包括细胞周期、范可尼贫血途径、基因复制、核苷酸切除修复、同源重组、错配修复和嘧啶代谢通路;Purple模块内基因显著富集于ECM-受体相互作用、黏着力和细胞黏附分子(CAMs)通路。富集结果表明,共有10条与肌肉生长发育相关的信号通路(表3)。
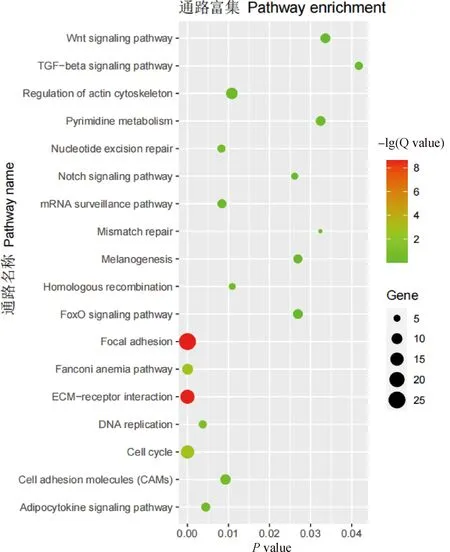
气泡大小表示基因数量;颜色表示富集分析的显著性The size of the bubbles indicate the number of genes; the color indicates the significance of the enrichment analysis图3 目标模块KEGG富集分析Fig.3 KEGG enrichment analysis of target modules
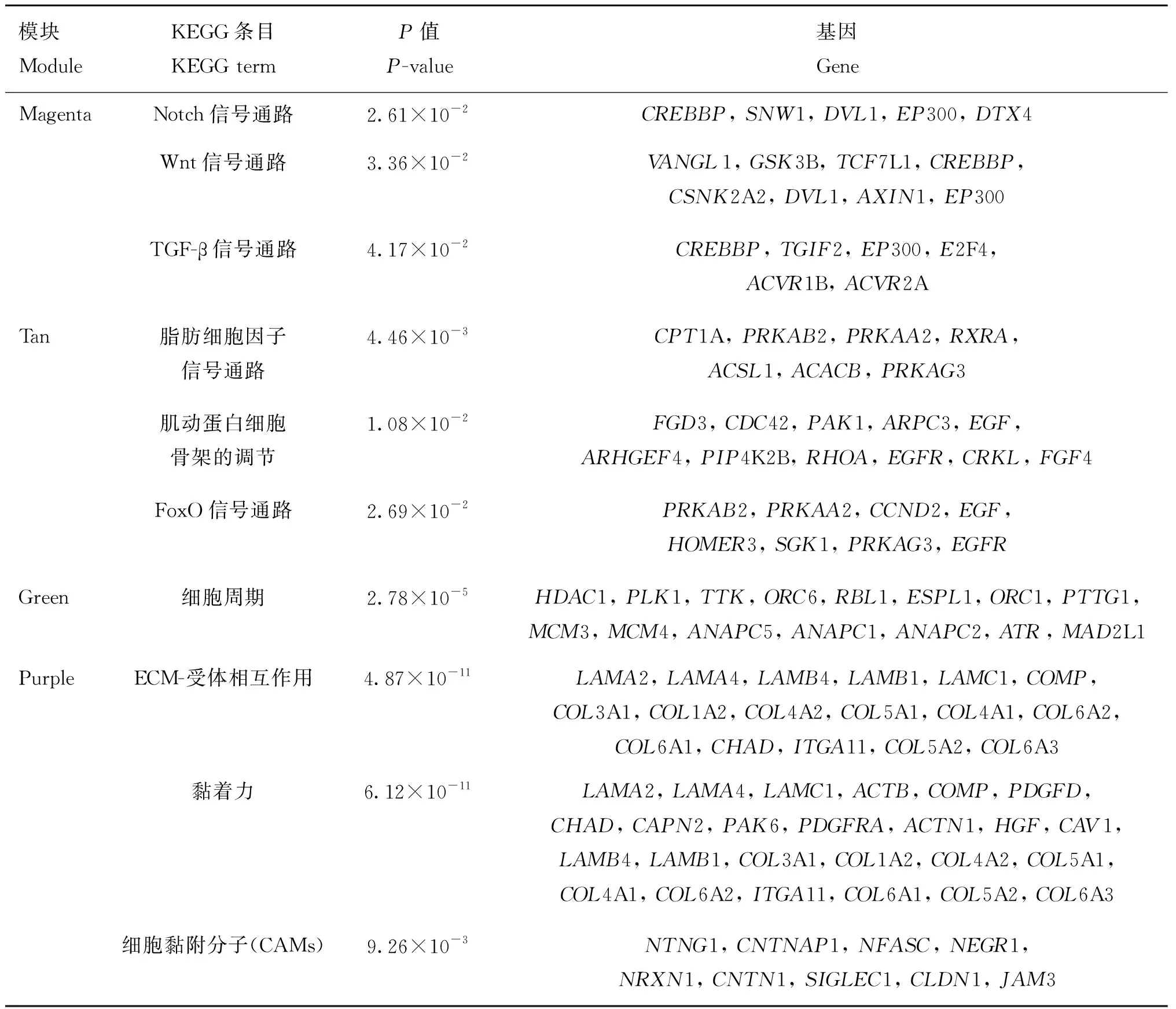
表3 目标模块KEGG富集结果Table 3 KEGG enrichment results of target modules
2.5 枢纽基因PPI网络构建
对枢纽基因进行PPI网络构建,以0.4为互作标准进行筛选。结果显示(图4a),前20个蛋白之间形成2个子网络,8个基因的子网络均来自Green模块,12个基因的子网络均来自于Purple模块。其中12个基因的子网络存在强互作,对子网络中的每个基因分别构建PPI网络,发现COL1A2、COL6A1、COL6A2、COL6A3和SPARC的互作蛋白大多数包括在1个子网络内,表明这5个基因可能在该网络中发挥核心作用,作为重要候选基因(图4b~f)。
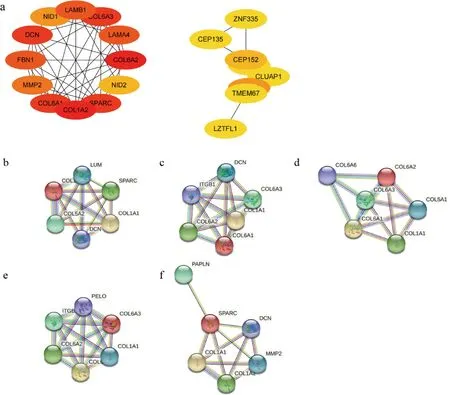
a.前20个枢纽基因PPI网络互作图;b~f.分别为COL1A2、COL6A1、COL6A2、COL6A3和SPARC基因PPI网络互作图a. Top 20 PPI network hub genes interaction diagram; b-f. PPI network COL1A2, COL6A1, COL6A2, COL6A3 and SPARC genes interaction diagram图4 PPI网络枢纽基因互作图Fig.4 PPI network hub genes interaction diagram
2.6 枢纽基因相关性分析
利用SPSS软件计算104个个体的表型数据与58个枢纽基因表达量的Spearman相关性,统计相关性大于0.4的基因。与PPI分析获得的网络节点基因取交集,筛选到与EP相关的1A2和基因,相关性见图5。

a.COL1A2基因与EP的相关性;b.SPARC基因与EP的相关性a. Correlation between COL1A2 gene and EP; b. Correlation between SPARC gene and EP图5 候选基因相关性分析图Fig.5 Candidate gene correlation analysis diagram
3 讨 论
胴体性状是极为复杂的经济性状,是优质肉鸡产肉性能选育的核心指标。屠体率和全净膛率是衡量产肉性能的主要指标,屠体率在80.0%以上及全净膛率在60.0%以上,被认为是产肉性能良好的标志。黄得纯等曾报道,138和168日龄清远麻鸡的屠体率和全净膛率分别为92.1%、64.8%及91.7%、65.6%。本研究结果表明,126日龄天农麻鸡的屠体率和全净膛率的均值分别达到89.0%和67.7%,产肉性能良好。
基于基因表达量与表型数据的WGCNA分析可以充分利用表型信息,将基因和表型之间的关联转化为多个基因集和表型之间的关联,可以有效地反映基因之间的相互作用。本研究利用104个样本的RNA-Seq数据,通过WGCNA分析挖掘胴体性状相关候选基因,有利于鉴定到与表型相关的基因集,相比于初步的差异基因鉴定更有利于构建调控网络。结果筛选出58个枢纽基因,涉及肌动蛋白细胞骨架的调节、ECM-受体相互作用、黏着力等通路,且与肌肉生长发育、脂质合成和能量代谢等有关。其中ECM-受体通路中细胞外基质参与细胞的增殖、分化和迁移,调节鸡的生长发育,大量研究报道,细胞外基质在维持骨骼肌结构和完整性及调节细胞间物质运输等过程中发挥重要作用。黏着力是复杂的细胞质膜相关的大分子组装体,通过招募大量黏着力相关蛋白与肌动蛋白细胞骨架形成的物理连接,是信号传导过程中许多信号分子组装的平台,与细胞的生长、发育密切相关。本研究ECM-受体相互作用和黏着力通路中检测到1A2、3A1、6A1、6A2、6A3、4和1与表型显著关联。4和1是细胞外基质糖蛋白家族的成员,是基底膜的主要非胶原成分。它们参与多种生物过程,包括细胞黏附、分化、迁移、信号传导、神经突生长和转移等。胶原蛋白VI的形成是由6A1、6A2和6A3基因编码的3条ɑ链组成的异源三聚体。研究表明,胶原蛋白VI分子组装时产生的微纤维与肌肉功能的维持有关,通过各种分子相互作用在肌细胞和周围基质之间提供重要的联系。本研究结果与Bordini等的研究报道一致,均鉴定到ECM-受体相互作用通路和4A1、4A2、2、4等枢纽基因,可能通过细胞外基质组成的改变以某种方式激活级联生物反应,影响肌肉生长发育,并参与改变IV型胶原蛋白的结构。此外,枢纽基因2可通过脂肪细胞因子信号通路和FoxO信号通路发挥调节作用。该基因通过部分磷酸化转录因子起到调节基因表达,同时参与调节脂肪酸和胆固醇的从头合成。2基因对秦川牛部分体尺及肉质性状有显著影响,是秦川牛新品种早期选择的候选基因。宋娇等以爱拨益加鸡和北京油鸡为试验素材,发现2基因表达量与肌内脂肪沉积显著相关。因此,推测2基因是屠体率性状的候选基因。
本研究将以上鉴定到的天农麻鸡胴体性状相关的58个枢纽基因进行PPI网络分析,鉴定到5个与全净膛率相关的重要候选基因(1A2、6A1、6A2、6A3和),同时结合Spearman相关性分析结果,确定1A2和基因表达量和全净膛率存在极显著的正相关。在前人的报道中,是一种Ca结合糖蛋白,与形态发生、重塑、细胞迁移和增殖有关。基因是胶原蛋白的钙化、细胞外基质的合成和软骨内成骨过程中细胞形状变化所必需的。在鸭胸骨骨化中同样发挥重要作用。1A2是胶原蛋白家族的成员,胶原蛋白是动物结缔组织中的一种重要蛋白质,也是细胞外基质的主要成分,可以为细胞生长提供支持。且与2A1和1A2之间存在相互作用。因此,1A2和是影响全净膛率性状的重要候选基因。1A2和可能通过协同表达影响蛋白互作,从而影响肌肉、骨骼等多组织的生长发育。
4 结 论
天农麻鸡126日龄的屠体率和全净膛率分别达到89.0%和67.7%,产肉性能良好;研究利用WGCNA和通路富集分析鉴定到ECM-受体相互作用和黏着力信号通路是影响胴体性状的主要通路,其中1A2和是影响全净膛率性状的重要候选基因,为获得天农麻鸡胴体性状分子标记、解析复杂性状分子调控机制奠定重要基础。

