基于3D深度残差网络和多模态MRI的脑胶质瘤自动分级
王瑞,刘志强,齐崇,孟蓝熙,李少武
1.北京市神经外科研究所/首都医科大学附属北京天坛医院,北京 100070;2.国家癌症中心/国家肿瘤临床医学研究中心/中国医学科学院北京协和医学院肿瘤医院放疗科,北京 100021
前言
脑胶质瘤是最常见的脑部肿瘤[1],根据2016版世界卫生组织(World Health Organization,WHO)中枢神经系统肿瘤分类标准[2],脑胶质瘤可以分为I~IV级:I级和II级为低级别脑胶质瘤(Low Grade Gliomas,LGG),III 级和IV 级为高级别脑胶质瘤(High Grade Gliomas,HGG)。其中LGG生长速度较慢,通常手术辅助其他治疗手段预后较好,而HGG生长迅速、恶性程度高、侵袭性强,患者在手术切除后仍有很高的病死率,术后需要进一步辅助放疗和化疗,生存预后差。因此,脑胶质瘤的准确分级在确定治疗方案和预测预后方面起着重要作用。磁共振成像(Magnetic Resonance Imaging,MRI)已被广泛应用于脑胶质瘤的无创诊断、监控和随访[3]。与单模态MRI 相比,多模态MRI:包括T1加权像(T1)、T1对比增强加权像(T1C)、T2加权像(T2)、T2液体衰减反转恢复像(FLAIR)、灌注加权像、弥散加权像、弥散张量加权像和波谱等,可以提供更丰富的肿瘤信息,为肿瘤的精确分级提供基础。目前,脑胶质瘤分级的标准方法基于组织病理学分析,存在以下局限性:(1)活检是一种侵入性手术,有潜在的风险;(2)由于MRI 图像上的肿瘤信号不均匀,活检可能存在固有的取样误差;(3)不同检验者之间可能存在观察的不确定性;(4)组织病理学分析费时,可能会延误诊断。为克服以上缺点,发展计算机辅助脑胶质瘤的自动分级技术尤为重要。如何利用多模态MRI 提供的丰富信息,快速实现脑胶质瘤精确的自动分级是具有挑战性的问题。
传统的模式识别和机器学习方法(如线性判别分析[4-5]、K-均值聚类[6]、决策树[7]、支持向量机[8-9]等)已经应用于单模态或多模态MRI 图像实现脑胶质瘤的分级,传统的方法需要手动勾画感兴趣区域(Region of Interests,ROIs),基于ROIs 提取肿瘤形状和统计特征、MRI 图像强度特征、以及图像纹理特征等用于图像分类。这些方法训练模型前需手动勾画ROIs和特征提取,这个过程耗费时间和精力、并且依赖经验,从而影响分级效率和准确性。
深度卷积神经网络在图像识别方面有很好的应用效果[10-14],与传统方法相比,训练模型的过程中可自动提取全面的图像特征,在肿瘤自动分级应用方面有很大的潜力,尤其是深度残差网络(Residual Network,ResNet)[15-16],它具有容易优化、增加深度提高准确性而不引起梯度消失的特点,同时考虑不同层面的MRI 图像具有序列信息,二维网络提取特征会丢弃掉部分信息,而三维(3D)网络可以更好地捕获序列信息和空间维度特征,而且不同模态的MRI具有不同的图像信息。本研究基于3D深度残差网络和多模态MRI图像实现脑胶质瘤的自动分级。
1 资料和方法
脑胶质瘤自动分级工作流程(图1)主要包括:(1)多模态MRI 图像预处理;(2)预处理后的多模态MRI 图像和胶质瘤分级标签(LGG 或HGG)输入3D深度残差网络模型进行训练和验证,确定最终的模型;(3)新患者的多模态MRI 图像进行预处理;(4)预处理后的多模态MRI 图像输入到已经训练好的3D深度残差网络模型得到分级结果(LGG或HGG)。
1.1 数据
本研究中用于训练和测试3D深度残差网络模型的数据来自BraTS2020公共数据集[17-19],包括369例脑胶质瘤,其中HGG 293例、LGG 76例。每例术前MRI图像均包含T1、T1C、T2和FLAIR 4个模态的图像(图2)。这些数据来自于多家机构的不同扫描设备,由1~4名医生手动勾画肿瘤区域,包括增强肿瘤区(Enhancing Tumor,ET)、瘤周水肿区域(Peritumoral Edema,ED)、坏死和非增强肿瘤核心区域(Necrotic/Non-enhancing Tumor Core,NCR/NET),并由神经放射科医生确认,根据组织病理学分析确定胶质瘤分级并作为金标准用于模型训练和测试。多模态MRI图像已经与标准空间的参考图像进行配准,并将颅骨剥离分离出大脑区域,将大脑图像重新采样至(1×1×1)mm3体素分辨率,所有肿瘤区域的并集生成MASK结构。
1.2 数据预处理
脑胶质瘤多模态MRI 图像3D 空间每个维度都具有复杂的成像特征,为了充分利用所有空间维度信息,本研究利用3D空间表示脑胶质瘤的异质性,即按照3D 脑组织的边界轮廓对MRI 图像和MASK 结构进行3D 裁剪,并重新采样到64×64×64 的体素,多模态MRI图像和MASK 结构作为模型输入。为了减少非标准MRI 强度值伪影,对多模态MRI 图像进行归一化处理,如公式(1)所示:
其中,x表示每个模态MRI的强度值,μ表示MRI图像中整个大脑非肿瘤区域强度值的平均值,σ表示对应的标准差。归一化后的强度值截断在[-5,5]区间,并最终归一到[0,1]之间,图3是归一化后的多模态MRI图像强度值分布。
1.3 3D深度残差网络模型
本研究采用的3D 深度残差网络模型如图4所示,图4a 是20 层的3D 残差网络模型框架,图4b~d 分别是3 个残差模块:Res-Block 0、Res-Block 1、Res-Block 2[16]。多模态的3D-MRI 图像和肿瘤区3DMASK 作为模型输入,经过卷积核是3×3×3、特征图数量是16、步长(Stride)是1 的3D 卷积(Conv3D),连接正则归一化Batch Norm 和激活函数ReLU,连接残差块Res-Block 0 和Res-Block 1 的组合、两次残差块Res-Block 2 和Res-Block 1 的组合,特征图数量从64过渡到256,图像尺寸从64×64×64 降到16×16×16,连接正则归一化Batch Norm、激活函数ReLU、平均池化(Average Pooling)和Flatten,经过全连接层Fullyconnected layer后,通过分类器Softmax classifier实现高低级别胶质瘤分类。
1.4 训练和测试
369 例脑胶质瘤(293 例HGG 和76 例LGG)数据样本分别被随机分成训练样本(64%),验证样本(16%)和测试样本(20%)。训练过程中通过优化输入数据结构和模型超参数,最终确定4个MRI模态的整个脑部区域和1个肿瘤MASK区域同时作为输入,网络模型的输入数据维度是5×64×64×64,设置超参数BatchSize 为2,选择Adam 优化器沿着梯度下降的方向优化模型,即每迭代2个训练样本更新一次优化权值,设置最大迭代次数Epochs 为100,设置初始学习率(Learning Rate,LR)为1e-4。
本研究采用二值交叉熵函数Binary Crossentropy作为损失函数,如公式(2)所示:
其中,yi是第i个样本的标签,HGG 标记为1,LGG 标记为0,N是参与计算的样本数量,p(yi)代表第i个样本预测为HGG的概率。
训练过程中自动调整LR,即训练迭代次数增加10 次后,验证数据集上的损失函数不再减小,LR 减小为原来的一半。为避免出现过拟合现象,训练过程中应用数据增强和Early-Stopping 技术,数据增强是训练过程中将数据进行随机翻转或旋转以增加训练数据的多样性,Early-Stopping是训练迭代30次后,验证数据集上的损失函数不再减小,停止继续迭代,保留在验证数据上损失函数最小的模型作为最终的模型。
对于测试样本,将每例患者的多模态MRI 图像经过1.2 节中的预处理后输入到已经训练好的3D 深度残差网络模型,即可以得到肿瘤的自动分级结果。
1.5 评价
分类结果的评价基于准确率(Accuracy,ACC)、敏感性(Sensitivity,SEN)、特异性(Specificity,SPE)、受试者工作特征(Receiver Operating Characteristic,ROC)曲线下面积(Area Under Curve,AUC)。定义高低级别胶质瘤混合矩阵(表1),即患者被识别为HGG 为正例(Positive)事件,被识别为LGG 为负例(Negative)事件。预测为HGG,实际为HGG,即为正确的正例事件(True Positive,TP);预测为LGG,实际为LGG,即为正确的负例事件(True Negative,TN);预测为HGG,实际为LGG,即为错误的正例事件(False Positive,FP);预测为LGG,实际为HGG,即为错误的负例事件(False Negative,FN),其中计算概率阈值设置为0.5,即预测概率≥0.5 时被识别为高级别HGG,否则被识别为LGG。
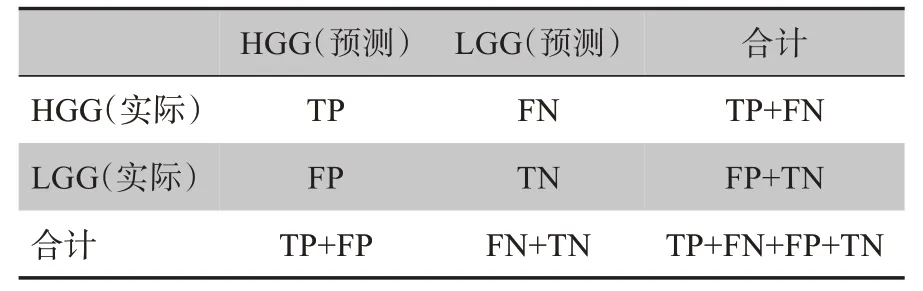
表1 高低级别脑胶质瘤混合矩阵Table 1 Confusion matrix of high and low grade gliomas
ACC 表示预测正确的样本占所有样本的比例,如公式(3)所示:
SEN 表示实际为正例的样本中,被预测为正例的样本比例,即HGG 样本中被预测为HGG 的比例,如公式(4)所示:
SPE表示在实际为负例的样本中,被预测为负例的样本比例,即LGG 样本中被预测为LGG 的比例,如公式(5)所示:
2 结果
本研究的验证数据集包括随机选出的59 例脑胶质瘤样本,其中HGG 48 例、LGG 11 例;测试数据集包括随机选出的75 例脑胶质瘤样本,其中HGG 62例、LGG 13 例。图5是3D 深度残差网络预测脑胶质瘤分级表现的ROC 曲线,对于验证数据集,SEN、SPE、ACC 和AUC 分别为0.94、0.91、0.93、0.97;对于测试数据集,SEN、SPE、ACC 和AUC 分别为0.95、0.62、0.89和0.93。
本研究分别探究了不同的图像区域、不同的MRI 模态组合作为输入数据,以及使用不同的数据增强方式和网络模型超参数组合时数据的分类结果。(1)表2是不同的图像区域作为输入数据的分类结果,其中参考模型代表1.2 小节数据预处理中的输入数据,肿瘤区域代表只提取多模态MRI 图像中的肿瘤MASK 区域;(2)表3是不同的MRI 模态组合作为输入数据的分类结果,其中参考模型代表4个模态(T1、T1C、T2和FLAIR)MRI和MASK 区域;(3)表4是不同数据增强方式的分类结果,其中参考模型代表使用翻转和旋转的组合作为数据增强方式;(4)表5是使用不同网络模型超参数组合的分类结果,其中参考模型代表使用1.3和1.4小节描述的超参数。

表2 不同图像区域作为输入数据的分类结果Table 2 Classification results with different image regions as input
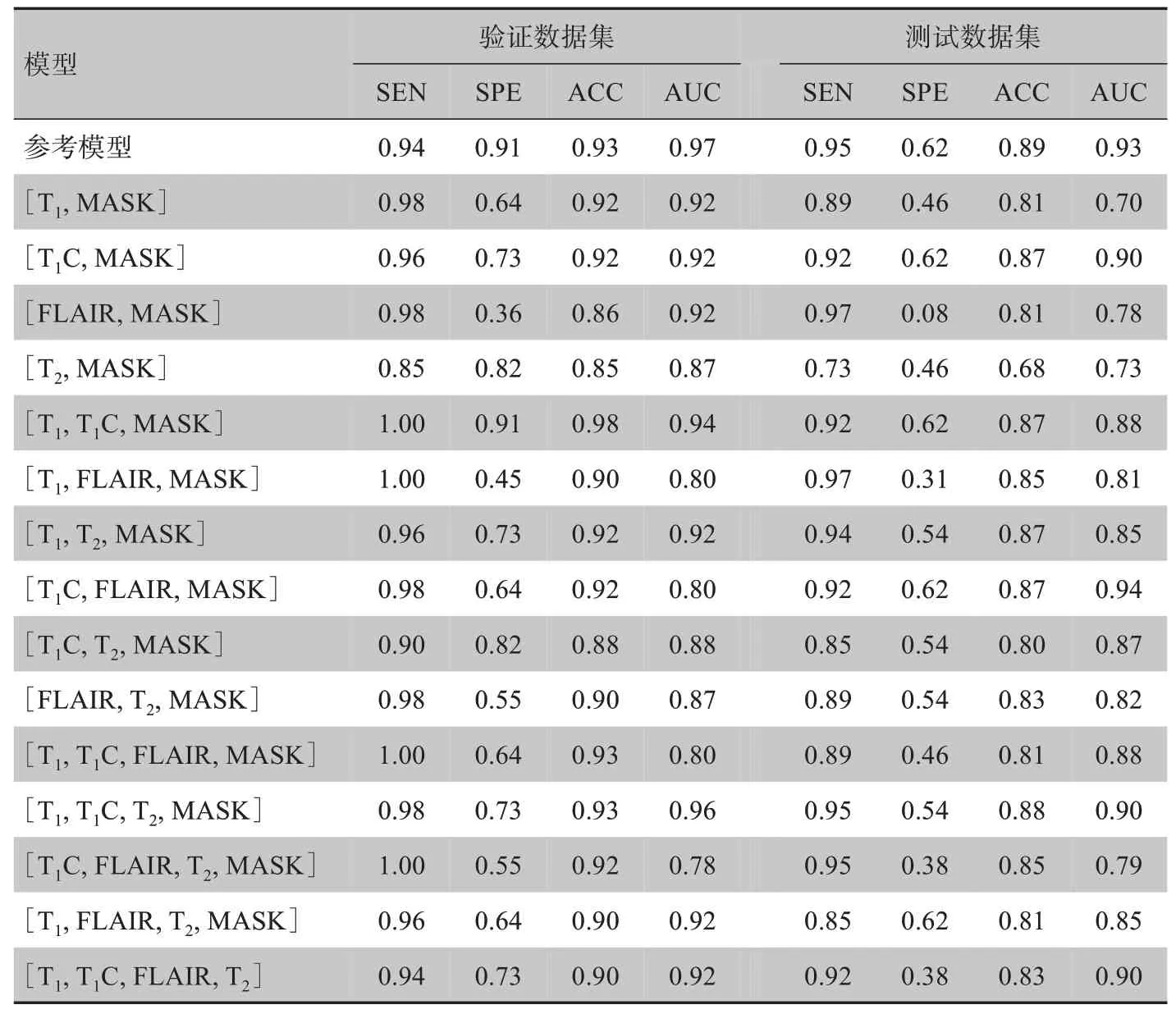
表3 不同MRI模态组合作为输入数据的分类结果Table 3 Classification results with different combinations of MRI modality as input
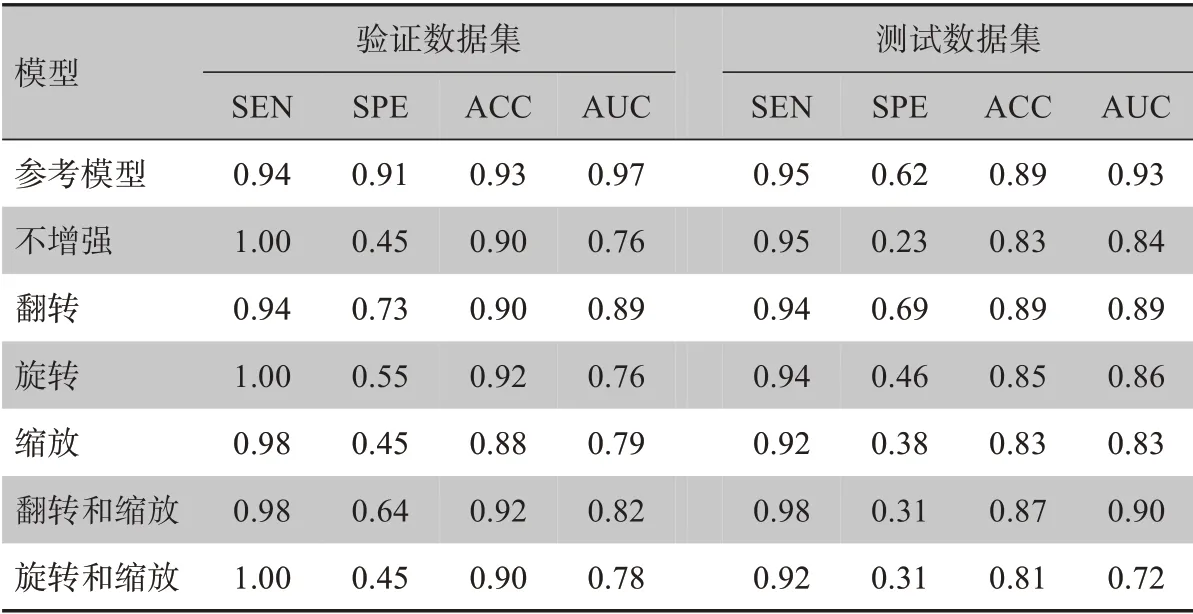
表4 不同数据增强方式的分类结果Table 4 Classification results of different data augmentation manners

表5 不同网络模型超参数组合的分类结果Table 5 Classification results of different model superparameter combinations
3 讨论
脑胶质瘤的准确分级在精确制定治疗方案和预测预后方面起着重要作用,侵入性活检存在潜在风险和固有的取样误差,不同检验者的观察存在不确定性且组织病理分析费时费力,自动分级技术可以克服活检存在的以上缺点。相比于传统的模式识别和机器学习方法,基于深度学习方法是近几年的研究热点,非常具有潜力。本文将3D 深度残差网络应用在多模态MRI 数据上实现了对高低级别脑胶质瘤的自动分级,文中利用的BraTS2020数据集是目前公开可以利用的多模态MRI 数据样本,包含的数据丰富,是测试算法的优质数据库。本文采用了20 层的3D 深度残差网络模型,二值交叉熵函数BinaryCrossentropy 作为损失函数,从不同角度(即不同的图像区域、MRI 模态组合作为输入数据,使用不同的数据增强方式和网络模型超参数组合)来优化模型,初步结果表明多模态MRI 和肿瘤MASK 区域的组合比只考虑多模态MRI肿瘤MASK 区域的结果要好(表2);如果只考虑单模态MRI图像序列,T1C的预测效果最好,如果考虑2 个模态MRI 图像序列,[T1C,T1]或[T1C,FLAIR]的组合预测效果较好,如果考虑3 个模态的MRI 图像序列,[T1,T1C,T2]的组合预测效果较好(表3);训练过程中数据进行增强处理可以提高预测精度,旋转和翻转组合的方式预测效果最好(表4)。同时,进一步探究了通过增加网络深度评价模型的表现,从初步实验结果(表6)可以看到通过将网络深度增加到56 层[15-16],测试数据集的ACC和AUC并没有提高。
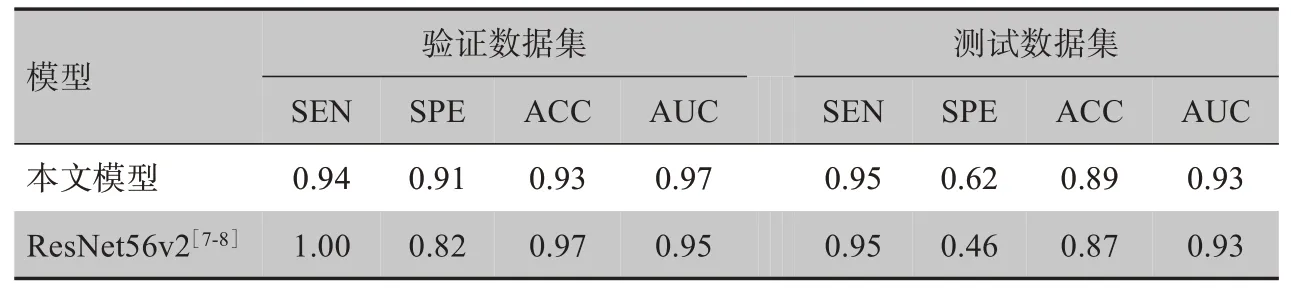
表6 不同深度的残差网络模型的分类结果Table 6 Classification results of residual networks with different depths
不同研究者利用深度学习网络在脑胶质瘤自动分级方面进行了不同的探索(表7)。Yang 等[20]利用2D 的GoogLeNet 和AlexNet 网络,在113 例5 折交叉验证数据集上,考虑T1C 模态作为输入从头开始训练,GoogLeNet 网络在验证数据集的ACC、测试数据集的ACC 和AUC 分别为0.87、0.91 和0.94,AlexNet网络在验证数据集的ACC、测试数据集的ACC 和AUC 分别为0.87、0.86 和0.90。Naser 等[21]利 用VGG16 网络,在110 例LGG 的5 折交叉验证数据集上,考虑T1、FLAIR、T1C 的MRI 图像数据作为输入进行迁移学习,实现对II 级和III 级胶质瘤的分级ACC为0.89。Ge 等[22]基于2D CNN 模型,在295 例BraTS2017 数据集上,考虑4 个模态MRI 图像作为输入,在测试数据集上实现LGG和HGG分类的ACC为0.91。Shahzadi 等[23]使用VGGNet-LSTM 模型,在60例BraTS2015数据集上,考虑单模态FLAIR图像作为输入数据,在测试数据集上实现LGG 和HGG 分类ACC为0.84。与前述研究相比,本文在BraTS2020数据集上,考虑4个模态的MRI数据和肿瘤MASK区域作为模型输入从头开始训练,在验证数据集上,ACC和AUC分别为0.93和0.97,在测试数据集上,ACC和AUC 分别为0.89 和0.93。本研究采用的数据是目前公开可以利用的数据,虽然与其他研究者用的数据不同,但初步结果表明与其他研究者的结果相近。

表7 不同模型预测脑胶质瘤分级结果比较Table 7 Comparison of the performances of different models in glioma grading
本文利用BraTS2020 公开数据集,应用3D 深度残差网络在多模态的MRI 图像上实现了对高、低级别脑胶质瘤的自动分级。研究采用的网络模型不局限于对脑胶质瘤的自动分级,还可以实现对其他肿瘤的分级。同时可以应用在更多的医学图像处理领域,比如可用于脑胶质瘤基因状态预测和患者生存期预测等方面,从而为精准医疗的实现提供更多助力。

