电子病历命名实体识别技术研究综述
吴智妍,金 卫,岳 路,生 慧
山东中医药大学 智能与信息工程学院,济南 250355
电子病历(electronic medical records,EMR)[1]由于快捷、易于统计和处理的特点在大数据分析在医学中的应用中取代了传统的纸质病历。目前大部分的电子产品医学病历都采用非结构化的文本保存,其中非结构化文本蕴含着大量未被发现的病例记录以及丰富医疗工作中专业知识的内容。运用自然语言处理技术(natural language processing,NLP)对电子病历所蕴藏知识挖掘是实现电子病历结构化和信息提取主要关键技术。其中命名实体识别[2](name entity recognition,NER)是NLP 技术对电子病历的最基础任务,电子病历通常包括“症状”和“体征检查和检验”“药物”“疾病和诊断”以及“身体部位”五大类实体。对EMR 的命名实体识别可以挖掘出大量的医学知识,分析其中的病情、体征以及与诊断之间的关联,节省了大量人工成本,帮助医学决策,医疗科学研究,提升医学信息系统服务质量都具有非常重要的意义[3-5]。
许多学者对有关电子病历命名实体的研究成果进行总结归纳,并对其研究现状进行分析,推动电子病历数据挖掘领域的发展。杨锦锋等人[6]介绍了电子病历文本特点,总结了基于词典和规则的方法和机器学习两种方法对电子病历命名实体识别任务的研究。崔博文等人[7]对包括命名实体识别在内的电子病历信息抽取研究进展工作进行了论述。吴宗友等人[8]基于词典和规则、基于统计学习以及基于深度学习三类方法对电子病历命名实体识别给予论述,并探讨其对糖尿病、心脑血管疾病中的运用。
从电子病历的自由文本中提取所蕴含丰富的表达性数据将有助于临床研究。命名实体识别任务是电子病历信息抽取的第一步,准确地识别各类实体类别有助于后续信息提取工作。因此,提高EMR 医学命名实体的识别精确度和准确度仍有发展的空间。区别以上综述,本文深究深度学习方法常用网络模型与预训练模型应用于电子病历命名实体识别任务。本文归纳梳理了常用电子病历文本研究的数据集、语料标注标准和评价指标,简要地罗列基于字典和规则、传统机器学习的命名实体识别方法在电子病历中的应用,详细介绍NLP领域中深度学习方法对医学命名实体识别特征提取常见的网络结构,以及介绍了融合传统机器学习与深度学习相结合的混合模型用于电子病历实体识别,由于深度学习方法中预训练模型在命名实体识别任务中产生优异的结果,阐述了深度学习方法的通用领域预训练模型,并且概述针对医学领域的预训练模型进一步提升医学文本识别的准确度。针对训练数据短缺导致模型泛化能力弱提出解决方法,最后分析目前电子病历命名实体识别面临的挑战和未来的发展趋势。
1 常用数据集、语料标注标准与评价指标
1.1 常用数据集
数据集是研究的基础,电子病历实体命名识别研究数据大多来自各种医疗机构,或是医疗信息资源平台,再通过人为的标注进行研究,此外还有一些常用权威的数据。
(1)I2B2 2010数据集
I2B2系列评测数据在国外的有关研究成果中取得了至关重要的地位。其中应用最为普遍的是在2010年的评测数据集。I2B2 系列评测数据美国国家集成生物学和医院信息系统学研究服务中心(informatics for integrating biology and the bedside)提供,I2B2 进行的一系列开放评测工作,大大地推动了基于英文电子病历的抽取研发。I2B2 2010 数据集涵盖包含871 份出院小结,3大类具体形式分为医学现象(medical problem)、检测(test)和诊断(treatment)并且提供了标注语料库标准,为后期研究者建立语料库奠定了基础。
(2)CCKS 2017数据集
全国知识图谱与语义计算大会(China conference on knowledge graph and semantic computing,CCKS)由中国中文信息学会语言与知识计算专家委员会组织和举办。CCKS 2017数据集是在中文电子病历实体识别研究中最常见的数据集,同时是中文电子病历的国内医疗实体识别测评挑战赛所采用的公共数据集。CCKS2017的数据集人为地标注了表1五类实体数据。
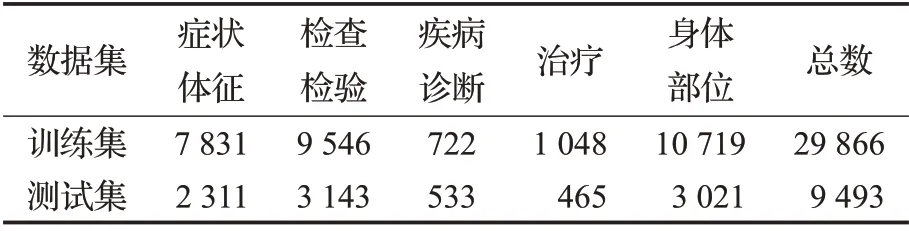
表1 CCKS 2017语料统计数据Table 1 CCKS 2017 corpus statistics
(3)CHIP 2020数据集
CHIP 2020 数据集测评任务一——中文医学文本命名实体识别,由北京大学计算语言学教育部重点实验室、郑州大学信息工程学院自然语言处理实验室、哈尔滨工业大学(深圳),以及鹏城实验室人工智能研究中心智慧医疗课题组联合构建。总字数达到220 万,包含47 194 个句子,938 个文件,平均每个文件的字数为2 355。数据集中涵盖了504 种最常用的儿科病症、7 085种人体部位、12 907种临床表现、4 354种医学程序等九大类别的医疗实体。数据集中的语料来自临床儿科学,训练集包含1 500条医疗记录,验证集为5 000条,测试集Test1、Test2分别包含3 000、3 618条医疗记录。
(4)瑞金医院糖尿病数据集
数据集将是2019 年阿里云天池实验室所推出,内容来自中文糖尿病研究领域的权威杂志,时间跨度超过将七年。内容涵盖了中国目前许多糖尿病的科学研究和热点,数据集的标注者都具有医学背景。尽管因为发布日期较短等因素,目前暂未获得普遍应用,但依然是一个具有权威性的中文开源大数据集。依托于该数据集,包括医生、科研人员、企业开发者就能开展用于临床诊断的知识库,知识图谱,辅助诊断等产品开发,进一步探索研究糖尿病的奥秘。
(5)其他数据集
除这些公共数据收集外,有部分学者还利用了一些内部数据或临床数据收集,进行了电子产品病史的实体鉴别研究工作,例如,曲春燕等人[9]通过与哈尔滨医科大学第二附属医院建立的合作关系,收集了来自医院35个主要科室和87个细分科室的992份电子医疗记录,各科医疗人员参与数据标注工作,为数据集的质量提供保障。杨晓辉医生[10]曾对新疆某三甲医院提出的500余名冠心病患者的出院小结经行预处理,包括了300个诊疗记录作为训练集,测试集中包括了200 篇的诊疗记录。参照2014年美国临床信息学研究中心I2B2发布的冠心病危险因素标注语料库,制定了标注指南并开发了危险因素语料库标注工具,由两名临床医生完成了预标注和正式标注工作。最后在XML 文件中存入入院记录、治疗方案、检验结论、出院小结、出院医嘱内容。表2总结了用于命名实体识别的数据集。
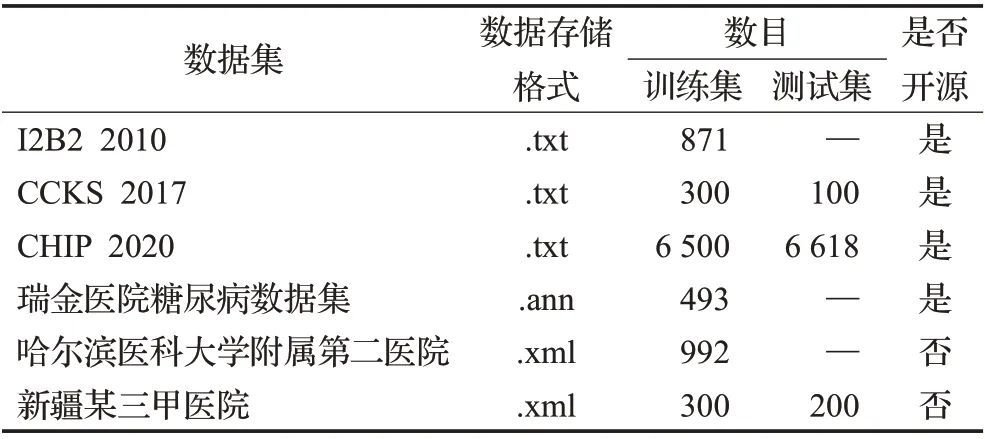
表2 电子病历实体命名识别常用数据集Table 2 Common datasets for naming and identifying entities in electronic medical records
1.2 实体识别语料标注标准
语料库的建立是命名实体研究的基石。美国国家集成生物技术和临床信息系统研发中心已于2006年根据对不同病例及风险因素识别特征等情况建立了相应的语料库,而中国的知识图谱与语义计算大会(CCKS)也针对中文电子病历系统建立了文本语料库资源。哈尔滨工业大学的研发队伍在借鉴了国外电子病历标注规范的基础上,也提供了一个较为完善的中文电子病历系统命名与实体标注方法[9-12]。目前,对命名实体的标签标注主要使用BIO 表达方式[13-14],中文文本命名实体识别相较于英文命名实体识别来说更加困难,因为英文中每个词由空格区分,而中文中词与词之间是没有明显的边界的。后来Uchimoto 等人[15]将独立元素加入之中成为BIESO 表达方式有利于中文实体语料标注。B(Begin):指示命名实体组成的起始词汇;I(Intermediate):指示由命名实体中间组成部分的词;E(End):指示命名实体构成的结尾词汇;S(Single):指示单个词汇组成的命名实体;O(Other):指示与命名实体所构成不相干的词汇。BIESO语料标注方式如表3所示。

表3 BIESO数据标注示例Table 3 BIESO data annotation example
1.3 评价指标
电子病历的命名实体识别必须同时判断实体界限以及实体类型,所以,只有在对实体界限和实体类别的辨认结果都无误后,才能够认为命名实体鉴别的结论是准确的。电子病历命名实体识别使用的评估指数为精确度(Precision)、召回率(Recall)及其F1值。F1值是综合精确度与召回率指数的综合评估指数,用于综合反映整体的指标,是目前使用最为广泛的评测标准。其中,TP(true positive)为识别完全正确、FP(false positive)为不为该实体但模型判定为实体、FN(false negative)为应该被识别但模型实际没有识别。
1.4 本章小结
本章介绍了常用的电子病历文本数据集:I2B2 2010 数据集、中文电子病历CCKS 2017 数据集、CHIP 2020数据集、瑞金医院糖尿病数据集,还有两个他人研究未公开的数据集。以及简述语料库建立的标注标准和命名实体识别是否准确的评价指标。
2 传统命名实体识别方法
2.1 基于字典和规则方法
最早命名实体识别技术采取了基于字典匹配和规则的方法,基于字典方法先是构建医学术语字典,之后再利用匹配算法进行名称实体识别[16]。针对电子医学病历等专业性较强文献,该方法对标识作语料即领域字典的规模与质量发挥着非常重要的功能,为了提高领域医疗词典的个性化信息内容,可对电子医学病历文献、领域医疗文献等先采用分词管理,然后透过抽取带有较大词频——逆向文件频率的几个词,加入到领域字典中。2013 年Xu 等人[17]创建了336 个出院小结,并分词和标注同类实体语料,参考2010 年I2B2 数据集进行实体类别识别,把诊断划分为药物(medication)和过程(procedure),标注工作由2 名医师负责,而且增加了人体部位(anatomy)这个类型。为了展开中医病历名称实物研究,针对肿瘤相关的生物信息系统,Wang等人[18]在肿瘤病案命名系统中进行了一系列的研究,该论文共包含复旦大学中山医院恶性肿瘤患者的115份手术记录,并根据临床医学需求定义了12 个实体类别,其中两个医生分别标记,最后将961个实体标记出来。词典匹配法虽然有很高的识别率,但该方法需要人员具有专业医学知识,以及构建语料库需投入大量的人力。
与词典方式不同,基于规则的方法是一般是在分析实物的特征后,然后构造人工规律,利用实物的上/下文信息进行匹配。Kraus等人[19]建立大量临床记录中的药物、剂量等医学实体。使用基于规范的方式来完成命名实体识别工作,在较小型的语料测试中具有较好的有效性且迅速。
2.2 统计机器学习方法
根据上述问题,研究者们给出了统计机器学习的方法,并逐步形成了对命名实体识别的主要方式。基于数据训练的方法主要是通过在给定的数据中抽取出相应的词特征,并利用机器学习的算法迭代更新参数,由此来构建模型,然后再把测试数据集注入到已经训练好的词模型,由模型算出下一个词中所命名实体的概率,若概率超过模型中某值时,则判断该词为命名实体,否则不是命名实体。传统的机器学习模型,如隐马尔可夫(hidden Markov model,HMM)[20]、最大熵模型(maximum entropy,ME)[21]、支持向量机(support vector machine,SVM)[22]和条件随机场(CRF)[23]。
De Brujin 等人[24]使用HMM 模型在I2B22010 数据集上取得0.85的F1值。张坤丽等人[25]采用最大熵(ME)模型以及基于规则的方法按照顺产、剖宫产、引产、堕胎、保胎治疗,以及其他6 类对中文产科电子病历系统进行了分级,分类的F1 值达到88.16%。Doan 等人[26]特别突出了语义特征在NER的重要性,实验结果表明,基于SVM 的NER 系统在考虑语义特征的情况下能够获得最佳F1值达到90.05%。Ju[27]利用SVM从生物医疗文本中标识特定种类的名称。Tang[28]提出基于SVM 的NER系统,来标识在医疗服务收费摘要中的临床实体。另外,他还获得了2 种不同类别的词表特性(基于聚类的表达特征和分布表达特性),并将它们与SVM的临床NER 系统集成在一块。支持向量机的多种分类模型能够有效地利用医疗问题的上下文、句子、标题等多种特征。王世昆在文献[29]给出了一个基于CRF 的中药实体命名识别模型,表明了该模型对于明清时代提出的中国古代医药症状与疾病机理的认识有着突出的优越性。Ye 等人[30]利用词性、字符、词汇特征和词的边界等特点,运用CRF技术用到了中文电子病历进行分析,取得了较好的结果。Liu 等人[31]首先构建了一个医学词典,并且采用条件型随机场CRF算法,并深入地探究了各种分类特点在中文临床文本NER任务中的意义。
2.3 本章小结
基于词典和规则的命名实体识别方式不论是通过人工方法整理规范,或是对语料库和命名实体库的整合,均需耗费巨大的财力与时间;且泛化能力极差,使用到其他的应用领域则必须重新制定词典,制定过多的规则会导致规则之间互相冲突。
相对于之前的方法,基于统计机器学习方法识别能力有了很大的提升。在开放性数据集中,由于新词和语言表达的多样性,采用统计机器学习虽然可以从小型标注数据集获得较好的结果,但在大量的语料库中,遇到更加复杂的语义,特别是在处理特定的词语组合和同义词等问题,其效果仍具有明显的劣势。
3 深度学习方法
近年来,深度学习的方法越来越成熟,通过构建深层结构,可以自动提取文本中的局部和全局特征。常见的深度学习中模型一般有卷积神经网络(convolutional neural network,CNN)[32]、循环神经网络(recurrent neural network,RNN)[33]、长短期记忆网络系统(long short term memory network,LSTM)[34]、双向长短期记忆网络系统(bi-directional long-short term memory,Bi-LSTM)[35]和自注意力机制(self-attention mechanism)[36],其本质是通过使用大量无监督数据构建多层神经网络模型。
3.1 卷积神经网络
卷积计算神经网络(CNN)是Lecun 在1989 年提出[32],由输入层、卷积层、池化层、全链接层四部分组成。CNN在NLP中对词向量输入进行的特征提取是一重要方法。在词嵌入层,CNN 将EMR 中的所有单词转换为词矢量,并把生成的单词矢量矩阵作为CNN 的层。在卷积层,利用不同的过滤器对向量阵的卷积操作,可以得到不同的局部表示。最大池化完成后,将提取到的多个局部表示进行了首尾连接。最后,通过全连接层,得到了EMR与疾病的关系。图1为CNN对文本特征提取示意图。
因为CNN 共用卷积核,对更高维数据处理并无压迫,并且运行速度极快。然而CNN在提取特征时,受限于卷积核的大小,只能集中于提取部分单词的信息,提取到的是EMR的局部特征。但是,EMR中的每个单词都包含了它的上下文的语义信息,CNN 并不能够提取整个EMR的序列信息。CNN的另一个问题是模型参数多,需要大量数据训练,当数据不足时就有问题。Wu等人[37]先对中文临床文本词向量预训练,再通过CNN 命名实体识别,提高了标准模型的准确性。Yang等人[38]使用带有单层卷积层的CNN 方法对中文EMR 进行疾病诊断;Li等人[39]提出了基于微调的CNN对中文儿科EMR进行疾病诊断。Yin 等人[40]利用CNN 获得字符的特征数据,并通过自注意力机制的文字间的依赖关联等特性,来确定了医学电子病历的相关实体。
3.2 循环神经网络
循环神经网络(RNN),由输入层、循环层和输出层构成,是一种浅层、不断重复的结构,是应用于自然语言处理(NLP)深度学习中最常使用的算法。经网络的模块A,开始读出某个输入Xi,并产生某个值hi,如图2所示。循环可以让讯息能够在当前步中传送到下步,而RNN 也可被认为是一种神经网络的多次重复,因为每个神经网络组都会将消息传递给下一次。
ArunKumar 等人[33]提出了最先进的深度学习循环神经网络(RNN)模型来预测国家累计确诊病例、累计治愈病例和累计死亡人数。基于集成的CNN-RNN 框架Zhou 等人[41]分析患者-医生生成的数据,便于患者在线查询。Al-Rakhami 等人[42]提出了卷积神经网络(CNN)和循环神经网络(RNN)的组合架构,用于通过胸部X光片诊断COVID-19。RNN 通过线形顺序结构形式不断地从前往后收集输入数据,但是这个线形顺序结构形式在相反传递的时刻面临着优化困难等问题,而且由于相反传递路线很长,极易造成严重的阶梯消失或阶梯爆炸现象。
3.3 长短期记忆网络
长短期记忆网络(LSTM)提出有效克服了长距离依赖问题,并对模型梯度消失问题有一定程度上缓解。LSTM 作为RNN 的变体现广泛应用于自然语言处理(NLP)领域[34]。LSTM也在RNN的基础上引进了“门控”的选择性机制,分别为遗忘门、输入门和输出门,从而有选择性地保留或删除信息,以能够较好地学习长期依赖关系。在LSTM 里将原来记忆里的值乘上某个值后再加上输入的值再放到单元里,它的记忆与输入都是相加的,所以不像RNN在每个时间点都会被覆盖掉,只要前一时刻的信息一旦被格式化掉,影响就消失了,但是在LSTM里的影响一直会存在,除非遗忘门把记忆里的信息清洗掉。LSTM网络基本结构图如图3所示。
Liu等人[43]使用LSTM实体识别取得F1值为87.66%。LSTM能够解决一百个量级的排序,但是对于一千个量级,甚至更长的排序则依然会变得非常棘手,因为计算量过于费时。而且每一个LSTM的工作单元里都有4个全连通层(MLP),所以一旦LSTM 的工作时段跨越较大,而且网络范围又非常深,这个运算量就会巨大而且费时。LSTM模式中只存在于单向传输,模式中实际只应用到了“上文”的信息内容,而并未顾及到“下文”的信息内容。在现实情景中,实体命名识别可以要应用到所有输入顺序的所有信息内容。
所以,目前电子病历实体名称识别一般采用的是双向长短时记忆网络技术(Bi-LSTM)。单向的LSTM 模型能够捕捉到从前向后传输的信号,而正向和反向LSTM信息可以同时被Bi-LSTM捕捉,使得对文本信息的利用更全面,效果也更好。
由图4中可以看出BiLSTM模型中能够让LSTM同时处理前向和后向两个方向序列,并具有各自的隐藏层,在特定的时间步长下,每一个隐藏层都能同时捕捉到过去(向前)和将来(向后)的信息[35]。这样,才能提取出比较全面的实体特征,提高网络的预测性能。Huang等人[44]采用BiLSTM 进行通用领域的命名实体识别任务,取得了优秀的效果。李纲等人[45]使用BiLSTM模型的融合Word2Vec 和外部词典信息,有效地识别中文电子病历中的疾病实体,实体识别F1 值高90.14%。屈倩倩等人[46]将《伤寒论》分为五类实体,即“症状、病名、方剂、中药名称、时间”,融合自注意力机制采用BERT 模型词嵌入,再通过BiLSTM 方法提取实体特征,将特征输入CRF模型,以达到95%的F1值。Zhu等人[47]混合了临床报告和相关维基百科页面的语料库上运用BiLSTMCRF 模型测试2010 年I2B2/VA 挑战赛数据集,模型实体识别获得F1 值达到88.60%。Yan 等人[48]提出基于ALBERT-BiLSTM-CRF 的半监督的中文电子病历NER模型,与其他模型相比,使用半监督方法可以提高识别的准确性,该模型精确度、召回率和F1值分别为85.45%、87.81%和86.61%,医疗命名实体识别更加准确和全面。然而BiLSTM 模型如果不计算出前一个时刻的结果,就没法计算下一个时刻的结果,因此会造成无法并行计算。
3.4 自注意力机制
2017 年文章Attention is all you need[36]提出的自注意力机制(self-attention mechanism)降低了对外部信号的依赖性,更善于抓住与数据或特征之间的内在关联。Self-attention 并非基于Transformer 的传统注意力机制的改变,而是一个全新的注意力机制,其设计思想来自RNN和CNN。自注意力机制应用在NLP领域,在文本中的运用主要是利用计算单词中间的交互影响,来克服长距离依赖问题。Self-attention 机理,相对于传统Attention 机制原理而言是将输入映射在三种不同的空间上,在文本中自己生成如图5所示key、value、query并且key=value=query,由文本和文本的自己求相似率再与文本自己相乘运算而得到。通过Self-attention 机理不但能够获取传统Attention 机理中不存在的源头端与目标端词与词相互之间的依赖关系,而且同样还能够有效地获得源头端或目标端自身词与词相互之间的依赖关系。
吴倩倩等人[49]利用自注意力机制提高CT图像中肾脏小肿瘤自动分割准确率。巩敦卫等人[50]在BiLSTM的隐藏层引入自注意力机制对CCKS 2017开源数据集与自建的糖尿病中文电子病历中疾病、身体部位、症状、药物、操作五类实体识别率均取得最好性能,其中身体部位相关的F1值高达97.54%。罗熹等人[51]结合多头自注意力机制准确地捕获电子病历字符间潜在的依赖权重、语境和语义关联等多方面的特征,将Embedding 层的输出独立并联地依次注入在BiLSTM 层和多头自注意力模式中,再将这两种模式的输入输出经过相互融合后注入CRF 层得出的最终结果,从而有效地提升了中文电子病历命名实体的识别能力。张世豪等人[52]对CHIP2020数据集中文医学病历运用多通道自注意力机制深入发掘句子的全局语义特性,F1值可达到85.23%。Yin等人[40]利用CNN获得的文本字符间特征信息,并使用自注意力机制捕获文字间的依赖关联特征,来确定在电子病历中的相关实体。自注意力机制能够从始而终地获取全局与局部联系。Self-attention机制模型复杂度远低于CNN 与RNN 网络,参数少运行时间更为快速,最突出的特点是可以并行运算大大提高了计算效率。然而,自注意力机制无法学习序列中的顺序关系,目前可以通过与预训练模型相结合来改善该不足。
3.5 混合模型方法
近年来,研究者利用合理地集成词典、规则、统计机器学习、深度学习等新技术,以提高NER模型稳定性与实体识别能力。
Wei等人[53]单一疾病名实体识别引入条件随机场和双向递归神经网络,其最终F1 值达到了84.28%。龚乐君和张知菲[54]给出了一个词典和CRF 双层标准的电子病历实体识别方式,F1值达97.2%。陈德鑫等人[55]使用CNN-BiLSTM 模型识别医学相关实体。李丽双等人[56]提出CNN-BLSTM-CRF模型识别生物医学实体。Li等人[57]从中国电子病历数据库中提取语料库,去除“急性上呼吸道感染”以外的7 大类疾病测试模型的有效性,再进行八大类以及数据集中63种疾病来测试模型的泛化能力,根据原始语料库构建医学词典,作为分词的外部专家知识,再使用单层CNN嵌套的5倍交叉验证方法进行训练,提高了文本分类的准确度。Tang等人[58]提出基于注意力机制的CNN-LSTM-CRF模型对CCKS2017_CNER数据集临床文本分为疾病、症状、检查、治疗、身体五大类实体识别,经过测试识别症状、检查F1值分别达到95.60%、93.57%。目前,大多数的基于实体识别的任务处理方法都是利用BiLSTM对字或词向量特征提取,然后将其输入到CRF 模型中来进行序列参数的优化。与以前只采用CRF 模式模型相比,BiLSTM-CRF 无需人为进行特征处理,利用LSTM 网络抽取文本特征,实现了对句子层次的理解。近几年,在电子病历中应用BiLSTM-CRF模型的研究日益受到研究者重视[59-60]。
Zhu等人[61]在包括临床治疗报告及与临床治疗范围有关的Wikipedia网页的语料库上先练习以上文有关单词嵌入模式,之后训练BiLSTM-CRF 模式。沈宙锋等人[62]对中文电子病历通过XLNet 预训练模型进行词向量表示,解决了同一词语在不同语境下的含义,利用BiLSTM-MAH 模型对临床文本的特征提取,最后输入条件随机场CRF 识别全局最优序列,在CCKS-2017 命名实体识别数据集数上取得了精确度92.07%、召回率91.21%、F1 值91.64%的优异结果。Yang 等人[63]共收录了240 例肝细胞癌患儿的病历记载,内容主要涉及了入院记录与出院小结,以及基于BiLSTM-CRF 训练实体识别的模型医学与实体识别,取得了85.35%的F1值。Jagannatha等人[64]将BiLSTM网络和CRF技术相结合,应用在了1 154份肿瘤患者的病历中,结果达到了整体82.10%的F1值。
3.6 本章小结
由上述可知,深度学习方法因不需要太多的人工设定,已被广泛地用于医学命名实体识别并且取得了很好的效果。对于高维数据CNN 处理起来毫无压力;RNN可以有效提取EMR前后信息;LSTM和BiLSTM网络能够较好地学习长期依赖关系,提取出比较全面的实体特征,提高网络的预测性能;引入自注意力机制(selfattention)使得识别过程中可以并行运算且大大提高了计算效率。传统方法与深度学习方法各模型相结合提高命名实体识别能力,BiLSTM-CRF是目前主流的特征提取模型。表4 对比分析各模型自身的特点与优势及局限性,表5总结各混合模型在电子病历命名实体识别的结果。

表4 基于深度学习的命名实体识别方法对比Table 4 Comparison of named entity recognition methods based on deep learning

表5 混合方法命名实体识别的评价指标结果Table 5 Results of evaluation metrics for hybrid approach to named entity identification
4 预训练模型
在NLP模型的深度学习中,词嵌入通常被用作第一个数据预处理层,即每个词都需要嵌入到一个数学空间中,才能得到向量词的表达式。因为模型学习到的单词向量可以捕获一般的语义和句法信息,单词嵌入可以在各种NLP任务中产生优异的结果[65-66]。
4.1 通用领域预训练模型
通用领域中常用的词向量表示有两种:一种为非语境的词嵌入One-hot、Word2vec、Glove模型,另一种为结合上下文词嵌入ELMo、BERT、XLNet模型。
4.1.1 One-Hot编码
One-Hot 编码又名“独热编码”,One-Hot 编码使每个词都对应唯一的单词向量,且任意两个词之间是独立的。尽管One-Hot 编码能够很好地解决离散数据的难题,而且能够对特征进行一定的扩展。但语料库的大小决定了向量维度,随着词库的规模越来越大,向量维数也会随之暴涨,从而引发维度灾难。由于One-Hot编码是一个词袋模型,每个词向量之间相互独立,具有高稀疏性、离散型性的特征,因此词之间的关联性会被忽略,带来“语义鸿沟”问题。
4.1.2 Word2Vce模型
2013 年由Mikolov 等人[67]提出了Word2Vce 词向量表示模型,Word2Vce是一种浅层神经网络预处理模型,将大规模未标注的语料库转换为低维、稠密词向量。Word2Vce 模型充分利用语境信息,通过联系需要理解词语的上下文即可理解该词语在文本中的意思。
杨红梅等人[63]基于Skip-gram 方法的Word2Vce模型将500 份电子病历训练出128 维词向量,再使用Bi-LSTM+CRF模型对其命名实体识别达到总体0.805 2的F1 值。黄艳群等人[68]研究以Word2Vce 模型训练北京市某三甲医院144 375万条住院患者EMR,以低维的词向量表达病人特征(疾病、药物、实验室指标),从某种意义上解决了病人初始结构特征的高维特征,同时有效地利用特征低维向量来显示特征之间的关系。Word2Vce模型克服了“维度灾难”现象,但Word2Vec 窗口是以局部语料库为基础的,所以当在文本中使用Word2Vec 词矢量表示时,则会失去上下文中各实体之间的关联,仅提取语句层次的信息,造成同一实体在上下文中的标注不一致。
4.1.3 Glove模型
随后,Pennington[69]又提出窗口基于文字全局语料Glove 模型,其收集词语在文本共现的全文信息并对词语进行单词矢量表达,同时考虑了局部信息和整体的信息,以此增加了对文字中同一实体标注的准确性。
吴迪和赵玉凤[70]提出融合LDA 和Glove 模型的病症文本聚类算法,将电子病历命名实体分为“呼吸科”“消化科”“神经科”“骨科”“皮肤科”和“其他”6 大类。马满福等人[71]采用Glove 模型与LCN 神经网络相结合用于医疗问题的特征提取,对某三甲医院的15 000份住院病历进行词向量训练,采用Glove词向量对词汇的共现率进行统计,得到的词特征信息更加完整,效果比其他的方法更好,准确率稳定在89%左右。巩敦卫等人[50]通过维基百科、医疗论坛、相关医学书籍获取大量医学文本,分词训练后使用Glove 模型得到100 维的字词向量,作为下游任务的输入层。Glove模型则和Word2Vce模型很相似,将同一文字中不同语义的词汇表达为一个向量表示,而不会考虑同一词语在不同的语境下产生的不同意义。
4.1.4 ELMo模型
前面介绍模型虽可以捕捉每个词的表征,但它们无法捕捉上下文中的高层次概念,如句子结构、语义和词语歧义等。为了解决这个问题。Peter等人[72]在2018年提出了ELMo 模型,将一个句子或段落输入模型,它可以通过上下文语境动态地改变词向量编码,表达多义词的多种语义。
在某些特定的领域中,专有的词典的词向量ELMo模型比通用的ELMo[73]具有更好的性能。在临床NER任务中,Johnson 等人[74]使用ELMo-LSTM-CRF 模型在医学语料库重症监护数据集III(MIMIC III)上进行了预训练,结果显著优于具有通用ELMo 的相同模型。Yang 等人[75]对2010 年I2B2/VA 挑战赛数据集命名实体识别提出了一种基于注意力的ELMo-BiLSTM-CRF 模型,取得88.78%的F1值。实验证明,ELMO模型在医疗领域数据上的表现也超过之前传统语言模型。ELMO模型采用LSTM 的特征提取方法,具有较大的学习周期。基于上下文利用向量拼接方法进行特征的融合效果较差,且有伪双向特性。
4.1.5 BERT模型
Devlin 在2018 年 提 出BERT 模 型[76],在 医 学 领 域BERT 模型对电子病历命名实体识别表现非常出色,如今大部分电子病历命名实体识别研究在词向量表达时都会使用BERT模型。BERT模型能够在大量的无标记非结构化文本中获取大量的语义信息,同时还利用了多层的Transformer为输入序列的各个词汇建模上下文语义信息,使同一词汇在不同的上下文中根据含义不同而有不同的词向量表达。图6所示对某电子病历“慢性胃炎,饮食不节”通过BERT模型词向量表达。
李正民等人[77]提出了基于BERT 技术的BERTBiLSTM-IDCNN-CRF 多特征融合技术,对CCKS 2020中文电子病历的6类命名实体进行了研究,该模型获得最佳F1值达到89.68%。该模型在嵌入层使用BERT生成词向量并且自动学习丰富的语义信息,使得命名实体识别能力有明显的提高。Vunikili等人[78]对2020年ber-LEF的癌症文本进行命名实体识别(NER),在不使用任何特征工程或基于规则的方法的情况下,通过BERT模型对西班牙语临床报告中提取肿瘤形态,取得了73.4%的F1分数。为了确保训练效率,BERT模型对输入的句子最大长度限制为512个字。相对于传统的语言模式,BERT 的每次训练只能得到15%的标记被预测,从而使模型的学习过程变得更为复杂。
4.1.6 XLNet模型
XLNet[79]是CMU 与谷歌Brain 于2019 年推出的一种新的NLP预训练模式,在模型中输入句子[患,有,冠,心,病],随机生成一种排列[冠,病,患,心,有] 。根据新的排列方式得到图7所示的XLNet的掩码机制图,第一行为“患”,因为在新的排列方式中“患”在第三个,根据从左到右方式,“患”字可以获得信息“冠”“病”字(在图3中“冠”“病”位置用蓝色实心表示),其他字则被遮掉(图3中其他字位置用空心表示)。以此类推。第二行字为“有”,因为“有”在新排列是最后,就能看到“冠”“病”“患”“心”字,于是“冠”“病”“患”“心”字所在位置是蓝色实心。第三行字为“有”,因为“有”在新排列中第一个,看不到其他信息位置,所有字全部遮盖掉。
Yan 等人[80]证明在NER 任务中预训练模型XLNet优于BERT 模型,比较在CoNLL-2003 数据集XLNet-BiLSTM-CRF、BERT-CRF 和BERT-BGRU-CRF 模型命名实体识别任务,Yan提出的XLNet-BiLSTM-CRF模型取得97.64%的F1 值。Wen 等人[81]收集了1 000 条中医医嘱,证实了XLNet预训练模型在医学领域的命名实体识别任务中的优异性,与单一或联合模型在同一数据集上的实验结果相比,XLNet 预训练模型的F1 值分别提高了9.65%和8.71%。XLNet 模型可以提取丰富的语境信息,从而在长文本输入类型的NLP 中表现出多种优势,可以根据句子的语义来充分表示每个标记。但XLNet预训练模型相对于BERT需要更大的计算能力以及高质量的语料。
4.2 生物医学领域预训练模型
前文介绍的预训练模型通用于一般领域的语料库。然而,由于生物医药领域的文字中含有大量的专业术语和专有名词,因此基于一般语料库的预训练模型在医疗文本的挖掘过程中常常表现不佳。近来研究者们提出了针对医学命名实体识别任务预训练模型可分为以BioBERT为代表的基于医疗领域数据进行继续预训练;基于医疗领域数据从头进行预训练Med-BERT 模型;以及MC-BERT 为代表的基于医疗领域数据的自监督训练三类。
4.2.1 BioBERT
Lee 等人[82]在2020 年提 出的BioBERT 预训练模型是为了更有效地进行医学文本挖掘任务,该模型与BERT模型结构几乎相似。BioBERT预训练模型如图8所示,首先运用BERT 的加权对BioBERT 进行初始化,在模型训练阶段采用已经通过英语维基百科和Books-Corpus 语料库训练完成的BERT 模型,进一步运用PubMed摘要和PMC全文文章对BERT模型训练。
BioBERT 预训练模型对于命名实体识别任务采用精确率、召回率、F1值作为评价指标。Lee等人[82]采用了多种组合和不同数量的通用领域语料库和生物医学语料库对BERT、BioBERTv1.0、微调BioBERTv1.1 进行对比,结果表明在NCBI disease、2010 i2b2/VA、JNLPBA等8 个数据集中BioBERT 的得分均高于BERT,微调BioBERTv1.1 比BioBERT 平均F1 值提高0.62%。Yu 等人[83]实现EMR 中的临床问题、治疗和检查进行自动标注,使用BioBERT模型对I2B2 2010挑战数据集词向量表示,再通过BiLSTM-CRF模型完成电子病历命名实体识别任务,达到87.10%的F1 值。Symeonidou 等人[84]提出基于BioBERT-BiLSTM-CRF 的迁移学习的NER 模型,实验数据表明增加BioBERT 预训练模型相比于前人的NER模型在三个不同的医学数据集上F1值分别提高了6.93%、10.46%、13.31%。并且由于引进迁移学习,Elsevier’s gold set 数据集只需要大约7 000 个标记的语句就可以达到80%以上的F1 分数的结果,这意味着迁移学习和BioBERT预训练模型提高了实体命名识别的准确性。Naseem等人[85]提出了一种新的医学命名实体识别(BioNER)方法,通过分层融合的BioBERT 模型对医学文本进行词向量表示,在BC5DR 数据集上识别药物实体F1值达到94.24%。实验对比表明分层融合的BioBERT词向量模型能够更有效的捕捉上下文,处理语义、新词等问题。
BioBERT 是第一个基于生物医学语料库的预训练模型,同时是将BERT 技术运用到临床医学中的关键。BioBERT能够识别出BERT无法识别的医学命名实体,并能准确地确定实体的边界,使得可以识别更长的命名实体,仅仅只对结构进行小型的改动,BioBERT 就可以比先前的模型更好地识别医学实体。
4.2.2 Med-BERT
针对生物医学领域的Med-BERT 预训练模型由Rasmy等人[86]于2021年提出,不同于之前先采用通用域语言模型再结合混合域方法的预训练模型,如图9所示Med-BERT 则是从一开始就使用PubMed 的摘要与PubMed Central(PMC)全文文章作为预训练语料库,并且对于一些医学领域的专业术语或通用域中所认为的新词该模型也能完整表示,在大部分的任务中能取得与BioBERT相同的效果。
Rasmy等人[86]评估了Med-BERT对糖尿病心力衰竭和胰腺癌的预测。实验结果表明Med-BERT对疾病预测任务中的有效性,有助于减少数据标签的负担,大幅提高基础模型在小样本上的性能。杨飞洪[87]采用Med-BERT模型对中文电子病历命名实体识别(CEMRNER)数据集“身体”“疾病和诊断”“药物”“检验”“影像检查”“手术”6种类型实体识别。Med-BERT在CEMRNER数据集总体F1值达到82.29%,其中对“药物”实体的识别率最高,取得了90.48%的F1 值。Gan 等人[88]融合无监督增强文本和标签策略Med-BERT 模型对CCKS2021 数据集中文电子病历词嵌入表示,提高了实体识别命名的准确性。
相比于只采用摘要的Med-BERT模型,由于全文文章的篇幅较大所含的噪声更多,语料库包含摘要与全文文章的Med-BERT 词向量表示效果往往会有所降低。尽管训练全文文章对模型有所帮助,但想要运用更多的信息,还需花费更多的训练时间。研究者通过多次实验得出,通过将训练时间延长60%,选用摘要与全文文章的Med-BERT 比仅仅使用摘要结果更好。Med-BERT是一种全新的在特定的生物医学领域从头学习的神经语言模型,比以往的各种预训练语言模型效果更好,是目前生物医学领域预训练的前沿技术。
4.2.3 MC-BERT
MC-BERT模型由阿里巴巴Zhang等人[89]在2020年提出针对中文医学文本的预训练模型。MC-BERT 将BERT模型作为基础模型,用医学数据对模型进行训练,同时利用生物医学语料与生物医学实体知识同时注入到表示模型中进行训练,进一步引入了领域知识。生物医疗领域的短语通过自动词条和阿里巴巴知识图谱获取,此外使用一个二分类的模型(fasttext)用来识别是否真的是生物医疗领域的短语。
MC-BERT 生成器仅分成k条路径,降低了系统的复杂性。在一个句子中,元控制器首先将抽样的词语替代一小节标签来打破该语句。然后,在每个位置创建k个候选选项。生成器将受损的语句用作输入,代入k个候选词进行预测来纠正每个词。MC-BERT测试类似于实际应用中的多项选择型完型填空题如表6所示,基于上下文的深度语义信息做出相应的预测。
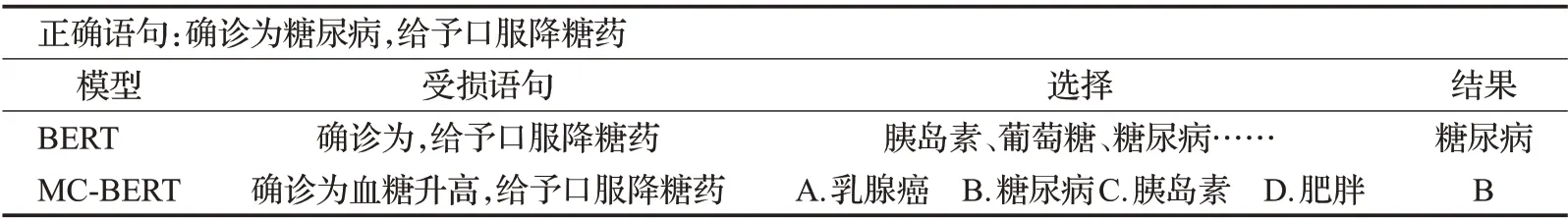
表6 MC-BERT测试示意表Table 6 MC-BERT test schematic table
总之,MC-BERT 能够很好地兼顾了模型学习中的语义信息和训练效率。对于命名实体识别任务,在cEHRNER 和cMedQANER 数据集测试下,与其他经典的预训练模型相比,MC-BERT 平均F1 值达到90%。MC-BERT是一个预先训练好的具有生物医学领域知识的语言词向量模型,与此同时也是对中文医学文本处理方向的一个开端。
4.3 本章小结
本章从通用领域与生物医学领域两个角度介绍词向量模型,通用词向量模型的发展不断提高医学命名识别能力,表7 对通用领域词向量模型进行综合分析,从特点和优缺点方面进行对比总结。随着医学文本的数量急剧增长,通用领域的预训练模型无法更好地表示生物医学领域中的专业术语,医疗词汇的长尾分布也很难从普通语料中学习,4.2 节介绍的在生物医学领域中BioBERT、Med-BERT、MC-BERT三种主要预训练模型能够有效表达生物医学词汇,进一步提高命名实体识别准确度,三种医学领域预训练模型性能分析如表8所示。

表7 通用领域预训练模型总结Table 7 Summary of general domain pre-trained models
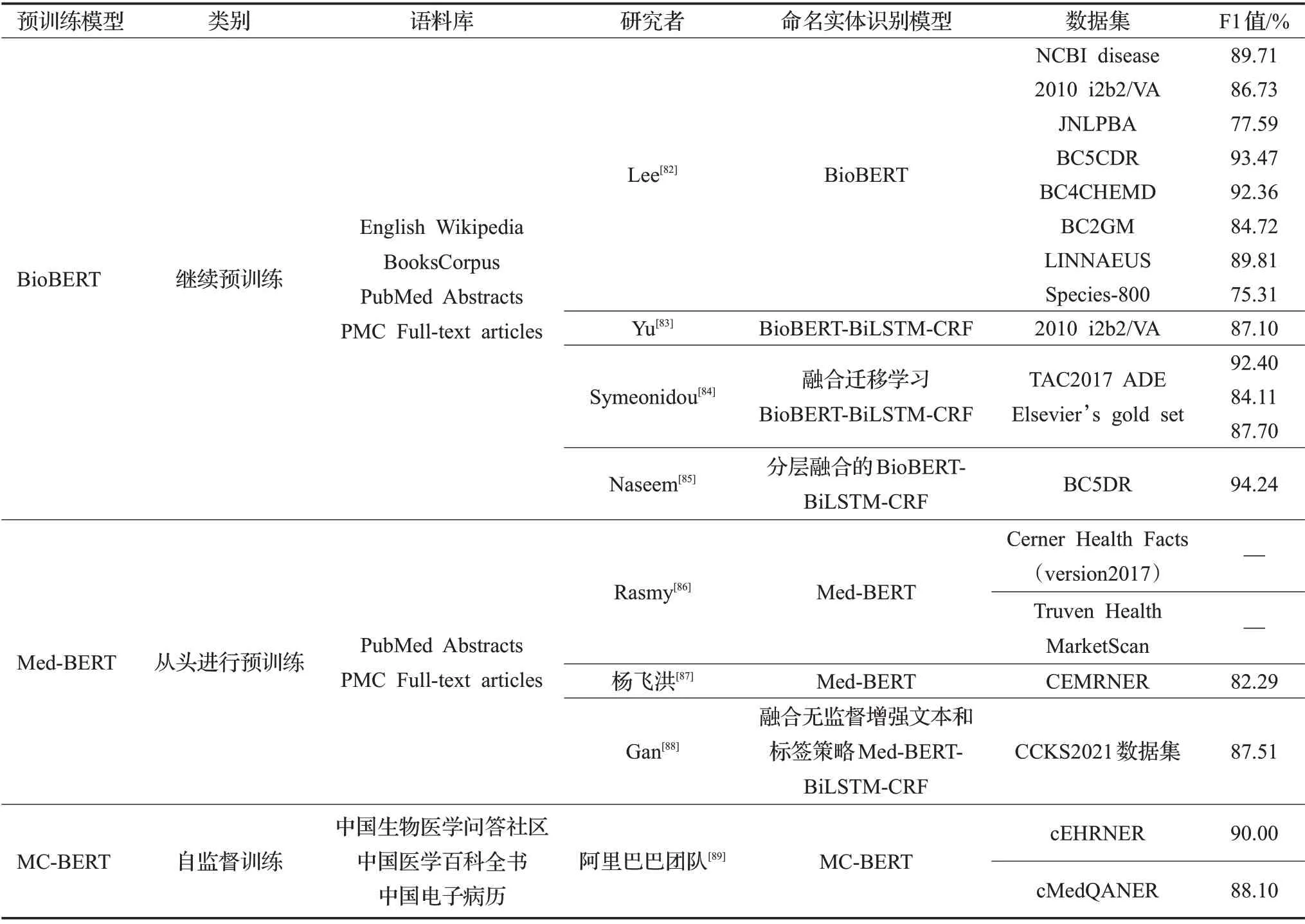
表8 医学领域预训练模型性能分析Table 8 Performance analysis of pre-trained models in medical field
5 小样本问题的处理
传统的深度命名实体识别方法,依赖于大规模且具备已标注的训练数据。电子病历系统由于牵涉患者隐私的特点,使得生物医学命名实体识别数据集相对匮乏,大部分的生物医学挖掘模型都不能完全发挥深度学习的能力。如今,引入迁移学习、小样本学习和多任务学习有效解决了标注数据稀疏以及模型泛化能力差的问题。
5.1 迁移学习
迁移学习是将一个任务或领域学到的知识转移到另一个任务或领域应用。唐观根[90]提出弱迁移方法,将新闻领域数据特征迁移到医疗领域,实现总体88.76%F1值,缓解了中文电子病历语料库缺乏的问题。Giorgi[91]将训练源数据集SSC所有参数运用基于深度神经网络的迁移学习提高了目标领域GSC“药物”“疾病”“物种”“基因/蛋白质”四种生物医学实体类别,利用迁移学习减少医学命名实体识别所需的手工标记语料的数量。Lee 等人[92]使用MIMIC 作为源数据集训练ANN模型,对于i2b2 2014目标数据集,应用迁移学习后仅使用5%数据的作为训练集,命名实体识别F1 值达到了93.21%。将迁移学习引入到命名实体的识别中,通过使用源领域资源数据进行目标领域任务模型创建,以增加对目标领域的标注信息量和减少目标领域建模中对标注数据量的要求等,很好地解决了命名实体识别任务存在数据匮乏的问题。
5.2 小样本学习
小样本学习旨在使用少量标记的数据(即先验知识)完成新的任务。Hofer团队[93]从MIMIC III和i2b2 2010数据集中随机抽取10 个样本作为训练集,识别i2b2b 2009命名实体,获得平均F1值78.07%。Lara-Clares[94]采用小样本学习对数据集MEDDOCAN shared task 500例样本进行29类命名实体识别达到90%F1值。由于深度学习模型含有许多隐藏层和神经网络,需要海量数据训练提高模型的准确性,在医学领域标注数据较少并且需要专家花费大量精力,因此通过小样本学习缓解数据稀疏问题。在识别电子病历中各类实体任务中引进小样本学习,通过少量的标记数据对该任务建立模型,对不同的类别的实体进行区分,同时能在不改变模型参数的前提下对新的类别进行识别,大大减少了人工标记所带来的成本。
5.3 多任务学习
通常用一个复杂模型解决单个任务,多任务学习目的是通过多个相关的任务包含的知识同时并行学习提升模型泛化效果。在多任务学习中,多任务间的参数共享会减弱网络对单个任务的学习性能,减少过拟合,提高泛化效果。另外,当学习某项任务时,与此项任务无关的部分将会被视为噪声,噪声的加入则能改善其泛化能力。Yang 等人[75]对电子病历中疾病实体识别和症状的严重程度抽取双任务采用多任务学习,运用疾病实体以提高患者症状严重程度的性能。Xue等人[95]把BERT模式结合在多任务学习中,增加了共享参数层的特征表达能力,实验结果表明,实体识别的F1值达到了96.89%,与之前的模型相比,F1值提高了1.65%。电子病历命名实体识别与修饰识别和关系抽取具有较强的关联性,结合这三个任务采用多任务模型,利用实体和关系之间紧密的交互信息,提升命名实体识别的准确率。
5.4 本章小结
综上所述,迁移学习与小样本学习能够在有限的数据中完成命名实体识别任务,有效处理深度学习所需标注语料不足的难题。多任务学习通过参数共享并行学习相关任务的知识以提高准确预测命名实体识别和处理其余任务的能力,有效解决因数据稀缺导致模型泛化能力较弱的问题。在未来的研究中,迁移学习、小样本学习和多任务学习如何应用在医疗领域具有重要意义。
6 总结与展望
将自然书面语言的非结构电子病历转换成结构化数据是医学信息研究当下一大热点,在医学领域,命名实体识别的首要目标是确定电子医疗记录中的治疗方法、疾病类型、检查方法、症状等实体。随着深度学习技术的迅速发展,使用词向量融合传统机器学习与深度学习相结合尤其是BERT-BiLSTM-CRF 的混合模型用于电子病历实体识别已成为主流方法并取得了较好的效果,研究人员提出了针对生物医学领域BioBERT、Med-BERT、MC-BERT 预训练模型进一步提高识别准确率,引入迁移学习、小样本学习和多任务学习有效解决了标注数据稀疏以及模型泛化能力差的问题。当前,电子病历命名实体识别仍面临诸多挑战:
(1)公开数据集匮乏。到目前为止,虽然国外电子病历命名实体识别已经取得很大的进展。但国内使用最普遍的是全国知识图谱与语义计算大会(CCKS)公开数据集,关于中文电子病历的开源数据集稀缺,同时没有公开的关于中文生物医学技术语言资料的支持,这就给进行中文电子病历研究工作造成了一定的障碍。
(2)标注成本高。在生物医学文献挖掘任务中,由于生物医学文献语料库和一般领域文献语料库在用词和表述方面都有着很大不同。并且新的医学实体数量迅速增加,需要识别大量未注册的词汇,医疗领域对标注人员的专业性要求较高,使得医学标注大规模的语料成本非常高。
(3)医学实体多义性。目前电子病历实体识别中存在着医疗实体边界模糊,具有多义性,存在口语化、医疗实体词缩写等问题,这也是生物医学命名实体识别的主要障碍之一。
为解决目前存在的问题,未来研究可着重于以下几个方面:
(1)小样本数据识别。在训练数据不足或标注语料短缺的情况下,构建小样本数据的网络至关重要,引入迁移学习、小样本学习、多任务学习、图卷积网络、对抗生成网络、数据增强等方法,这有利于完成命名实体识别任务并提高识别准确率。
(2)加强小样本方法运用。虽然小样本学习在医学领域中得到了普遍的认可,但在标注数据较少的情况下这一方法尚未得到充分利用。在未来,创建基于小样本方法的生物医学NLP 的标准化的公共数据集为生物可以引领该领域的研究,除此之外,多次重复实验与比较研究在多个数据集性能设置也有助于命名实体识别评估。
(3)应用XLNet 预训练模型。目前XLNet 预训练模型还未广泛应用于电子病历命名实体识别任务中,XLNet预训练模型利用随机排列、双流自注意力机制以及循环机制方法产生包含语境信息的动态词向量,充分利用上下文信息有效解决医学实体一词多义的问题,提升命名实体识别的效果。
通过本文综述,基于深度学习的命名实体识别技术有效地从非结构文本中提取医疗实体,并从中挖掘出医疗知识,从而有效地推动医学学科的发展。

