基于支持向量机的重组大肠杆菌产脂肪酶MAS1的发酵建模与优化
肖阳,王永华,杨博*
(1.华南理工大学生物科学与工程学院,广东广州 510006)(2.华南理工大学食品科学与工程学院,广东广州510640)(3.广东优酶生物制造研究院有限公司,广东佛山 528226)
脂肪酶(EC 3.1.1.3)具有催化水解、酯化、酯交换等多种功能[1-3]和特异的底物选择性、高效的催化效率等特性[4],在造纸、油脂加工、制药、皮革加工等工业领域广泛利用[5]。目前,大多数已报道的脂肪酶均表现出对sn-1和sn-3位的偏好性,只有极少数来源于南极假丝酵母(Candida antarctica)、洋葱假单胞菌(Pseudomonas cepacia)、荧光假单胞菌(Pseudomonas fluorescens)和海洋放线菌(Streptomycessp.)W007[6]的脂肪酶表现出具有非区域选择性[7]。来源于海洋放线菌(Streptomycessp.)W007的脂肪酶MAS1具有耐受甲醇及高温等特点,可高效催化合成富含n-3 PUFA 甘油三酯[8-10]和生物柴油等[11-13]。徐婉莉等[11]利用固定化脂肪酶MAS1在叔丁醇体系中催化黑兵蝇幼虫油甘油解形成月桂酸单甘酯,其底物转化率最高为97.88%,月桂酸单甘酯的含量最高为70.8%。汪秀妹等[12]在真空环境下,利用固定化脂肪酶催化多不饱和脂肪酸与 sn-甘油-3-磷脂酰胆碱酯化成对人体具有多种益处的溶血卵磷脂,其sn-甘油-3-磷脂酰胆碱转化率为 93.12%,溶血卵磷脂含量为 90.77 mol%。吕雯等[13]利用固定化脂肪酶MAS1酯化大豆油脱臭馏出物与甲醇形成生物柴油,生物柴油产率为97.08%。
目前,重组脂肪酶MAS1主要在毕赤酵母中进行异源表达,蓝东明等[14]通过共表达PDI、HAC1和免疫球蛋白结合蛋白的策略成功使 MAS1活力提高了1.7倍,随后通过发酵条件优化,最终在发酵144 h时,发酵液上清中酶活力达到440 U/mL。瞿曼等[15]通过单因素实验优化了毕赤酵母表达MAS1的诱导温度和诱导pH,并进行了双碳源流加策略的优化,酶活力较原来提高了4.83倍。大肠杆菌表达系统相比毕赤酵母表达系统具有发酵周期短发酵成本低等优点,以大肠杆菌作为脂肪酶MAS1异源表达系统有望满足产业化应用的不同需求,然而通过统计学方法进行发酵条件优化的研究还鲜有报道。
统计学方法如响应面、正交设计[16]、神经网络、支持向量机(Support Vector Machines,SVM)应用于微生物发酵条件已证明具有十分重要的价值。名晓东等[17]用响应面方法在摇瓶水平优化了重组大肠杆菌产卤代烷脱卤酶培养基成分,优化后的卤代烷脱卤酶酶活力比优化前提高了 93.78%。Rani等[18]采用正交实验优化了拟根孢霉MTCC 1467产木聚糖酶的发酵参数,优化后木聚糖产率提高了 65.1%。然而微生物发酵过程是非线性的[19],响应面、正交试验设计等得到的线性模型拟合效果较差,而神经网络、SVM等算法因具有较强的非线性拟合能力而在发酵条件优化中更有价值。Sathish等[20]用神经网络算法和遗传算法(Genetic Algorithm,GA)优化了枯草芽孢杆菌RSP-GLU产谷氨酰胺酶发酵条件,将谷氨酰胺酶的产量提高了47%。
虽然神经网络算法在发酵建模领域被广泛应用,但是神经网络算法仍然无法解决收敛速度慢、易陷入局部最小值等问题[21];关于神经网络算法的神经元个数的选取也没有特定的标准,因缺乏对神经网络结构的了解,使得所建立的神经网络模型出现缺乏泛化能力和过拟合的问题[22];神经网络算法对于数据样本的大小也有一定的要求,当数据样本比较小时,神经网络算法拟合效果较差[19]。基于结构风险最小化的SVM利用简单的统计学理论解决了上述问题,它的非线性拟合性更强,且更加适合小样本数据模型的构建[23],可通过引入最小二乘法提高了模型的训练速度[24],因而被广泛应用于数学模型的构建和自动控制等领域。Zhu等[25]通过粒子群优化(Particle Swarm Optimization,PSO)算法优化了基于最小二乘支持向量机(Least Squares Support Vector Machines,LSSVM)构建的海洋溶菌酶发酵模型,将优化后的模型进行了广义预测控制,将酶活性提高了20%,酶产率提高了30%。GA基于自然选择和遗传学的机制,通过计算机模拟选择、交叉和变异的过程以求解优化问题的最优解,是一种集概率、随机和引导搜索机制的优化算法[26]。SVM和GA的结合,可通过SVM来描述各种因素间复杂的关系,在此基础上用GA找出最优解。Gao等[27]将SVM与实数编码遗传算法(Real-code Genetic Algorithm,RGA)相结合,成功使发酵过程中的青霉素滴度提高了22.88%。
目前,脂肪酶MAS1已被证明可以合成多种对人体有益的成分,例如具有杀菌功效的月桂酸单甘脂[11]和对人体有多种益处的溶血卵磷脂[12]等;在清洁能源领域,脂肪酶MAS1也可以催化油脂产业的废料高效转化成生物柴油[13]。因此脂肪酶MAS1在未来具有十分广泛的应用前景,但已报道的由毕赤酵母发酵的脂肪酶MAS1产量较低,难以满足下游应用行业的需求。为了降低生产成本和促进脂肪酶MAS1的产业化,本研究利用大肠杆菌异源表达脂肪酶 MAS1,基于Box-Behnken实验设计得到发酵数据,建立了重组大肠杆菌表达脂肪酶MAS1发酵模型。利用GA对诱导温度、诱导pH和IPTG浓度等发酵条件进行优化,以获得重组大肠杆菌高效表达MAS1的发酵条件,为重组脂肪酶工业化应用提供研究基础,其建模和优化流程如图1所示。
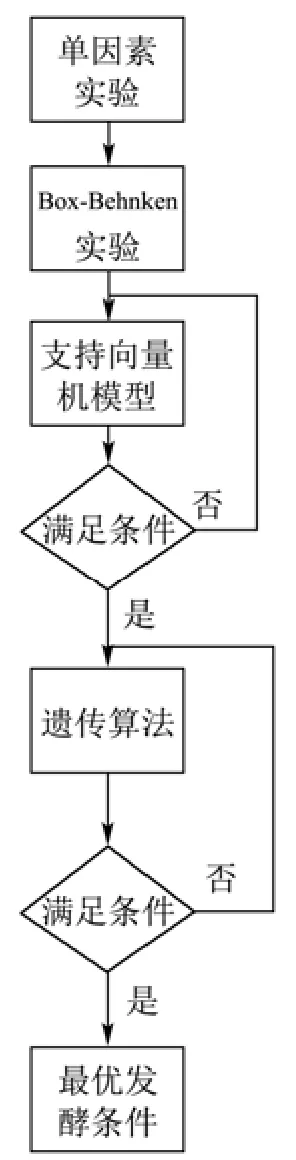
图1 MAS1发酵建模与优化流程Fig.1 The flow chart of the MAS1 fermentation modeling and optimization
1 材料与方法
1.1 材料与试剂
1.1.1 菌株与质粒
重组大肠杆菌表达菌株BL21(DE3)/pET22b-MAS1由华南理工大学生物化工实验室构建和保藏。
1.1.2 生化试剂
酵母粉、蛋白胨购自广东环凯生物科技有限公司;葡萄糖、磷酸二氢钾、磷酸氢二钾等购自国药集团化学试剂有限公司;氨苄青霉素钠购自生工生物工程(上海)股份有限公司;橄榄油购自上海麦克林试剂有限公司。
1.1.3 主要仪器设备
7 L发酵罐,上海保兴生物设备工程有限公司;H1850R台式高速冷冻离心机,湘仪离心机仪器有限公司;SCIENTZ-ⅡD超声波细胞粉碎机,宁波新芝生物科技股份有限公司;ZHSY-50WE水浴恒温振荡培养箱,上海知楚仪器有限公司;IS-A叠加式恒温振荡器,苏州捷美电子有限公司;SPX-150生化培养箱,中仪国科(北京)科技有限公司;UV-8000型双光束紫外可见分光光度计,上海元析仪器有限公司。
1.1.4 培养基
固体培养基(1 L):5 g酵母粉、10 g胰蛋白胨、10 g NaCl、18 g琼脂粉,补加终浓度为100 μg/mL氨苄青霉素钠。
摇瓶种子液培养基(1 L):5 g酵母粉、10 g胰蛋白胨、10 g NaCl,补加终浓度为100 μg/mL氨苄青霉素钠。
发酵培养基(1 L):10 g无水葡萄糖(单独灭菌),11.5 g酵母粉,19.5 g蛋白胨,4 g (NH4)2SO4、0.94 g一水柠檬酸,18 g K2HPO4·3H2O,3 g KH2PO4,1.2 g MgSO4(单独灭菌),微量元素1 mL。
微量元素溶液(1 L):2.8 g FeSO4·7H2O、1.71 g MnSO4·H2O、2.8 g CoSO4·7H2O、1.13 g CaCl2、0.2 g CuCl2·2H2O、0.14 g ZnCl2。
1.2 方法
1.2.1 OD600检测
将取得的发酵液用纯水稀释适当倍数,于UV-8000紫外分光光度计下测定其吸光度。
1.2.2 种子液制备方法
将保藏于-80 ℃冰箱的BL21(DE3)/pET22b-MAS1活化于含100 μg/mL氨苄抗性的LB固体培养基上,37 ℃过夜培养。接种单菌落到5 mL LB培养液中,37 ℃、200 r/min培养12 h。将上述的培养液按1%(V/V)的接种量接种至含有150 mL LB液体培养基中,37 ℃、180 r/min振荡培养至对数生长中后期,OD600=3~6。
1.2.3 发酵罐培养条件
7 L发酵罐装液量为3 L,接种量为5%(V/V),分批培养温度为37 ℃,通气量为3 L/min,发酵过程中溶氧不低于20%,流加氨水调节pH值为7.0。分批培养 4 h后开始流加葡萄糖,葡萄糖流速控制在12.5 g/h。在对数生长期添加异丙基-β-D-硫代半乳糖苷(IPTG),发酵22 h时停止补料。诱导阶段每3 h取样,测定细胞湿重和酶活。诱导时间分别设置为对数生长前期(2 h)、对数生长中期(4 h)、对数生长后期(7 h);诱导温度分别设置为 15、20、25、30和37 ℃;诱导pH值分别设置为6.0、6.5、7.0、7.5和8.0;IPTG终浓度分别设置为0.2、0.4、0.6、0.8和1.0 mmol/L。
1.2.4 细胞破碎
取15 mL发酵液,于4 ℃、8 000 r/min下离心5 min,倒掉上清液。菌体用等体积 pH值 7.5的10 mmol/L磷酸盐缓冲溶液重悬,随后再次离心,重复两次后,用15 mL pH值7.5的10 mmol/L磷酸盐缓冲溶液重悬菌体,于200 W下超声破壁15 min。将破碎液于4 ℃、8 000 r/min离心5 min,所得上清液即为MAS1粗酶液。
1.2.5 MAS1水解酶活检测
称取4 g橄榄油乳化液于50 mL具塞三角瓶当中,随后加入5 mL 20 mmol/L pH值7.0的磷酸盐缓冲溶液,设置一个空白组和三个平行组。向空白组中加入15 mL无水乙醇,与平行组放置于65 ℃、150 r/min的水浴锅中预热 5 min。随后向反应体系中加入稀释适当倍数的1 mL酶液,反应10 min,平行组分别加入15 mL无水乙醇将酶液灭活。待冷却到室温之后,用0.05 mol/L的NaOH溶液滴定,并记录滴定体积。水解酶活计算方法如下:
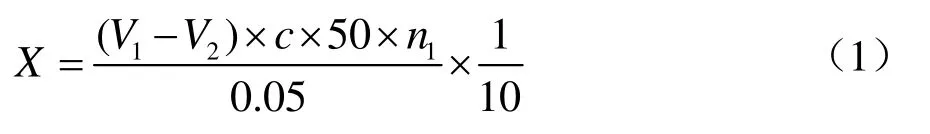
式中:
X:样品的酶活力,U/mL;
V1:滴定样品所用NaOH的体积,mL;
V2:滴定空白时所消耗的NaOH的体积,mL;
c:滴定所用NaOH的体积,mL;
50:0.05 mol/L的NaOH溶液1.00 mL相当于脂肪酶50微摩尔;
10:反应时间,min;
n1:稀释倍数。
1.2.6 发酵液中菌体湿重的测定
取2 mL发酵过程中的发酵液于4 ℃、12 000 r/min下离心3 min,去上清,所得菌体于分析天平上称重,重复测三次。
1.2.7 实验设计与预测模型的建立
本文采用R语言rsm包设计Box-Behnken实验,采用MATLAB 2020a软件的LIBSVM 和LIBSVMFaruto Ultimate工具箱构建SVM模型。在构建模型的过程中,以诱导pH、诱导温度、诱导物IPTG终浓度作为自变量,以发酵液中MAS1酶活力作为因变量构建SVM模型,使用从实验中收集的发酵数据训练出该模型,以获得BL21(DE3)/pET22b-MAS1的发酵预测模型。
1.2.7.1 BL21(DE3)/pET22b-MAS1生长曲线的绘制
将BL21(DE3)/pET22b-MAS1按方法1.2.3于37 ℃下进行培养,培养过程中不诱导,每隔1 h取发酵液样品,按方法1.2.1测OD600。
1.2.7.2 单因素试验设计
以BL21(DE3)/pET22b-MAS1为出发菌株,以诱导温度(15、20、25、30、37 ℃)、诱导pH值(6、6.5、7、7.5、8)和 IPTG 浓度(0.2、0.4、0.6、0.8、1.0 mmol/L)为单因素进行试验。
1.2.7.3 响应面实验设计
采用R语言rsm包设计Box-Behnken响应面实验,以1.2.7.2三因素得到最优水平为基础,设计三因素三水平试验设计表。
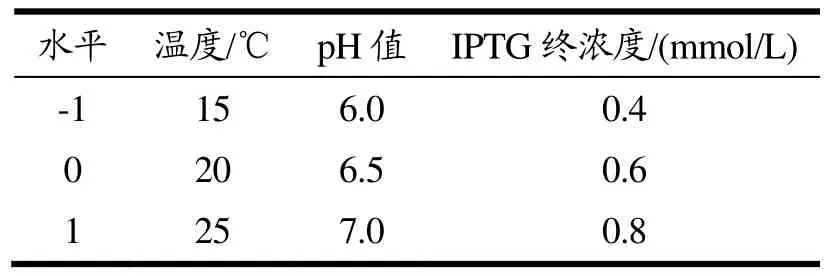
表1 Box-Behnken试验设计因素与水平Table 1 Factors and levels of Box-Behnken experiments design
1.2.7.4 SVM最优c值和g值的确定
在SVM模型构建过程中,有两个参数的选择会影响到SVM模型的精度,一个是惩罚系数c值,另一个参数为SVM径向基核函数g值。c值过大或过小会造成模型泛化能力的减弱,g值的大小会影响到支持向量的数目,进而影响到支持向量机的训练与预测的速度[28]。本研究采用了 MATLAB 2020a的LIBSVM-FarutoUltimate工具箱的SVMcgForClass函数对SVM模型的c值和g值求最优解。为了约束寻优范围,将c值和g值的范围约束在-2~2之间。
1.2.7.5 SVM模型的构建
使用MATLAB 2020a的LIBSVM工具箱进行模型的构建。将Box-Behnken实验得到的数据随机划分为2组,分为训练集和测试集。训练集包含12组数据用以训练SVM模型,测试集包含3组数据用以验证SVM模型。其关键参数c和g值采用1.2.7.4得到的最优结果。由于本研究构建的模型为支持向量回归模型,因此”-S”参数选择3,p设为0.1,考虑到变量较多,故核函数选择了径向基核函数。
1.3 GA求解SVM模型的最优解
采用MATLAB 2020a的GA工具箱对上述构建好的SVM 模型求其最优解。遗传算法的初始种群设置为50,交叉概率为0.8,终止代数为100,以1.2.7.5得到的SVM模型作为适应度函数,以1.2.7.2得到的单因素实验数据所确定的诱导温度、诱导 pH值及IPTG浓度的水平作为变量范围,其余为默认参数,通过选择、变异、交叉操作进行参数寻优。
1.4 数据处理
以上实验均设置3组平行,采用Microsoft Excel 2019软件计算所有数据的平均值和标准差。采用Origin 2018软件作图,R语言进行分析,显著性水平p<0.05。
2 结果与讨论
2.1 BL21(DE3)/pET22b-MAS1生长曲线的绘制
由图2可知,BL21(DE3)/pET22b-MAS1的对数生长前期、中期、后期分别为发酵2 h、4 h、7 h左右。
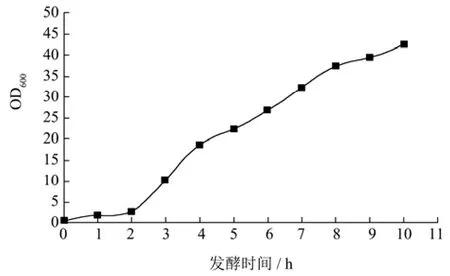
图2 BL21(DE3)/pET22b-MAS1的生长曲线Fig.2 Growth curve of BL21(DE3)/pET22b-MAS1
2.2 诱导时间对BL21(DE3)/pET22b-MAS1产MAS1的影响
Seyfi等[29]在以葡萄糖为碳源的培养基中用大肠杆菌Rosetta-gami B(DE3)生产重组蝎蛋白时,分别在对数生长前期、对数生长中期和对数生长后期进行诱导,结果发现在对数生长前期和中期蛋白含量表达不高,但在对数生长后期进行诱导时,重组蝎蛋白的表达量达到最大,为 3.5 g/L。因此分别选择了在BL21(DE3)/pET22b-MAS1的对数生长前期(2 h)、对数生长中期(4 h)和对数生长后期(7 h)添加IPTG进行诱导,以探究不同对数生长时期诱导对BL21(DE3)/pET22b-MAS1发酵表达MAS1的影响。
由图3a和图3c可知,在对数生长前期和中期进行诱导,最大细胞湿重分别只能达到76.31和95.33 g/L,均低于在对数生长后期诱导达到的最大细胞湿重141.27 g/L。同时葡萄糖的积累量随着发酵时间的延长也在快速增长,在发酵至22 h时,在对数生长前期诱导和对数生长中期诱导时葡萄糖分别积累至38.61 g/L和28.93 g/L,可能是在对数生长前期和中期进行诱导时,细胞生物量未达到足够高的密度,部分细胞未形成完整的碳链骨架,此时添加IPTG会影响细胞正常的生理功能,造成细胞代谢活力下降,对葡萄糖的利用率降低,造成未代谢的葡萄糖在发酵液中大量积累。如图3b,在对数生长后期进行诱导,有利于BL21(DE3)/pET22b-MAS1细胞的生长和目的蛋白的表达,当发酵至13 h,此时酶活力达到了1 733.33 U/mL,比对数生长前期和对数生长中期诱导分别提高了153.66%和77.47%。结果表明,在BL21(DE3)/pET22b-MAS1对数生长后期进行诱导可以提高MAS1的表达水平。
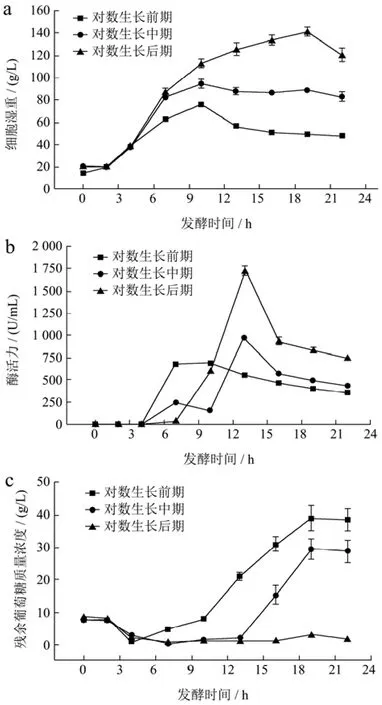
图3 诱导时间对BL21(DE3)/pET22b-MAS1发酵过程的影响Fig.3 The effect of induction time on the fermentation process of BL21(DE3)/pET22b-MAS1
2.3 诱导物 IPTG浓度对于 BL21(DE3)/pET22b-MAS1表达MAS1的影响
IPTG作为乳糖的类似物,在重组大肠杆菌表达外源蛋白过程当中具有十分重要的作用。其浓度的高低可以影响到重组大肠杆菌表达外源蛋白的效率。舒畅等[30]利用大肠杆菌 BL21(DE3)发酵生产谷氨酰胺转氨酶时,分别添加终浓度为0.2、0.4、0.6、0.8和1.0 mmol/L的IPTG进行诱导,结果发现当IPTG浓度为0.6 mmol/L时,谷氨酰胺转氨酶比酶活达到最大,为1.32 U/mg。本实验在25 ℃、pH值7.0、对数生长后期诱导的条件下,分别添加终浓度为0.2、0.4、0.6、0.8和1.0 mmol/L的 IPTG,探究了不同诱导浓度对 BL21(DE3)/pET22b-MAS1表达MAS1的影响。由图4a、4b和4c可知,随着IPTG浓度的增大,对细胞生长的抑制作用增强,当IPTG浓度为1.0 mmol/L时,最大细胞湿重只达到99.81 g/L,葡萄糖积累量也随时间的增加而迅速增长至31.41 g/L。而在0.2 mmol/L条件下,最大细胞湿重可达122.51 g/L,酶活力最高为936.67 U/mL,葡萄糖积累量呈现平稳增长趋势,发酵至22 h时,残糖量为10.36 g/L。当IPTG浓度为0.6 mmol/L时,发酵13 h酶活力达到了最大,为2 063.47 U/mL。这一结果表明IPTG诱导物为0.6 mmol/L有利于MAS1的表达。
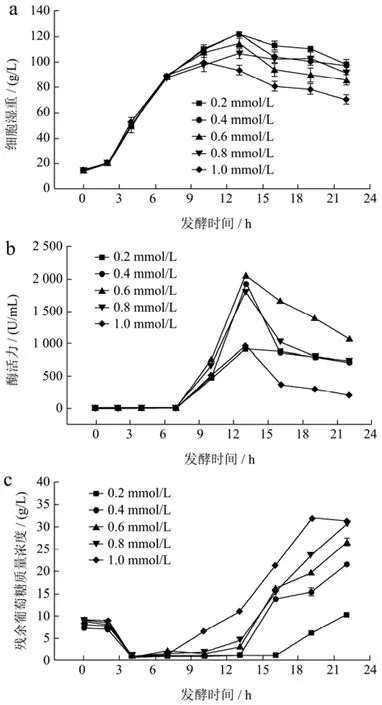
图4 IPTG浓度对于BL21(DE3)/pET22b-MAS1发酵过程的影响Fig.4 The influence of IPTG concentration on the fermentation process of BL21(DE3)/pET22b-MAS1
2.4 诱导pH值对于BL21(DE3)/pET22b-MAS1表达MAS1的影响
pH值作为发酵过程当中重要的参数,它可以影响到重组大肠杆菌细胞的生长和蛋白的表达。当外部环境的pH值发生变化时会造成细胞内部的pH值也发生改变,从而影响到细胞内部分代谢酶的活性进而造成细胞内部生理状态的改变,因此维持发酵过程当中的pH值恒定对于发酵具有十分重要的意义[31]。Shahzadi等[32]在优化大肠杆菌产纤维素酶的发酵条件时,发现当诱导pH值大于或小于7时,重组纤维素酶表达量都会降低,可见诱导pH值对重组菌表达蛋白具有重要的影响。分别控制诱导pH值为6.0、6.5、7.0、7.5、8.0,探究了不同pH值下BL21(DE3)/pET22b-MAS1发酵表达MAS1的影响。由图5a、5b和5c可知,当诱导pH值为6.5时,细胞湿重和酶活力均达到最大,分别为 146.83和2 103.69 U/mL,葡萄糖的积累速率显著慢于其他诱导pH值下葡萄糖的积累速率,发酵至22 h时,葡萄糖残余含量仅为11.63 g/L。当pH值为6.0和7.0时,最大细胞湿重分别为93.5 g/L和113.5 g/L,最大酶活力分别为688.42 U/mL和1 909.58 U/mL,均显著低于pH值6.5条件下的最大细胞湿重和最大酶活力。于此同时pH值6.0条件下,葡萄糖快速积累,发酵至22 h时葡萄糖残余含量为31.34 g/L。这一结果说明诱导pH值为6.5时更有利于BL21(DE3)/ pET22b-MAS1表达MAS1。
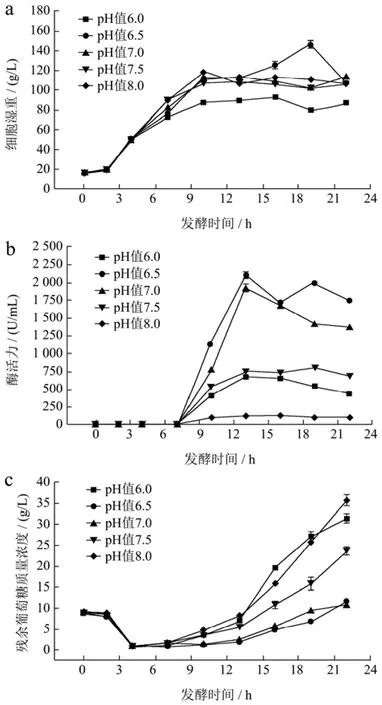
图5 诱导pH对BL21(DE3)/pET22b-MAS1发酵过程的影响Fig.5 The effect of induced pH on the fermentation process of BL21(DE3)/pET22b-MAS1
2.5 诱导温度对于 BL21(DE3)/pET22b-MAS1表达MAS1的影响
温度对于大肠杆菌的生长和外源蛋白的表达具有重要的影响,较高的温度有利于大肠杆菌的生长,较低的温度有利于大肠杆菌表达外源蛋白[33]。因此,常见的大肠杆菌发酵控温工艺,多采用在生长阶段维持较高的温度,而在诱导阶段维持较低的温度。另外,低温条件下诱导可以提高质粒在大肠杆菌中的稳定性[31]。Liang等[34]在探究重组大肠杆菌产丙酮酸氧化酶变温发酵策略时,发现当诱导温度为 37 ℃时,大肠杆菌生长速度最快,但丙酮酸氧化酶活性仅为890 U/L,而诱导温度分别为28 ℃和32 ℃时,丙酮酸氧化酶活性达到1 099 U/L和1 189 U/L。本研究在对数生长后期添加0.6 mmol/L IPTG、诱导pH维持6.5条件下,分别维持诱导温度为15、20、25、30、37 ℃。由图6b可知,最佳诱导温度为20 ℃,酶活达到最高为2 011.56 U/mL。如图6a和6c,当温度逐渐升高时,BL21(DE3)/pET22b-MAS1最大湿重是逐渐增长的,在25 ℃下培养时细胞湿重最大为126.67 g/L,但是超过 25 ℃时,最大湿重呈现下降的趋势;当诱导温度为37 ℃时,最大细胞湿重仅为88.36 g/L,且葡萄糖积累的速率开始加快,发酵至22 h时,发酵液中的葡萄糖含量积累至 44.5 g/L,同时最大酶活力也仅为110.6 U/mL,可能是因为在高于 25 ℃诱导时BL21(DE3)/pET22b-MAS1表达蛋白质和生长速度快,这增加了细胞的代谢负担。Paul等[35]发现较高温度下诱导重组大肠杆菌表达麦芽糊精葡萄糖苷酶会产生较多包涵体,从而导致大肠肝菌生长速率的下降。当诱导温度为15 ℃时,最大湿重仅为85.61 g/L,仅为20 ℃诱导下最大湿重的72.55%,说明诱导温度过低时不利于BL21(DE3)/pET22b-MAS1的生长,发酵至22 h时,发酵液中的葡萄糖残余含量为44.2 g/L,且最大酶活为 873.74 U/mL。这一结果说明,低温有利于BL21(DE3)/pET22b-MAS1表达MAS1,但是温度过低则会导致BL21(DE3)/pET22b-MAS1生长受到抑制并且影响到MAS1的表达。
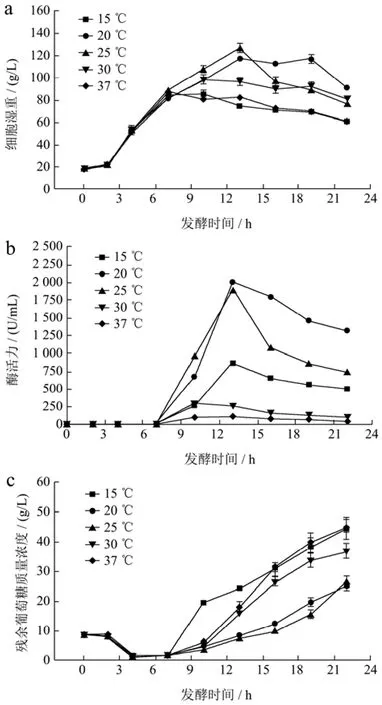
图6 诱导温度对BL21(DE3)/pET22b-MAS1发酵过程的影响Fig.6 The influence of induction temperature on the fermentation process of BL21(DE3)/pET22b-MAS1
2.6 SVM模型的构建
利用R语言的rsm包设计了Box-Behnken实验方案,随后依据Box-Behnken实验方案得到了对应的数据。利用MATLAB 2020a软件的Mapminmax函数对Box-Behnken实验方案结果进行归一化处理,然后用randperm随机函数将数据集随机分为两组,其中测试集函数由12组实验数据构成,测试集由3组实验数据构成。通过 MATLAB 2020a软件的 LIBSVMFarutoUltimate工具箱的SVMcgForClass函数对SVM模型的c值和g值进行寻优,确定c值和g值分别为1.5157和0.33。随后使用MATLAB 2020a的Libsvm工具箱构建 BL21(DE3)/pET22b-MAS1产 MAS1的SVM模型。由表2和表3可知,训练集的平均相对误差为4.26%,测试集的平均相对误差为3.32%,综合训练集和测试集误差来看,SVM模型预测结果与实验结果平均相对误差为4.07%,拟合效果较好。
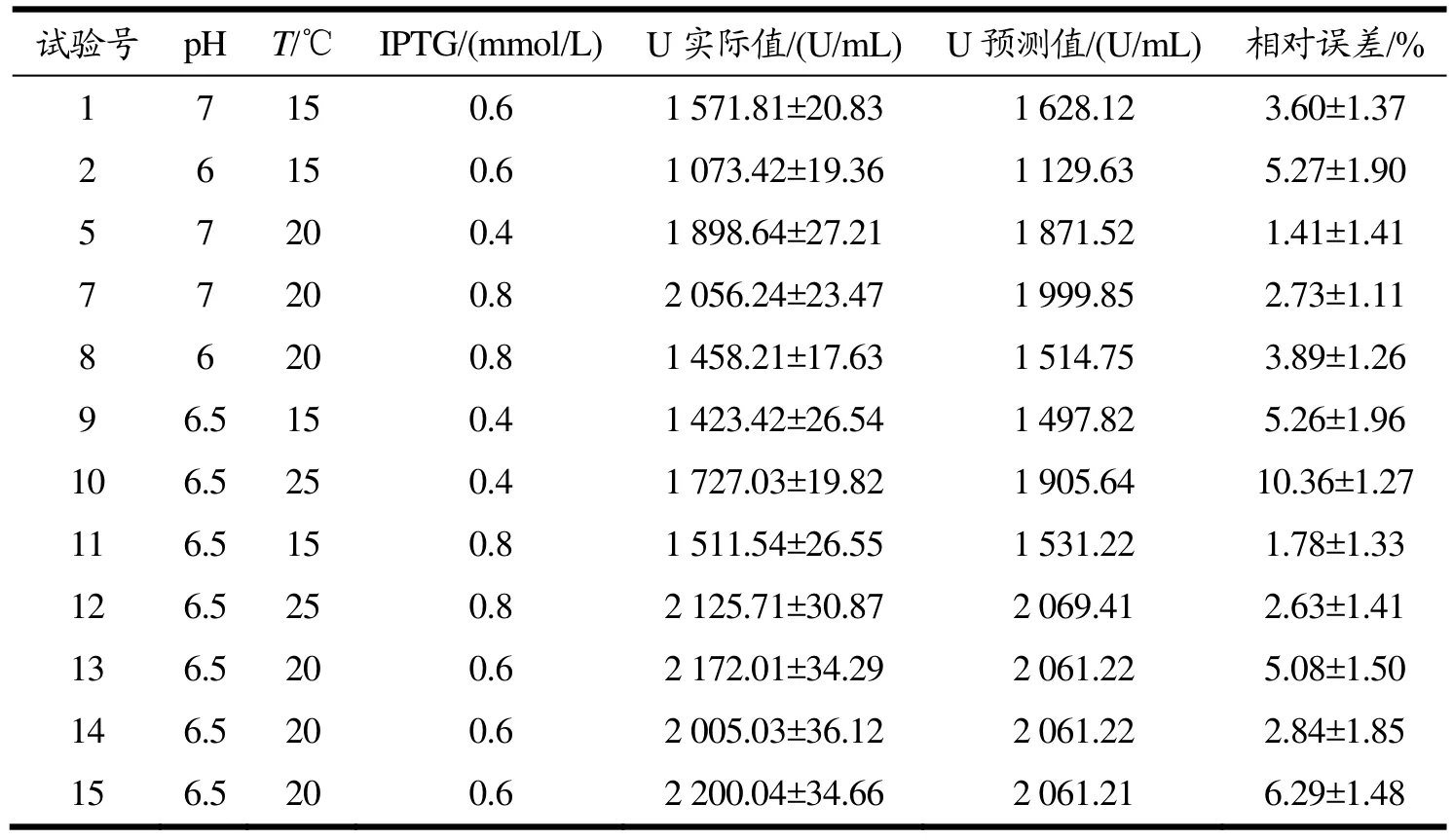
表2 SVM模型训练数据样本设计与实验结果Table 2 SVM model training data sample design and experimental results

表3 SVM模型测试数据样本设计与实验结果Table 3 SVM model testing data sample design and experimental results
2.7 GA寻优
从MATLAB 2020a软件的GA工具箱对SVM模型寻优结果(如图7)来看,当迭代至66代时,GA找到了最优结果:BL21(DE3)/pET22b-MAS1发酵产MAS1的最佳发酵条件为诱导pH值6.73、诱导温度22.923 ℃、诱导物IPTG浓度0.648 mmol/L,预测此时MAS1的酶活达到了最大为2 276.99 U/mL。
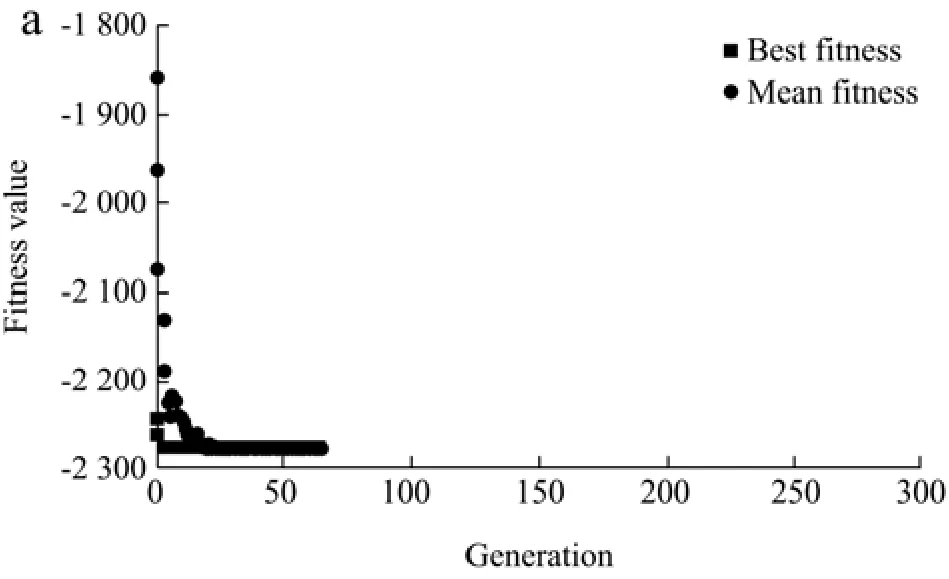
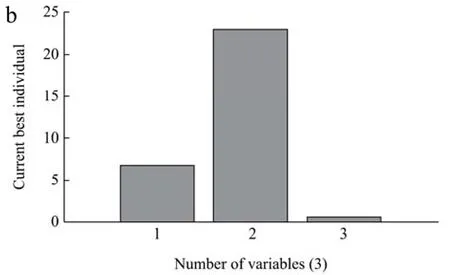
图7 遗传算法寻优结果图Fig.7 Genetic algorithm optimization result graph
2.8 模拟结果的验证
对SVM和GA得到的BL21(DE3)/pET22b-MAS1发酵产MAS1的最佳发酵条件进行发酵验证,设置诱导pH值6.7、诱导温度23 ℃、诱导物IPTG浓度为0.65 mmol/L,重复三次,实验结果分别为2 310.32、2 307.44、2 330.29 U/mL。图8的电泳结果表明了该策略具有可行性。王鲜芳等[36]将用SVM建立了谷氨酸动态时变发酵模型,用免疫遗传算法(Immune Genetic Algorithm,IGA)对一些关键操作变量进行了优化,在发酵28 h时谷氨酸浓度达到了12.05 g/L,比未优化前提高了29.57%。Zhang等[37]用SVM建立了以纤维素乙醇废水为原料红酵母发酵模型,采用 GA对SVM模型寻优,最终将生物量和脂质分别提升至11.87和2.18 g/L。说明了SVM-GA算法可在发酵条件优化中良好应用。
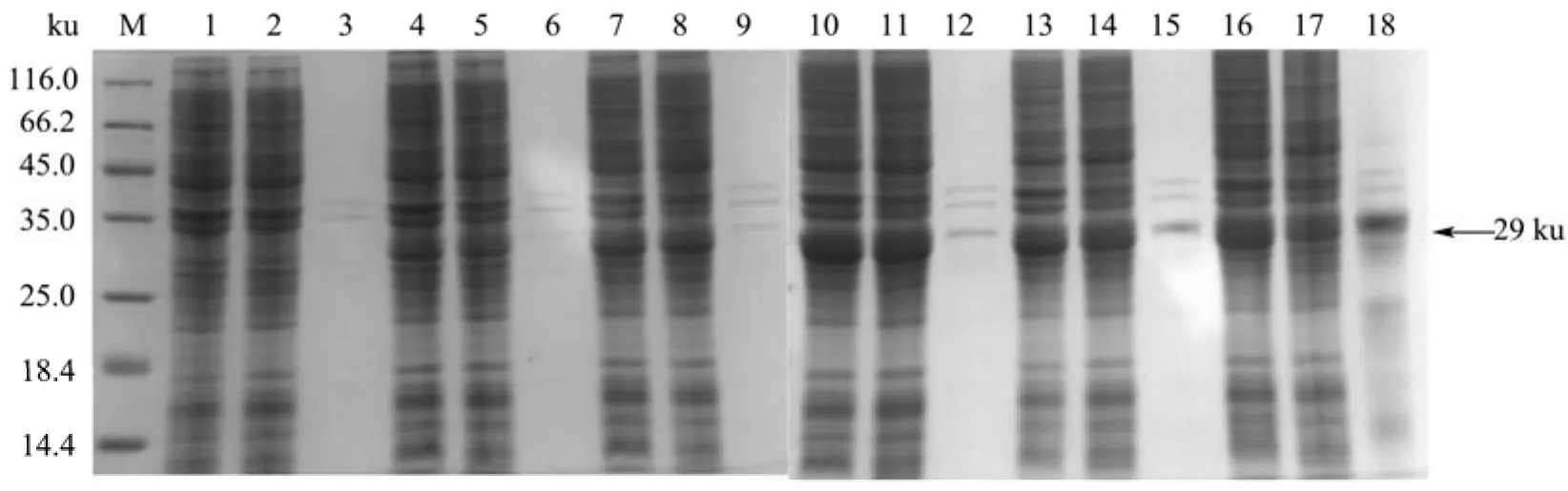
图8 MAS1的SDS-PAGE检测Fig.8 SDS-PAGE detection of MAS1
3 结论
本研究通过对BL21(DE3)/pET22b-MAS1产MAS1的发酵条件进行模型构建,使用 GA对三个重要的发酵参数进行了优化。研究结果表明,通过SVM-GA优化后的最佳发酵条件为诱导pH值6.7、诱导温度23 ℃、诱导物IPTG浓度为0.65 mmol/L,优化后MAS1酶活力达到了2 316.02 U/mL,较优化前提高了33.61%。本研究通过一系列的发酵条件优化,MAS1的发酵表达水平有了明显的提高,丰富了MAS1发酵条件优化的手段。目前在发酵罐进行的发酵条件优化限于诱导时间、诱导pH、诱导物IPTG浓度和诱导温度,后续可进行补料策略的优化以进一步提升MAS1的表达水平,为实现MAS1的工业化应用奠定基础。

