多光谱法研究钙黄绿素-U(VI)配合物与鲱鱼精DNA的作用机理
刘德春 杨文彬 肖啸 杨懿 谢红威 李威 颜奥琦
摘要:结合UV-Vis吸收光谱、荧光光谱和黏度测试法,系统研究了Tris-HCl(pH=7.25)缓冲溶液中U(VI)-钙黄绿素(CA)配合物与鲱鱼精DNA(hsDNA)的相互作用机理。通过摩尔比率法确定U(VI)-CA-hsDNA体系的结合比nU(VI)∶nCA∶nhsDNA=1∶2∶1。以双倒数法计算出此体系在不同反应温度下的结合常数,热力学计算表明U(VI)-CA-hsDNA的反应是疏水力驱动进行的自发行为。结合荧光分析、Scatchard法分析和溶液黏度变化分析,证明U(VI)-CA配合物对hsDNA的荧光猝灭为动态和静态混合猝灭过程,作用方式为嵌插和非嵌插的混合结合模式。实验表明U(VI)-CA配合物严重影响hsDNA的生物功能。
关键词:鲱鱼精DNA放射性U(VI)钙黄绿素荧光猝灭
中图分类号:O657.61文献标志码:A文章编号:1671-8755(2023)01-0022-09
Abstract:ViathemeasurementapproachesofUV-Visabsorptionspectrum,fluorescencespectrumandviscosity,theinteractionmechanismbetweenU(VI)-calcein(CA)complexandherringspermDNA(hsDNA)intheTris-HClbuffersolution(pH=7.25)wassystematicallyinvestigated.BindingratioofU(VI)-CA-hsDNAwasdeterminedasnU(VI)∶nCA∶nhsDNA=1∶2∶1bymolarratiomethod.BindingconstantsofU(VI)-CA-hsDNAcomplexatdifferenttemperatureswereobtainedbydoublereciprocalmethod,andtheevaluatedthermodynamicdataindicatedthattheformationofU(VI)-CA-hsDNAcomplexwasspontaneousanddrivenbyhydrophobicforce.Combinedwiththefluorescenceanalysis,scatchardmethodanalysisandsolutionviscositychangeanalysis,thefluorescencequenchingmodeofhsDNAbyU(VI)-CAcomplexwasprovedtobeamixeddynamic-staticquenchingprocess,andtheactionmodewasamixedcombinationmodeofintercalationandnonintercalation.TheexperimentalresultsshowthattheU(VI)-CAcomplexseriouslyaffectsthebiologicalfunctionofhsDNA.
Keywords:HerringspermDNA;RadioactiveU(VI);Calcein;Fluorescencequenching
铀在工农业生产和科技领域有着广泛应用。作为重要的放射性核素之一,即使在低浓度下也具有严重的毒性[1],其不当的后处理或排放会对地面和含水层造成严重的环境威胁,因此,铀及其各种衍生物的分析监测受到广泛关注[2]。含铀废物在水中易溶解、输运和沉淀,若处理不当而排入水体中,在微生物作用下将产生更多有毒金属有机物,通过食物链富集进入人体,从而对人体健康造成严重影响[3]。因此,探索放射性铀及其有机小分子活性配合物与生物大分子的作用方式,对了解放射性核素危害人体健康的致病机理具有重要意义。
生物体细胞核中的重要遗传物质DNA大分子由糖磷酸骨架和4个核碱基组成[4],它控制着蛋白质的合成和储存遗传信息[5],在基因转录、基因表达、癌变和突变中起着至关重要的作用。因此,DNA常作为研究各种抗肿瘤和抗癌药物的生物活性靶分子[6]。研究各种小分子及其金属离子的配合物与DNA的相互作用机理,对于设计能够识别特定DNA位点的新型高效药物具有重要意义[7]。近年来,有学者开展了小分子及其各种金属离子配合物与DNA结合的研究[8]。然而,放射性U(VI)及其各种有机小分子活性配合物与DNA的相互作用机理却鲜有报道。研究铀及其有机小分子活性配合物与DNA的结合方式,可以从分子水平上了解铀及其小分子活性配合物对人体健康的影响,准确推进针对靶向癌细胞和正常细胞的无毒高效药物的作用机理研究和开发[9]。UV-Vis吸收和荧光光谱具有测量简单、快速且灵敏的特点,是研究小分子与生物大分子之间作用机理的最有效方法[10]。因此,本实验在生理pH值条件下利用多光谱法研究了U(VI)-钙黄绿素活性配合物与鲱鱼精DNA的相互作用机理,为放射性铀在环境科学、生物医学和毒理学领域的毒性预防、评价及相关药物的开发提供理论依据。
1实验
1.1试剂
鲱鱼精DNA(BR级,纯度>99.0%,分子量为17736g·mol-1),上海源叶生物科技有限公司;三羟甲基氨基甲烷(BR级)、NaOH(AR級)、HCl(AR级)、茜素红(AR级)、钙黄绿素(AR级),成都市科隆化学品有限公司;硝酸铀(AR级),北京化工厂。所有实验溶液均使用生物反应最优pH值的Tris-HCl缓冲溶液制备。
1.2实验方法
1.2.1UV-Vis吸收光谱测定
UV-Vis吸收光谱测量在UV-3150分光光度计(日本京都岛津公司)和1.0cm石英比色皿上进行。吸收光谱的测定分3步进行,首先在系列10mL比色管中用Tris-HCl缓冲溶液配制固定浓度的U(VI),CA和hsDNA溶液(cCA=1×10-3mol·L-1,cU(VI)=1×10-3mol·L-1和chsDNA=1×10-5mol·L-1),以相应pH值的缓冲溶液为空白参比溶液,扫描三者之间在20℃下相互作用的吸收光谱以确定最佳反应pH值。其次,配制最佳反应pH值下的U(VI),CA和hsDNA溶液(浓度同上一步),在石英比色皿中加入3.5mL相关溶液,采用滴定法测定U(VI)与CA相互作用的吸收光谱。最后,以摩尔比法得到U(VI)与CA反应的结合比,配制此结合比下的一定浓度的U(VI)-CA配合物溶液,采用滴定法测定其与hsDNA在不同温度下相互作用的吸收光谱。每次扫描光谱前均将比色皿中的样品溶液充分混合并恒温5min,体积效应忽略不计,下同。
1.2.2荧光光谱测定
荧光光谱测量在F-4600荧光分光光度计(日本日立高科技公司,1.0cm石英比色皿)上进行。测量过程分为3个步骤:首先,不同反应温度下U(VI)-CA-hsDNA三相作用体系荧光猝灭光谱的测定采用1×10-3mol·L-1浓度的U(VI)-CA配合物溶液滴定1×10-5mol·L-1浓度的hsDNA溶液。其次,以3×10-4mol·L-1茜素红作为荧光探针,固定hsDNA浓度为1×10-6mol·L-1,在室温25℃下测定hsDNA和U(VI)-CA配合物不同混合摩尔比(0,0.2,0.4,0.6,0.8和1.0)的荧光光谱。最后,通过测量U(VI)-CA-hsDNA溶液从30℃升温至90℃的最大特征荧光峰(扫描步长为10℃)的变化来确定U(VI)-CA-hsDNA配合物的熔点温度(Tm),这里固定U(VI)-CA-hsDNA配合物的浓度为1×10-3mol·L-1。所有荧光光谱测定条件设置为中等速度,狭缝宽度5nm,幅度为2,波长310nm。
1.2.3U(VI)-CA-hsDNA反应体系黏度的测定
使用乌氏黏度计(0.6mm)在25℃水浴中进行反应体系溶液的黏度变化测量。hsDNA和U(VI)-CA配合物溶液的浓度均固定在1×10-4mol·L-1。加入2mLhsDNA溶液于10mL比色管中,分别加入不同量(0~8mL)的U(VI)-CA溶液,以Tris-HCl缓冲溶液稀释制备系列不同浓度的U(VI)-CA-hsDNA三相体系溶液。分别在黏度计中加入不同浓度的U(VI)-CA-hsDNA溶液,重复3次测定各样品的流动时间t。溶液比黏度η0和η值由η=(t-t0)/t0进行计算[11],其中t和t0分别代表在添加不同浓度U(VI)-CA配合物和单独缓冲溶液存在下hsDNA的流出时间;η和η0分别代表添加和没有添加U(VI)-CA配合物的hsDNA溶液的比黏度。
2结果及分析
2.1溶液pH值对U(VI)-CA-hsDNA配合物形成的影响
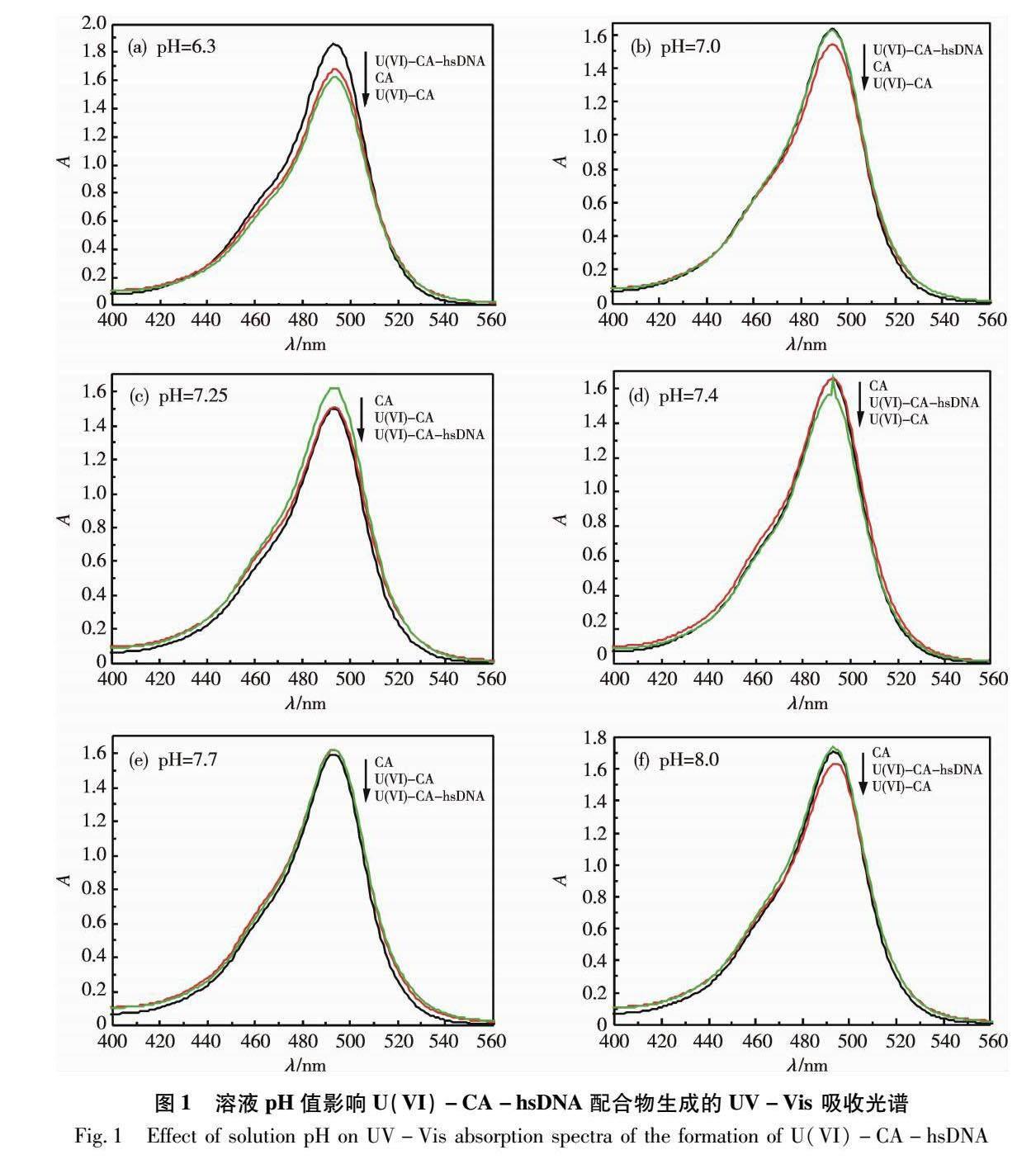
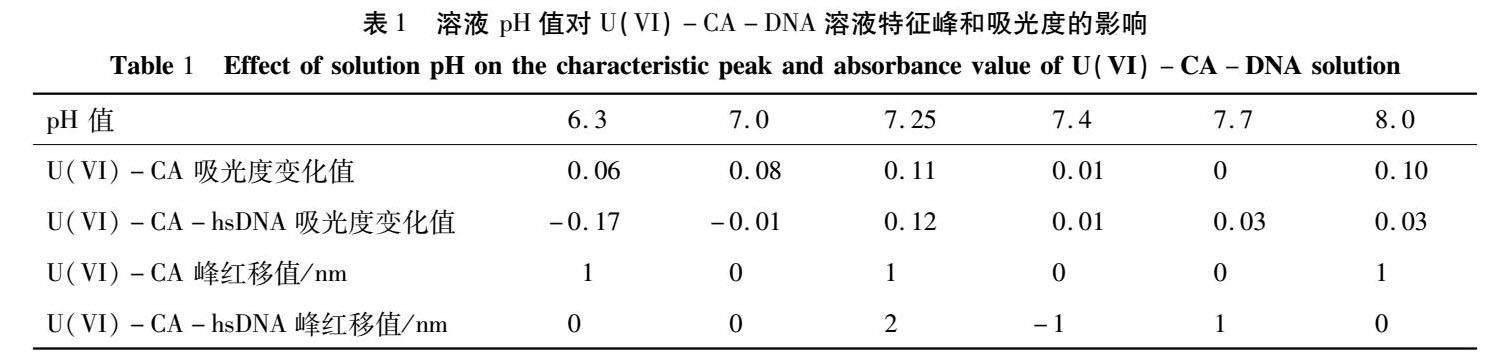
CA在不同的溶液pH值条件下与放射性U(VI)离子或hsDNA反应时,其UV-Vis吸收光谱特征峰的吸光度值和位置会发生明显变化,从而可确定体系反应的最佳pH值。如图1所示,CA溶液在323nm和493nm出现两个特征吸收峰。将各反应体系在493nm处的特征吸收峰的吸光度变化值和红移值列于表1中,结果发现U(VI)-CA和U(VI)-CA-hsDNA系统在pH值7.25时存在最大的吸光度降低值和红移值,表明此pH值为U(VI)-CA-hsDNA配合物形成的最佳值,它接近人体生理pH值,符合hsDNA大分子的生物学条件。因此,后续实验均在pH=7.25下进行。
2.2U(VI)-CA-hsDNA配合物形成的UV-Vis吸收光谱分析
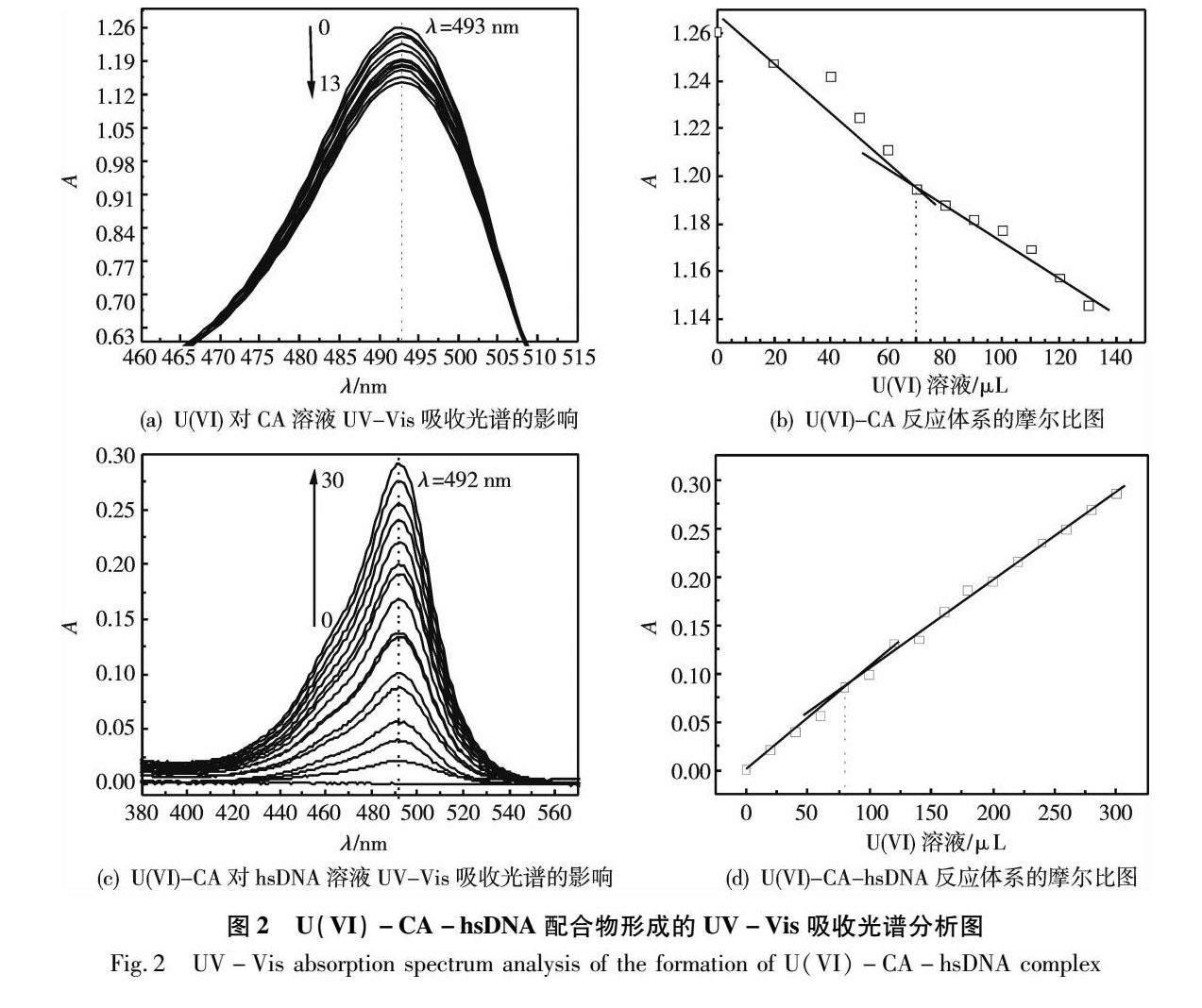
CA与U(VI)的结合率可以通过分光光度滴定法的UV-Vis吸收光谱计算得到[12]。实验发现U(VI)-CA配合物在493nm特征峰处的吸光度值随U(VI)量的增加而显著降低,并产生一定程度的红移(图2(a),每滴10μL,0-13对应0~130μL)。通过摩尔比法可求得此特征峰处U(VI)-CA配合物的结合比nU(VI)∶nCA=1∶2(图2(b))[13]。采用相同的方法,在λ=492nm处可确定U(VI)-CA-hsDNA三相体系的结合比nU(VI)∶nCA∶nhsDNA=1∶2∶1(图2(c)和图2(d),cU(VI)-CA=5.0×10-5mol·L-1,chsDNA=1.0×10-6mol·L-1,每滴10μL,0-30对应0~300μL)。根据朗伯-比尔定律(吸光度A=εbc,b=1cm),U(VI)-CA和U(VI)-CA-hsDNA配合物在20℃下的表观摩尔吸光度(ε)分别为5.11×104L·mol-1·cm-1和7.33×104L·mol-1·cm-1。
2.4U(VI)-CA配合物对hsDNA的荧光猝灭机理
图5为不同温度下滴加U(VI)-CA配合物的hsDNA溶液的荧光光谱(λex=310nm,λem=391nm,pH=7.25)。从图5可见,hsDNA溶液的荧光强度随着U(VI)-CA量的增加而显著降低,但最大发射波长随温度的升高而产生明显的红移现象,表明hsDNA的构象产生了改变(图5(a)、图5(b)和图5(c))。同时,在475nm附近出现了一个等荧光强度点,表明U(VI)-CA配合物对hsDNA产生了嵌插作用[17]。由于hsDNA蛋白随温度升高而变性,导致U(VI)-CA-hsDNA溶液的荧光强度随温度升高而降低[18]。基于摩尔比法,计算出U(VI)-CA-hsDNA配合物在不同温度下的结合比nhsDNA∶nU(VI)-CA=1∶1(图5(d)),与UV-Vis吸收光谱的分析结果相同。
为了进一步探索U(VI)-CA对hsDNA的猝灭机理,假设荧光猝灭是动态猝灭过程,因此可用Stern-Volmer方程来描述[19]:
2.5U(VI)-CA配合物與hsDNA之间的结合模式分析
由于茜素红(AR)分子可以嵌入DNA链双螺旋的碱基对中,从而增加其荧光强度。因此,AR可用作荧光探针来研究U(VI)-CA配合物与hsDNA之间的相互作用。AR对U(VI)-CA-hsDNA体系的影响可通过荧光Scatchard方程进行分析[24]:
式中:r是指每个核苷酸结合配合物的量;c是游离配合物的浓度;n是r的最大值;K是单个位点固有的结合常数。根据r的定义,可得:
U(VI)-CA是否嵌插入hsDNA的螺旋双链中,可以通过测定U(VI)-CA-hsDNA不同温度下的变性温度(Tm)来确定。因为DNA的变性温度一般在70℃左右,如果嵌入剂嵌入DNA的双链碱基对中,则可以提高DNA螺旋双链的稳定性和Tm值;反之,非嵌入结合会导致Tm值增加不明显[27]。测定U(VI)-CA-hsDNA配合物在不同加热温度下的系列荧光光谱,以最大特征峰处的荧光值比Ft/F0为纵坐标、加热温度为横坐标绘制U(VI)-CA-hsDNA配合物的热变性曲线如图8所示。实验发现在U(VI)-CA配合物存在下,hsDNA的Tm为80℃,表明U(VI)-CA配合物已嵌入hsDNA的双螺旋碱基对中[28]。
雖然光学探针可以提供必要但不充分的线索来探索二者的结合模式,但流体动力学测量对溶液中结合模式的长度变化却很敏感[29]。即,hsDNA与U(VI)-CA配合物的结合模式可以通过黏度测量进一步验证。当嵌入剂嵌入DNA后会增长DNA的双螺旋链,导致DNA溶液的黏度显著增加。反之,小分子配合物部分或非嵌入DNA的双螺旋链中却会降低DNA溶液的黏度,这是因为非嵌入结合不会明显增加DNA溶液的黏度[30]。如图9所示,hsDNA溶液的相对黏度值随着U(VI)-CA配合物的增加量而降低,并最后趋于平衡。进一步证明U(VI)-CA与hsDNA的相互作用模式确实为嵌入和非嵌入结合模式。
3结论
本文结合UV-Vis吸收光谱、荧光光谱、热变性和黏度测量法研究了U(VI)与钙黄绿素和U(VI)-CA配合物与hsDNA之间的相互作用,获得了U(VI)-CA-hsDNA的结合比(摩尔比1∶2∶1)、结合作用力和结合方式等信息和数据。实验结果表明U(VI)-CA配合物和hsDNA的结合是受焓驱动的自发作用,前者对hsDNA的猝灭过程是由动态和静态猝灭相结合引发的,二者之间的作用方式为采用嵌插和非嵌插混合的结合模式。本文结果为了解放射性核素对人体的致病机理和寻找新的能够识别特定DNA位点的新型抗肿瘤药物提供了理论和实验数据。
参考文献
[1]WANGD,XUYB,XIAODF,etal.UltrathinironphosphatenanosheetsforhighefficientU(VI)adsorption[J].JournalofHazardousMaterials,2019,371:83-93.
[2]SUMANS.Hybridnuclearrenewableenergysystems:Areview[J].JournalofCleanerProduction,2018,181:166-177.
[3]FENGMB,ZHANGP,ZHOUHC,etal.Waterstablemetalorganicframeworksforaqueousremovalofheavymetalsandradionuclides:Areview[J].Chemosphere,2018,209:783-800.
[4]JASHB,MLLERJ.Concomitantsitespecificincorporationofsilver(I)andmercury(II)ionsintoaDNAduplex[J].Chemistry—AEuropeanJournal,2018,24(42):10636-10640.
[5]ZHANGGW,GUOJB,ZHAON,etal.Studyofinteractionbetweenkaempferol-Eu3+complexandDNAwiththeuseoftheNeutralReddyeasafluorescenceprobe[J].SensorsandActuatorsB:Chemical,2010,144(1):239-246.
[6]LAURIAA,BONSIGNORER,TERENZIA,etal.Nickel(II),copper(II)andzinc(II)metallointercalators:structuraldetailsoftheDNA-bindingbyacombinedexperimentalandcomputationalinvestigation[J].DaltonTransactions,2014,43(16):6108-6119.
[7]WANGR,HUAX,PANJH,etal.InteractionofisoeugenolwithcalfthymusDNAanditsprotectiveeffectonDNAoxidativedamage[J].JournalofMolecularLiquids,2019,282:356-365.
[8]HUY,XIEMY,WUXY.InteractionstudiesofsodiumcyclamatewithDNArevealedbyspectroscopymethods[J].SpectrochimicaActaPartA:MolecularandBiomolecularSpectroscopy,2019,220:117085.
[9]KAVITHAB,SRAVANTHIM,REDDYPS.DNAinteraction,docking,molecularmodellingandbiologicalstudiesofo-VanillinderivedSchiffbasemetalcomplexes[J].JournalofMolecularStructure,2019,1185:153-167.
[10]QIAOCY,BISY,SUNY,etal.StudyofinteractionsofanthraquinoneswithDNAusingethidiumbromideasafluorescenceprobe[J].SpectrochimicaActaPartA:MolecularandBiomolecularSpectroscopy,2008,70(1):136-143.
[11]HANKH,JEONGS,HONGIK,etal.Predictionofsolubilityparameterfromintrinsicviscosity[J].JournalofIndustrialandEngineeringChemistry,2013,19(4):1130-1136.
[12]WUQY,ZHAIFW,LIUY,etal.Interactionsbetweenuranium(VI)andphosphopeptide:experimentalandtheoreticalinvestigations[J].DaltonTransactions,2016,45:14988-14997.
[13]PEKELN,GVENO.Investigationofcomplexformationbetweenpoly(N-vinylimidazole)andvariousmetalionsusingthemolarratiomethod[J].ColloidandPolymerScience,1999,277:570-573.
[14]PURCELLM,NEAULTJF,TAJMIR-RIAHIHA.Interactionoftaxolwithhumanserumalbumin[J].BiochimicaetBiophysicaActa(BBA)-ProteinStructureandMolecularEnzymology,2000,1478(1):61-68.
[15]OJHAH,MURARIBM,ANANDS,etal.InteractionofDNAminorgroovebinderhoechst33258withbovineserumalbumin[J].ChemicalandPharmaceuticalBulletin,2009,57(5):481-486.
[16]WANGBL,PANDQ,ZHOUKL,etal.Multispectroscopicapproachesandmolecularsimulationresearchoftheintermolecularinteractionbetweentheangiotensinconvertingenzymeinhibitor(ACEinhibitor)benazeprilandbovineserumalbumin(BSA)[J].SpectrochimicaActaPartA:MolecularandBiomolecularSpectroscopy,2019,212:15-24.
[17]BRUNAM,HARRIMANA.Dynamicsofelectrontransferbetweenintercalatedpolycyclicmolecules:Effectofinterspersedbases[J].JournaloftheAmericanChemicalSociety,1992,114(10):3656-3660.
[18]LIUJM,YUEYY,WANGJ,etal.Studyofinteractionbetweenhumanserumalbuminandthreephenanthridinederivatives:Fluorescencespectroscopyandcomputationalapproach[J].SpectrochimicaActaPartA:MolecularandBiomolecularSpectroscopy,2015,145:473-481.
[19]LAKOWICZJR.Principlesoffluorescencespectroscopy,3rdedition[M].NewYork:Springer,2006.
[20]XUH,GAOSL,LVJB,etal.Spectroscopicinvestigationsonthemechanismofinteractionofcrystalvioletwithbovineserumalbumin[J].JournalofMolecularStructure,2009,919(1/3):334-338.
[21]YUXY,YANGY,LIURH,etal.Theinvestigationoftheinteractionbetweenedaravoneandbovineserumalbuminbyspectroscopicapproaches[J].JournalofLuminescence,2011,131(7):1510-1514.
[22]ABOU-ZIEDOK,AL-SHIHIOI.CharacterizationofsubdomainIIAbindingsiteofhumanserumalbumininitsnative,unfolded,andrefoldedstatesusingsmallmolecularprobes[J].JournaloftheAmericanChemicalSociety,2008,130(32):10793-10801.
[23]WAREWR.Oxygenquenchingoffluorescenceinsolution:Anexperimentalstudyofthediffusionprocess[J].TheJournalofPhysicalChemistry,1962,66(3):455-458.
[24]LEPECQJB,PAOLETTIC.Afluorescentcomplexbetweenethidiumbromideandnucleicacids:physicalchemicalcharacterization[J].JournalofMolecularbiology,1967,27(1):87-106.
[25]TONGCL,HUZ,WUJM.Interactionbetweenmethyleneblueandcalfthymusdeoxyribonucleicacidbyspectroscopictechnologies[J].JournalofFluorescence,2010,20:261-267.
[26]LIHB,LEIST,TANGYQ,etal.Drugeffectofthulium(III)-arsenazoIIIcomplexonherringspermDNA[J].JournalofChemistry,2018,2018:1-9.
[27]PRATVIELG,BEMADOUJ,MEUNIERB.CarbonhydrogenbondsofDNAsugarunitsastargetsforchemicalnucleasesanddrugs[J].AngewandteChemieInternationalEdition,1995,34(7):746-769.
[28]ZHAON,WANGXM,PANHZ,etal.Spectroscopicstudiesontheinteractionbetweentryptophanerbium(III)complexandherringspermDNA[J].SpectrochimicaActaPartA:MolecularandBiomolecularSpectroscopy,2010,75(5):1435-1442.
[29]CHENCY,CHENK,LONGQ,etal.StructuralcharacterizationandDNA-bindingpropertiesofSm(III)complexwithofloxacinusingspectroscopicmethods[J].Spectroscopy,2009,23(2):103-111.
[30]NAFISIS,SABOURYAA,KERAMATN,etal.StabilityandstructuralfeaturesofDNAintercalationwithethidiumbromide,acridineorangeandmethyleneblue[J].JournalofMolecularStructure,2007,827(1/3):35-43.

