大豆种质耐碱性状全基因组关联分析
滕卫丽,史飞飞,林 峰,刘晨煦,高 鹏,赵慧艳,赵 雪,韩英鹏,李文滨
(东北农业大学,大豆生物学教育部重点实验室,农业农村部东北大豆生物学与遗传育种重点实验室,哈尔滨 150030)
大豆作为我国五大作物之一,是重要的高蛋白粮饲兼用及油料作物。当前大豆产量不能满足人们日益增长的生活需求,土壤盐碱化是影响大豆产量重要因素之一。土壤中存在的盐类影响植物正常生长发育,降低产量[1],研究认为由Na2CO3、NaHCO3等碱性盐比NaCl、Na2SO4等中性盐对作物造成的胁迫危害更严重[2]。我国是受土壤盐渍化危害影响比较严重的国家,有逐年扩大趋势[3-4]。筛选耐碱大豆种质,找到与耐碱有关且稳定表达的位点,对培育耐碱大豆新品种、有效利用盐碱化土壤具有重要意义。
种子芽期和苗期是作物在盐渍环境下生长的关键及敏感阶段,避开敏感期,作物在各发育时期均可较好生长发育。大豆耐碱相关性状是复杂的数量性状,受多基因控制[5]。随着现代分子生物技术发展,耐碱基因发掘和使用成为近年植物抗逆研究热点。目前,许多学者已检测到多个芽期和苗期耐碱相关位点和QTL。Zhang 等对257 份大豆品种在芽期作Na2CO3溶液处理,通过测定主根长度、幼苗生物量等芽期耐碱指标评估品种耐受性,结合135个SSR标记进行互作关联分析,检测到86个耐碱性相关QTL[6]。张新草利用混盐碱溶液模拟盐碱胁迫,对281份大豆品种进行芽期胁迫鉴定,调查发芽率、根长、幼苗长等8个芽期耐盐碱指标,经全基因组关联分析,关联到268个SNP标记,其中优异增效等位变异有44 个,占SNP 标记总数16.42%[7]。Tuyen等利用F6RIL群体和F2群体进行QTL分析,在17号染色体上检测到耐碱性显著的QTL,占F6和F2群体STR(耐盐碱等级)总变异的50.20%和13.00%[8]。Tuyen 等进一步以剩余杂合系(RHL46)为材料,利用表型评估和SSR 标记分析,在标记Satt447和GM17-11.6之间3.33 cM处定位到耐碱QTL[9]。张金龙以NJRIZM6 和NJRILM6 两个RIL群体为试验材料,利用NaHCO3溶液进行碱胁迫处理,对STR、Na+浓度、K+浓度等7个苗期耐碱指标进行QTL定位,在两个群体中分别定位到22 个、19个与耐碱性状相关的QTL位点[5]。
目前对玉米、水稻和大豆等作物耐盐碱鉴定时期虽集中在芽期和苗期,但主要采用单一时期和单一成分的盐或碱开展试验。黑龙江省作为大豆主产地之一,盐碱土类型主要为苏打盐碱土,且盐碱土面积有逐年增加趋势[3],因此利用混合成分模拟实际碱胁迫环境,开展多时期大豆种质碱胁迫鉴定研究,对实际生产意义重大。
本研究针对黑龙江省大庆市苏打盐碱地,以混合碱模拟其碱胁迫环境,评价255份大豆种质芽期和苗期耐碱相关性状,结合全基因组关联分析,获得关联位点,挖掘候选基因,以期为分子标记辅助选育大豆耐碱品种提供依据。
1 材料与方法
1.1 试验材料
选用255份大豆种质构成的自然群体为试验材料(见表1),试验所用材料均由东北农业大学提供。

表1 255份大豆供试材料Table 1 Total 255 soybean test materials
1.2 试验设计
1.2.1 芽期试验设计
针对黑龙江省苏打盐碱地,以混合碱模拟李宪伟研究中采用的大庆地区中度苏打盐碱土壤基本情况[10],按其土壤中主要成分HCO3-与CO32-质量比为8.37∶1,计算HCO3-与CO32-物质的量比为10.56∶1,配置碱溶液,pH 为8.9~9.1;借鉴Tuyen等试验方法[8],采用浓度梯度方式在人工气候箱25 ℃、相对湿度60%条件下开展试验,隔天更换相应25 mL溶液,试验中每天调查发芽种子数,用于计算活力指数,种子发芽标准参考徐玲秀[11]。
1.2.2 芽期耐碱性状鉴定
芽期耐碱性状鉴定指标包括:吸胀率(Imbibition rate-48h,IR-48 h)、胚根干重(Radicle dry weight,RDW)、子叶下部干重(Dry weight below cotyledon,DWBC)、活力指数(Vigour index,VI)[12],相对碱害指标(Alkali tolerance,AT)包括:相对吸胀率(Alkali tolerance-imbibition rate,AT-IR)、相对胚根干重(Alkali tolerance-radicle dry weight,ATRDW)、相对子叶下部干重(Alkali tolerance-dry weight below cotyledon,AT-DWBC)、相对活力指数(Alkali tolerance-vigour index,AT-VI)[13]。
1.2.3 苗期试验设计
苗期同样以混合碱模拟碱胁迫环境,借鉴Tuyen 等试验方法[8],将大小均匀的大豆种子播种于装有1.15 kg土的盆中,待所有品种达到V2期进行定苗,对长势一致的大豆植株进行碱胁迫处理。根据李宪伟描述的中度苏打盐碱土壤情况[10],计算每次每盆需加入碱溶液或自来水318.5 mL,待最大浓度180 mmol·L-1处理2 d后进行表型测定。
1.2.4 苗期耐碱性状鉴定
苗期耐碱性状鉴定指标包括:茎粗(Stem diameter,SD)、叶绿素(Soil and plant analyzer develotrnent,SPAD)含量、主根长(Main root length,MSL)、地上部鲜重(Shoots fresh weight,SFW),相对碱害指标(Alkali tolerance,AT)包括:相对茎粗(Alkali tolerance-stem diameter,AT-SD)、相对叶绿素(Alkali tolerance-soil and plant analyzer develotrnent,AT-SPAD)、相对主根长(Alkali tolerancemain root length,AT-MSL)、相对地上部鲜重(Alkali tolerance-shoots fresh weight,AT-SFW)[5]。
1.2.5 基因型鉴定与分析
利用Illumina 基因组分析仪Ⅱ进行测序,完成基因型鉴定,将最小等位基因频率MAF<0.05 的SNP 通过BLINK 和FarmCPU 软件过滤淘汰,最终得到1 355 928个标记用于进一步关联分析。
1.2.6 全基因组关联分析
利用BLINK 和FarmCPU 两种方法对大豆种质芽期和苗期碱胁迫下相关性状表型值进行关联分析,为更好降低假阳性率,识别真正的标记关联性状,利用Bonferroni 校正方法,将显著P值设置为P<3.6875e-8(P=0.05/n,n为总标记数),绘制曼哈顿图显示SNP 与表型性状之间相关性。
1.2.7 数据处理与分析
采用Microsoft Excel 2021 处理表型数据和作图,利用Soybase 和Phytozome 数据库获得基因功能注释和基因在组织中表达数据,利用CMplot R包和Prism 8 分别绘制曼哈顿图和基因组织表达热图。
2 结果与分析
2.1 大豆种质耐碱性状表型和相关性分析
大豆自然群体耐碱相关性状均值、峰度、偏度等见表2。与碱胁迫相比,无碱条件下芽期和苗期相关性状表型值更高。芽期和苗期多数耐碱性状偏度和峰度绝对值小于1,且在8 个性状中,群体变异系数也不同,其中,叶绿素含量对照组群体变异系数最小,为7.06%,活力指数处理组变异系数最大,为123.70%。
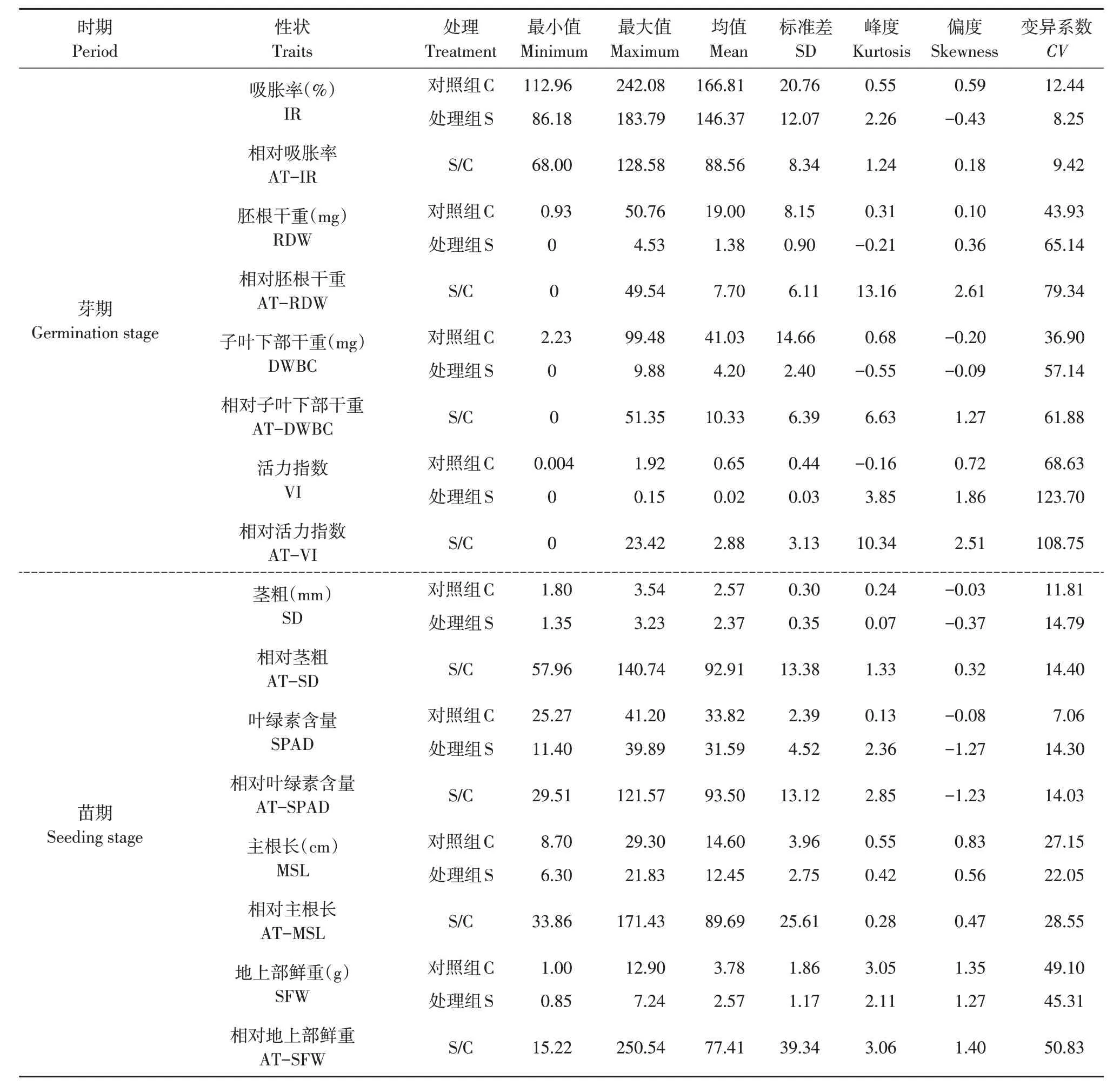
表2 大豆种质耐碱性状表型分析Table 2 Phenotypic analysis of alkali tolerance traits in soybean germplasm
为消除不同大豆种质间基因型背景差异,以芽期和苗期性状相对值为相关耐碱指标分析相关性(见图1)。在芽期阶段,相对胚根干重与相对下部干重、相对活力指数呈极显著正相关,其中相对胚根干重与相对下部干重相关程度最高,相关系数为0.90;相对活力指数与相对下部干重呈极显著正相关,相关系数为0.22。在苗期阶段,相对叶绿素与相对主根长呈显著负相关;相对茎粗与相对地上部鲜重呈显著正相关,相关系数为0.16。在芽期测得的耐碱性状与苗期测得的耐碱性状之间未观察到显著相关性,表明大豆两个发育阶段耐碱相关性状在很大程度上受独立遗传控制。
大豆种质耐碱性状分布如图2所示。相对吸胀率、相对叶绿素等芽期和苗期耐碱相关指标近似于正态分布,表明耐碱性状由多基因控制,适合进行关联分析。

图2 大豆种质耐碱性状分布Fig.2 Distribution of alkali tolerance traits of soybean germplasm
2.2 大豆种质耐碱性状全基因组关联分析
本研究利用Bonferroni 校正方法识别真正的标记关联性状,因Bonferroni 校正方法较严格,借鉴Li等GWAS分析果实大小性状时适当调整阈值的方法,将阈值由3e-8降至1e-5,以消除Bonferroni方法校正过于严格导致的假阴性[14]。利用BLINK 和FarmCPU 两种方法对大豆种质芽期和苗期耐碱性状进行全基因组关联分析(见图3 和4),两种方法共检测到64 个与大豆耐碱性状关联的SNP 位点(见表3)。

图3 大豆芽期耐碱相关性状全基因组关联分析曼哈顿图Fig.3 Manhattan plots of genome wide association analysis of alkali tolerance related traits in soybean at germination stage

图4 大豆苗期耐碱相关性状全基因组关联分析曼哈顿图Fig.4 Manhattan plots of genome wide association analysis of alkali tolerance related traits in soybean at seedling stage

表3 与大豆耐碱性状相关联位点Table 3 Loci associated with alkali tolerance traits in soybean

续表
这些位点主要分布在除12、13、16、20 号染色体外其他16 条染色体上,呈不均匀分布。其中,有2个SNP位点与芽期相对吸胀率相关联,分布在2、14 号染色体上,表型贡献率为0.36%~0.44%;9 个SNP 位点与相对胚根干重相关联,分布在3 号和7 号染色体上,表型贡献率为0.09%~9.62%,其中6 个SNP 位点贡献率高于1%,位点7:8915107 贡献率最高。BLINK 和FarmCPU 两种方法在芽期下部干重共检测出8个关联SNP位点,其中,BLINK方法单独检测出1个显著关联位点,为9:37033917,贡献率为2.41%,还有7 个关联SNP位点由两种方法同时检测出,分布在3、4、8、9号染色体上,表型贡献率为0.11%~8.16%,其中3:37525453 贡献率最高,该位点与相对下部干重显著关联。两种方法共检测出9个与相对活力指数相关联SNP位点,其中FarmCPU方法单独检测出8个SNP 位点,分布在1、6、7、8、17、18、19 号染色体上,还有1 个相同位点由BLINK 和FarmCPU方法同时检测出,分布在6号染色体上。两种方法共检测出11 个与苗期相对叶绿素含量相关联的SNP位点,其中有2个SNP位点由FarmCPU方法单独检测出,均位于5号染色体上,还有9个关联位点由BLINK 和FarmCPU 方法同时检测出,分布在2、5、15、18 号染色体上,贡献率为0.10%~1.70%。两种方法共检测出16个与苗期相对茎粗相关联SNP位点,其中有8个位点由FarmCPU方法单独检测出,分布在2、3、5、8、9、10、18号染色体上,还有8个位点由两种方法同时检测出,分布在4 号和8 号染色体上,贡献率为0.15%~1.35%。两种方法同时检测出3 个与相对主根长相关联的SNP 位点,分布在6、9、14 号染色体上,贡献率为0.24%~1.65%。两种方法共检测出6个与苗期相对地上鲜重相关联SNP 位点,其中,FarmCPU 方法单独检测出1个与相对地上鲜重关联位点,位于11号染色体上,还有5个关联SNP位点由两种方法同时检测出,位于11号和17号染色体上,贡献率为0.47%~1.91%。
2.3 大豆耐碱性候选基因预测
将检测到的64 个SNP 位点在上下游100 kb 区间内挖掘基因,共挖掘到26 个有功能注释基因。根据Soybase和Phytozome数据库中基因功能注释数据(见表4),发现这些基因主要在编辑蛋白激酶超家族蛋白、可逆糖基化多肽方面发挥作用。结合Phytozome 数据库中已收录基因表达量数据,发现上述基因主要在花、叶、根等9 个组织中表达(见图5)。

图5 候选基因组织表达热图Fig.5 Heatmap profiles of the candidate genes in tissues
分析上述基因功能,发现有7个基因与耐逆性相关。与芽期相对下部干重相关联基因Glyma.03G160100、Glyma.03G160200 均具有编码脂肪酸ω-羟化酶的功能,脂肪酸ω-羟化酶参与角质合成,角质与植物抵御外界胁迫有关[15],其中Glyma.03G160200 在根瘤、根、根毛中表达量较高。与相对下部干重相关联基因Glyma.09G149900在根中表达水平最高,该基因与bHLH 有关,bHLH 转录因子在抗非生物胁迫方面发挥重要作用[16-17]。与相对活力指数相关联基因Glyma.19G067400 在花、叶等9个组织中均有较高水平表达,具有编码Arf-GTPase 激活蛋白的功能,ArfGTPase 激活蛋白与植物应答非生物胁迫有关[18]。与芽期相对活力指数关联基因Glyma.18G272200、Glyma.01G057100 分别在(根瘤、根、根毛)和(豆荚、根)中表达量较高,两个基因分别具有编码可逆糖基化多肽2 和UDP-葡糖6-脱氢酶家族蛋白的功能,可逆糖基化多肽2 可能直接参与植物防御反应[19],UDP-葡糖6-脱氢酶家族蛋白与细胞壁合成有关[20],细胞壁能提高植物耐盐性[21]。与苗期地上部鲜重相关联基因Glyma.11G238400 编码硫酸盐转运蛋白,在叶和茎尖分生组织中表达量较高,硫酸盐转运蛋白可能参与相应非生物逆境[22]。
3 讨 论
芽期和苗期是大豆在盐渍环境下生长关键和敏感阶段,高浓度碱延迟种子萌发和出苗,导致植株茎秆变细,干重降低,老叶逐渐枯黄发干甚至脱落[11]。本研究在大豆芽期和苗期进行混碱鉴定,对耐碱相关性状进行GWAS 分析,以期发掘重要标记和遗传位点,挖掘候选基因,为分子标记辅助选育大豆耐碱品种提供依据。
目前,BLINK 和FarmCPU 等方法在挖掘全基因组遗传变异方面发挥重要作用。本研究利用这两种方法检测到64 个与大豆耐碱性状相关联SNP 位点,其中7 个位点与已知QTL 位点物理距离小于5 Mb[23]。与相对胚根值和相对下部干重值关联位点3:37525453,与相对胚根值关联位点3:37513903、3:37490361,这3 个关联位点与耐盐相关的ss715585865 分别相距377.48、389.03、412.57 kb[24],本研究发现位点3:37525453与相对下部干重显著关联,贡献率较高,为8.16%,因此该位点可靠性较高,可用于进一步基因挖掘。与相对下部干重显著关联位点9:37033917 与苗期耐碱性状相关的qCa09.1 相距240.70 kb[5],相对活力指数关联位点17:1589196 与盐胁迫相关BARC-031827-07220 相距196.70 kb[25],相对主根长关联位点14:9834716 在碱胁迫性状相关的9 806 588-9 879 926 区间内[5],相对茎粗关联位点9:43068852与碱胁迫下苗期性状相关qCa09.3相距887.82 kb[5]。由此可知,3:37513903、3:37490361、9:37033917等7个位点相对稳定,可靠性较高,具有重要参考价值。
在GWAS 分析过程中发现,与FarmCPU 方法相比,BLINK方法的运算速度更快,在检测功效方面,FarmCPU方法更具明显优势,可挖掘更多与耐碱相关的位点,如19:19200072、18:55518213 等,与Liu 等对相关性状进行GWAS 分析时发现Farm-CPU方法的检测功效较高的观点相似[26]。
本研究通过功能分析,发现7个基因与植物耐逆性相关。其中与相对下部干重相关联的Glyma.03G160100、Glyma.03G160200 具有相同的拟南芥同源基因,功能注释为脂肪酸ω-羟化酶。脂肪酸ω-羟化酶,属于细胞色素P450超家族,与角质合成有关,角质在保护植物免受胁迫具有重要作用[15]。Mao 等研究发现细胞色素P450 单加氧酶基因突变体过表达可增强转基因拟南芥盐胁迫耐受性[27]。Xia 等以蒺藜苜蓿为材料再次证实MtP450 枢纽基因提高植物盐胁迫下耐受性[28]。Glyma.03G160200在根瘤、根、根毛中表达量较高,是由重要位点3:37525453 挖掘出的基因,可将其作为与耐碱相关的候选基因。与相对下部干重显著关联的基因Glyma.09G149900,功能注释为碱性螺旋-环-螺旋(bHLH)DNA 结合超家族蛋白。bHLH 转录因子在调控植物的耐盐性方面,分为正负调控两种,如Zhou 等研究发现OrbHLH2 基因超表达会提高植株在高盐胁迫下的种子萌发率[16],Verma 等研究发现,高盐胁迫下bHLH 转录因子编码基因AtMYC2负调控脯氨酸的合成,进而降低转基因植株耐盐性[17]。基因Glyma.09G149900 在根中表达水平很高,是由重要位点9:37033917 挖掘出的基因,可将其作为与耐碱相关的候选基因之一。与相对活力指数相关联的基因Glyma.19G067400,功能注释为编码Arf GTPase 激活蛋白,刘芳等以美洲商陆为材料,发现ArfGTPase 激活蛋白基因PaAGAP 参与植物逆境反应[18]。根据Glyma.19G067400 在花、叶、根瘤、豆荚等9个组织中均有较高水平表达,且是由显著关联位点19:19200072 挖掘出的基因,可将此基因也作为耐碱候选基因。与相对活力指数相关联基因Glyma.01G057100 和Glyma.18G272200、与苗期地上部鲜重相关联基因Glyma.11G238400的功能注释参与植物耐逆性[19-22],但尚未发现挖掘这3个基因的位点与其他耐盐碱位点重叠或相近。在功能分析方面,上述7 个基因与拟南芥、蒺藜苜蓿、美洲商陆等作物的非生物胁迫相关,但目前尚未在大豆碱胁迫方面发现这些基因的相关报道,可能是由于所用材料类型不同,遗传背景存在差异导致。为进一步探究这些基因在大豆耐碱方面的作用,后期将验证候选基因功能,为大豆在耐碱方面分子育种提供有效依据。
综上,根据挖掘基因位点情况,查询基因功能注释及分析基因表达量等,可将Glyma.03G160200、Glyma.09G149900 和Glyma.19G067400 等3 个 基 因作为大豆碱胁迫性状相关重要候选基因。
4 结 论
a.BLINK和FarmCPU两种方法共同检测出64个与大豆芽期和苗期耐碱性状关联SNP 位点,其中3:37513903、3:37490361、9:37033917 等7 个 位 点相对稳定,可靠性较高。
b. 在上下游区间100 kb 进行基因挖掘,根据Soybase和Phytozome筛选到26个有功能注释基因。
c. 根据挖掘基因位点情况、功能注释和基因表达量,推测3 个基因与耐碱性相关,分别为Glyma.03G160200、Glyma.09G149900 和Glyma.19G067400。

