林麝肺成纤维细胞线粒体全基因组结构特征及遗传变异分析
唐可欣 伍茜 付文龙 程建国 吴杰 周磊 王翔 赵位 任梓薇 李依濛 刘洁 罗燕


摘 要 林麝(Moschus berezovskii)是一种具有较高经济价值和药用价值的保护野生动物。为深入研究林麝线粒体基因组的结构特征及遗传变异,基于Illumina NovaSeq平台对林麝肺成纤维细胞FMD-C1线粒体进行全基因组测序,并进行特征注释、结构预测、序列分析和系统发育重建。结果显示,FMD-C1线粒体基因组序列全长为16 351 bp,共有13个蛋白编码基因(protein coding gene,PCG),22个tRNA,2个rRNA和两段非编码区,具有明显的AT偏好性。将序列上传至GenBank,登录号为MW879208。与NCBI上林麝的其余序列比对,FMD-C1的4个PCGs中检测出5个单氨基酸突變和3个插入氨基酸;D-loop区检测出19个差异碱基和2个碱基缺失。系统进化分析发现与林麝亲缘关系最近的是安徽麝。研究结果可为林麝的选育和遗传多样性保护提供参考依据,也为进一步探讨林麝肺部疾病线粒体水平的致病机制奠定基础。
关键词 林麝;线粒体基因组;蛋白编码基因;非编码区;系统进化
林麝(Moschus berezovskii)是麝属中体型最小的一种。雄麝香腺中分泌的麝香是一种高级动物香料,也是一味名贵中药材,具有较高的经济价值和药用价值[1]。由于近几十年栖息地破坏和人为捕杀,种群数量大幅减少,麝现已被列为中国一级重点保护动物[2]。为解决麝数量骤降以及推广麝香的可持续利用,中国现大力发展人工养麝产业,但麝养殖种群发病率高、死亡率高,导致人工养麝产业发展缓慢。其中,肺部疾病是林麝主要死因之一,对其背后的病理机制探究目前多集中在病原菌分离鉴定等方面[3-4]。
线粒体是广泛存在真核生物细胞内的重要细胞器,参与细胞供能、信息传递和细胞凋亡等过程,与多种疾病、衰老过程、发育及生物的性状密切相关[5]。线粒体基因组(mitochondrial DNA,mtDNA)独立于核基因组之外,具有分子质量小、独立复制、母系遗传、突变速率快等可成为理想遗传标记的特点,是研究物种起源和遗传进化的重要分子工具[6-7]。除此之外,线粒体产生的活性氧(reactive oxygen species,ROS)在以肺部为代表的多种疾病发病机制中发挥广泛的作用,过量的ROS还可以反作用在mtDNA上引起碱基突变[8-9]。由于林麝驯化期短,其警觉、易应激的特点使其疾病难以在病危前被察觉,并且麝的分类至今仍在探讨阶段[10-12]。因此本研究对林麝肺成纤维细胞mtDNA进行测序和分析,进一步丰富林麝mtDNA的信息,巩固林麝的系统分类,为林麝选育和遗传资源保护奠定基础。通过比较林麝mtDNA的差异,以期为林麝的肺部疾病前期发现和多样性保护等研究提供科学依据。
1 材料与方法
1.1 试验材料
林麝肺成纤维细胞系FMD-C1由四川农业大学动物检疫实验室建立并保存。线粒体基因组提取试剂盒购于北京索莱宝科技有限公司。
1.2 方 法
1.2.1 线粒体提取 将5×107个林麝肺成纤维细胞FMD-C1消化洗涤后离心,将收集到的细胞用1.0 mL预冷的Lysis Buffer重悬,冰浴研磨后转移至离心管,1 000×g离心5 min后收集上清并转移至新的离心管中重复离心,然后将收集到的上清再次转移至新的离心管中,12 000×g离心10 min,收集沉淀,经洗涤后将最终得到的高纯度线粒体沉淀用100 μL的Store Buffer重悬。全程4 ℃低温操作。浓度及纯度经NanoDrop 2000检测合格后,送至南京派森诺基因科技有限公司进行全基因组测序。
1.2.2 全基因组测序与组装 采用全基因组鸟枪法策略构建文库。然后利用第二代测序技术,基于Illumina NovaSeq平台对文库进行双末端测序。将平均质量值小于20和长度小于50 bp的序列过滤后,采用A5-miseq 2015和SPAdes 3.9.0对其余高质量数据从头拼装,构建contig和scaffold序列。根据拼接序列的测序深度提取序列,将高测序深度的序列同 NCBI 上的 nt 库进行 blastn比对,挑出各拼接结果的线粒体序列。将拼接结果整合,确定contig间的位置关系,填补gap。使用pilon 1.18软件对结果进行校正后,得到最终的林麝线粒体序列。
1.2.3 序列分析 采用 cgview(http://cgview.ca/)可视化软件绘制线粒体全基因组圈图。将拼接得到的完整线粒体基因组序列上传至在线服务器MITOS(http://mitos2.bioinf.uni-leipzig.de/index.py)进行功能注释和tRNA二级结构预测。再分别计算线粒体全基因组和各特征的碱基组成并分析。从NCBI下载已发布的林麝线粒体序列,将蛋白编码基因(protein coding gene,PCG)翻译后进行氨基酸差异比对,并比对两段非编码区的碱基位点突变情况。
1.2.4 系统发育分析 从NCBI上下载28个哺乳动物的mtDNA作为参考序列,包括麝科、牛科、鹿科、叉角羚科和其他哺乳动物。运用软件MEGA 7.0以邻接法构建系统进化树,bootstrap重复次数设置为1 000。
2 结果与分析
2.1 线粒体基因组结构与组成
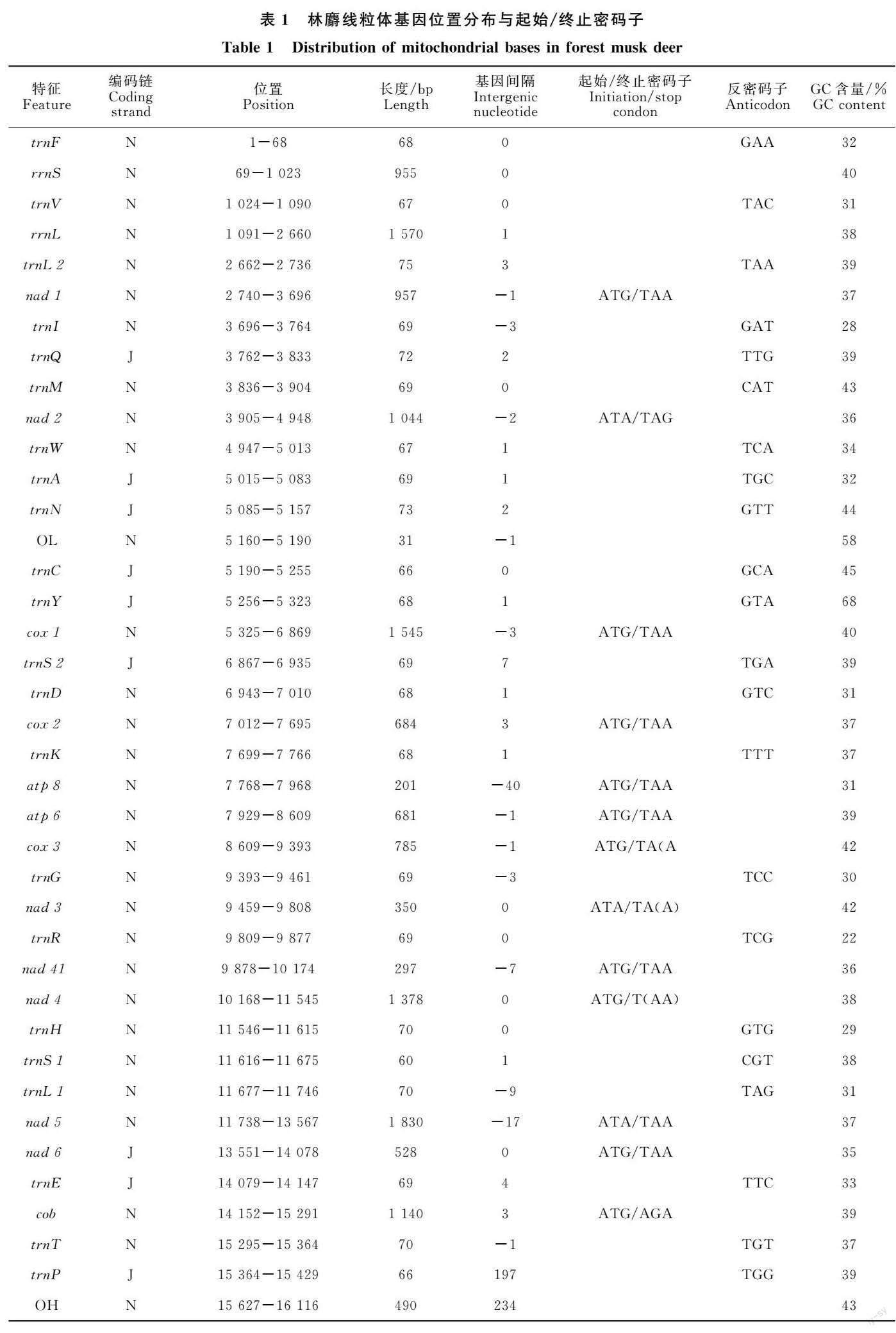
林麝肺成纤维细胞FMD-C1线粒体基因组序列经注释后提交GenBank(登录号:MW879208)。如图1所示,林麝线粒体基因组序列全长为16 351 bp,共有13个PCGs,22个tRNA,2个rRNA和两段非编码区。林麝FMD-C1线粒体的碱基组成为A(33.96%)、G(12.92%)、C(25.03%)和T(28.10%)。林麝线粒体各特征的分布和碱基组成情况如表1所示,线粒体基因排列紧凑,部分基因间存在重叠现象,除trnY之外,各基因的GC含量均小于50%,表明林麝线粒体基因组具有AT偏好性。
2.2 rRNA与tRNA编码基因结构特征
林麝肺成纤维细胞FMD-C1线粒体包含两个rRNA,分别为12S rRNA和16S rRNA,两者均位于正义链上,之间夹着1个trnV基因。与大多数麝类似,林麝线粒体中也包含22个tRNA基因,其序列长度为66~75 bp,tRNA的总序列长度达1 511 bp。22个tRNA基因中,有2个丝氨酸转运RNA和2个亮氨酸转运RNA;8个tRNA(trnQ、trnA、trnN、trnC、trnY、trnS2、trnE和trnP)在反义链上,其余14个tRNA均位于正义链。tRNA预测显示(图2),FMD-C1的 21个tRNA均可形成三叶草型二级结构,而 trnS1(AGY)由于缺失二氢尿嘧啶臂(D臂)而不能形成典型的三叶草结构。
2.3 蛋白编码基因
林麝线粒体包含13个PCGs,基因长度为201~1 830 bp,占基因组总长度的69.84%,除nad6外,其他12个蛋白基因均编码于正义链上。将林麝线粒体13个PCGs进行编码氨基酸差异比对,结果如图3所示,除cox2外的其余12个PCGs均有差异氨基酸出现。大部分差异氨基酸出现在登录号为KY792714的林麝上。对于FMD-C1,仅在4个PCGs中检测出差异氨基酸,分别是位于nad1的I102V、nad2的M217T、nad5的T41M、I580V和2~4位3个插入氨基酸KVI以及cob的I232V。
2.4 非编码区
林麝肺成纤维细胞FMD-C1线粒体的非编码区包含2 段,分别是D-loop (displacement-loop region) 区和OL (origin of L-strandreplicationregion)区。其中D-loop区位于trnP和trnF之间,共922 bp。统计两段非编码区的突变位点发现,OL区在林麝线粒体中无突变发生。D-loop区的碱基位点突变情况如图4所示,出现差异的90个碱基中,大部分突变发生在登录号为KY792714的林麝上,且同源树显示6条D-loop序列形成以KY792714和其余序列的两个分支。在其余5条序列中,则仅有30个位点出现碱基不一致的情况,其中FMD-C1的单一突变占19个,并在149~150 bp出现2个碱基缺失。
2.5 线粒体基因组系统进化分析
系统发育重建结果如圖5所示,该进化树由两个大分支构成,苏门答腊猩猩单独形成一个分支,在进化树上与林麝的亲缘关系最远。同科物种在进化树上聚类。5条林麝mtDNA序列于进化树顶部聚类,与安徽麝共同组成一个分支,而登录号为KY792714的林麝与喜马拉雅麝等处于同一分支,置信度均为100。根据聚类情况可知与FMD-C1亲缘关系最近的物种是安徽麝。
3 讨 论
mtDNA常常被作为遗传多样性分析的工具和相关疾病的分子标记。随着高通量测序技术的快速发展,其应用范围在林麝基因组学的研究中也越来越广。本研究通过高通量测序获得林麝肺成纤维细胞线粒体FMD-C1的全基因组序列。该序列为双链环状闭合分子,包含37个基因,基因排列顺序与其余林麝完全一致,总体碱基含量呈现出AT偏好,这可能与自然突变和选择压力等原因有关[6,13]。22个tRNA中,除trnS1外均能形成典型的三叶草结构。两个rRNA(rrnS和rrnL)均为单拷贝,基因内部无间隔区,也符合后生动物的特征[6]。
研究证实,线粒体损伤与炎症性疾病和自身免疫性疾病的发生有关,异常线粒体特征如mtDNA突变的发生率增加等也是导致多种肺部疾病的重要原因[8,14-15]。mtDNA的改变不仅可以作为独立的病理过程,而且还可以与现有的病理机制协同作用,从而诱发、促进或加剧肺部疾病[8]。Gazdhar等[16]的研究证明,mtDNA突变在肺纤维化过程中累积,成为ROS和随后纤维化的重要驱动因素,而ROS释放增加反过来继续损害mtDNA。且由于其不包含内含子或组蛋白,靠近ROS的产生位点和复制遵循不对称分裂等特点,mtDNA比核DNA更容易受到损伤,突变率高出10~20倍[15,17]。低效的氧化磷酸化(oxidative phosphorylation,OXPHOS)易引起ROS的产生,mtDNA编码的13种蛋白质均与OXPHO有关,因此本研究对13个PCGs进行单氨基酸突变分析[18]。从结果来看,不同功能的PCGs的碱基组成和选择压力不同。登录号为KY792714的林麝氨基酸差异率高,与其余林麝呈现显著异常,因此应除去该条序列进行讨论。与其余林麝PCGs编码蛋白对比,FMD-C1在nad1、nad2、nad5和cob中检测出单氨基酸突变,内容均为I与V、M与T之间的替换,经预测对蛋白质结构无明显影响,跨膜蛋白的分布也与其余序列一致,因此在FMD-C1的mtDNA中未检出引起PCGs明显功能障碍的突变位点。
目前,麝的分类仍在探讨中,早期麝科动物的分类主要依据外形、地理分布和解剖学特征,此后一些学者结合单基因序列比较来对麝科动物的系统分类进行研究,但研究结果仍存在差异,如彭红元等[10]提出,麝科动物的分类还存在着喜马拉雅麝和安徽麝等是不是有效种的争议。随着分子生物学技术的发展,借助于mtDNA可以提高线粒体遗传树和物种树之间的一致性概率。线粒体中的非编码区在调节线粒体DNA 复制和转录中起到非常重要的作用,该区域由于不编码基因,可以容纳和积累碱基突变[19-20]。非编码区一般包括两段,其中D-loop 区突变速率快且多态性丰富,因此在生物的遗传多样性和起源进化研究中具有重要意义。从D-loop区的碱基突变情况来看,与PCGs相同,大部分突变发生在登录号为KY792714的林麝上。除去该条序列,FMD-C1的该区域也存在着较为丰富的变异,有一定的遗传多样性。而登录号为EU043465和NC_012694的林麝D-loop区序列完全一致,可见两个种群间基因交流较多。
但实际上,单基因系统发育通常与涉及多个数据集的研究有很大不同,因此,完整的mtDNA逐渐被用于构建可靠的系统发育,以确定具有准确时间尺度的物种或更高分类群之间的进化关系[21]。本研究的系统进化树展示了明确的分类关系,鼷鹿相对于其他反刍动物依然占据基础位置,这与Hassanin等[22]的研究相同,不同的是麝科更靠近鹿科而不是牛科。本研究中,林麝与安徽麝的亲缘关系相近,这与Pan等[12]的研究一致。而对于登录号为KY792714的林麝线粒体mtDNA与喜马拉雅麝等聚类更近的情况,结合与其余林麝PCGs的氨基酸差异比较和D-loop区碱基突变位点分析结果,推测是该麝在分类时方法选择不同或是未鉴定的杂交个体,建议根据核基因组分析重新判断其作为林麝mtDNA的可靠性[23]。
结合线粒体基因组研究的意义,本研究结果为林麝的遗传多样性保护与育种提供了参考依据。同时也为进一步探讨林麝肺部疾病线粒体水平的致病机制奠定基础。
参考文献 Reference:
[1] 杨 威,伍 茜,程建国,等. 林麝干扰素α基因克隆、表达及转录调控分析[J]. 生物技术通报,2022,38(1):21-31.
YANG W,WU X,CHENG J G,et al. Cloning,expression and transcriptional regulation of interferon-α in forest musk deer[J].Biotechnology Bulletin,2021,38(1):1-11.
[2] 王 霞,袁旭东,周洪艳,等. 重庆金佛山国家级自然保护区林麝(Moschus berezovskii)种群数量初步调查及地理分布[J]. 中国农学通报,2022,38(1):21-31.
WANG X,YUAN X D,ZHOU H Y,et al. Moschus berezovskii in Chongqing Jinfo Mountain national nature reserve:population size and geographical distribution[J]. Chinese Agricultural Science Bulletin,2020,36(19):44-47.
[3] 伍 茜,李雄婕,喻 东,等. 林麝源支气管败血波氏杆菌FMDBb1株的分离鉴定及全基因组序列分析[J]. 微生物学通报,2021,48(12):4731-4741.
WU X,LI X J,YU D,et al. Isolation,identification and whole-genome analysis of Bordetella bronchiseptica FMDBb1 from forest musk deer (Moschus berezovskii)[J].Microbiology China,2021,48(12):4731-4741.
[4] 刘春燕. 林麝Moschus berezovskii养殖种群死亡原因及其生命生理特征研究[D]. 上海:华东师范大学,2008.
LIU CH Y. A study on reasons of death and physiological features for breeding forest musk deer(Moschus berezovskii)[D]. Shanghai:East China Normal University,2008.
[5] 侯俊秀. 冬蟲夏草菌的线粒体基因组测序及比较线粒体基因组学研究[D]. 太原:山西大学,2016.
HOU J X. Asssembly of the mitochondrial genome of Ophiocordyceps sinensis and its comparative genomic analysis[D]. Taiyuan:Shanxi University,2016.
[6] 刘 凯,冯晓宇,马恒甲,等. 钱塘江三角鲂线粒体基因组测序及其结构特征分析[J]. 浙江农业学报,2020,32(9):1591-1608.
LIU K,FENG X Y,MA H J,et al. Complete sequence and gene organization of mitochondrial genome of Megalobrama terminalis from Qiantang River[J]. Acta Agriculturae Zhejiangensis,2020,32(9):1591-1608.
[7] KRAMER P,BRESSAN P. Our(mothers) mitochondria and our mind[J]. Perspectives on Psychological Science,2018,13(1):88-100.
[8] CLOONAN S M,CHOI A M. Mitochondria in lung disease[J]. The Journal of Clinical Investigation,2016,126(3):809-820.
[9] ARAVAMUDAN B,THOMPSON M A,PABELICK C M,et al. Mitochondria in lung diseases[J]. Expert Review of Respiratory Medicine,2013,7(6):631-646.
[10] 彭红元,陈伟才,张修月. 麝的分类研究概述[J]. 玉林师范学院学报,2010,31(02):66-72.
PENG H Y,CHEN W C,ZHANG X Y. A review on the classification of musk deer[J]. Journal of Yulin Normal University (Natural Science),2010,31(2):66-72.
[11] 竭 航,郑程莉,王建明,等. 林麝分子遗传学研究进展[J]. 中国中药杂志,2015,40(22):4319-4323.
JIE H,ZHENG CH L,WANG J M,et al. Research progress on molecular genetics of forest musk deer[J]. China Journal of Chinese Materia Medica,2015,40(22):4319-4323.
[12] PAN T,WANG H,HU C,et al. Species delimitation in the genus moschus (ruminantia:Moschidae) and its high-plateau origin[J]. Plos One,2015,10(8):e134183.
[13] 夏立萍,徐 鸣,郭宝英,等. 等边浅蛤线粒体全基因组测序和系统发育研究[J]. 浙江海洋大学学报(自然科学版),2021,40(2):93-100.
XIA L P,XU M,GUO B Y,et al. Complete mitochondrial genome sequencing and phylogeny analysis of macridiscus multifarius[J].Journal of Zhejiang Ocean University (Natural Science),2021,40(2):93-100.
[14] RILEY J S,TAIT S W. Mitochondrial DNA in inflammation and immunity[J].EMBO Reports,2020,21(4):e49799.
[15] ANNESLEY S J,FISHER P R. Mitochondria in health and disease[J].Cells,2019,8(7):680.
[16] GAZDHAR A,LEBRECHT D,ROTH M,et al. Time-dependent and somatically acquired mitochondrial DNA mutagenesis and respiratory chain dysfunction in a scleroderma model of lung fibrosis[J].Science Report,2014,4:5336.
[17] SON J M,LEE C. Mitochondria:multifaceted regulators of aging[J].BMB Reports,2019,52(1):13-23.
[18] SINGH A,KUKRETI R,SASO L,et al. Oxidative stress:a key modulator in neurodegenerative diseases[J].Molecules,2019,24(8):1583.
[19] 張惠锋,王华伟,谢振荣,等. 结直肠癌患者瘤内线粒体DNA非编码区突变异质性研究[J]. 中华肿瘤防治杂志,2019,26(7):473-478.
ZHANG H F,WANG H W,XIE ZH R,et al. Intratumoralheterogeneity of non coding region of mitochondria DNA in patients with colorectal cancer[J]. Chinese Journal of Cancer Prevention and Treatment,2019,26(7):473-478.
[20] 于 萍,曹 婷,赵春萍,等. 五指山猪线粒体基因组全序列测定与分析[J]. 西北农业学报,2015,24(7):16-22.
YU P,CAO T,ZHAO CH P,et al. Complete mitochondrial genome sequence and analysis of wuzhishan pig[J]. Acta Agriculturae Boreali-occidentalis Sinica,2015,24(7):16-22.
[21] 肖 帆,张利平,朗 侠,等. 不同品种绵羊D-loop区遗传多样性研究[J]. 西北农业学报,2021,30(8):1122-1129.
XIAO F,ZHANG L P,LANG X,et al. Genetic diversity of mitochondrial DNA D-loop region in different breeds of sheep[J]. Acta Agriculturae Boreali-occidentalis Sinica,2021,30(8):1122-1129.
[22] HASSANIN A,DOUZERY E J. Molecular and morphological phylogenies of ruminantia and the alternative position of the moschidae[J].Systematic Biology,2003,52(2):206-228.
[23] YANG C,WANG W F,DU X J,et al. Mitochondrial genome of captive Alpine musk deer,Moschus chrysogaster (Moschidae),and phylogenetic analyses with its coordinal species[J]. Mitochondrial DNA. Part B,Resources,2021,6(2):598-600.
Abstract Moschus berezovskii(FMD) is a kind of nationally protected species with high value. In order to further study the structural characteristics and genetic variation of mitochondrial genome of FMD,the whole-genome of FMD-C1 mitochondria from FMD fibroblasts was sequenced based on Illumina novaseq platform. Then its features were annotated. Structure prediction,sequence analysis and systematic development reconstruction were carried out. The results showed the length of whole-genome sequence was 16 351 bp. With obvious AT preference,there were 13 protein coding genes (PCGs),22 tRNAs,2 rRNAs and two non-coding regions. The sequence was submitted to GenBank and the accession number MW879208 was obtained. Compared with other FMD mitochondrial whole-genome sequences of NCBI,5 single amino acid mutations and 3 inserted amino acids were detected in 4 PCGs of FMD-C1; 19 differential bases and 2 bases deletion were detected in D-loop region. Phylogenetic analysis showed that moschus anhuiensis was the closest relative to FMD.The results provided a reference for the breeding and genetic diversity protection of FMD,and also laid a foundation for further exploring the pathogenic mechanism of mitochondrial level of lung diseases in FMD.
Key words Moschus berezovskii; Mitochondrial DNA; Protein coding gene; Non-coding region; Phylogeny

