含羞草叶片转录组测序及其黄酮合成途径解析
毛仁俊 白朕卿 何志贵 李秋月 吴佳文 陈浩 阎岩


摘 要 为了解析含羞草中黄酮类物质的生物合成途径,利用Illumina platform 2000TM测序平台对含羞草叶片进行转录组测序,共获得94 182个Unigene,平均长度为695 bp,N50值为1 159 bp。共有30 243个Unigene注释于50个GO功能组中,其中“代谢过程”“催化活性”以及“细胞”注释的Unigene数量较多。KEGG通路分析鉴定出49个Unigene注释在黄酮生物合成途径,分别编码查尔酮合成酶(CHS,12个Unigene)、查尔酮异构酶(CHI,4)、黄烷酮-3-羟化酶(F3H,3)、黄烷酮-3-羟化酶(F3H,9)、黄烷酮-3,5-羟化酶(F3,5H,1)、二氢黄酮醇4-还原酶(DFR,5)、黄酮醇合成酶(FLS,3)以及无色花色素还原酶(LAR,12)基因。从转录组数据库中鉴定出7 382个以单核苷酸和三核苷酸为主要类型的SSR标记。随机选出10对SSR引物进行扩增,其中有7对能够扩增出清晰的条带。
关键词 含羞草; 转录组测序; 代谢途径; 黄酮生物合成
含羞草(Mimosa pudica Linn)是豆科含羞草属的一种多年生草本或小灌木,主要分布于热带和亚热带地区,具有较高的观赏价值。含羞草以全草入药,在印度、孟加拉国、菲律宾以及中国广西、云南等少数民族地区是一种常用药材[1-2]。中国古代医学典籍《生草药性备要》、《本草求原》以及《岭南采药录》中均记载了含羞草味甘、性寒,具有清热解毒、消肿止痛的功效。现代药理学研究表明含羞草具有愈合创伤[3]、抗菌消炎[4]、安神止痛[5]、降低血压[6]以及缓解糖尿病病症[7]等多种功效。此外,含羞草作为触感性研究的模式植物,具有显著的触感特性,即在受到强光、震动、风吹、碰触等外界刺激时会迅速闭合叶片、收紧枝条(图1)。
含羞草中含有儿茶素、槲皮素、豆甾醇以及对香豆酸等黄酮类物质,也含有生物碱、皂苷以及酚酸类化合物[8-9],这些化合物是其发挥药效功能的物质基础。Zhang等[1]通过核磁共振氢谱(1H-NMR)、核磁共振碳谱(13C-NMR)以及质谱(MS)的方法从含羞草中分离出异红草素、异牡荆素及牡荆素等黄酮类物质,并对其活性进行分析。Yuan等[10]利用红外光谱(IR)、质谱(MS)以及核磁共振(NMR)的方法首次从含羞草中分离出黄酮类化合物6,7,3′,4′-四羟基-8-C-[α-L-鼠李糖-(1→2)]-β-D-葡糖黄酮碳苷和5,7,3′,4′-四羟基-8-C-[β-D-芹菜糖-(1→4)]-β-D-葡糖黄酮碳苷。乔文涛等[11]通过正交分析法对含羞草中黄酮类物质的研究表明,以10倍量的80%乙醇提取,利用大孔吸附树脂进行分离,再以不同浓度的乙醇进行梯度洗脱,最后经过浓缩得到高浓度的黄酮。
目前,关于含羞草的研究主要集中于药效功能[1,12]、触感性效应[13-14]以及生理活性等方面[15-16],而分子水平的研究匮乏,组学研究鲜见报道。早期对于含羞草触感性的研究已经证明含羞草枝条基部的叶枕是調节其叶片开合的主要器官[17],也鉴定出钾-L-苹果酸,镁钾反式乌头酸等与其触感性相关的化合物[13-14]。然而,近年来触感性相关研究进展缓慢,制约其发展的重要因素是分子水平研究不足,次生代谢物合成途径尚未解析。因此,本研究利用转录组测序技术对含羞草叶片进行分析,解析黄酮生物合成途径,注释途径中的关键酶基因,以期为深入研究黄酮途径中关键酶基因的功能以及进一步开发含羞草药用价值提供参考。
1 材料与方法
1.1 植物材料
含羞草样品于2019年6月采集于广西药用植物园。试验样品经过延安大学生命科学学院阎岩教授鉴定为豆科(Leguminosae)含羞草属(Mimosa Linn)植物含羞草(Mimosa pudica L.)。采集6株长势良好且无病虫害的含羞草植株地上部分,选取叶片用于后续试验分析。将含羞草叶片分为两份,一份用锡箔纸包裹后立即置于液氮中速冻,随后置于超低温冰箱保存,另一份样品阴干后用于黄酮类物质的测定。
1.2 总黄酮和儿茶素含量的测定
以亚硝酸钠-氯化铝-氢氧化钠显色法测定含羞草叶片中总黄酮的含量[18]。精确称取过40目筛的含羞草叶片粉末1.0 g,置于具塞烧瓶中,加入25 mL体积分数为 70%的乙醇,称量。加热回流1 h,冷却后再次称量,并用70%乙醇补足质量,过滤后待用。取1 mL样品溶液置于25 mL容量瓶中,加入6 mL超纯水和1 mL 5%的亚硝酸钠溶液,混匀后静置6 min,再加入10% 硝酸铝溶液1 mL,加入10 mL 4.3%氢氧化钠,用70%乙醇定容,静置15 min。利用分光光度计(UV-2450,Shimadzu)测定波长510 nm处的吸光度值,以芦丁(纯度>98%,Sigma,USA)为对照品制作标准曲线计算总黄酮含量。以香荚兰素比色法[19]测定儿茶素的含量。取含羞草叶片粉末1.0 g置于具塞烧瓶中,加入25 mL 70%甲醇,称量。加热回流0.5 h,放冷后再次称量,用70%甲醇补足质量,过滤,取续滤液待测。以儿茶素(纯度>98%,Sigma,USA)为对照品绘制标准曲线计算儿茶素含量。
1.3 RNA的提取和测序
利用RNeasy Plant Mini试剂盒(Qiagen,Hilden,Germany)提取含羞草叶片的总RNA,提取方法参照试剂盒说明。通过Nanodrop核酸蛋白检测仪(Thermo Scientific,MA,USA)和Qubit 2.0核酸定量仪(Life Technologies,CA,USA)分析样品RNA的质量。cDNA文库的构建参照RNA-Seq文库构建试剂盒(Illumina,USA)说明书进行。测序工作由北京百迈客生物科技有限公司利用Illumina HiSeq2000TM测序平台完成。
1.4 转录组数据的拼接、组装及功能注释
剔除测序结果中的接头读序、低质量读序以及包含ploy-N的读序,获得高质量读序。利用Trinity软件[20]对序列进行组装,获得Unigene和转录本(Transcript)。利用Blast2GO进行GO功能注释[21],利用KOBAS 2.0进行KEGG代谢通路注释[22]。通过BLASTX将Unigene分别与Nr(NCBI非冗余蛋白数据库)、Swiss-Prot(Swiss-蛋白序列数据库)、eggNOG(基因进化谱系:无监督的同源群体)、KEGG(京都基因和基因组百科全书)和KOG数据库(蛋白直系同源数据库)进行相似性比对,获取Unigene的功能注释和分类信息。
1.5 SSR标记的开发与鉴定
使用MicroSatellite(http://pgrc.ipk-gatersleben.de/misa/)軟件对转录组中的序列长度大于1 kb的Unigene进行串联重复位点检测。以单核苷酸重复不少于10次、二核苷酸重复不少于6次、三核苷酸、四核苷酸、五核苷酸、六核苷酸不少于4次为标准,统计分析SSR分子标记的种类与数量[23]。从转录组开发出的SSR数据库中随机选出10对引物进行PCR扩增验证,引物序列信息见表1。
2 结果与分析
2.1 总黄酮与儿茶素含量测定
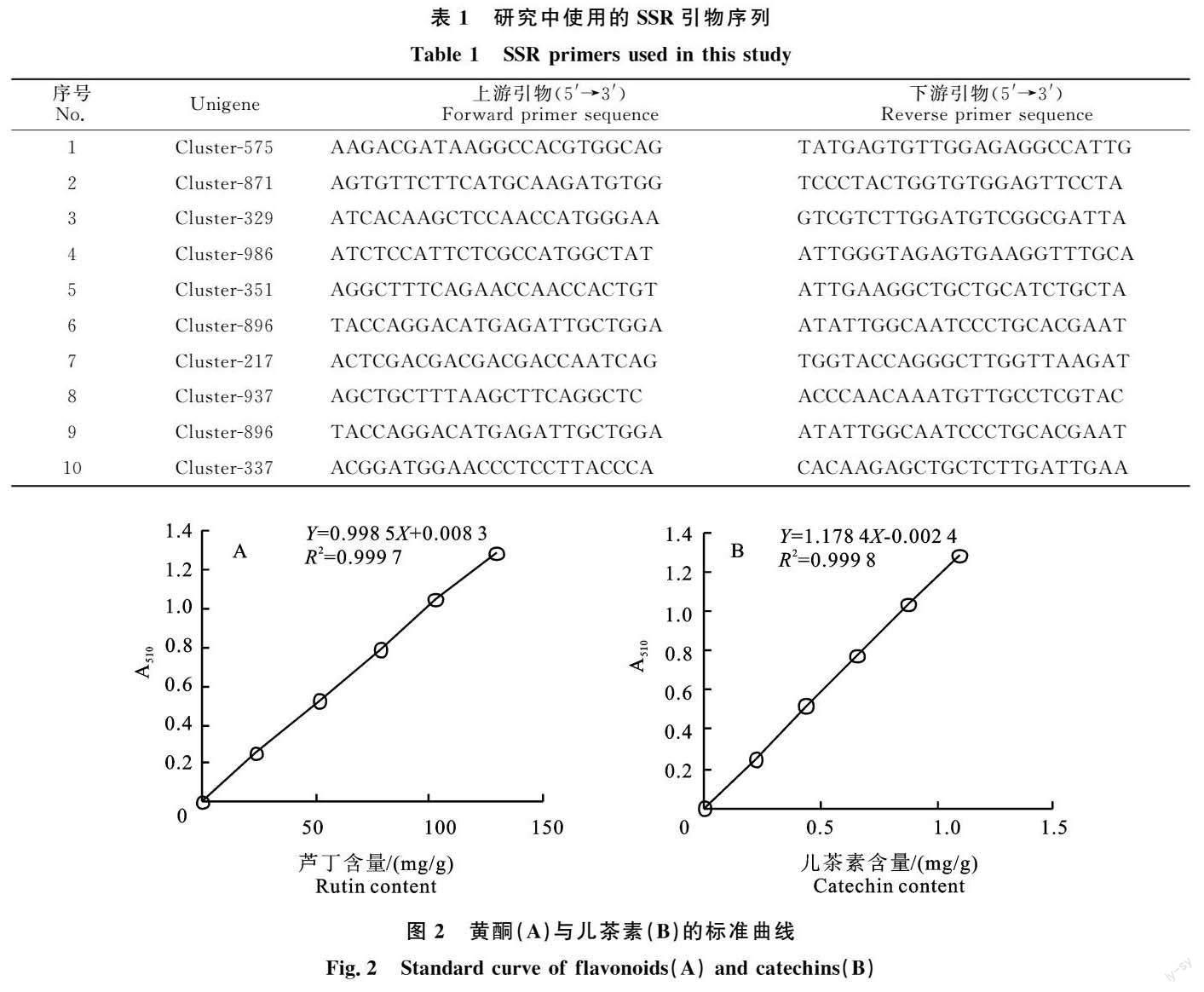
利用分光光度法测定含羞草叶片中总黄酮和儿茶素的含量。以不同浓度芦丁为横坐标,对应的吸光度值为纵坐标绘制标准曲线,得到总黄酮标准曲线为Y=0.998 5X+0.008 3,R2=0.999 7,线性范围0~128.9 mg/g(图2-A)。测定结果表明3份含羞草样品中总黄酮含量为75.71 mg/g±0.42 mg/g;以同样方法获得儿茶素标准曲线为Y=1.178 4X-0.002 4,R2=0.999 8,线性范围0~1.1 mg/g,3份样品中儿茶素含量为0.223 mg/g±0.012 mg/g(图2-B)。
2.2 序列组装
3个样本共获得67 400 933个高质量读序,对比成功的读序数量为55 702 861,3个样本中比对成功的读序比例均超过82%(表2)。本次测序总数据量为20.08 GB。拼接后共获得211 405个转录本,平均长度为1 178 bp,N50值为1 962 bp。共获得94 182个Unigene,平均长度为695 bp,N50值为1 159 bp。样本的平均GC含量为45.86%,Q30值均大于93.35%(表2)。测序错误率小于0.1%的碱基数量百分比为92.48%。Unigene长度分布结果表明,长度为1 000~2 000 bp和300~500 bp的Unigene占比较大,分别为37.30%(35 134个)和11.46%(10 795个)。这些指标表明本次测序结果良好,得到大量序列信息,能够满足后续生物信息学分析的要求。转录组数据已提交至美国国家生物技术信息中心数据库,获得序列号PRJNA634474。
2.3 Unigene的功能注释
以E-value≤e-5且HMMER E-value≤e-10为标准进行BLAST,共获得47 090个有注释信息的Unigene。进一步分析表明分别有13 921、30 243、19 427、26 970、29 902、33 103、44 256和46 480个Unigene注释在COG、GO、KEGG、KOG、Pfam、Swissprot、eggNOG和Nr数据库中。注释到长度大于1 kb的Unigene共有15 775个,占所有注释Unigene的33.5%(表3)。Nr注释的物种分类表明,比对到木豆(Cajanus cajan)的Unigene最多,为3 207个,占比6.90%,随后为大豆(Glycine max)3 147个、橡胶树(Hevea brasiliensis)2 709个、羽扇豆(Lupinus angustifolius)2 608个以及麻风树(Jatropha curcas)2 144个(图3)。
2.4 GO功能分类
按照GO功能分类,30 243个Unigene可分入细胞组成(CC)、分子功能(MF)和生物过程(BP)3大类,50个功能组(图4)。细胞组成中的Unigene可进一步分为15个功能组,其中以“细胞”(3 887个Unigene)和“细胞部分”(13 813)数量较多;分子功能中的Unigene同样被分入15个功能组,主要注释在“催化活性”(16 651)和“结合”(14 544);在生物过程中,Unigene被分入20个功能组,以“代谢过程”(15 764)和“细胞过程”(1 416)中的Unigene数量较多(图4)。
在KOG数据库中比对Unigene信息,预测 Unigene功能并进行分类统计。结果表明共有94 182个Unigene获得注释信息,可分为25类(图5)。其中“一般功能预测(R)”注释的Unigene数量最多,为6 252个,占Unigene总数的23.18%。随后依次为“翻译后修饰、蛋白质转换、分子伴侣(O)”(2 979,11.05%)、“信号转导机制(T)” (2 689,9.97%)、“翻译-核糖体结构和生物发生(J)”(1 385,5.14%),而“细胞活动性”(N)(9,0.03%)、“细胞外部结构(W)”(89,0.33%)及“核结构(Y)”(113,0.42%)注释到的Unigene数量较少(图5)。
2.5 KEGG途径分类注释
为进一步研究含羞草的代谢途径,将获得功能注释的Unigene进行KEGG通路分析。结果表明共有9 155个Unigene注释在“新陈代谢”、“遗传信息处理”、“环境信息处理”、“细胞过程”以及“有机系统”5个大类、28个小类(图6)。注释在“新陈代谢”中的Unigene数量最多,共有3 885个,其中又以“碳代谢(700)、氨基酸生物合成(619)”、“淀粉及糖类代谢(508)”注释到的Unigene数量较多。共有3 243个Unigene注释在“遗传信息处理”,其中有603个Unigene注释在“核糖体”,数量最多,“内质网蛋白加工”中注释的Unigene次之(482个)。“环境信息处理”中注释到的Unigene较少,其中“ABC转运器”途径仅有92个Unigene(图6)。
2.6 含羞草黄酮生物合成途径的解析
黄酮是含羞草中重要的药效成分,本研究重点关注含羞草黄酮生物合成途径(ko00941)。注释分析的结果表明共有9 155个Unigene注释在136条KEGG途径,其中49个Unigene注释在黄酮生物合成途径,分别编码查尔酮合成酶(CHS)、查尔酮异构酶(CHI)、黄烷酮-3-羟化酶(F3H)、黄烷酮-3-羟化酶(F3H)、黄烷酮-3,5-羟化酶(F3,5H)、二氢黄酮醇4-还原酶(DFR)、黄酮醇合成酶(FLS)以及无色花色素还原酶(LAR),涵盖了途径中所有已知的关键酶。其中,编码CHS和LAR基因的Unigene数量最多,均为12个,而编码F3,5H的Unigene最少,仅有1个。如图7所示,苯丙氨酸解氨酶(PAL)催化苯丙氨酸转化为反式肉桂酸,经过肉桂酸-4-羟化酶(C4H)、对香豆酸-辅酶A连接酶(4CL)的作用将苯丙烷代谢途径(KEGG 数据库通路 ID:ko00940)与黄酮生物合成途径连接。香豆酰辅酶A在CHS的作用下形成查尔酮,随后在CHI作用下形成柚皮素,再经过F3H的催化形成二氢山奈酚。二氢山奈酚是黄酮合成途径下游的一个节点物质,能够在不同酶的作用下生成多种黄酮类物质。例如,二氢山奈酚在F3,5H的作用下生成二氢杨梅素,随后在LAR的催化下生成儿茶酚。儿茶酚是含羞草中一种主要的黄酮类物质。另外,二氢山奈酚在F3H的作用下生成二氢槲皮素,进而经过FLS的催化生成类黄酮(图7)。本研究也鉴定出一些Unigene注释在氮素代谢(ko00910)、萜类骨架的生物合成(ko00900)和苯丙素生物合成(ko00940)等次生代谢途径中,其中注释在苯丙素生物合成途径的Unigene数量最多,为212条(表4)。
2.7 SSR标记的开发
在含羞草转录组数据库中共鉴定出7 382个SSR标记。根据SSR的类型可分为单核苷酸(3 554)、二核苷酸(1 637)、三核苷酸(1 978)、四核苷酸(160)、五核苷酸(30)以及六核苷酸重复(23)。其中单核苷酸A/T(1 692/1 784)和三核苷酸GAA/TTC(172/125)为主要重复类型。从开发出的SSR标记中随机选取10对引物,并进行PCR扩增验证,结果如图8所示,7对引物对能够扩增出清晰条带,泳道4的引物扩增出的条带较暗,而泳道9、10的2对引物未能成功扩增出条带。
3 讨论与结论
转录组测序具有分析效率高、覆盖面广,且不需要了解物种基因组信息便可获得大量转录水平的数据,在植物代谢途径解析[24]、SSR标记位点挖掘[25]以及基因功能研究等[26]领域已有广泛应用。含羞草作为一种非模式植物,转录水平研究匮乏,参与黄酮生物合成的相关基因未见报道。本研究利用转录组测序技术对含羞草叶片进行测序分析,共获得20.08 GB数据,丰富了含羞草的转录组数据。原始数据经过拼接后得到94 182个Unigene,GO富集分析将Unigene注释于50个GO功能组。共有9 155个Unigene注释在136条KEGG代谢通路,其中包括黄酮生物合成、苯丙素生物合成以及萜类骨架生物合成等重要的次生代谢途径。
利用转录组数据开发SSR标记具有广阔的应用前景。肖亮等[27]从葛根转录组中鉴定出25 452个SSR标记,选取28对引物对不同葛根资源进行了遗传分析,表明不同葛根种质具有很高的遗传多样性。梅利那等[28]基于转录组分析开发出大量马尾松的SSR标记。以多态性较高的24对引物对27份马尾松进行鉴定,发现供试样品遗传相似系数为0.32~0.92,UPGMA分析将样品分为3大类。该研究结果为构建马尾松遗传图谱、绘制指纹图谱提供了参考。本研究结果表明含羞草具有丰富的SSR位点,以单核苷酸(A/T)和三核苷酸(GAA/TTC)为主要类型。杨晓等[29]对秦艽,徐志军等[30]对花生SSR的分析也得出相似的结果。开发出的SSR标记能够为今后研究含羞草遗传多样性、种群结构以及分子辅助育种提供参考,也为后续有针对性的开发药用型、观赏型含羞草资源提供了分子水平的依据。
黄酮生物合成途径是植物体内重要的次生代谢途径之一。利用转录组测序能够高效获取黄酮合成相关的转录本,为解析黄酮合成途径,分析基因功能提供参考。马丹丹等[31]利用转录组测序技术对罗布麻进行研究,鉴定出查尔酮合成酶、查尔酮异构酶等参与黄酮合成的基因;姚运法等[32]分析黄秋葵花和果荚中的类黄酮代谢途径,结果表明黄秋葵花是通過F3H、DFR、ANS以及葡萄糖基转移酶将柚皮素催化生成花青素苷,而黄秋葵果荚则是通过F3,5H将柚皮素催化成为二氢杨梅素,表明黄酮途径在花和荚果中存在差异。本研究以转录组分析为基础,鉴定出含羞草中黄酮合成途径中的8个关键酶基因CHS、CHI、F3H、F3H、F3,5H、FLS、LAR和DFR的Unigene,初步构建了黄酮生物合成途径。此外,本研究也鉴定出参与异喹啉生物碱合成、萜类骨架生物合成以及苯丙素生物合成相关的Unigene。该结果与含羞草中含有L-含羞草碱、多酚类物质的情况相符,为解析含羞草中其他药效物质的合成途径提供了新的线索。
本研究对含羞草叶片进行了转录组测序分析,获得大量转录水平数据。GO富集分析表明Unigene主要注释在“代谢过程”、“催化活性”以及“细胞”三个功能组。KEGG通路分析鉴定出49个Unigene注释在黄酮生物合成途径,编码该途径中的查尔酮合成酶、查尔酮异构酶、黄烷酮-3-羟化酶、黄烷酮-3-羟化酶、黄烷酮-3,5-羟化酶、二氢黄酮醇4-还原酶、黄酮醇合成酶以及无色花色素还原酶基因。本研究结果丰富了含羞草的转录组数据,构建了黄酮生物合成通路,为深入研究黄酮合成途径关键酶基因的功能以及探索含羞草叶片开合的分子机制奠定了基础。
参考文献 Reference:
PANG D D,LIU Y F,SUN Y N,et al.Full-length transcriptome and gene structure analysis of kucha(Camellia sinensis) [J].Acta Agriculturae Boreali-occidentalis Sinica,2021,30(4):563-571.
[27] 肖 亮,尚小红,曹 升,等.基于转录组测序的葛根SSR标记研究与利用[J].西北植物學报,2019,39(1):59-67.
XIAO L,SHANG X H,CAO SH,et al.Utilization of simple sequence repeat(SSR) markers development from a de novo transcriptome assembly in Pueraria thomsonii Benth[J].Acta Botanica Boreali-Occidentalia Sinica,2019,39(1):59-67.
[28] 梅利那,范付华,崔博文,等.基于马尾松转录组的SSR分子标记开发及种质鉴定[J].农业生物技术学报,2017,25(6):991-1002.
MEI L N,FAN F H,CUI B W,et al.Development of SSR molecular markers based on transcriptome sequences and germplasm identification in Masson pine(Pinus massoniana)[J].Journal of Agricultural Biotechnology,2017, 25(6):991-1002.
[29] 杨 晓,马子豪,马 婕,等.粗茎秦艽种子萌发过程的转录组及关键因子分析[J].中草药,2021,52(1):219-226.
YANG X,MA Z H,MA J,et al.Transcriptome and key factors analysis of Gentiana crasicaulis in seed germination process[J].Chinese Traditional and Herbal Drugs,2021,52(1):219-226.
[30] 徐志军,赵 胜,徐 磊,等.基于 RNA-seq 数据的栽培种花生 SSR 位点鉴定和标记开发[J].中国农业科学,2020,53(4):695-706.
XU ZH J,ZHAO SH,XU L,et al.Discovery of microsatellite markers from RNA-seq data in cultivated peanut(Arachis hypogaea)[J].Scientia Agricultura Sinica,2020,53(4):695-706.
[31] 马丹丹,周佳熠,马鹏举,等.罗布麻转录组数据的注释和黄酮合成相关基因的挖掘[J].基因组学与应用生物学,2019,38(10):4580-4587.
MA D D,ZHOU J Y,MA P J,et al.Assembly on the data of Apocynum venetum transcriptome group and mining of genes involved in flavonoid synthesis[J].Genomics and Applied Biology,2019,38(10):4580-4587.
[32] 姚运法,张少平,练冬梅,等.黄秋葵花和果荚转录组测序及类黄酮代谢差异表达分析[J].西北植物学报,2018, 38(11):2000-2009.
YAO Y F,ZHANG SH P,LIAN D M,et al.Transcriptome sequencing and differential expression analysis of flavonoid metabolism in flowers and fruits of okra[J].Acta Botanica Boreali-Occidentalia Sinica,2018,38(11):2000-2009.
Abstract To understand the flavonoid biosynthesis pathway of M.pudica,Illumina platform 2000TM platform was used to assemble the transcriptome of M.pudica leaf,a total of 94 182 unigene were obtained,with average length of 695 bp,the N50 value was 1 159 bp.Totally of 30 243 unigene were annotated in 50 GO functional classification and dominated by “Metabolic process” “Catalytic activity” and “Cell”.A total of 7 382 SSR were explored from transcriptome database,among which mononucleotide and trinucleotide were the dominant types.Seven out of ten randomly selected primers could produce clear fragments.The results of KEGG analysis indicated that 49 unigene was identified in the flavonoid biosynthesis pathway,and chalcone synthase(CHS,12 unigene),chalcone isomerase(CHI,4),flavanone-3-hydroxylase(F3H,3),flavanone-3-hydroxylase(F3H,9),flavanone-3,5-hydroxylase(F3,5H,1),flavanone 4-reductase(DFR,5),flavonol synthase(FLS,3),and leucoanthocyanidin reductase(LAR,12) were encoded.
Key words Mimosa pudica Linn; Transcriptome sequencing; Metabolic pathway; Flavonoid biosynthesis

