基于线粒体COI基因对棘胸蛙养殖群体的遗传多样性分析
张燕萍 章海鑫 习宏斌 吴子君 廖再生


文章编号:1006-3188(2023)04-001-05
摘要:为研究江西省棘胸蛙的遗传多样性,利用线粒体COI分子标记对采自江西省内峡江、靖安、金溪、明月山、于都5个棘胸蛙养殖群体144个样本进行遗传多样性分析。结果显示:144条棘胸蛙线粒体COI长度为990bp,共检测出变异位点270个,包括简约信息位点250个,单突变位点20个,2个缺失和插入;共检测到35个不同的单倍型,单倍型多样性为0.927;核苷酸多样性为0.0405,平均核苷酸差异数(K)为40.018。养殖群体除峡江和于都群体的遗传多样性较高,其余3个群体均较低;群体内发生了极大的分化,群体变异主要来源于群体内(78.08%)。
关键词:棘胸蛙;COI;养殖群体;遗传多样性
中图分类号:Q959.53;S966.3文献标识码:A
棘胸蛙(Quasipaa spinosa)又名石蛙、石鸡等,属脊椎动物门、两栖纲、无尾目、叉舌蛙科、棘胸蛙属[1],具有肉质细嫩、味道甘美、营养丰富的特点,而且具有较高的经济和科研价值,所以受到越来越多的人关注。为了满足市场需求及保护野生资源,江西20世纪80年代初开始人工养殖棘胸蛙,人工养殖过程中近亲繁殖,导致我省棘胸蛙出现生长速度慢、抗病力差、个头小等问题,缺乏对养殖群体进行遗传多样性分析。
细胞色素氧化酶亚基I(Cytochrome coxidase subunit I,COI)是线粒体中13个蛋白编码基因之一,该基因功能比较保守,同时又有一定的进化速率,包含的遗传进化信息量较大,在物种内变异较小,种间变异较大,很少存在插入和缺失,且容易被通用引物所扩增,所以在水生生物种群遗传多样性及遗传结构分析、系统发育等发面得到了广泛应用[2-3]。
本研究基于线粒体DNACOI,对江西省内5个棘胸蛙养殖群体进行遗传多样性与遗传结构分析,旨在为解决棘胸蛙人工养殖过程中由于遗传多样性下降而导致养殖效益降低的现状,同时为我省棘胸蛙的品种选育与改良工作提供理论基础,为棘胸蛙种质资源恢复与可持续利用提供科学依据。
1 材料与方法
1.1 样品采集
棘胸蛙采自江西省吉安地区峡江群体(XJ)22尾、宜春地区明月山群体30尾、宜春地区靖安群体32尾、赣州地区于都群体30尾、抚州地区金溪群体30尾。共计144尾,剪肌肉用95%乙醇保存,运回实验室,-20℃冷冻保存备用。
1.2 实验方法
1.2.1 基因组DNA的提取
取少量棘胸蛙的肌肉,利用生工生物工程(上海)有限公司提供的动物基因组提取试剂盒提取。提取后的DNA利用1.0%的琼脂糖凝胶电泳检测其完整性,-20℃冷冻保存备用。
1.2.2 PCR扩增
用于线粒体COI通用引物,引物序列为F(5′–GAAAGACGGGCCTTGATCCC–3′)和R(5′–GCCATTGAGAGGAGTACAGGG–3′),送至上海生工生物工程有限公司合成。PCR擴增体系为:10×buffer(MgCl2)2.5μL,dNTP(mix)(10mmol/L)1μL,引物(10mmol/L)各1μL,模板DNA(50ng/L)2μL,Taq DNA Polymerase(5U/μL)0.2μL,加超纯水至25μL。PCR反应程序:95℃预变性5min;94℃变性30 s,63℃退火30 s,72℃延伸 30 s,10个循环;95℃变性 30 s,58℃退火 30 s,72℃延伸 30 s,30个循环;72℃ 延伸 10 min,4℃保存。
1.2.3 PCR产物检测和测序
PCR产物取5μL用1.0%琼脂糖凝胶电泳检测PCR产物的长度和浓度合格后,将PCR产物送往生工生物工程(上海)有限公司纯化与双向测序,测序引物同上。
1.3 数据处理与分析
测得的上下游序列利用DNA序列用SeqMan软件进行拼接并手工校对。Clustal W(1.83)软件[4]分析比对;利用DNAsp 5.0[5]软统计单倍型及变异位点(S)、计算单倍型多样性(Hd)、核苷酸多样性(Pi)、平均核苷酸差异(K)等多样性参数。用MEGA(version 6.0)软件[6]中的Kimura双参数法计算各单倍型间的遗传距离;采用NJ(Neighbor-joining)法,Bootstrap置信值估算重复次数1000次,对基因序列数据进行系统分析。用Ariequin3.11[7]软件计算两两群体间的分化指数(Fst),进行分子方差分析(analysis of molecular variance,AMOVA)。
2 结果与分析
2.1 棘胸蛙COI基因序列PCR扩增
PCR扩增产物的琼脂糖凝胶电泳检测,见图1。由图1可知,棘胸蛙COI序列条带电泳图谱条带清晰、明亮,线粒体COI序的片段大于1000bp。这可说明提取的棘胸蛙肌肉DNA质量较高,能够用于后期的群体遗传学研究(见图1)。
2.2 棘胸蛙线粒粒体COI序列特征
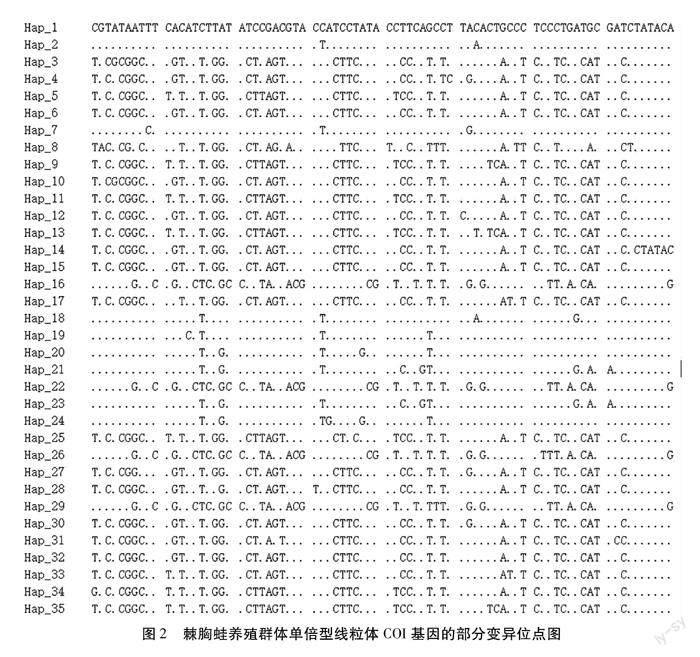
序列经比对后,获得的棘胸蛙样品的线粒体COI序列长度为990bp,检测到插入和缺失位点2个,检测到变异位点270个,其中简约信息位点250个,单突变位点20个(图2)。碱基平均含量:A(27.1%)、T(31.3%)、G(16.8%)和C(24.8%),A+T含量(58.4%)显著高于G+C含量(41.6%),表现为明显的碱基组成偏倚性,与一般脊椎动物线粒体DNA序列特征基本一致。
2.3 单倍型分析
基于5个棘胸蛙养殖群体144条COI序列,共界定出35种单倍型,结果见表1,各群体包含单倍型类型差异较大,于都群体分布的单倍型最多(13种)、金溪群体分布的单倍型最少(9种)。分布最广泛的Hap_5、Hap_6以及Hap_9单倍型分别出现为15次、29次和13次,其中Hap_6为共有单倍型。
2.3 遗传多样性分析
群体内的遗传多样性可以通过单倍型多样性、核苷酸多样性和平均核苷酸差异数等数据来体现。通过DnaSP5软件计算各群体的遗传多样性参数见表2。144个样本合计,单倍型多态性(Hd)为0.927,核苷酸多样性(Pi)为0.04050,5个养殖群体的Pi值均超过0.01,说明这五个养殖群体的变异程度均较大;平均核苷酸差异数(K)为40.018,各群体平均核苷酸差异在21.060~39.391。
2.4 遗传结构
通过利用MEGA 4.0软件中的Kimura 2-parameter模型,计算出5个棘胸蛙群体之间以及群体内部的遗传距离(D),结果如表3所示。5个棘胸蛙养殖群体内的遗传距离在0.025~0.049之间分布,内部平均遗传距离为0.045。在5个养殖群体中,遗传距离最近的为于都群体和金溪群体,遗传距离最远的为明月山群体和靖安群体。
2.5 遺传差异及遗传分化
通过运用Arequin 3.5软件计算5个棘胸蛙养殖群体的遗传分化,结果见表4。结果表明棘胸蛙两两群体间遗传分化系数(FST)值在0.00382~0.40090之间,其中宜春地区的明月山群体(MY)与宜春地区靖安群体(JA)发生的遗传分化最大,而与抚州地区金溪群体(JX)发生的遗传分化最小,YD、MY、JA群体间,JA与JX群体间,XJ与JA群体间均发生极大的分化(FST>0.25,P<0.001),但XJ与YD及JX群体间遗传分化处于中等水平(0.1 利用AMOVA软件进行棘胸蛙群体间和群体内的遗传变异分析,分析结果如表5所示。分析结果显示,5个棘胸蛙养殖群体中,群体间的变异为27.92%,群体内的变异为72.08%,群体内的变异明显大于群体间的变异,表明变异主要来自棘胸蛙群体内部。 2.6 单倍型系统进化树 基于COI基因序列的棘胸蛙群体的单倍型数据,构建NJ系统发育树,见图3。35个单倍型分成4个单系,独有单倍型hap14、hap15、hap35形成一小单系,属于明月山和金溪群体;独有单倍型hap1、hap2、hap7、hap18、hap19、hap20、hap21、hap23、hap24、hap26、hap29和共享单倍型hap16、hap22形成一大单系,属于于都、峡江、靖安群体。共享单倍型hap9、hap13、hap5、hap25、hap11和独有单倍型hap34形成一单系,属于峡江、明月山、金溪、靖安群体。其他单倍型聚为一大单系,来自所有群体。 3 讨论 3.1 棘胸蛙的序列特征 本文中5个144尾棘胸蛙群体mtDNACOI基因序列中,碱基A、T、G、C的平均含量分别为27.1%、31.2%、16.8%、24.8%,5个群体中A+T碱基的平均含量(58.4%)大于G+C碱基平均含量(41.68%),在水产动物中此类现象比较常见,符合脊椎动物mtDNA碱基组成分布不均一的特点[8]。 3.2 棘胸蛙的遗传多样性 本试验中测定的棘胸蛙5个群体的COI序列中共检测270个变异位点,占全部序列的27.27%,共定义了35个单倍型。本研究中单倍型多样性指数和核苷酸多样性指数分别为0.927、0.0405,与路庆芳[9](2008)研究的野生群体(Hd:0.968,Pi:0.1168)相比,Hd和Pi均有下降,这一点与魏朝宇[10]研究的养殖群体(Hd:0.94568,Pi:0.0341)比野生群体低的结果一致。养殖群体遗传多样性下降,可能是因为人工养殖过程中,存在近亲繁殖的现象,导致养殖群体遗传多样性下降。 3.3 棘胸蛙群体的遗传变异 根据COI序列变异得到的NJ系统进化树结果显示(图3),共享单倍型hap6分布于5个群体中,所含样本数最多29个,推测为古老单倍型。总体来说,江西省各个县区棘胸蛙养殖群体的单倍型呈现出一种混杂的分布格局,错综交织。 群体间的遗传分化指数(Fst)用来表示群体间的遗传分化程度,Fst值越大,表明两个群体间的分化程度越高;反之,分化程度越低[8]。根据各群体的Fst值,判断峡江与于都群体间、明月山与靖安群体间遗传分化很小,可以忽略不计;峡江与明月山、金溪群体间遗传分化处于较高水平;明月山与于都、靖安群体间,于都与靖安、金溪群体间,靖安与金溪群体间遗传分化很大。 群体间的遗传距离能反映群体间的亲缘关系,值越大亲缘关系越疏远。利用MEGA软件计算群体间遗传距离(表3),XJ与YD(0.034)、XJ与JX(0.035)、JX与MY(0.037)之间的遗传距离较小,表明亲缘关系较近,而MY与YD、JA 2群体间的遗传距离较远,说明MY群体与YD群体及JA群体之间的亲缘关系较远。 参考文献 [1]詹忠根,刘悦,黄伟素,秦钢,程宏毅,郑荣泉.棘胸蛙种质资源研究进展[J].中国农学通 报,2016,32(02):77-81. [2]毕相东,杨雷,侯俊利,白东清,董少杰,孙金辉,李永仁,侯林.COIs基因在海洋动物 分子系统学研究中的应用[J].水产科学,2008(02):105-108. [3]梁宏伟,孟彦,罗相忠,李忠,邹桂伟.基于线粒体COI基因的6个黄鳝群体遗传多样性 [J].中国水产科学,2018,25(04):837-846. [4]Thompson J D,Gibson T J,Plewwniak F,et al. The Clustal X windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Research,1997,25(24) :4876-4882. [5]HarrisonRG.AnimalmitochondrialDNAasagenetic markerin populationand evolutionary biology.TrendsEcol Evol,1989,4:6-11. [6] Tamura K,Dudley J,Nei M and Kumar S (2007).MEGA4:Molecular Evolutionary Genetics Analysis (MEGA) software version 4.0.Mol.Biol.Evol.24:1596-1599. [7] ExcoffierL,LavalG,Schneider S.Arlequin (version 3.0):Anintegratedsoftware packagefor populationgeneticsdataanalysis.EvolutionaryBioinformatics Online,2005,l:47-50. [8]杨金权,胡雪莲,唐文乔.长江及其南部邻近水域刀鲚的种群遗传结构及种群历史[J].上海海洋大学学报,2008,17(5):513-519. [9] 路庆芳.利用线粒体DNA分子标记探讨棘胸蛙种群遗传结构[D].浙江师范大学,2008. [10]魏朝宇.棘胸蛙养殖群体遗传多样性评价及其应用分析[D].贵州大学,2022. 基金项目:江西省重点研发计划项目(20192BBF60023) 作者简介:张燕萍(1979-),女,博士,主要从事水产养殖、渔业资源与渔业生态环境研究。E-mail:zhangyanpingxie@163.com.

