基于宏基因组测序的稻蟹共作稻田根际土壤微生物群落功能分析
宋 宇,王 鹏,韦月平
(辽东学院,辽宁丹东 118003)
土壤微生物几乎参与了土壤生态系统全部的养分转化过程[1],是土壤养分循环和植物对养分利用过程中重要参与者,对维持土壤肥力和植物的生长有重要的作用[2]。根际离根轴表面数毫米范围之内,是土壤-根系-微生物相互作用的微区域。有研究发现根系分泌物的改变驱动了有益微生物在根际部位的富集,从而增加宿主吸收养分、增强宿主抵抗胁迫的能力[3]。农作物可通过施用化肥达到增产的目的,但长期大量施用化肥造成土壤板结及破坏土壤微生物区系结构,影响土壤养分的转化[4]。水稻是世界上重要的粮食作物,现代人们通过在稻田中引入鱼、鸭等生物进行复合种养达到了改善土壤环境和提高水稻产量的目的[5-6]。徐敏等[7]研究稻蟹共生系统不同水稻栽培模式土壤理化性状和有效养分的变化规律,结果表明养蟹稻田可显著提高土壤中有机质、碱解氮、速效磷和有效钾含量并能提高水稻产量。土壤微生物的早期研究大都基于平板菌落计数技术、PCR-DGGE、T-RFLP和焦磷酸测序等不可培养的分子分析,如今随着基于Illumina Miseq和Illumina Novaseq测序平台的高通量测序技术的发展,土壤微生物群落结构、多样性和功能的研究得到不断深入。宋宇等[8]探讨了不同稻田共作模式对土壤细菌群落结构的影响,初步揭示了河蟹的引入增加了稻田土壤细菌群落多样性,丰富了细菌群落组成;又进一步研究了河蟹对水稻不同生育期根际土壤细菌群落结构的影响,结果表明河蟹引入生物有机肥稻田后土壤细菌的群落结构会随着生育期发生一定变化,而这种变化和土壤理化因子密切相关[9]。目前对水稻根际微生物群落的研究多为基于Illumina Miseq平台上的群落结构和多样性的研究[10-13],而对基于Illumina Novaseq宏基因组测序平台上的的群落功能分析还少见。本文就是以生物有机肥稻田为对照,基于宏基因组测序来分析施入生物有机肥的稻蟹共作稻田根际土壤微生物群落功能差异来揭示河蟹的引入对生物有机肥稻田的水稻根际土壤微生物代谢功能的影响,为共作稻田的栽培模式提供理论依据。
1 材料与方法
1.1 田间试验和土壤样品采集
供试水稻是‘越光’大米,试验地点是丹东东港市北井子镇盖家坝有机种植农场(39°54′N, 123°50′E),东港市属北温带湿润地区大陆性季风气候。受黄海影响,具有海洋性气候特点。正常年全年平均气温8.4 ℃,降雨量888 mm。设置施入生物有机肥的稻蟹共作稻田和空白对照的施入生物有机肥稻田的水稻大田试验,两种样地各占地约1 000 m2。对照组生物有机肥稻田的土壤样品标记为YJTCS,该稻田施用日照益康有机农业科技发展有限公司生产的海藻生物有机肥(有机质含量≥30%),其分别在3月份和7月份按 1.35 kg/hm2施用。稻蟹共作稻田的土壤样品标记为HXTCS。该稻田在和对照组等时等量施入生物有机肥的同时又在4月末放入河蟹苗,按 0.5 kg/hm2投放,10月末收蟹。于2020年10月15日(水稻成熟期)在两种稻田上各设置3个10 m×10 m的采样区域,每个区域按照5点采样法选取5个点采用破坏性取样法将水稻整株挖出,勿伤害根系,抖落大块土壤后用无菌菌刷刷取根际区域约1 mm紧密粘附在根表面的土壤,每5株为一个重复,混匀后装入50 mL离心管中,在 -80 ℃冷冻保存后送至上海美吉生物医药科技有限公司进行DNA提取和宏基因组测序。
1.2 土壤DNA提取和宏基因组测序

1.3 宏基因组测序分析
使用fastp[14](https://github.com/OpenGene/fastp,version 0.20.0)对reads 3′端和5′端的adapter序列进行质量剪切;使用基于succinct de Bruijn graphs原理的拼接软件MEGAHIT[15](https://github.com/voutcn/megahit,version 1.1.2)对优化序列进行拼接组装,在拼接结果中筛选≥300 bp的contigs作为最终的组装结果;使用MetaGene[16](http://metagene.cb.k.u-tokyo.ac.jp/)对拼接结果中的contigs进行ORF预测。选择核酸长度≥100 bp的基因,并将其翻译为氨基酸序列;用CD-HIT[17](http://www.bioinformatics.org/cd-hit/,version 4.6.1)对所有样品预测出来的基因序列进行聚类(参数为:90% identity、90% coverage),每类取最长的基因作为代表序列,构建非冗余基因集;使用SOAPaligner[18]软件(http://soap.genomics.org.cn/,version 2.21),分别将每个样品的高质量reads与非冗余基因集进行比对(95% identity),统计基因在对应样品中的丰度信息;使用Diamond[19](http://www.diamondsearch.org/index.php,version 0.8.35)将非冗余基因集的氨基酸序列与非冗余蛋白库(Non-Redundant Protein Sequence Database) NR数据库进行比对(BLASTP比对参数设置期望值e-value为1e-5),并通过 NR 库对应的分类学信息数据库获得物种注释,然后使用物种对应的基因丰度总和计算该物种的丰度。
1.4 功能注释方法及数据库
使用 DIAMOND(https://github.com/bbuchfink/diamond)将非冗余基因集序列与EggNOG数据库进行比对(参数:blastp;E-value ≤ 1e-5),获得基因对应的 COG(Clusters of orthologous groups of proteins,直系同源蛋白簇),然后使用 COG 对应的基因丰度总和计算该 COG 的丰度。使用 DIAMOND(https://github.com/bbuchfink/diamond)将非冗余基因集序列与 KEGG的基因数据库(GENES)进行比对(参数:blastp;E-value ≤ 1e-5),根据KO、Pathway对应的基因丰度总和计算该功能类别的丰度。使用 CAZy 数据库的对应工具 hmmscan 将非冗余基因集与 CAZy 数据库进行比对,比对参数设置期望值 e-value 为 1e-5,获得基因对应的碳水化合物活性酶注释信息,然后使用碳水化合物活性酶对应的基因丰度总和计算该碳水化合物活性酶的丰度。
2 结果与分析
2.1 微生物门水平群落组成分析
NR注释结果显示样品中微生物以细菌为主,约占98.09%,其次为古菌约占1.76%,真菌及其他约占0.15%。由图1所示,两种土壤样品在微生物门水平上共有10个主要菌群,分别是变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、绿弯菌门(Chloroflexi)、酸杆菌门(Acidobacteria)、Gemmatimonadetes_d_Bacteria、广古菌门(Euryarchaeota)、Unclassified_d_Bacteria、硝化螺旋菌门(Nitrospirae)、拟杆菌门(Bacteroidetes)和疣微菌门(Verrucomicrobia)。其中变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、绿弯菌门(Chloroflexi)、酸杆菌门(Acidobacteria)为优势菌门,共占比89.11%。稻蟹共作稻田(HXTCS)中的变形菌门、放线菌门和广古菌门的相对丰度分别为39.82%、22.79%和 0.97%低于对照组(YJTCS)的42.26%、27.17%和1.98%。而绿弯菌门、酸杆菌门和疣微菌门的相对丰度分别为15.95%、10.15%和1.13%则高于YJTCS的12.93%、7.19%和0.73%。

图1 微生物门水平群落组成
2.2 2种稻田根际土壤微生物群落的LEfSe分析
为了提供更多关于2种种养模式稻田的根际土壤微生物群落的信息,使用LEfSe来识别YJTCS和HXTCS中LDA得分高于4.0的从门到属水平差异丰度分类群。LEfSe采用线性判别分析(LDA)来估算每个物种丰度对差异效果影响的大小。如图2所示,对根际细菌群落的LEfSe分析表明2种稻田中有16个细菌类群存在显著差异,只有4个在对照组(YJTCS)中富集,而稻蟹共作稻田(HXTCS)则主要富集绿弯菌门、酸杆菌门及隶属于二者的相关目、科、属等12个分类群。如图3所示,对根际古菌群落的LEfSe分析表明2种稻田中有13个明显丰富的分类群。对照组(YJTCS)中只富集3个而稻蟹共作稻田(HXTCS)则富集深古菌门、广古菌门及隶属于它们的相关纲、目、科及属等10个分类群。如图4所示,对根际真菌群落的LEfSe分析表明2种稻田中有11个明显丰富的分类群,对照组(YJTCS)中富集6个,而稻蟹共作稻田(HXTCS)则富集5个分类群。通过2种稻田根际土壤微生物群落的LEfSe分析结果表明河蟹引入生物有机肥稻田后细菌和古菌群落会富集更多的差异分类群。
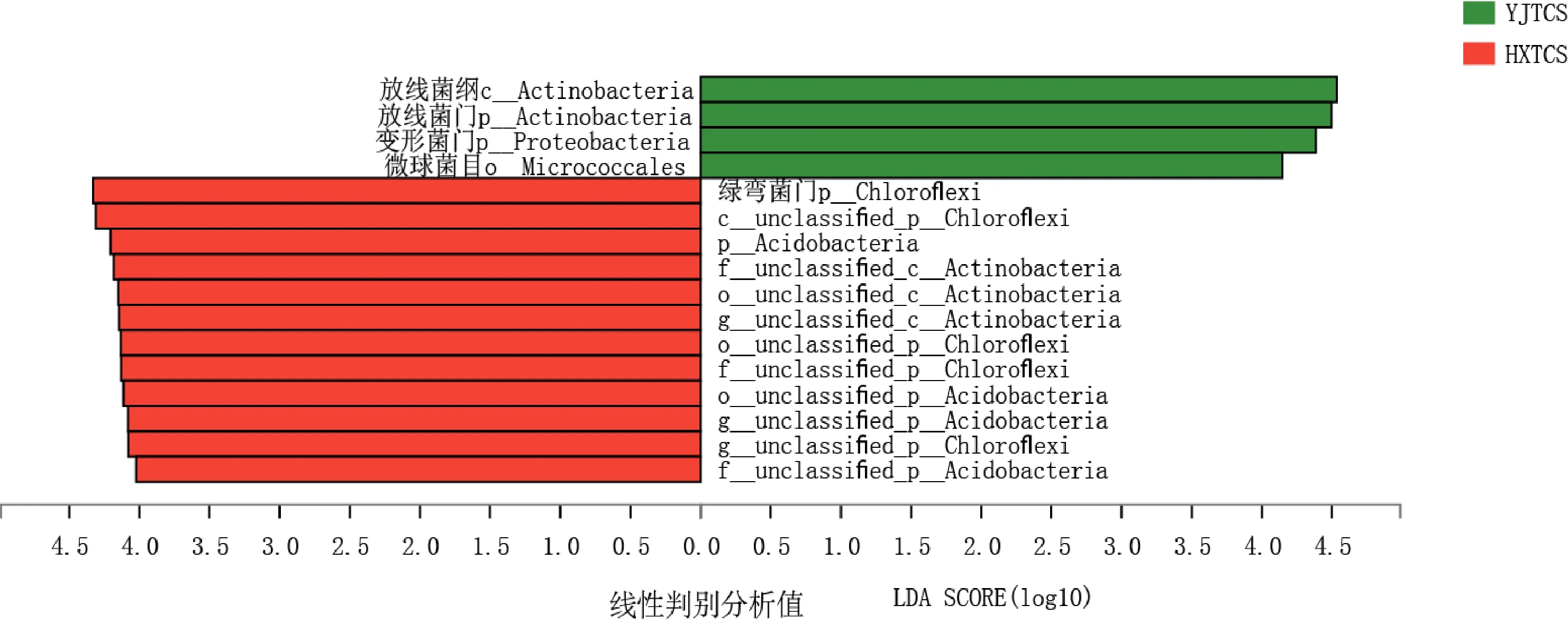
图2 2种稻田根际土壤细菌群落的LEfSe分析结果

图3 2种稻田根际土壤古菌群落的LEfSe分析结果
2.3 基于宏基因组土壤微生物功能注释
2.3.1 CAZy功能基因注释 CAZy(Carbohydrate-Active enzymes,碳水化合物活性酶)可分为6大蛋白功能模块,即辅助氧化还原酶(AAs)、碳水化合物结合模块(CBMs)、碳水化合物酯酶(CEs)、糖苷水解酶(GHs)、糖基转移酶(GTs)和多糖裂解酶(PLs)。如表1所示,HXTCS在6大蛋白功能模块基因的相对丰度均高于对照组YJTCS。其中GHs、GTs和PLs差异显著(P<0.05)。
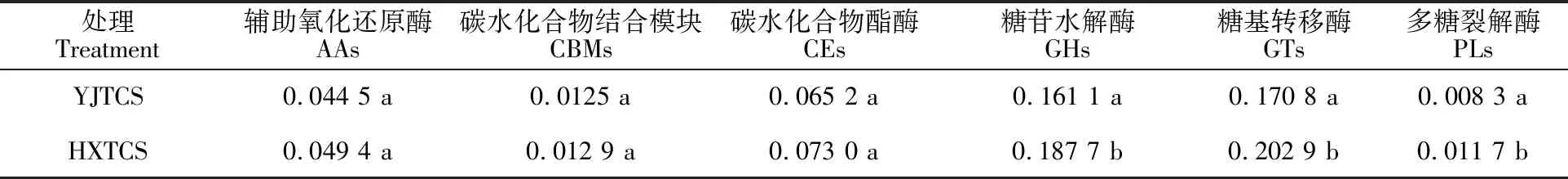
表1 碳水化合物活性酶相对丰度
2.3.2 COG功能基因注释 将测序结果EggNOG数据库进行对比(表2),COG(Clusters of Orthologous Groups,蛋白相邻类的聚簇)将基因功能分为25个大类,包含于四大功能:细胞过程和信号(Cellular processes and signaling)、信息存储和处理(Information storage and processing)、代谢(Metabolism)和未知的功能特征(Poverty characteristics)。功能注释结果表明HXTCS在相对丰度排名前20的基因功能中有19个基因功能的相对丰度均高于对照组YJTCS。其中氨基酸转运与代谢和脂质转运与代谢基因的相对丰度在2种稻田中差异显著 (P<0.05)。

表2 COG功能蛋白注释丰度
2.3.3 碳代谢途径基因注释分析 宏基因组测序结果发现两种土壤样品共有16个碳代谢相关途径,如表3所示,HXTCS在所有碳代谢途径的基因注释的相对丰度均高于对照组YJTCS。其中丙酮酸代谢和原核微生物的碳固定途径的相对丰度在2种稻田中差异显著(P<0.05)。
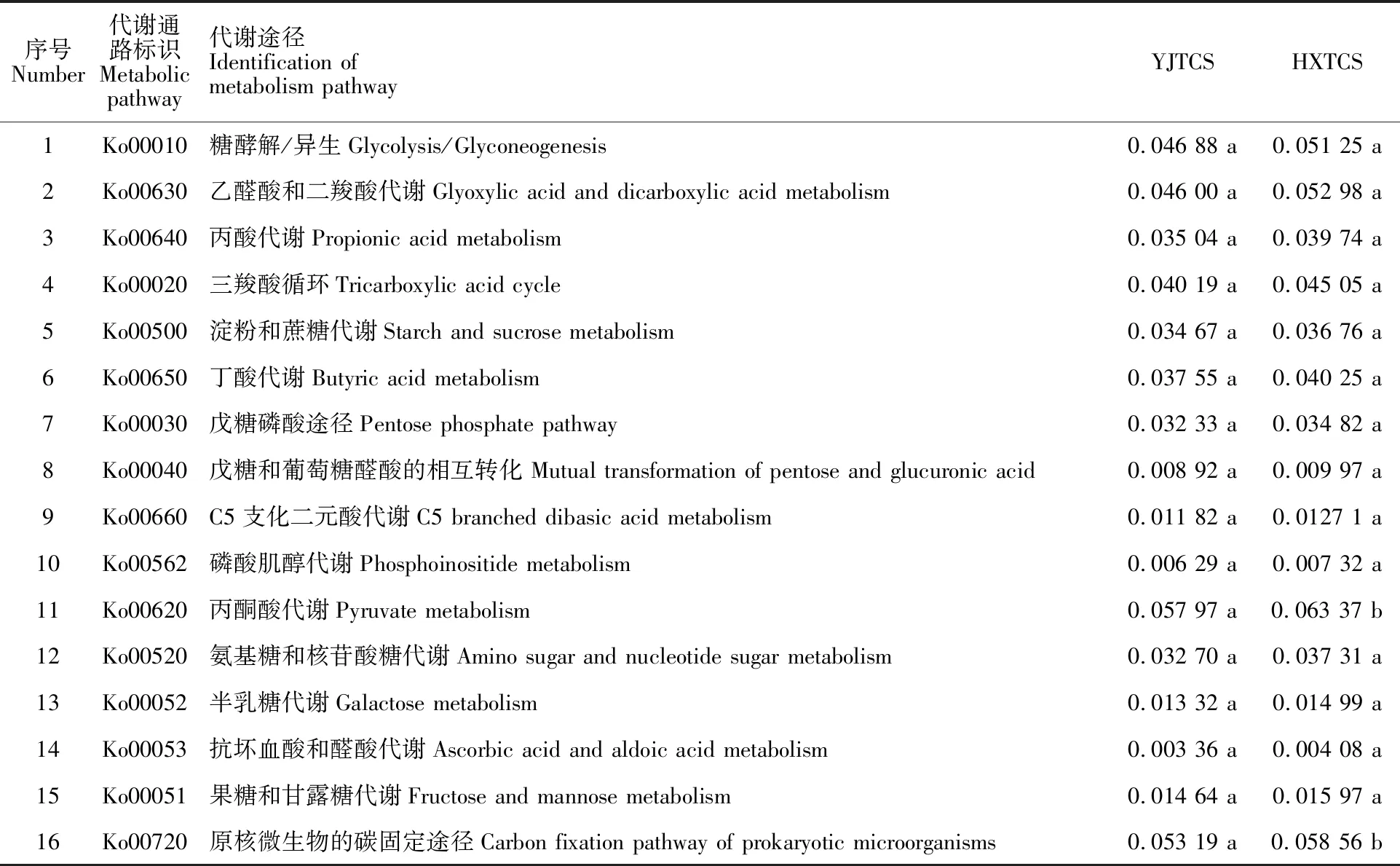
表3 碳代谢途径基因注释丰度
2.3.4 氮代谢功能分析 氮代谢在土壤氮素循环中起到重要作用。本研究主要针对硝化作用、反硝化作用、固氮作用、硝酸盐还原作用和谷氨酰胺合成等5个氮转化过程(图5)。宏基因组测序分析结果显示硝化作用因相关酶基因缺失表现不完整。铁氧还蛋白-硝酸还原酶(EC:1.7.7.2)、硝酸还原酶(醌)(EC:1.7.5.1)、硝酸还原酶(细胞色素)(EC:1.9.6.1)、亚硝酸还原酶(NO生成)(EC:1.7.2.1)、一氧化氮还原酶(细胞色素C)(EC:1.7.2.5)、氧化亚氮还原酶(EC:1.7.2.4)是反硝化过程的关键酶。固氮酶(EC:1.18.6.1)是固氮作用的关键酶。亚硝酸还原酶(EC:1.7.7.1)、亚硝酸还原酶(NADH)(EC:1.7.1.15)、亚硝酸还原酶(细胞色素,氨形成)(EC:1.7.2.2)是硝酸盐还原作用的关键酶。谷氨酰胺合成酶(EC: 6.3.1.2)、谷氨酸合酶(NADPH)(EC:1.4.1.13)、谷氨酸合酶(NADH)(EC:1.4.1.14)、谷氨酸合酶(铁氧还蛋白)(EC:1.4.7.1)、谷氨酸脱氢酶(EC:1.4.1.2)、谷氨酸脱氢酶[NAD(P)+] (EC:1.4.1.3)、谷氨酸脱氢酶[NADP+](EC:1.4.1.4)是谷氨酰胺合成作用的关键酶。

图5 土壤氮素循环过程
如图6所示,在反硝化作用中,硝酸还原酶(醌)基因narH和narY、铁氧还蛋白-硝酸还原酶基因narB、硝酸还原酶(细胞色素)基因napY、亚硝酸还原酶(NO生成)基因nirS、一氧化氮还原酶(细胞色素C)基因norB和氧化亚氮还原酶基因nosZ在HXTCS的相对丰度均高于YJTCS。其中铁氧还蛋白-硝酸还原酶基因narB和硝酸还原酶(细胞色素)基因napY在HXTCS中的相对丰度都显著高于YJTCS(P<0.05)。在固氮作用中,固氮酶基nifD和nifK在HXTCS中的相对丰度略低于YJTCS。在硝酸盐还原作用中,亚硝酸还原酶(NAD基因nirB和nirD在HXTCS中的相对丰度和YJTCS相当,而亚硝酸还原酶nirA基因和亚硝酸还原酶(细胞色素,氨形成)基因nrfA在HXTCS中的相对丰度要高于YJTCS。在谷氨酸合成作用中,谷氨酰胺合成酶基因glnA和GLUL、谷氨酸合酶(NADPH)基因gltB和gltD、谷氨酸合酶(NADH)基因GLT1、谷氨酸合酶(铁氧还蛋白)基因GLU和gltS、谷氨酸脱氢酶基因gudB和rocG、谷氨酸脱氢酶 [NAD(P)+] 基因GLUD1-2、谷氨酸脱氢酶[NADP+]基因gdhA在HXTCS的相对丰度均高于YJTCS。因此,河蟹引入生物有机肥稻田后促进了根际土壤氮循环中的反硝化作用、硝酸盐还原作用及谷氨酸的合成作用。

图6 氮代谢途径中主要酶基因的相对丰度
3 讨论与结论
为进一步探讨河蟹对生物有机肥稻田的水稻根际土壤微生物群落的影响,利用宏基因组测序技术对土壤微生物群落和功能进行研究。通过NR物种注释发现,微生物均以细菌为主,其次为古菌,真菌和病毒等占比很小。微生物群落门水平的组成分析表明河蟹有机肥稻田的绿弯菌门和酸杆菌门的相对丰度均高于对照组。研究证实,大多数绿弯菌门具有反硝化能力[20]。酸杆菌门具有降解植物残体多聚物、参与铁循环、具有光合能力和参与单碳水化合物代谢的功能[21]。2种稻田根际土壤微生物群落的LEfSe分析结果表明稻蟹共作稻田中富集更加丰富的深古菌门(Bathyarchaeota)和奇古菌门(Thaumarchaeota)及隶属于它们的相关目、属。深古菌门是地球上数量最多的古菌群之一,通过宏基因组学研究证实,它们的生理活动与蛋白质和纤维素的降解及以二氧化碳的固定有关[22]。有研究报道奇古菌门菌门是一类可同时参与自然界C、N元素循环的古菌[23]。
河蟹引入生物有机肥稻田后,增加了碳水化合物活性酶基因的6大蛋白功能模块即辅助氧化还原酶(AA)、碳水化合物结合模块(CBM)、碳水化合物酯酶(CE)、糖苷水解酶(GH)、糖基转移酶(GT)和多糖裂解酶(PL)的相对丰度。其中GH、GT和PL与对照组YJTCS的相对丰度差异显著。糖苷水解酶(GH)包括大量参与淀粉、纤维素、木聚糖和几丁质等多糖代谢的酶。碳水化合物结合模块(CBM)可与多糖结合,提高糖苷水解酶(GH)降解复杂碳水化合物的催化效率。碳水化合物酯酶(CE)可降解植物纤维。糖基转移酶(GT)可裂解酸性多糖中的糖苷键。多糖裂解酶(PL)主要作用于阴离子多糖,通过β消除机制降解糖苷键。因此,河蟹的引入有助于增强水稻根际土壤微生物对碳水化合物和多糖类物质的降解活性。COG功能注释结果表明,河蟹的引入也增强了HXTCS在相对丰度排名前20的基因功能中的19个基因功能的相对丰度,尤其是增强了土壤氨基酸和脂质的转运功能。
碳代谢途径基因注释分析结果显示,HXTCS在宏基因组测序发现的16个碳代谢相关途径的基因注释的相对丰度均高于对照组。其中HXTCS在丙酮酸代谢和原核微生物的碳固定途径中的相对丰度均显著高于对照组。糖酵解是微生物对碳水化合物进行代谢的一种主要方式,是厌氧微生物获取能量的主要方式[24]。三羧酸(TCA)循环是糖、脂肪酸和蛋白质三大分解代谢的终极途径,可产生大量的ATP供微生物生长。丙酮酸是糖代谢中具有关键作用的中间产物,可通过三羧酸循环实现体内糖、脂肪和氨基酸间的互相转化,因此,丙酮酸在三大营养物质的代谢过程中起重要的枢纽作用。磷酸戊糖途径是除了糖酵解之外的另一葡萄糖代谢途径,不仅能产生大量能量,还可以产生核糖和赤藓糖等中间产物用于脂肪酸等物质的合成。由于CO2浓度升高导致的全球变暖及由此引发的一些列严重问题已引起各国政府的高度关注,各国都在探索有效的固碳减排途径,现在已越来越重视农业生态系统在碳固定中的作用[25]。农田土壤是一个具有碳固定潜能的潜在碳库,通过农田土壤进行碳固定来缓解碳排放压力是未来国民经济发展的一个重要的战略选择[26]。碳代谢途径基因注释分析结果表明,河蟹的引入能提升原核微生物的碳固定的能力。
氮代谢在土壤氮素循环中起到重要作用。氮代谢功能分析结果表明,河蟹引入生物有机肥稻田后促进了根际土壤氮循环中的反硝化作用、硝酸盐还原作用及谷氨酸的合成作用。反硝化作用是将硝酸盐还原为分子态氮的过程,是氮循环的关键一环,在自然界有重要意义,可消除因硝酸积累对生物的毒害作用。硝酸盐还原是一个耗能的过程,硝酸盐可被还原为铵并能同化为氨基酸[27]。谷氨酸的合成是无机氮向有机氮转化的重要环节。谷氨酰胺合成酶和谷氨酸合酶是谷氨酰胺合成中重要的酶,其酶基因相对丰度高则有助于谷氨酸的合成。土壤中N2固定主要是通过固氮酶来实现,HXTCS中固氮酶基因的相对丰度低于对照组,结果表明,河蟹的引入促进了土壤中反硝化和硝酸盐还原作用及谷氨酸的合成但并未改善土壤微生物对N2的固定。
利用宏基因组测序技术分析稻蟹共作稻田根际土壤微生物群落的功能,结果说明河蟹引入生物有机肥稻田后,增强了根际土壤碳水化合物活性酶的活性和主要代谢通路的代谢活性,有助于碳代谢及促进根际土壤氮循环中的反硝化作用、硝酸盐还原作用及谷氨酸的合成作用。但该技术只能证明微生物群落基因组中存在实现某项功能所需的基因,但基因的存在并不能证明基因一定会表达,因此还需通过宏转录组和宏蛋白组进一步深入研究探讨。

