小麦SPL家族基因在非生物胁迫下的表达分析
贺金秋,苗敬南,马 超,樊紫薇,王超丽,李欢欢,赵 月,刘文轩
(河南农业大学生命科学学院,河南郑州 450002)
SPL(SQUAMOSA promoter binding protein)是植物特有的转录因子家族之一,具有高度保守的SBP结构域,该结构域由70~80个氨基酸构成,具有2个锌指位点(Cys-Cys-His和Cys-Cys-Cys-His)[1-2]。研究表明,SPL家族基因不仅参与植物生长发育[3-6],还参与植物激素和光信号转导、生物和非生物胁迫响应等过程[7]。
前人已在拟南芥、水稻和玉米中分别鉴定到17、19和31个SPL基因家族成员[8]。在小麦中,多位学者也对SPL家族基因进行了全基因组鉴定,除Li等[9]鉴定到48个SPL基因外,其他学者均鉴定到56个SPL基因[10-13]。部分小麦SPL基因的功能也得到深入研究。Liu等[14]利用基因编辑技术将TaSPL8基因敲除后,发现小麦植株叶片直立,叶枕结构缺失;Li等[11]研究发现,过表达TaSPL13基因会影响小麦的花序结构,导致小花和籽粒数量增加;Cao等[15]研究发现,小麦TaSPL16基因可促进拟南芥早开花,并影响种子大小。SPL基因家族部分成员含有miR156结合位点,在转录水平上受miR156负调控[16]。在模式植物拟南芥和水稻中,miR156-SPL模块参与调控植物开花、株型结构、果实发育以及生物和非生物胁迫响应等过程[17-18];在小麦中,miR156-SPL模块通过与DWARF53基因相互作用调节小麦植株结构[19]。小麦SPL基因参与小麦不同组织(器官)的生长发育。然而,关于小麦SPL基因家族在缺氮、缺磷、高盐、低温、干旱、高温等逆境条件下的研究却鲜有报道。
本研究利用最新的中国春小麦参考基因组信息,基于全基因组搜索的方法鉴定小麦SPL基因家族成员,并根据转录组数据对其在逆境胁迫条件下的表达模式进行系统分析,以期为研究小麦SPL基因家族在响应非生物胁迫过程中的功能提供参考。
1 材料与方法
1.1 小麦SPL基因家族成员的鉴定
从Pfam数据库(http: //pfam.xfam.org/)下载SBP结构域的隐马尔可夫模型(HMM)文件(Pfam ID: PF03110),用HMMER 3.1软件从小麦族多组学数据库WheatOmics 1.0(http://202.194.139.32/)下载最新的小麦基因组文件(IWGSC RefSeq v2.1)[20],从中筛选获得所有包含小麦SBP结构域的蛋白序列,利用DNAMAN软件和SMART在线软件(http://smart.embl-heidelberg.de/)对所有候选序列进行分析,去除不完整读码框序列和冗余序列。从TAIR数据库(https://www.arabidopsis.org)下载拟南芥SPL蛋白序列,从水稻基因组注释工程(http://rice.plantbiology.msu.edu/)下载水稻SPL蛋白序列。
1.2 系统发育分析
用ClustalX软件对小麦、拟南芥和水稻SPL蛋白的氨基酸序列进行多序列比对,参数为默认值。用MEGA 8.0软件采用邻接法构建系统发育树,bootstrap值设置为1 000。
1.3 miR156靶位点预测
从miRBase数据库(https://www.mirbase.org/)检索小麦所有miR156序列,将miR156和SPL基因家族所有成员的转录本序列上传到psRNATarget在线软件(https://www.zhaolab.org/psRNATarget/analysis),对miR156靶位点进行预测。
1.4 小麦SPL家族基因的表达模式分析
从小麦族多组学数据库WheatOmics 1.0下载小麦SPL基因在缺氮、缺磷、高盐、低温、干旱、高温胁迫以及热旱共胁迫下的表达谱数据。每个基因的相对表达量用TPM(transcript per million)值来表示,用Excel对数据进行log2转换,用基迪奥生物云工具(https://www.omicshare.com/tools/)中的热图工具绘制热图。与对照相比,表达量变化倍数大于2,被认为是胁迫响应基因。随机挑选4个SPL基因,进一步对缺磷、高盐(200 mmol·L-1NaCl)、低温(4 ℃)、干旱和高温(42 ℃)胁迫条件下这些基因的表达模式进行qRT-PCR验证,具体处理方法:挑选大小均匀的中国春小麦种子,用75%的酒精消毒3 min,无菌水冲洗4~5次后将种子摆放于含有霍格兰营养液的水培盒中,置于光照培养箱(24 ℃光照16 h,18 ℃黑暗8 h)中进行培养,每隔两天换一次营养液。正常生长一周后,将其分别进行胁迫处理,以未处理的样品作为对照(CK)。缺磷和高盐胁迫处理时,先倒掉营养液,然后分别加入缺磷霍格兰营养液及高盐霍格兰营养液继续培养;干旱胁迫是将正常生长一周的幼苗从营养液中移出,置于滤纸上,在室温条件下脱水干旱。将未处理和处理不同时间的叶片取样后用液氮冷冻保存。每个处理进行3次重复。
用Trizol试剂(天根,北京)提取RNA,用HiScript II 1st Strand cDNA Synthesis Kit (+gDNA wiper)试剂盒(诺唯赞,南京)将RNA反转录为cDNA第一条链,以小麦Actin基因为内参,以cDNA为模板,用SYBR Green Realtime PCR Master Mix(Takara,大连)在CFX96定量PCR仪(Bio-Rad,美国)上进行qRT-PCR。PCR扩增体系参照试剂盒说明书进行,PCR扩增程序:95 ℃ 3 min;95 ℃ 5 s,60 ℃ 20 s,72 ℃ 20 s,40个循环。用2-ΔΔCT法计算基因的相对表达量,每个样品3个技术重复。qRT-PCR所用引物序列如表1所示。

表1 qRT-PCR所用的引物Table 1 Primers used in this study for qRT-PCR
2 结果与分析
2.1 小麦SPL基因家族成员的鉴定
从最新小麦基因组(IWGSC RefSeq v2.1)对用SBP结构域(Pfam ID:PF03110)进行检索,共发现73个蛋白含有SBP结构域。用DNAMAN和SMART软件进行结构域分析,同时去除重复蛋白序列,最终获得56个含有SBP结构域的SPL基因家族成员。
2.2 系统进化分析
利用MEGA 8.0软件对小麦(56个)、水稻(19个)和拟南芥(17个)SPL蛋白进行系统进化分析。结果表明,SPL蛋白被划分为7个亚家族(图1)。Class III亚家族中含有的SPL蛋白最多,有23个,其中小麦12个;其次为Class VII亚家族,有21个SPL蛋白,其中小麦17个;Class V和Class VI亚家族中的SPL蛋白最少,均不含拟南芥SPL蛋白。
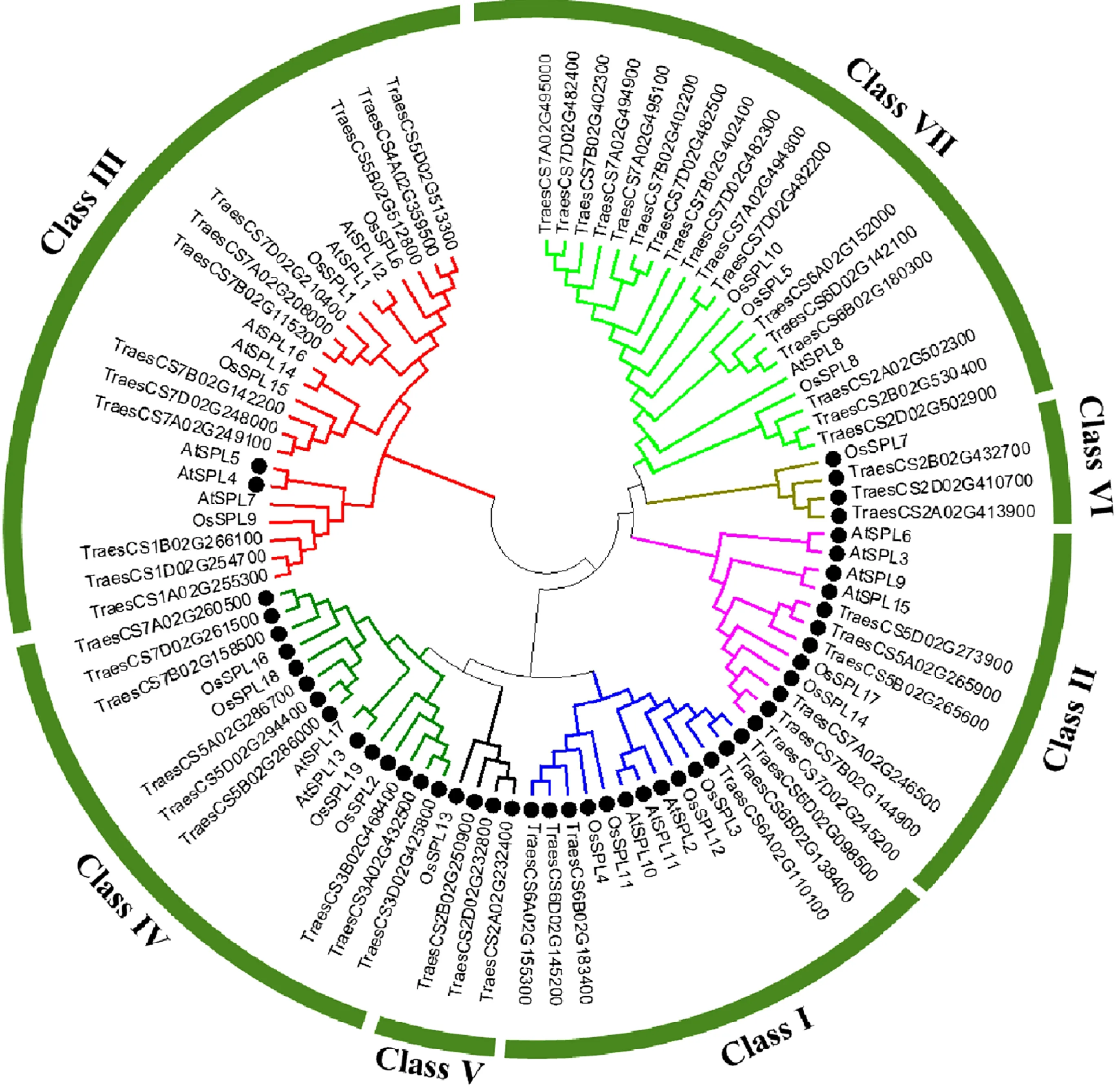
黑圆点表示这些基因含有miR156靶位点。Black dots indicate the genes containing the miR156 target sites.图1 小麦、拟南芥和水稻SPL蛋白的系统进化树Fig.1 Phylogenetic tree of SPL proteins in wheat, Arabidopsis and rice
2.3 miR156靶位点的序列比对分析
从miRBase数据库中检索小麦miR156序列(UGACAGAAGAGAGUGAGCACA),并将该序列和56个小麦SPL基因的转录本序列上传到psRNATarget在线软件,对包含miR156靶位点的小麦、拟南芥和水稻SPL基因进行预测,发现这些基因主要分布在Class I、Class II、Class IV、Class V和Class VI亚家族中(图1),说明miR156调控SPL基因表达的分子调控机制在不同物种之间具有较高的保守性。小麦有27个SPL基因含有miR156靶位点,除了Class V亚家族中3个基因的miR156靶位点位于3’UTR区外,其余24个基因均位于CDS区(图2)。序列比对发现,Class II和Class IV亚家族中15个小麦SPL基因的miR156靶位点与tae-miR156序列之间只有第8个碱基不互补,其他碱基均互补;Class V亚家族3个小麦SPL基因不互补碱基对最多,在第1、2、8位均不互补。

图2 27个小麦SPL基因miR156靶位点的序列比对分析Fig.2 Multiple alignment of tae-miR156 target sites of the 27 wheat SPL genes
2.4 小麦SPL基因在非生物胁迫中的表达模式
2.4.1 缺氮、缺磷和高盐胁迫下的表达模式
从小麦族多组学数据库中获得56个小麦SPL基因在缺氮(材料为生长15 d的小麦幼苗,处理后取叶片)、缺磷(材料为生长14 d的小麦幼苗,处理后分别取叶片和根部)、高盐胁迫(材料为生长9 d的小麦幼苗,处理后取根部)的RNA-seq数据,并分析这些基因的表达模式。结果(图3)发现,叶片中有12个小麦SPL基因响应缺氮胁迫,其中在缺氮胁迫处理1 h后,有2个基因下调表达,分别是TraesCS5B02G286000和TraesCS7A02G208000,后者的下调幅度最大;复氮培养24 h后,仅TraesCS2A02G232400上调表达,有9个基因下调表达。
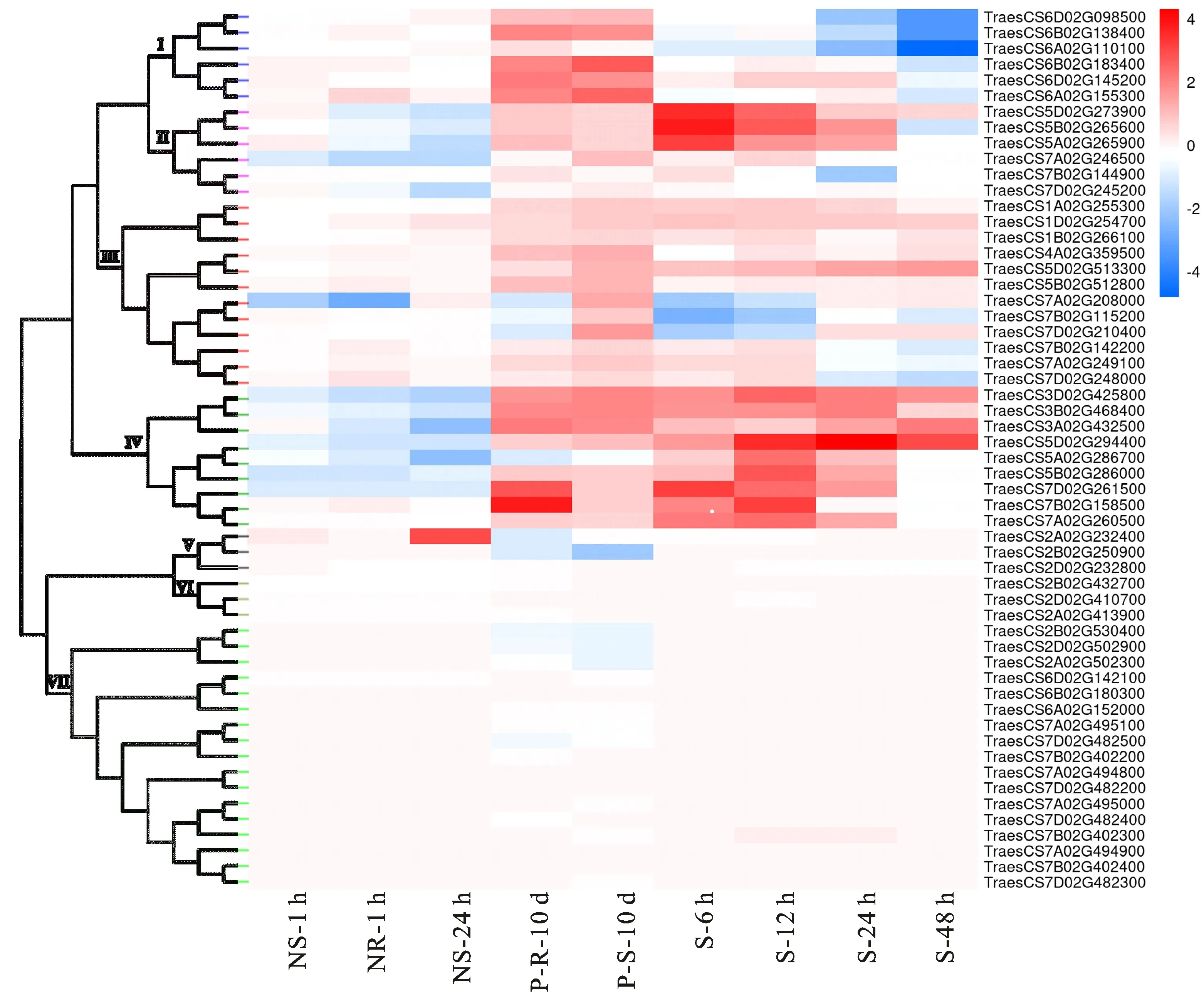
NS-1 h:缺氮处理1 h的叶片;NR-1 h:复氮处理1 h的叶片;NR-24 h:复氮处理24 h的叶片;P-R-10 d:缺磷处理10 d的根;P-S-10 d:缺磷处理10 d的叶片;S-6 h:高盐处理6h的根部;S-12 h:高盐处理12 h的根部;S-24 h:高盐处理24 h的根部;S-48 h:高盐处理48h的根部。图中每个单元格对应的数据为胁迫处理样品与对照样品中基因表达水平差异倍数的对数值。NS-1 h: Leaves treated with nitrogen deficiency for 1 h; NR-1 h: Leaves treated with nitrogen recovery for 1 h; NR-24 h: Leaves treated with nitrogen recovery for 24 h; P-R-10 d: Roots treated with phosphorus deficiency for 10 d; P-S-10 d: Leaves treated with phosphorus deficiency for 10 d; S-6 h: Roots treated with high salt for 6 h; S-12 h: Roots treated with high salt for 12 h; S-24 h: Roots treated with high salt for 24 h; S-48 h: Roots treated with high salt for 48 h. The data corresponding to each cell in the figure is logarithmic value of fold change in gene expression level between stress treatment and CK.图3 小麦SPL基因在缺氮、缺磷和高盐胁迫下的表达模式Fig.3 Expression profiles of wheat SPL genes under nitrogen deficiency, phosphorus deficiency and high salt stresses
缺磷胁迫处理10 d后,共有16个小麦SPL基因表达量变化显著。根中有1个SPL基因(TraesCS7A02G208000)下调表达,10个SPL基因上调表达,其中TraesCS7B02G158500上调幅度最大;叶片中有13个基因上调表达,其中TraesCS6B02G183400上调幅度最大,仅TraesCS2B02G250900下调表达。
在高盐胁迫处理后6、12、24和48 h四个不同时间点,根部共有22个SPL基因表达量发生变化,且多数为上调表达,其中TraesCS5D02G294400上调表达幅度最高,在处理24 h时表达量最高。进一步分析发现,在缺氮、缺磷、高盐胁迫下表达量发生显著变化的SPL基因主要分布在Class I~Class Ⅳ亚家族。
2.4.2 低温、高温、干旱胁迫以及热旱共胁迫下的表达模式
从小麦族多组学数据库获得56个小麦SPL基因在低温(材料为生长14 d的小麦幼苗,处理后取叶片)、高温、干旱胁迫以及热旱共胁迫下的RNA-seq数据,并分析这些基因的表达模式。结果(图4)表明,低温胁迫处理后,小麦幼苗叶片中有6个SPL基因显著上调表达,其中TraesCS3A02G432500和TraesCS3D02G425800上调幅度最大;高温胁迫处理下,有14个小麦SPL基因的表达量变化显著,其中根中有3个,叶片中有1个,籽粒中有12个,除TraesCS6B02G183400在根中上调表达外,其余基因均下调表达,其中TraesCS6A02G110100下调幅度最大;干旱胁迫处理下,有13个小麦SPL基因的表达量变化显著,相较于叶片和籽粒,小麦根中响应干旱胁迫的SPL基因较多,其中9个上调表达,1个下调表达;热旱共胁迫处理下,小麦的根、叶片及籽粒中共有21个SPL基因响应热旱共胁迫,其中TraesCS3B02G468400在叶片中上调幅度最大。响应热旱共胁迫的SPL基因主要分布于Class Ⅰ、Class Ⅲ和Class Ⅳ亚家族。
对响应不同胁迫的小麦SPL基因进行归类,共发现36个基因响应胁迫处理(表达量变化倍数大于2)。除TraesCS1B02G266100、TraesCS1D02G254700、TraesCS2D02G232800、TraesCS4A02G359500、TraesCS5B02G265600、TraesCS7A02G246500、TraesCS7A02G260500、TraesCS7B02G144900、TraesCS7B02G142200、TraesCS7D02G245200基因外,其余26个基因均响应2种及以上胁迫,其中TraesCS3D02G425800响应7种胁迫(表2)。
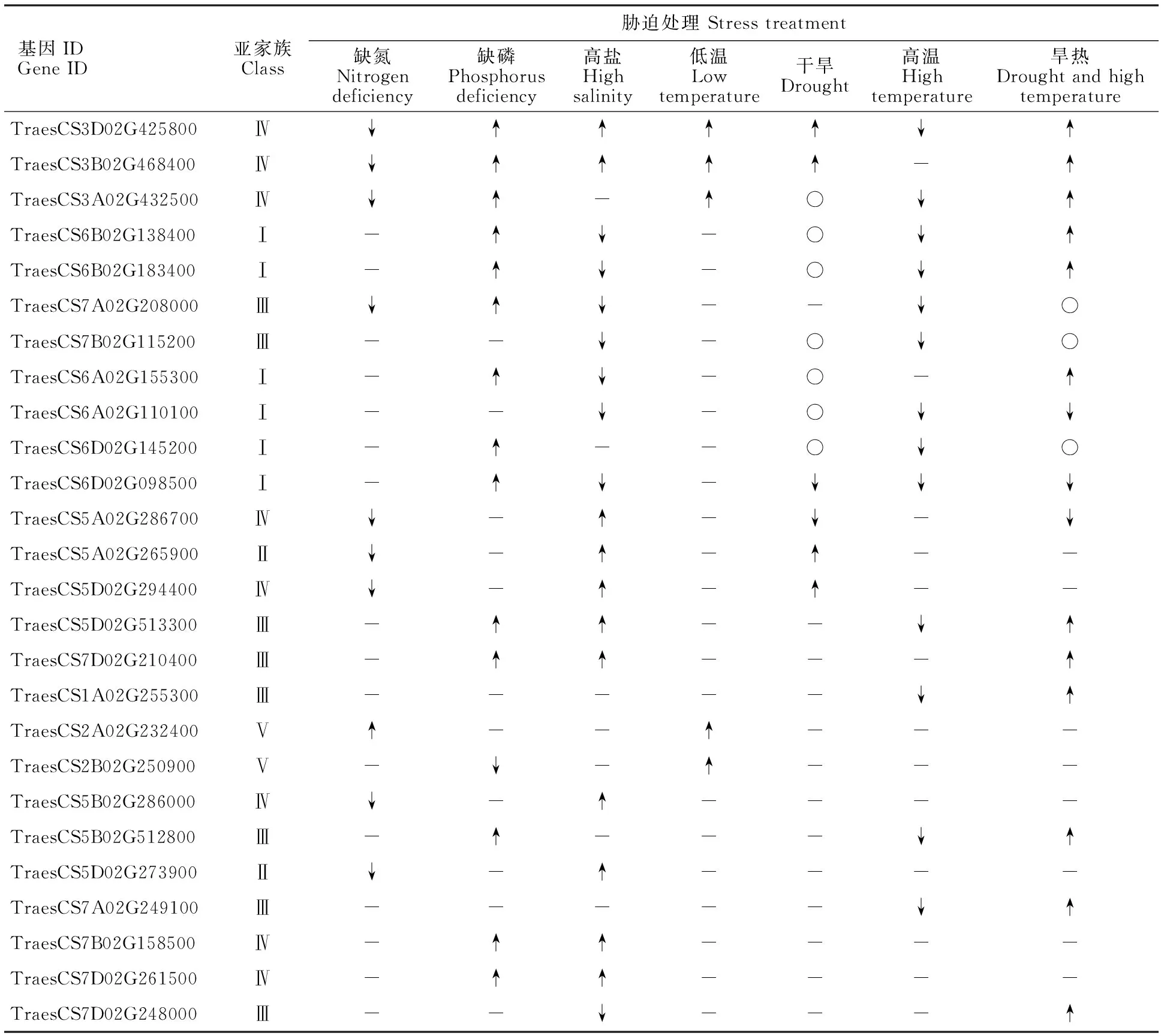
表2 26个响应多个非生物胁迫的小麦SPL基因Table 2 26 wheat SPL genes responding to multiple abiotic stresses
进一步分析发现,大部分位于相同亚家族的小麦SPL基因在同一胁迫下表达模式相似,说明小麦SPL基因可能存在功能冗余。如Class I亚家族中的TraesCS6A02G110100、TraesCS6B02G138400和TraesCS6D02G098500在高盐胁迫条件下均下调表达。但也存在例外,如Class Ⅳ亚家族中的TraesCS5A02G286700、TraesCS5B02G286000和TraesCS5D02G294400属于部分同源基因,但它们在干旱胁迫条件下分别表现出下调、上调和无变化三种表达模式。
随机挑选四个响应2种及以上非生物胁迫的小麦SPL基因(TraesCS7A02G208000、TraesCS6B0G138400、TraesCS6D02G145200和TraesCS7A02G249100),利用qRT-PCR验证它们在不同胁迫下的表达模式。从表3可以看出,缺磷胁迫处理下,TraesCS7A02G208000和TraesCS6B02G138400的表达量均呈先升后降的变化趋势,在处理9 d时表达量达到峰值;而TraesCS7A02G249100和TraesCS6D02G145200的表达量呈“降-升-降”的变化趋势,分别在处理6 d和9 d时表达量达到峰值,均与对照差异显著(图5)。高盐胁迫处理下,TraesCS6B02G138400和TraesCS6D02G145200的表达量均显著低于对照;TraesCS7A02G208000的表达量呈先升后降的变化趋势,在处理6 h时表达量达到峰值,且与对照差异显著;TraesCS7A02G249100的表达量无显著变化。低温胁迫处理下,TraesCS6B02G138400和TraesCS7A02G208000的表达量变化趋势相反,分别呈“升-降-升”和“降-升-降”的变化趋势,在处理1 h和12 h表达量达到峰值,均与对照差异显著;而其他两个基因的表达量在各处理时间点均无显著变化。干旱胁迫和高温胁迫处理下,4个小麦SPL基因的表达量均有所下降(除高温胁迫1 h后TraesCA7A02G208000的表达量显著高于对照外),部分处理时间的表达量与对照差异显著。qRT-PCR结果与转录组数据基本一致。
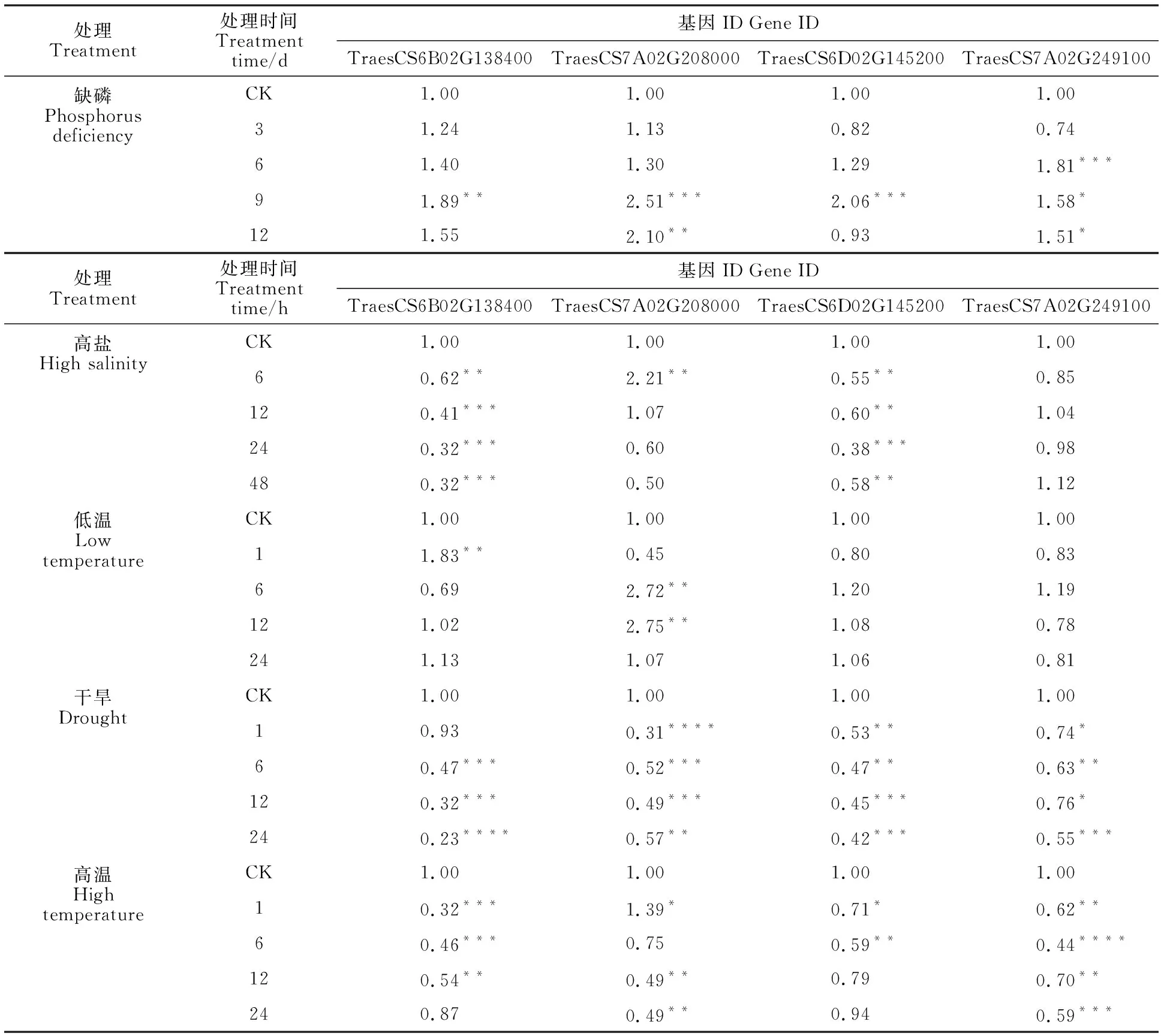
表3 不同胁迫处理下4个小麦SPL基因的相对表达量Table 3 Expression of the four wheat SPL genes under different stresses
3 讨论
本研究从最新的中国春小麦基因组(IWGSC RefSeq v2.1)中鉴定到56个小麦SPL基因,而拟南芥、水稻和玉米中分别鉴定到17、19和31个SPL基因[8]。说明小麦中SPL基因的数目远多于其他物种,原因可能是小麦为异源六倍体植物,在进化过程中经历了两次天然杂交和染色体加倍。本研究通过系统进化分析将56个小麦SPL基因分成7个亚家族,而Song等[10]、Li等[11]和Zhu等[12]分别将其分成5、8和10个亚家族,尽管每个亚家族中SPL基因的数目不一致,但序列相似的基因都聚在同一分支。本研究发现,Class Ⅴ和Class Ⅵ亚家族中没有对应的拟南芥SPL基因家族成员,推测Class Ⅴ和Class Ⅵ亚家族可能是在单子叶植物和双子叶植物分化后在单子叶植物中独立进化出的分支。
Cui等[21]研究表明,拟南芥AtSPL9基因通过miR156-SPL9-DFR途径响应高盐和干旱胁迫。本研究对不同胁迫下小麦SPL基因的表达模式进行分析,发现响应缺氮、缺磷、高盐、低温、高温、干旱胁迫和热旱共胁迫的小麦SPL基因家族成员主要集中在Class I~Class Ⅴ亚家族,而Class Ⅵ和Class Ⅶ亚家族成员基本上不响应上述胁迫。Zhu等[12]研究发现,大部分小麦SPL基因具有组织表达特异性,其中46个小麦SPL基因主要在穗部表达。本研究进行胁迫处理的样品取材部位主要为小麦叶片和根部,Class Ⅵ和Class Ⅶ亚家族成员在穗部是否特异表达需进一步试验验证。miR156是植物小RNA家族中表达丰度最大的家族,通过靶向SPL基因家族成员参与调控植株开花以及株型建成等过程[18]。在拟南芥中,miR156通过调控AtSPL3/4/5和AtSPL9的表达直接激活下游基因MADS-box,从而促进开花[22];miR156-SPL10通过介导细胞分裂反应,控制根的再生[23]。miR156-SPL模块也参与植物非生物胁迫,Ding等[24]发现,拟南芥miR156-SPL模块通过调控下游相关热响应基因的表达来适应环境温度变化;Kouhi等[25]发现,红花miR156-SPL模块可使红花幼苗在长期轻度干旱时继续存活,并提高开花率。本研究发现,27个小麦SPL基因可能受miR156调控,这为进一步探索miR156-SPL模块在小麦中的功能提供了参考。
本研究鉴定到36个与非生物胁迫响应相关的小麦SPL基因,其中TraesCS3D02G425800响应7种非生物胁迫,后续将通过过表达转基因技术或CRISPR-Cas9技术对该基因进行验证,深入研究该基因的功能。

