抗猪繁殖与呼吸障碍综合征基因编辑猪研究进展
李双喜 华进联
(西北农林科技大学动物医学院 陕西省干细胞工程技术研究中心,杨凌 712100)
国家生猪产业体系对我国生猪产业现状的调研显示,非洲猪瘟(African swine fever,ASF)、猪繁殖与呼吸障碍综合征(porcine reproductive and respiratory syndrome,PRRS)和猪流行性腹泻(porcine epidemic diarrhea,PED)是影响我国生猪生产的最重要的三大疫病。其中,PRRS是一种由猪繁殖与呼吸障碍综合征病毒(Porcine reproductive and respiratory syndrome virus,PRRSV)引起猪群发病的传染性病毒性疾病,在临床上主要表现为母猪的繁殖障碍和生长育肥猪的呼吸系统紊乱。虽然近两年在田间流行的类NADC30和类NADC34毒株尚未造成大面积疫情暴发,但对猪群健康的影响持续存在[1]。此外,从猪场经济学的角度来看,PRRS是对猪场经济影响最重要的疾病。目前对于该病的防控主要依赖于养殖场内外部生物安全体系,采取合理措施降低病毒的污染,切断病毒传播途径。
从保护易感动物的角度而言,抗病育种也是疫病防控的重大策略之一。近年来,随着基因编辑技术的发展和成熟,分子育种作为猪抗病育种的核心技术,已取得很多突破性进展。本文以PRRSV受体为切入点,综述猪抗PRRS育种的研究现状,以期为PRRS的防控和猪的抗病育种研究提供线索。
1 PRRS流行和防控现状
猪繁殖与呼吸障碍综合征于1987年首次在美国报道,该病在临床上的症状表现为母猪流产、早产、木乃伊胎等繁殖障碍,断奶猪的呼吸道症状、生长迟缓以及死亡率增加。当时由于病因不明,称其为“猪神秘病”。1991年,在欧洲首次分离到该病病原,称为猪繁殖与呼吸综合征病毒(PRRSV),并命名为Lelystad[2]。北美分离株VR‑2332于1999年分离,并进一步获得该毒株的全基因组序列[3]。根据基因序列的差异,PRRSV可分为欧洲株(PRRSV‑1,代表毒株Lelystad)和美洲株(PRRSV‑2,代表毒株VR‑2332)两个基因型,二者的核苷酸序列相似性为50%-60%,且均能够引起长期感染并且具有相似的临床症状[4]。在2020年国际病毒命名委员会发布的最新病毒分类报告中,这两个基因型作为两个种,分别命名为猪β动脉炎病毒1型和2型[5]。
我国于1995年底在华北地区规模化猪场首次暴发PRRS[6-7]。随后,PRRS在我国猪群中不断传播[8]。2006年,高致病性猪繁殖与呼吸综合征(highly pathogenic PRRS,HP‑PRRS)在我国暴发,在临床上表现为高热、高发病率和高致死率,造成大量猪只死亡,重创当时的养猪产业。HP‑PRRS自2006年后成为我国猪只中流行的优势毒株[9],并传入周边国家[10]。2014年,我国相继出现类NADC30毒株[11-12],成为我国优势流行毒株之一,且该毒株易与其他的毒株发生重组。同年,美国首次报道NADC34毒株[13],而我国于2017年在东北部地区检测到与之相似的毒株——类NADC34毒株[14],之后中部地区多个省份也陆续检测到了该类毒株[15-16]。与类NADC30毒株相似,类NADC34毒株也易与其他毒株发生重组。PRRSV极易变异和重组的特性,致使新毒株层出不穷,给该病的防控带来了严峻挑战。
疫苗是防控传染病最有效的措施之一。然而对于PRRS而言,灭活疫苗基本无效,减毒活疫苗目前在猪场免疫较为普遍,可对同源毒株提供临床保护,但是对异源毒株的交叉保护非常有限,甚至是无效的[17-18]。PRRS治疗方法也大多停留在研究阶段,尚未有可在临床上应用的有效治疗性药物。因此,在生产实践中常采用完善和加强猪场生物安全、合理使用疫苗、剔除阳性猪只、驯化阳性场等多项举措并施来防控PRRS,使猪群处于相对稳态[19]。抗病育种猪是PRRS防控的新策略。随着近年来CRISPR/Cas9基因编辑技术的发展和成熟,使得对动物基因组特定位置的编辑变得更加简便、精准和快速。国内外多个研究团队也利用该项技术对猪进行基因编辑,进行抗PRRS育种研究和探索。
2 抗PRRS基因编辑猪研究进展
基因编辑是指利用基因编辑工具对细胞、植物或动物基因组中进行特定基因序列的插入、删除或突变,从而改变该基因的表达。基因编辑工具以核酸酶为主要成分,最初的基因编辑工具为锌指核酸酶(ZFNs)基因编辑系统,该系统剪切效率低,且易产生非特异性脱靶切割[20]。2009年出现转录激活子样效应子核酸酶编辑系统(TALENs),与锌指核酸酶基因编辑系统相比,该系统识别位点多、剪切效率高,且操作简便,节省时间[21]。近年来CRISPR/Cas9基因编辑系统有了突飞猛进的发展,已成功应用于体内外基因治疗、动植物抗病育种等多个方面[22]。
目前,CRISPR/Cas9基因编辑系统是在基因编辑猪中应用比较广泛的基因编辑技术。国内外针对抗PRRS基因编辑猪的研究主要集中在改造PRRSV受体血红蛋白清道夫受体(haemoglobin scavenger receptor)分子。大量的研究证实,CD163可以介导PRRSV入侵细胞,且被认为是众多受体中最为重要的一个分子。CD169虽然也可介导病毒内化,但是研究表达该蛋白并非PRRSV感染所必需的,且敲除该基因对PRRSV感染无明显影响[23]。因此,近年来国内外多项研究致力于CD163敲除或功能域缺失对PRRSV感染的影响。
2.1 CD163蛋白的结构与功能
PRRSV在猪体内的靶细胞是猪肺泡巨噬细胞(porcine alveolar macrophage,PAM)。研究发现多种细胞受体可介导PRRSV进入PAMs,包括CD163(cluster differentiation 163)、唾液酸黏附素(sialoadhesin,Sn;CD169)、硫酸乙酰肝素(heparin sulphate,HS)、波形蛋白(vimentin)、CD151分子、树突状细胞表面的DC‑SIGN(CD209)以及肌球蛋白重链9(MYH9)。其中,CD163分子研究最为广泛。
CD163分子,又称血红蛋白清道夫受体,属于I型膜蛋白,分子大小为130 kD,包含9个清道夫受体超家族(scavenger receptor cysteine‑rich, SRCR)结构域,富含半胱氨酸[24]。猪源CD163基因含有7个外显子,编码的CD163蛋白包括N端信号肽,9个SRCR结构域、2个脯氨酸-丝氨酸-苏氨酸(PST)域和C端胞内域,其中PST结构域分别位于SRCR6和 SRCR9之后[25]。
CD163蛋白可作为多种分子的受体,参与维持机体的自身稳态。首先,在机体受到病原感染或组织发生损伤后,CD163蛋白发挥清道夫分子作用。CD163蛋白可通过其SRCR2‑SRCR3结构域结合血红蛋白-触珠蛋白复合物,该复合物经内吞进入巨噬细胞内体和溶酶体,最终清除游离血红蛋白,从而维持机体处于健康状态[26-28]。其次,CD163分子可作为肿瘤坏死因子样凋亡微弱诱导剂(TNF‑like weak inducer of apoptosis, TWEAK)受体,与后者结合后参与调控肿瘤细胞的增殖或凋亡[29]。同时,CD163分子属于成红血细胞黏附分子,它与成红细胞相互作用,在红细胞生成过程中发挥调节作用[30]。此外,2005年Fabriek等[31]研究发现表达CD163的巨噬细胞或可溶性CD163在炎性组织中大量存在,提示其通过巨噬细胞调节细胞因子的释放,在机体防御中发挥作用。
除了发挥以上生理功能之外,CD163分子还可作为PRRSV感染的受体。2007年,Calvert等[32]的研究首次发现CD163分子与PRRSV感染高度相关,是基因I型和II型PRRSV的受体。CD163主要介导PRRSV在细胞质中的脱衣壳和病毒基因组的释放,并且已经确定CD163的病毒结合区为SRCR5‑SRCR9,胞外N端的SRCR1‑3不影响CD163与PRRSV结合的受体活性。2009年,Van Gorp等[33]在CD163之前的研究基础上,通过构建缺失突变和嵌合突变体,进一步鉴定受体结合区域。攻毒感染试验揭示了SRCR5结构域在PRRSV感染中是必需的,而N端4个SRCR结构域和C端胞质区不参与病毒感染过程。除此之外,其余的CD163蛋白区域需要保留,但若将其替换为CD163‑L1(CD163b)的SRCR区域,则可引起感染效率降低(SRCR6及域间区)或不变(SRCR7‑SRCR9)。另外,该项研究还证实,识别CD163‑SRCR5的特异性抗体可降低PRRSV的感染。随后的研究解析了SRCR5的晶体结构信息,通过突变实验揭示在长环(long loop)区域的561位的精氨酸残基在PRRSV感染中发挥重要作用,进一步分析得知很可能是在病毒入侵过程中其参与CD163与PRRSV的结合[34]。病毒凋亡模拟(viral apoptotic mimicry)作为一种新型感染方式,在PRRSV感染中发挥着重要作用。研究发现PRRSV囊膜表面暴露有磷脂酰丝氨酸(PtdSer)模拟凋亡,T 淋巴细胞免疫球蛋白黏蛋白(T cell immunoglobulin domain and mucin domain,TIM)等可通过该物质识别PRRSV感染,进而通过CD163分子介导病毒入侵[35]。
2.2 以CD163为靶标的基因编辑猪抗PRRS研究现状
CD163分子在PRRSV复制周期的作用使其成为基因编辑猪抗PRRS研究的重要靶点。近年来国内外很多研究关注在缺失CD163受体的猪只对PRRSV的易感性。利用CRISPR系统结合体细胞移植技术,对猪CD163基因进行有针对性的编辑。待获得基因编辑猪后,利用猪只或者从猪只上分离PAMs,分析其对不同PRRSV毒株的易感性。
2016年,Whitworth等[36]首次使用CRISPR/Cas9编辑技术敲除已经报道的PRRSV受体发现,敲除CD163分子SRCR5结构域的猪能够完全抵抗II型PRRSV感染。随后,许多研究团队开始了针对CD163的抗PRRSV感染猪的基因编辑育种工作。次年,有研究发现完全敲除CD163分子或者缺失SRCR5结构域的猪只及其PAMs细胞对基因I型和II型PRRSV毒株均不易感,而利用人源类CD163分子(hCD 163L1)SRCR8区域替换猪源CD163分子 SRCR5区域,表达嵌合CD163分子的猪只和PAMs,只对基因I型毒株耐受,对基因II型毒株仍然易感[25]。然而,Chen等[37]发现hCD 163L1结构域替换猪源CD163分子SRCR5结构域的基因编辑猪对高致病性猪繁殖与呼吸综合征病毒(HP‑PRRSV)不易感。另一研究团队的结果表明,CD163分子SRCR5结构域缺失的猪只及PAMs和集落刺激因子因子1刺激的外周血单核细胞(PMMs)对基因I型高致病性毒株不易感。值得注意的是,除基因I型外,PMMs对基因II型的毒株也不易感[38-39]。
2018年,Yang等[40]研究也发现用HP‑PRRSV TP毒株感染CD163基因敲除猪后,猪只不表现出病毒血症、抗体反应、高热以及其他PRRS相关临床症状,提示其可完全抵御病毒感染。2019年,Guo等[41]利用双荧光筛选的方法提高了基因编辑猪的阳性率,并发现只缺失CD163分子SRCR5结构域41个氨基酸(缺失区域包含病毒受体结合位点)即可使猪抵抗基因II型毒株JXA1和MY毒株的感染。多基因同时敲除从而获得抵抗几种病毒感染的猪只更能迎合当下疫病防控的需求。2020年,Xu等[42]首次通过CD163和pANP双基因敲除获得了能同时抵抗基因II型PRRSV、TGEV和德尔塔冠状病毒的编辑猪,在肉质品质和繁殖性能上,与野生型无差异,但在双缺失型猪只的肌肉中检测到升高的铁。表1归纳总结了当前以CD163为靶标的基因编辑猪抗PRRS的主要进展。
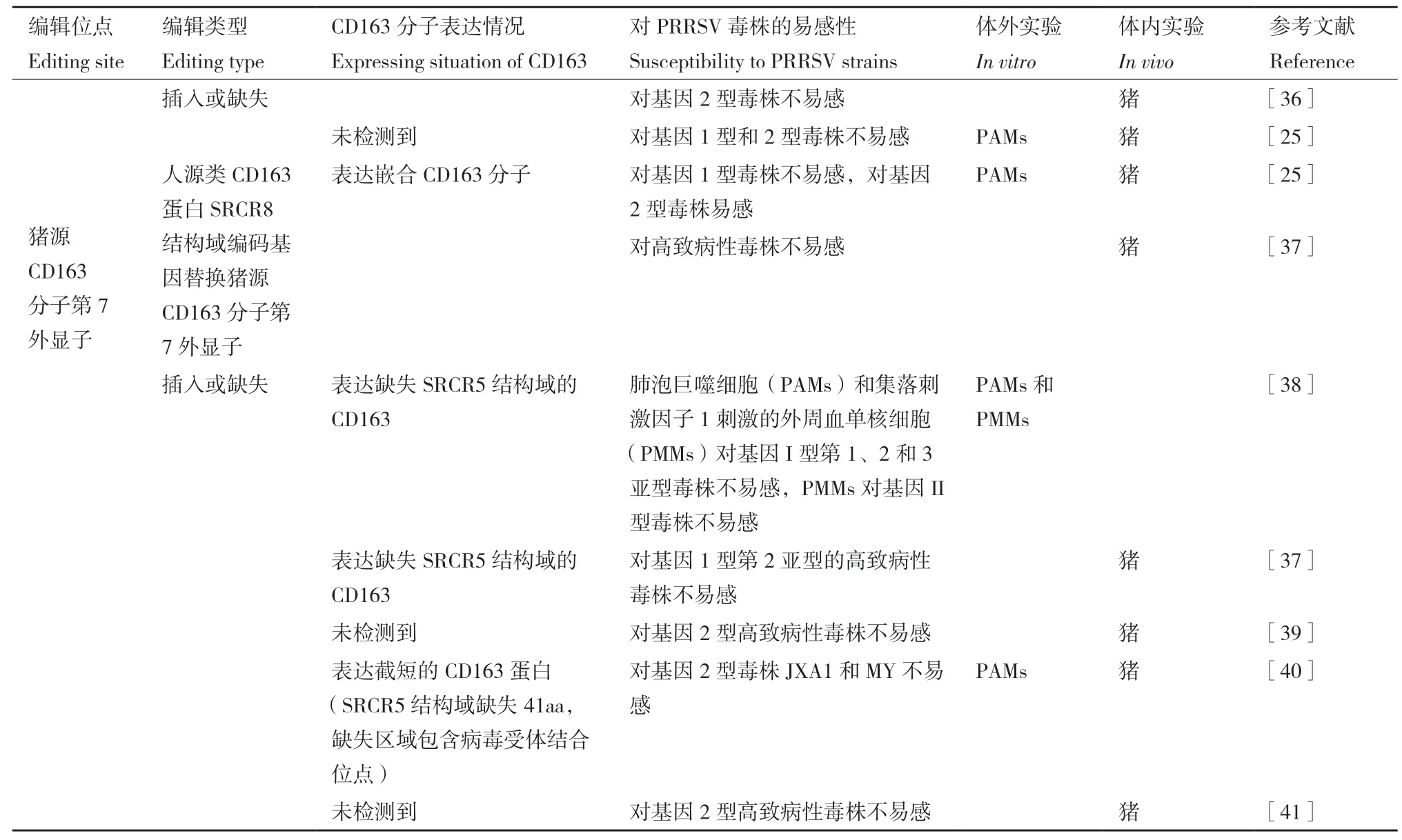
表1 以CD163为靶标的基因编辑猪抗PRRS的主要进展Table 1 Advances in anti-PRRS gene-editing pigs targeting CD163
除了靶向PRRSV受体CD163分子对猪抗PRRS进行探索之外,研究人员也在挖掘病毒感染过程中的关键宿主因子,以期筛选出新的靶标。利用全基因组基因敲除的猪源gRNA文库及高通量基因筛选技术筛选并在细胞水平上鉴定IL20RB、ATP6V0A1和STX10均可显著抑制PRRSV感染,为抗PRRS育种的研究奠定基础[43]。
在基因编辑方法上,近年来也取得一些重要进展。Xu等[44]开发了一种称为报告RNA富集的双引导RNA核蛋白(RE‑DSRNP)编辑技术,可用于快速构建无外源DNA基因编辑的克隆猪。该技术可提高编辑效率,避免质粒DNA随机整合到目标细胞的基因组,同时无单克隆细胞选择过程,缩短体细胞培养时间,降低体细胞异常比例。本实验室通过比较不同sgRNA的表达策略发现,连续的 sgRNAs表达盒策略可以显著提高猪源细胞的多基因编辑效率,并对仔猪全基因组测序,验证了该方法的安全性。近期,有研究团队通过把二元四环素诱导表达元件分别精确插入Rosa26和Hipp11位点,建立多西环素诱导SpCas9表达(DIC)猪模型。在该模型基础上,构建了一种基于Cas9条件敲除猪基因的方法。该方法可通过药物和sgRNA调控基因的沉默和过表达。DIC猪模型也可作为开发单一转基因整合的基本工具,对猪进行时空基因敲除,从而对一些复杂的疫病进行复制,进从而精准剖析蛋白功能。而且,DIC猪SpCas9蛋白表达的可调控性对探究该蛋白的剂量及谱系示踪研究至关重要[45]。
综上所述,关于抗PRRS猪育种已经取得许多重要研究进展,但是仍有一些问题是需要认真思考的。由于CD163的表达属于显性性状,遵循经典的孟德尔遗传定律,CD163-/-的亲本与野生型CD163+/+杂交会产生杂合CD163+/-胎儿,该胎儿能够表达正常的CD163蛋白。可喜的是,研究发现CD163基因敲除的母猪可以充分地保护仔猪免受PRRSV感染。尽管CD163基因敲除母猪产出的表达CD163分子后代在出生后对病毒存在易感性,但在母体内存在针对PRRSV的保护能够很大程度上减少经济损失以及满足动物福利的要求[46]。
3 展望
传染病防控三要素包括消灭传染源、切断传播途径以及保护易感动物这三方面,培育具有抗病基因的动物就是从保护易感动物这个角度入手进行研究,在农业领域,具有抗虫性、抗寒性、抗盐碱性的作物,如水稻、棉花等作物的生物措施已经得到了广泛的应用。植物与人的亲缘关系较远,具有天然的遗传屏障,相对较为安全,而在动物上的转基因操作出于伦理、人兽共患病等方面的考虑,科学界及社会则从未停止过争论。
近几年对于抗病猪的育种研究,研究人员取得了大量突破性的进展,包括抗PRRSV(CD163)单基因编辑猪、抗PRRSV/TGE(CD163/pAPN)的双基因编辑猪,都为未来猪病的防控开辟了新的道路。此外,一些特定品种的猪对某些疾病具有天然的抵抗能力,通过分析比较基因组差异,编辑差异位点也是抗病育种的一个思路。例如,2020年McCleary等[47]通过对比较易感非洲猪瘟病毒的家猪和不易感的非洲疣猪的基因组序列分析后发现,RELA基因存在3个单核苷酸多态性位点差异,通过基因编辑技术,将非洲疣猪的SNP替换到家猪上获得基因编辑猪,该猪感染非洲猪瘟病毒产生临床症状的时间晚于野生型猪,且排毒量也较低。2018年Xie等[48]应用CRISPR/Cas9为基础的定点整合(knock‑in)平台,结合RNA干扰技术,将筛选出的能有效抑制猪瘟病毒复制的shRNA基因精确整合至猪内源Rosa26基因启动子区域的下游,成功地筛选出能稳定表达抗病基因的细胞系。接着通过体细胞核移植技术和胚胎移植技术成功地获得抗猪瘟病毒的基因编辑猪。体内外攻毒实验均证实这些基因编辑猪能有效限制猪瘟病毒的感染并能显著地降低猪群中猪瘟疾病相关的临床症状和致死率。随后,该团队又在猪基因组上发现另一外源基因整合位点miR‑17‑92基因簇,通过将针对猪流行性腹泻病毒的shRNA整合到该位点可获得抗病基因编辑猪。这些研究都为抗PRRS基因编辑猪的探究提供了线索[49]。综上所述,基于基因编辑及多能干细胞手段赋予猪体抗病毒能力的方法是一种有效的抗病育种策略,或者筛选更加有效的抗病靶标及策略[50-52],且其具有非常广阔的应用前景。

