欧洲ELIXIR生物医学数据工具服务平台体系实践与启示*
刘盛宇 胡拯涌 段一凡 钱 庆 吴思竹
(中国医学科学院/北京协和医学院医学信息研究所 北京 100020)
1 引言
由于高通量、大数据、人工智能等技术的广泛应用,电子健康记录、临床数据和数字医学图像等生物医学数据量激增。然而,人工筛选和初级智能算法等传统分析手段无法处理这些复杂且维度高的数据[1]。因此,亟须发现、获取和利用能高效处理和分析挖掘相关数据的专业工具及服务。
美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)和欧洲生物信息学中心(European Bioinformatics Institute,EBI)在提供丰富的生物医学数据共享服务的同时,也支持数据处理和分析工具及服务,为数据利用提供了重要技术支撑。但在数据处理和分析工具管理方面,欧洲生命科学基础设施中心(European Life-Science Infrastructure for Biological Information,ELIXIR)更为规范和体系化,在工具注册、版本管理和用户支持方面均设定了更高的标准,包括算法透明度、可重复性、计算效率以及细致的版本控制和丰富的培训资源。ELIXIR建立了成熟的平台、标准和管理体系,促进研究人员访问、分析和整合生物医学数据,推动科学发现。深入研究ELIXIR的实践经验,对我国解决生物医学工具管理缺乏有效机制和方法、标准化不足等问题具有参考价值。鉴于此,本文探讨了ELIXIR数据工具服务平台的建设实践。
2 ELIXIR生物医学数据工具服务平台体系建设概述
2.1 建设概况
ELIXIR于2013年12月正式成立,由欧洲分子生物学实验室(European Molecular Biology Laboratory,EMBL)、21个成员国和2个候选国(塞浦路斯和奥地利)组成[2],是欧洲跨政府的生物医学资源整合机构。ELIXIR为了满足生物医学数据分析和整合研究需求,构建了生物医学数据工具服务平台体系,以及工具标准化注册、运行环境管理、工作流集成和工具验证等系列工具子平台,形成生物医学数据处理和分析工具(本文统称为工具)收、管、用体系[3]。
2.2 平台构成
ELIXIR生物医学数据工具服务平台体系提供了生物医学数据注册、标准化、深度分析及高效评估的一体化解决方案,由bio.tools、WorkflowHub、BioContainers、UseGalaxy和OpenEBench 5大子平台组成。每个平台各具特色功能。bio.tools平台致力于数据处理、分析和建模工具的标准化信息注册,并提供工具相关信息检索查询,已收录7 247个工具,涉及遗传学、病毒学和疫苗设计等9个领域。WorkflowHub平台专注于数据工作流的注册和共享,工作流主要执行生物医学数据处理或分析任务,如实现序列组装、序列质控等,已整合300余个工作流,覆盖基因组学、单核苷酸多态性、核糖核酸测序等355个领域。BioContainers平台是为工具提供标准化运行环境的容器解决方案,已支持8 982个工具的运行管理。UseGalaxy是专为生物医学研究人员设计的工具与工作流执行平台,已集成7 851个工具和工作流,其嵌入式设计使研究人员可以无缝执行相关工具和工作流。而OpenEBench平台侧重于多维度工具性能评估,已评估23 748个工具,并根据编程文件,如脚本和App等,对工具进行14种类别划分。
2.3 平台研发技术应用
在平台研发技术应用方面,5个子平台主要使用HTML、Shell和Python开发语言。然而,为了实现特定功能,各子平台还采用了其他开发语言。BioContainers平台利用Perl和Makefile语言自动化构建工具运行环境容器。UseGalaxy平台采用Jinja和TeX语言生成和格式化工作流代码模块。bio.tools平台使用XSLT语言处理XML格式的工具文件注册。而OpenEBench平台为满足多线程编程需求,使用Facebook开发的Hack语言。在平台服务方面,bio.tools、UseGalaxy和OpenEBench平台开发了自定义的应用程序接口(application programming interface,API),以提供更精细化的功能支持。而WorkflowHub和BioContainers平台注重接口可用性,采用了标准的API,如JSON API、RestfulAPI和TRS API,以加强平台之间的相互连接性,见表1。

表1 ELIXIR生物医学数据工具服务平台体系下各子平台属性
3 子平台架构和主要功能特点
3.1 生物医学数据工具注册平台bio.tools
bio.tools是生物医学数据工具的注册和检索平台,致力于实现工具信息的规范化管理。该平台存储了丰富的工具元数据描述,包括工具的唯一标识、基本信息、特征信息、功能信息、使用信息和相关学术资料。研究人员可以使用工具唯一标识符(biotools ID)、工具名称等主要元数据项来检索和发现工具[4-5]。
bio.tools平台为研究人员提供工具元数据注册功能,支持提交biotoolsID、输入/输出格式及数据类型等信息。为确保统一标准,bio.tools采用独特的biotoolsSchema数据模型,涵盖18个结构化生物医学词表,并基于生物医学数据分析和管理(EMBRACE data and methods,EDAM)本体框架实现细粒度描述。EDAM本体提供分层分类,包括工具类别、功能定义、输入/输出格式及数据类型、biotoolsID、支持的数据格式及访问链接等[6]。通过这些元数据标准,确保工具信息的高效整合,见图1。

图1 bio.tools平台工具注册服务模式
3.2 生物医学工作流资源集成平台WorkflowHub
WorkflowHub是生物医学数据工作流的注册和发现平台,提供丰富的工作流资源。支持多维检索,如按创建时间、名称、工作流类型(UseGalaxy、KNIME和Nextflow等)和发布团队进行搜索。研究人员可获取资金来源、工作流源文件和详细描述等信息。为注册集成工作流,WorkflowHub提供完善的注册机制,包括填写元数据信息、上传文件(支持本地和远程链接),并提供多次审核质控。每个工作流都有唯一的数字对象标识符(digital object unique identifier,DOI),确保唯一标识和定位,促进传播和引用。除鼓励研究人员上传工作流外,WorkflowHub管理团队还从GitHub、nf-core和myExperiment等权威渠道筛选和整合工作流,确保库中的工作流经过严格验证[7]。
WorkflowHub为研究人员提供了高效、规范的工作流注册平台,通过核心工作流标准化框架,支持同时注册多个工作流。该框架遵循RO-Crate、通用工作流语言(common workflow language,CWL)和Bioschemas 3个数据标准模型,确保工作流数据的规范化、结构化,提高Web服务器检索效率。Bioschemas标记28个关键属性,增强工作流的在线可查找性;CWL限定输入输出数据格式,规划任务执行顺序;RO-Crate将工作流的文件、目录结构、数据类型等描述整合到JSON-LD的元数据文件[8]。WorkflowHub平台服务模式,见图2,其中“工作流查看”板块(2A)展示基于Bioschemas的工作流检索服务,而“多个工作流注册”板块(2B)揭示依赖RO-Crate和CWL的多工作流并行注册机制。

图2 WorkflowHub平台服务模式
3.3 生物医学数据工具可执行环境容器化平台BioContainers
BioContainers平台利用容器化技术为工具提供了标准化运行环境,确保工具在不同操作系统中的稳定性。该平台主要包括Docker Containers、Conda Containers和Specifications 3大组件。Docker Containers依赖Dockerfile脚本自动设定工具运行环境。而Conda Containers则先利用Conda脚本创建Conda包,并基于此包制定Docker Containers,以确保容器的一致性与可追溯性。Specifications组件则定义了容器的相关规范,如许可证等。
BioContainers提供Web端服务器BioContainers Registry。该服务器对各种工具及其容器镜像进行索引,允许研究人员根据工具名称、类别或开发团队等关键信息进行筛选和查找[9]。每个工具都附带一份详细的描述文件(类似于README.md文件),内容包括工具名称、版本、功能概述及操作方法。借助这些明确指导,研究人员可以在本地轻松运行工具,避免复杂的环境配置步骤。
BioContainers允许研究人员选择合适的容器化技术并撰写配置脚本,以满足其自定义搭建工具时对运行环境的需求。社区管理人员负责对自定义容器进行审核和验证,经过验证的容器将存储在BioContainers核心数据库中,并且可以在BioContainers Registry中查找和使用。当工具运行环境容器需要更新时,研究人员可以通过“容器请求界面”提交更新需求。社区成员将评估和审核该需求,然后该更新将存入核心数据库,以供查找和应用。BioContainers运行环境容器架构,见图3,展示了BioContainers的完整组件,以及从研究人员的初步请求到容器的部署阶段。
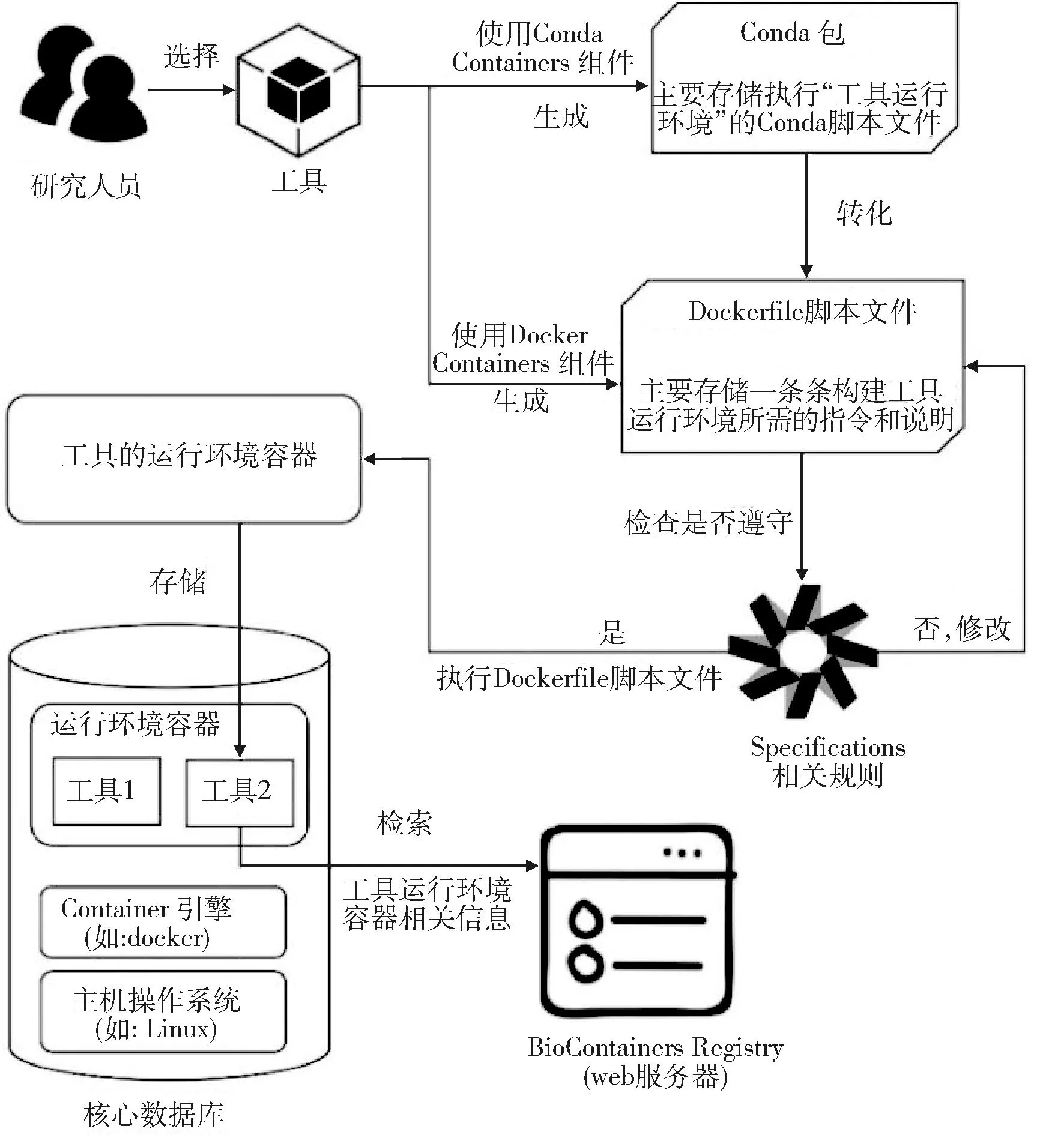
图3 BioContainers运行环境容器架构
3.4 生物医学数据工具/工作流分析平台UseGalaxy
3.4.1 平台功用 UseGalaxy是开源工具和工作流的集成分析平台,为非编程背景研究人员提供一系列准确且可复现的数据分析工具,支持团队协作和数据结果共享。使用UseGalaxy集成工具,研究人员可以完成多种生物医学分析任务,例如DNA/RNA序列比对、变异检测、基因组注释、蛋白质交互网络分析和代谢途径模拟。此外,UseGalaxy还允许研究人员创建、保存和根据项目需求调整自定义分析工作流。
3.4.2 平台技术架构和应用流程 UseGalaxy平台包含5大组件:公共UseGalaxy服务器、GalaxyToolShed存储库、UseGalaxy云服务模块、培训服务模块和UseGalaxy社区。公共服务器采用并行计算和高效工作流调度算法,适应高通量数据处理需求。GalaxyToolShed存储库持续更新,集合了材料科学、机器学习和系统生物学等领域的分析工具,设立严格的审核标准,确保工具和工作流的高质量,并与公共服务器无缝对接。UseGalaxy云服务模块提供弹性计算框架,满足学术用户和商业用户的个性化需求。培训服务模块为研究人员提供专门教学内容,确保平台的有效利用。UseGalaxy社区为研究人员、开发人员和教育工作者提供交流平台,通过论坛、交流会和年会等活动,分享和推广最新研究和技术成果[10]。UseGalaxy“用户工具或工作流分析流程”的技术架构和应用流程,见图4。

图4 UseGalaxy技术架构和应用流程
3.4.3 以用户需求为导向 UseGalaxy遵循用户需求导向的技术更新策略,及时将最新生物医学数据分析技术集成到平台服务中。平台支持数据类型批处理,并集成许多关于数据流开发建议,包括调整输入、处理不连续数据等,以提高数据处理的灵活性。平台引入的Galaxy-ML支持包括数据标准化、特征选择、模型定义、超参数优化和交叉验证在内的全套监督机器学习工具。结合最新生成式技术,UseGalaxy引入OpenAI GPT辅助工具为研究人员遇到的编程问题提供实时问答解决方案。
3.5 生物医学数据工具性能评估平台OpenEBench
3.5.1 平台功用 OpenEBench由ELIXIR及其合作伙伴和西班牙巴塞罗那超级计算中心(Barcelona Supercomputing Center,BSC)推出,是工具和工作流的标准化评估平台。在生物医学领域,基准评估为工具性能提供了客观标准。OpenEBench主要通过使用欧洲核苷酸序列数据库(European Nucleotide Archive,ENA)和欧洲基因组-表型组数据库(European Genome-phenome Archive,EGA)的基准测试数据集进行工具的测试和评估[11]。
3.5.2 工具评估步骤 OpenEBench的工具评估流程可概括为“预测结果生成”和“基准测试”两大阶段。在“预测结果生成”阶段,研究人员(用户)首先将选定的基准测试数据集以及待测工具导入数据分析平台(如UseGalaxy或Nextflow)。然后,该工具在数据分析平台上处理基准测试数据集并进行预测性比较,生成用于后续评估的“预测结果数据”。“基准测试”阶段由验证、指标计算和工具评估3个步骤组成。第1步,验证“预测结果数据”的准确性和完整性,并生成“验证结果数据”。第2步,根据确定的基准对“预测结果数据”进行深入对比,发现并生成关键性能指标,如匹配精度。第3步,将数据汇集到工具评估专题数据库中,生成可视化分析图表,显示最终结果。整个整合过程在虚拟研究环境中进行,以确保所有数据都符合统一且严格的评估标准。
3.5.3 工具评估具体流程 OpenEBench的工具评估由用户社区驱动,涉及初、中、高3个层级的社区成员,不同层级拥有不同权限。初级参与者负责基准测试结果的存储,确保实验的可重复性和数据来源的明确性。中级参与者除存储功能外,还能使用基准测试工作流评估其他成员的工具性能,包括使用参考数据集计算评估指标。高级参与者拥有平台全部权限,能直接启动OpenEBench中的整个工具评估流程。每个层级的评估操作都在前一层级评估设定的基础上进行扩展。高级参与者生成的数据将根据中级参与者设定的评估指标进行处理,并依照初级参与者制定的数据结构进行保存[12],见图5。

图5 OpenEBench工具评估流程
4 子平台之间和外部平台的交互模式
ELIXIR生物医学数据工具服务平台精心设计策略,以确保与其他平台间的高效交互,进而构建协同的研究环境,见图6。在资源方面,生物医学数据工具服务平台各子平台都采用统一的协同共享策略。特定工具和工作流被注册到bio.tools或WorkflowHub数据库后,BioContainers为这些工具和工作流提供稳定且适用于多个操作系统的容器化运行环境。配置完成后,这些工具和工作流在UseGalaxy上直接调用和执行。OpenEBench支持对工具和工作流使用前后的性能评估,以确保工具和工作流在生物医学数据分析任务中的完整性和互操作性,以及任务实现后的工具有效优化和改进。

图6 ELIXIR数据工具服务平台体系内部及外部交互
除了5个子平台之间构成有序的生物医学数据工具服务网络外,ELIXIR还建立了高性能计算平台和培训平台。高性能计算平台支持大规模基因组分析、蛋白质结构模拟等复杂任务,而培训平台则致力于指导研究人员高效应用分析工具和数据处理技术。数据工具服务平台遵循FAIR原则(即可发现、可访问、可互操作、可重用),与计算平台交互,构建标准化数据接口,确保数据符合FAIR特征,并采用高速数据通道技术保障平台间高效数据传输。培训平台定时更新相关培训和辅助学习资源,通过Webhooks和API endpoints为数据工具服务平台提供反馈机制,促进资源和服务的有效优化。此外,培训平台还集成典型工具与工作流应用,支持研究人员进行实践操作。
5 对我国生物医学领域数据工具服务平台建设的启示
5.1 注重工具服务平台生态体系建设
ELIXIR生物医学数据工具服务平台及其5个子平台构成了一个全面且互补的生物医学数据工具服务生态体系。这些平台实现了工具和工作流的规范化收录、存储、管理和应用,标准化组织和描述多种高效工具,提供迅速便捷的工具获取和发现功能。先进的容器化技术按需配置工具,改善工具间的兼容性问题,确保工具的可用性、有效性和可靠性。子平台的建设形成了从工具创建注册到使用反馈的全链条服务体系,规范了工具管理和服务流程,对我国生物医学领域的工具碎片化、开发冗余等问题有重要借鉴意义。
5.2 建立健全工具管理标准规范
ELIXIR的5个子平台在建设中均遵循统一标准,采用BioSchema提出的核心数据模型和工具格式,结合领域本体进行表达规范控制,确保数据一致性和工具间的互操作性。这种规范化策略简化了数据管理流程,提高了工具的适应性和跨平台、跨实验室的数据共享能力。当前,我国生物医学数据工具及平台建设缺乏统一标准,因此应借鉴ELIXIR的经验,加强国内生物医学数据和工具标准规范的研制和应用。这包括引进国际标准并建立健全具有本国特色的生物医学数据工具管理标准规范,以确保工具的高效和规范化管理与应用,进而提升生物医学科研效率并促进科研成果的产出。
5.3 统筹工具运行环境管理
整合存储和计算资源并实施统一管理策略能提高资源利用率。BioContainers使用标准化描述文件如Dockerfile,结合Docker和Singularity等容器技术,确保工具在各种环境中都能保持可移植性和一致性。这简化了部署和配置,减少了运行错误。按需调用和扩展配置能充分利用硬件资源,提高效率。我国生物医学数据增长迅速,科研需求迫切,但硬件和算力分布不平衡,导致分析困难,工具无法使用。ELIXIR提供底层运行环境支持和管理,为研究提供良好思路。
5.4 强化工具基准测试和验证
生物医学领域的数据处理和分析工具繁多,但质量和性能参差不齐,影响数据处理和分析结果的准确性。OpenEBench的基准测试框架科学、准确地评估工具性能,涵盖多种指标,并整合基准测试数据集,确保评估全面深入。这种综合评估方法提升了工具的信任度,为研发人员指明了优化方向。随着人工智能和机器学习在生物医学数据分析领域应用的增多,工具和模型的评测至关重要。我国可借鉴OpenEBench策略,结合实际情况构建基准数据集,建立评估体系,提升工具可用性。
5.5 重视技术跟进和融合
ELIXIR工具服务平台实现了对工具及工作流的版本管理,追踪新技术并整合。已集成OpenAI的GPT等最新技术到UseGalaxy平台,并对引进技术进行科学评估和实验验证。WorkflowHub工作流平台集成GitHub等开源工具的优质资源,不断更新和扩展,以满足科研人员需求。国内生物医学数据工具和平台建设应关注最新技术,优化性能,融入智能化技术和资源,助力创新性研究。
5.6 用户社区驱动创新发展
ELIXIR工具平台的特点是用户社区驱动发展策略,强调用户需求与工具开发的同步性。平台建立各子平台的用户社区,深入挖掘用户需求,确保工具与科研场景紧密结合。平台鼓励用户参与信息审核、工具检测和使用测评,积极获取用户反馈,不断迭代和优化工具产品,提高产品的可用性和易用性。此外,平台组织学术研讨、会议和培训,搭建技术与知识交流桥梁。国内生物医学数据工具和平台建设应重视用户社区参与度,建立交流机制和反馈通道,并根据科研用户需求优化工具产品。
6 结语
本文全面探讨了欧洲ELIXIR生物医学数据工具服务平台体系,并对其在工具注册标准化、运行环境容器化、分析架构及性能评估等领域的成果进行系统性论述。参照其经验,为我国生物医学数据工具规范化管理和相关平台建设提出建议。

