土壤环境中四环素抗性基因向病原菌的转移研究
彭双,宋丹,王一明*,林先贵
1. 江苏开放大学环境生态学院,江苏 南京 210017;2. 中国科学院南京土壤研究所/土壤与农业可持续发展国家重点实验室,江苏 南京 210008
随着畜禽养殖的发展,中国是已成为世界上最大的畜禽养殖国,每年约有38 亿吨畜禽粪便产生(Zhao et al.,2022)。将畜禽粪便还田仍是一种常见的废物处理方式,可提高土壤肥力并增加粮食产量(Li et al.,2021)。然而,随着抗生素在畜牧业中的广泛应用,畜禽粪便含有大量残留抗生素、多种抗生素抗性基因(ARGs)和重金属(Zhu et al.,2013;Sorinolu et al.,2021;Peng et al.,2022a)。越来越多的证据表明,施用粪肥会明显增加土壤中ARGs的种类和丰度(Peng et al.,2017;Rahman et al.,2018),导致抗生素抗性细菌和ARGs 扩散至蔬菜中(Zhang et al.,2019;Huang et al.,2021)。同时,畜禽粪肥还可能含有公共卫生相关病原体(Neill et al.,2018;Alegbeleye et al.,2020),它携带的人畜共患病原体可能会污染新鲜农产品(Li et al.,2020;Zhang et al.,2020),进而导致食用人群的胃肠道疾病。此外,如果这些人畜共患病原体同时携带ARGs和移动遗传元件(MGE),可能会对人类健康造成更为严重的威胁(Zhou et al.,2019)。因此,有必要探讨不同土壤环境条件下,肠道病原菌能否从施肥土壤处获得ARGs 并在土壤中存活,这对于指导动物粪便的合理施用具有重要的意义。
细菌获得抗生素耐药性有两种途径,包括通过自发基因突变产生ARG 和通过水平基因转移(HGT)获得ARG。HGT 可导致ARGs 在相同或不同种属细菌间传递,该过程具有速度快、周期短、范围广的特点,因此HGT 被视为ARGs 传播的主要途径(胡小婕等,2022)。因为致病细菌可以通过HGT 获得耐药性(Tang et al.,2023),所以ARGs的水平转移是评估其环境风险的重要参数之一。目前大多数关于土壤或堆肥环境中ARGs 水平转移的研究仅关注MGEs 的丰度变化特征及其与ARGs 以及细菌群落的相关性和影响因素(Zhou et al.,2021;Qiu et al.,2022;Zhang et al.,2022;Chen et al.,2023)。一些在实验室条件下开展的研究表明,土壤矿物硼钠石、小于100 nm 的塑料颗粒、有机污染物等物质可促进ARGs 的转移(Wu et al.,2020;Hu et al.,2022;Li et al.,2022;胡小婕等,2022)。然而,与实验室纯培养体系相比,自然状态下的土壤或水体环境非常复杂,其中的供体/宿主细菌及抗性质粒种类繁多,微生物的生理状态也不同,因此量化自然环境中的ARGs 转移频率相对较难。质粒介导的接合基因转移被认为是HGT 的主要机制(Sørensen et al.,2005;Thomas et al.,2005)。目前,已有研究在土壤环境中观察到质粒的接合转移(Krasovsky et al.,1987)。Macedo et al.(2022)将携带pKJK5 质粒的大肠杆菌(Escherichiacoli)与粪肥混合后施入土壤,发现质粒可以从粪便转移到土壤细菌,虽然转移的频率很低,且主要发生在施用粪肥后的4 d 内。Xu et al.(2021)向含有四环素的水培系统中接入携带RP4 质粒的大肠杆菌,通过荧光标记来追踪质粒在拟南芥内生菌中的传播,发现大肠杆菌被内化到植物组织中,RP4 质粒被转移到植物内生细菌中,来源于土壤的细菌群落抑制了大肠杆菌的内化,但显著促进了RP4 质粒向植物微生物组的传播。Fan et al.(2019)将携带抗性质粒RP4 的恶臭假单胞菌接种到土壤微宇宙中,在75 天的培养过程中,观察到RP4 的传播增加,宿主细菌的多样性增加,组成发生变化。上述研究通过向土壤/水培环境接入携带已知质粒的供体细菌的方式证明在环境条件下能够发生质粒的转移;然而,如果将环境中携带ARGs 的细菌视作供体,人类致病细菌是否可以通过HGT 从土壤环境或粪便获得ARGs 目前仍然未知。因此,在自然环境条件下进行相关探索,有助于我们了解环境中的ARGs 向致病菌转移的潜力和风险。
在肠道病原体中,沙门氏菌是通过食物和水传播疾病的常见病原体之一(Blanco et al.,2021)。有研究表明畜禽粪便携带的沙门氏菌在土壤中可存活968 d(Nicholson et al.,2005)。沙门氏菌在土壤中存活时间越长,它们污染水果或蔬菜的可能性就越高。为了了解沙门氏菌在不同类型土壤中的存活时长,我们在前期研究中分析了沙门氏菌在进入3种土壤后,其存活数量的动态变化,同时探讨了淹水环境对其存活的影响(Peng et al.,2022b)。由于沙门氏菌可在筛选培养基上形成黑色菌落,明显区别于其他细菌类群,为抗生素敏感型沙门氏菌是否能够从土壤或粪肥获得ARGs 的研究提供了可能性。四环素是动物粪便中最常见的抗生素(Gaballah et al.,2021),猪粪中常常能够检出高浓度的残留四环素和四环素抗性基因(Peng et al.,2022a)。此外,自20 世纪70 年代以来,土壤中几乎所有类别的ARGs 都显著增加,尤其是四环素抗性基因增加的最多(Knapp et al.,2010)。因此,在前期研究的基础上,本研究对进入土壤90 d 后依然存活的沙门氏菌进行了四环素抗性筛选,对分离的抗性疑似沙门氏菌进行了鉴定和全基因组测序;并在实验室纯培养体系下,分析了分离菌株携带的四环素抗性基因向E.coliO157:H7 的转移能力。本研究探讨了人畜共患病原体从土壤环境或者来源于土壤环境的细菌处获得ARGs 的潜力,研究结果可为科学评估土壤环境中ARGs 的转移风险提供参考。
1 材料与方法
1.1 实验设计和土壤样品采集
供试土壤采集自3 个中国科学院农业生态实验站,分别位于江西省鹰潭县(28°15′N,116°55′E)、江苏省常熟市(31°33′N,120°42′E)和河南省封丘县(35°00′N,114°24′E)。从长期施用化肥的田间采集土样(0—20 cm),土壤性质详见Peng et al.(2022b),土壤质地分别为红粘土(R)、壤土(L)和砂质壤土(S)。将土壤阴干后过5 mm 筛,将土壤含水量调节至土壤最大持水量的三分之一,取5 kg 土壤置于花盆(23 cm×22 cm)中。
鼠伤寒沙门氏菌Salmonellaentericasubsp.enteric serovar Typhimurium(CICC 21484)来自中国工业微生物菌种保藏中心(CICC),为四环素敏感型沙门氏菌。将单菌落接种到LB 肉汤中,并在37 ℃下培养约16 h。在4 ℃下离心收集菌体,用磷酸盐缓冲液(10 mmol·L-1,pH 7.2)洗涤3 次,悬浮在无菌去离子水中,调整菌体密度后与133.82 g 新鲜猪粪(干重为40 g)混合,使新鲜猪粪中的沙门氏菌浓度达到1011CFU·g-1。将猪粪与表层土壤混合,分别在自然条件下(接收雨水)和淹水状态下培养,每个处理重复4 次,详见Peng et al.(2022b)。在第90 天和120 天采集0—5 cm 土壤,带回实验室进行四环素抗性菌株的分离和鉴定。
1.2 土壤中四环素抗性疑似沙门氏菌菌株的分离和生理生化鉴定
将5 g 新鲜土壤样品添加到50 mL 磷酸盐缓冲液(10 mmol·L-1,pH 7.2)中,并在180 r·min-1条件下振荡30 min。取0.5 mL 土壤悬浮液涂布到添加四环素的木糖赖氨酸脱氧胆盐(XLD)琼脂(中国,海博)平板上,并在37 ℃下培养过夜。XLD平板中四环素浓度为8 µg·mL-1,在这个浓度下,土壤中的四环素抗性细菌数量约为106CFU·g-1干土(Peng et al.,2016)。沙门氏菌一般在XLD 上形成黑色菌落,该方法的检测限约为100 CFU·g-1干土。经平板涂布培养法检测,供试土壤和新鲜猪粪中均不含能够在XLD 培养基上形成黑色菌落的细菌。
使用从青岛海博生物购买的HBI 沙门氏菌生化鉴定条(HBIG01)、西蒙氏枸橼酸盐试验和吲哚试验对分离的抗性细菌进行生理生化鉴定,相关操作根据产品说明书进行,每株菌3 个重复。同时,用HE 琼脂、沙门氏菌显色培养基、SS 琼脂、亚硫酸铋BS 琼脂、麦康凯MAC 和三糖铁TSI 等筛选培养基培养,以鼠伤寒沙门氏菌SalmonellaTyphimurium (CICC 21484)作为对照,观察分离菌株在不同培养基上的形态特征。
1.3 DNA 提取、ARGs 检测和四环素抗性菌株16S测序鉴定
将分离的菌株划线接种到XLD(含8 μg·mL-1四环素)平板上,37 ℃过夜培养;挑单克隆到装有50 mL LB(含8 μg·mL-1四环素)的三角瓶中,振荡培养8h,将菌液转移到50 mL 无菌离心管中,8 000 g 离心10 min,弃上清,用无菌水洗两次,用细菌基因组DNA 提取试剂盒(TransGen Biotech,Beijing)提取DNA。土壤或猪粪DNA 分别用FastDNA®SPIN Kit for soil/feces(MP Biomedicals,Santa Ana,CA)试剂盒提取,用HT-qPCR 方法(Peng et al.,2022a)检测土壤、猪粪和四环素抗性菌株是否携带329 ARGs 和35 MEGs。
用细菌16S rRNA 基因通用引物27F/1492R(Popowska et al.,2012)扩增菌株的16S rRNA 基因,扩增产物经1%琼脂糖凝胶电泳验证,得到与预期相符的条带,并连接到pEASY-T3 载体上(TransGen Biotech,Beijing),然后克隆到Trans1-T1 感受态细胞中(TransGen Biotech,Beijing),挑取阳性转化子,PCR 验证其携带的条带大小,然后将正确的阳性转化子送交华大基因进行Sanger 测序鉴定。
1.4 全基因组测序和基因注释
全基因组测序工作由北京诺禾致源生物信息科技有限公司完成,采用单分子测序技术,测序平台为PacBio 和Illumina,使用SMRT Link v5.0.1 软件对reads 进行组装,得到初步组装结果,把reads比对到组装序列上,统计测序深度的分布情况,根据序列长度及比对的方法区分初步组装的序列为染色体序列还是质粒序列,并检验序列是否成环。使用GeneMarkS 软件(http://topaz.gatech.edu/)进行细菌的编码基因预测。基因岛可以编码多种功能,涉及共生关系和发病机理,还能提高生物的适应性。使用软件IslandPath-DIOMB 预测基因岛。整合在宿主基因组上的温和噬菌体的核酸称之为前噬菌体,通过软件phiSpy 预测前噬菌体。分别使用Diamond 软件和Resistance Gene Identifier(RGI)软件,与ARDB 和CARD 数据库进行比对,对ARGs进行注释。使用Diamond 软件,与VFDB 数据库进行比对,注释毒力因子信息。使用PlasmidFinder(https://cge.food.dtu.dk/services/PlasmidFinder/)注释质粒分型。T2 菌株的全基因组测序结果已经提交至NCBI 数据库(SUB12441302)。
1.5 四环素抗性菌株的抗性基因转移实验
将培养12 h 的T2 和四环素敏感型E.coliO157:H7(CICC 10907)菌液离心,收集菌体并用磷酸盐缓冲液(10 mmol·L-1,pH 7.2)洗涤3 次,用磷酸盐缓冲液将菌体重悬,制备成菌悬液,两种菌悬液的浓度均为109CFU·mL-1。参照Wang et al.(2019)的方法将等体积的T2 与E.coliO157:H7 菌悬液混合共培养,并添加盐酸四环素使混合液中的四环素浓度分别为0、0.05、0.5、1、5、8、10、20 和40 μg·mL-1。将混合菌液置于25 ℃培养12、24、120 h后,重新悬浮菌体,取100 μL 共培养菌液,分别涂布于添加四环素(终浓度为8 µg·mL-1)的XLD 和O157 显色培养基(青岛,海博),同时在不含四环素的两种平板上涂布混合菌液和O157 作为对照,以计算混合菌液中T2 和O157 的数量并确认平板中的抗生素有效。利用T2 和E.coliO157:H7 在含或不含四环素的XLD 和O157 显色培养基上形成不同颜色的菌落来区分供体、受体和接合子:XLD 平板上T2 菌落呈黑色,O157 菌落呈黄色;在O157 显色培养基上,T2 菌落呈蓝色,O157 菌落为浅红色。
2 结果与分析
2.1 土壤和猪粪中ARGs 的构成和含量
为了充分了解3 种土壤和猪粪中的ARGs 和MGEs 组成,用HT-qPCR 方法检测了猪粪和施用猪粪第1 天的土壤样品(未淹水处理组)中的329 ARGs 和35 MEGs。猪粪中检测到183 种ARGs,26 种MGEs;3 种土壤样品中分别检测到146(L)、152(S)、166(R)种ARGs 和20(L)、22(S)、24(R)种MGEs。土壤和猪粪中不同类型ARGs 的总相对丰度如图1 所示,猪粪中含量最多的是氨基糖苷类抗性基因(Aminoglycoside),占比34.8%,其次为四环素类抗性基因(Tetracycline),占比30.8%。土壤中四环素类ARGs 在3 种土壤中占比分别为47.3%(R)、30.6%(L)和21.4%(S),氨基糖苷类ARGs 占比分别为18.0%(R)、22.1%(L)和25.5%(S)。其中,tetW、tetO和ermB是土壤和猪粪样品中平均丰度最高的ARGs;tnpA-06/IS1216和tnpA-04/IS6100是平均丰度最高的MGEs。
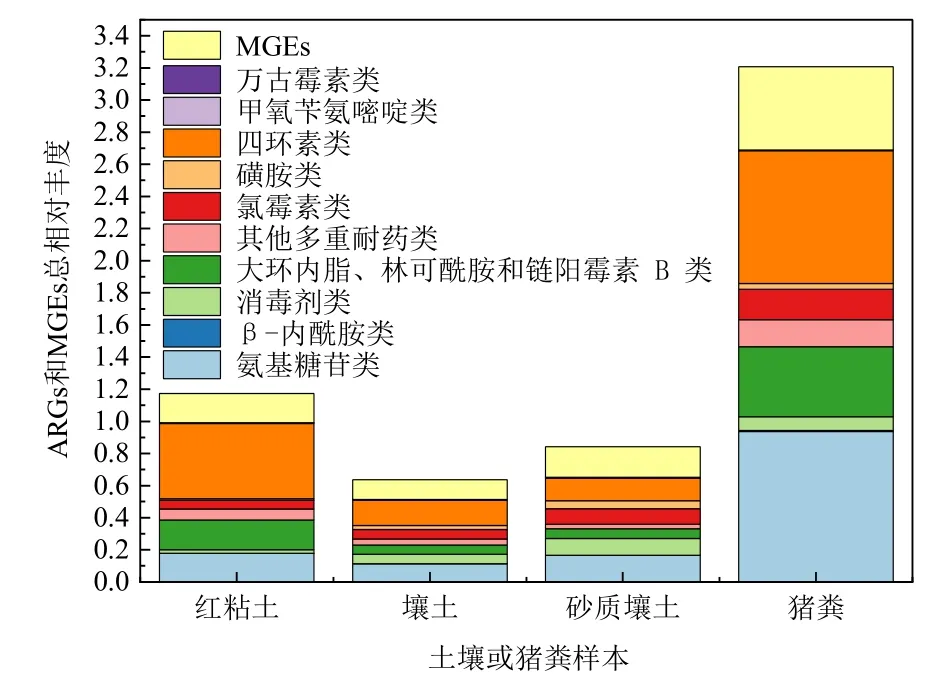
图1 猪粪和施用猪粪第1 天的土壤中ARGs和MGEs 的总相对丰度Figure 1 The relative abundance (ARG copies/16S rRNA gene copies) of ARGs and MGEs detected in pig manure and different types of soils under natural condition collected on the first day
2.2 土壤中四环素抗性疑似沙门氏菌菌株的分离和生理生化鉴定
在施肥后的第90 天,通过添加四环素的XLD平板筛选,仅在不淹水的红粘土中分离得到3 株能在XLD 平板上形成黑色菌落的四环素抗性疑似沙门氏菌,该抗性疑似沙门氏菌形成的菌落相对较小,生长较慢;该时期该处理土壤中存活的沙门氏菌数量为 (4.89±1.89)×104CFU·g-1干土。用沙门氏菌生化鉴定条和筛选培养基分别对分离到的这3 株抗性菌进行生理生化鉴定,结果如表1 所示,3 株抗性菌株可产生半乳糖苷酶,能分解邻硝基酚-半乳糖苷(ONPG),生成黄色的邻硝基酚,且不能利用西蒙氏枸橼酸盐,吲哚阳性,在这些生理生化特性上,与实验添加的沙门氏菌存在差异,根据这些特性初步判断3 株抗性菌为大肠杆菌。在施肥后的第120 天,未分离到具有四环素抗性的疑似沙门氏菌。
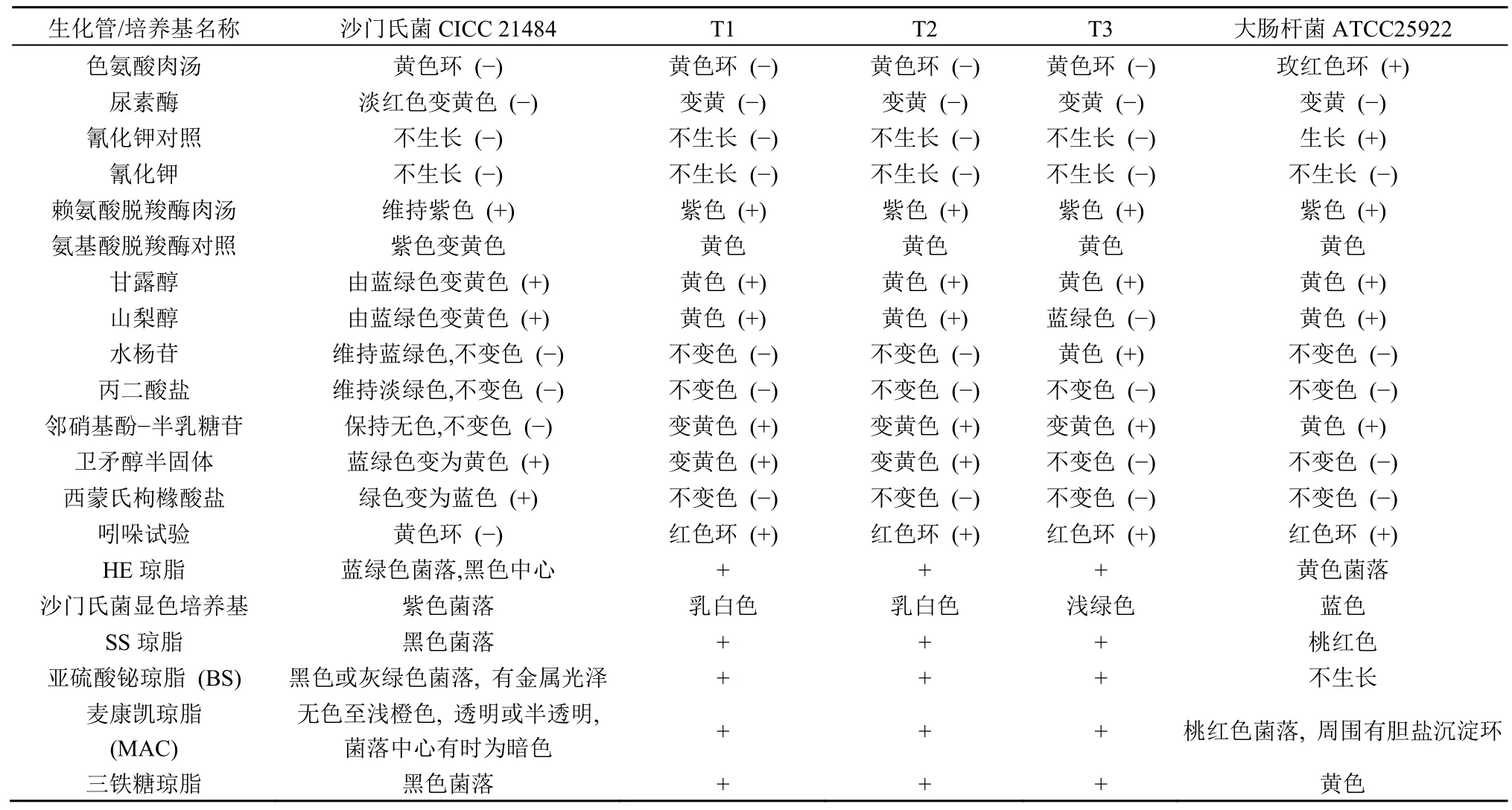
表1 沙门氏菌、大肠杆菌和3 株四环素抗性菌的生理生化鉴定结果Table 1 Physiological and biochemical identification results of Salmonella, E. coli and three tetracycline resistant strains
2.3 四环素抗性菌株的测序鉴定和ARGs 检测结果
对菌株的基因组进行16S rRNA 基因扩增,扩增产物经克隆测序后,将测得的序列在NCBI 数据库中比对,结果如表2 所示,3 个抗性菌株与大肠杆菌的相似度均达到99%,说明3 株抗性细菌均为能够产生H2S 的大肠杆菌,而不是获得四环素抗性的沙门氏菌。

表2 3 株四环素抗性菌测序和NCBI 比对结果Table 2 Sequencing and NCBI comparison results of three tetracycline resistant strains
通过HT-qPCR 检测方法,用329 对ARGs 和35 对MEGs 引物对3 株菌的DNA 进行了检测(部分ARG 种类有多条检测引物)。T1、T2 和T3 分别有55、64 和63 个ARG 引物检出阳性,其中包含了38、44 和43 种ARGs(表3)。T1、T2 和T3 分别携带7、11 和10 个MGEs,其中T3 携带的10 种MGEs 在T2 中均能检测到。如表3 所示,有37 种ARGs 和5 种MGEs 在3 株菌中共有,此外,3 株菌分别携带一些特有ARGs 和MGEs。根据3 株菌携带的ARGs 数量,选择T2 进行全基因组测序,以进一步了解该菌的基因构成和ARGs 分布位点。

表3 3 株四环素抗性菌携带的ARGs 和MGEs 检测结果Table 3 ARGs and MGEs carried by the three tetracycline resistant strains detected by HT-qPCR
2.4 抗性菌株T2 的全基因组分析
将 T2 菌株进行全基因组测序,共得到867 963 947 bp Clean Data,平均序列读长为8 951 bp,经生物信息学分析后,该菌株含有一个4.723 Mb 的染色体(GC 含量为50.88%)和两个长度分别为40.48kb(GC 含量为44.87%)和93.31 kb(GC含量为51.09%)的质粒,三者均为环状结构(图2)。共含有4690 个编码基因、30 个CRISPR 序列(均位于染色体上),15 个基因岛(其中1—13 位于染色体,14 和15 分别位于两个质粒上),8 个前噬菌体(均位于染色体上),170个非编码RNA(指tRNA、rRNA 及sRNA 等3 种),217 个散在重复序列。

图2 T2 菌株染色体和质粒圈图Figure 2 Chromosome and plasmid circle diagrams of strain T2
使用Diamond 软件,把T2 的4 690 个基因的氨基酸序列,与NR 数据库进行比对,把T2 的基因和其相对应的功能注释信息结合起来,总共有4 653 个基因在NR 数据库得到注释,其中有3 512个基因与Escherichia coli的基因相似,595 个基因与Achromobactersp 基因相似。该注释结果进一步验证了16S rRNA 基因测序鉴定结果,证实该菌株为大肠杆菌。该菌株携带的两个质粒序列经过blast比对,结果如表4 所示,两个质粒均与大肠杆菌携带的质粒序列高度相似,其中质粒1 携带的部分序列与沙门氏菌相似。使用PlasmidFinder 对两个质粒的分型进行检索,结果质粒1 为IncX1 型质粒(相似度98.93%),质粒2 为IncY 型质粒(相似度99.48%)。
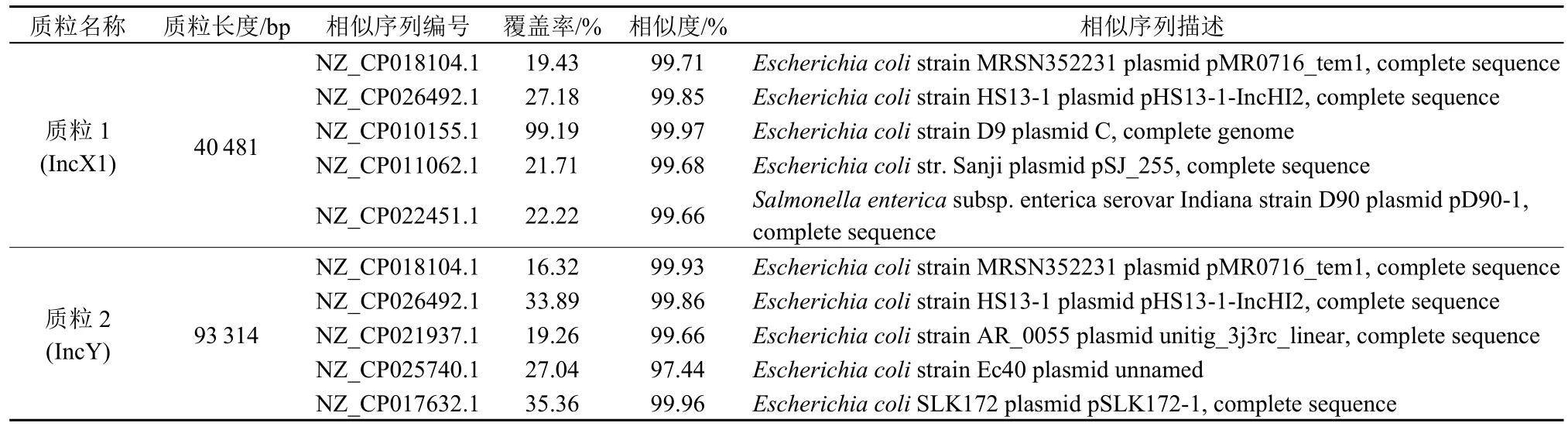
表4 T2 菌株携带的两个质粒序列比对结果Table 4 Sequence comparison results of the two plasmids carried by strain T2
全序列与COG、GO、KEGG、Pfam、Swiss-Prot、Nr 等通用数据库比对,其中,与COG 数据库比对得到了3 807 个注释基因,包含前噬菌体和转座子共197 个;与KEGG 数据库比对,得到4586 个注释基因,其中包含了多个对人类致病的基因,例如幽门螺杆菌感染中的上皮细胞信号转导基因、军团菌病、百日咳、沙门氏菌感染、肺结核基因等。再将序列与ARDB、CARD、VFDB 和PHI 等功能数据库比对,得到ARDB 注释基因34 个(相似度大于98%)、CARD 注释基因62 个(相似度大于91%)(表5),438 个VFDB 毒力因子,和440 个经PHI注释的病原体与宿主互作蛋白。注释的ARGs 中包括了3 种四环素抗性基因,均位于质粒2 上,但是两个数据库的比对结果存在差异。

表5 菌株T2 的全基因组序列与ARDB、CARD 比对后注释的ARGsTable 5 Antibiotic resistance gene annotated after comparing the whole genome sequence of strain T2 with ARDB and CARD
2.5 四环素抗性菌的基因转移
将T2 菌体与E.coliO157:H7(四环素敏感型)菌株共培养,并施加不同浓度梯度的四环素,观察T2 菌株能否将四环素抗性基因转移到E.coliO157:H7。实验结果表明,在0、0.05、0.5、1、5、8、10、20 和40 μg·mL-1四环素作用下,均未分离到获得四环素抗性的E.coliO157:H7。说明T2 与E.coliO157:H7 在共培养期间,T2 携带的四环素抗性基因没有转移到O157 菌株。
3 讨论
3.1 在土壤环境中,沙门氏菌未通过HGT 获得四环素抗性基因
细菌病原体对抗生素的耐药性发展使危及生命的细菌感染的治疗大大复杂化(O’Neill,2014),因此抗菌药物耐药性被认为是21 世纪全球卫生面临的主要挑战之一(Berendonk et al.,2015)。人类携带的抗性共生菌或致病菌被认为可以通过食物链或环境在人和动物之间直接或间接交换传播;动物和人类之间的联系,以及环境在促进这种联系方面的重要性,被称为“One Health”框架(Hernando-Amado et al.,2019)。在“One Health”框架下,了解ARGs 在环境中的归趋及其向人类致病菌的转移潜力对于评估ARGs 的潜在风险和控制ARGs 在环境中的转移扩散至关重要。本研究通过向3 种土壤施入含有大量沙门氏菌的粪肥的方式,尝试了解粪肥及土壤中的四环素抗性基因转移到沙门氏菌中的可能性。结果表明,在土壤中存在大量沙门氏菌受体的情况下,无论土壤是否处于淹水状态,沙门氏菌没有从粪肥和土壤处获得四环素抗性基因并在土壤中长期存活。研究表明,土壤细菌群落结构是 ARGs 含量的主要决定因素,而不是 HGT(Forsberg et al.,2014)。尽管土壤微生物携带多种ARGs,但仅有少数与人类病原体共有,而且这些基因在土壤群落内以及从土壤向其他细菌的传播并不常见(Sommer,2014)。ARGs 已被公认为环境中的新型污染物,虽然HGT 是ARGs 传播与扩散的主要途径,但其常受环境条件变化和共存物质的影响(胡小婕等,2022)。据报道,ARGs 的转移频率可能受各种非抗生素条件的影响,例如环境温度和pH、环境介质的特征、额外的电场和声场等(Shi et al.,2022)。因此,在自然条件下精确评估HGT 在环境ARGs 的扩散中发挥的作用,对于我们科学评估环境中ARGs 的转移风险至关重要。
绝大多数沙门氏菌可以产生硫化氢(hydrogen sulfide,H2S),因此,H2S 是初筛与鉴定沙门菌的重要生化指标。XLD 琼脂培养基是常见的用来鉴别和筛选沙门氏菌的培养基,在碱性的条件下,培养基中的硫代硫酸钠及柠檬酸铁铵与沙门氏菌产生的H2S 反应,使菌落的颜色呈黑色。我们从红壤中分离到3 株具有四环素抗性且能够产生H2S 的细菌,但是经过生理生化和测序鉴定,最终确定为大肠杆菌,而不是获得抗性的沙门氏菌。大肠杆菌是和临床关系最为密切的细菌之一,与畜牧兽医、公共卫生有关,长期以来一直作为粪源性细菌污染的卫生指标,是国际上公认的卫生监测指示菌。大肠杆菌一般不产H2S,近年来也偶见产H2S 大肠杆菌的报道(张晓玲等,2021)。有研究表明,H2S 的产生在大肠杆菌中是由质粒介导的(Bouanchaud et al.,1975),而且一些菌株可以通过质粒转移将此特性传递给不产H2S的大肠杆菌(Harnett et al.,1996)。H2S 是一种可自由穿梭细胞的小分子,扩散性强。已有研究指出,H2S 不仅是细菌硫代谢的副产物,还在大肠杆菌、沙门菌耐药过程中发挥重要作用,H2S 可利用其还原性清除抗生素氧化应激反应产生的活性氧物质(ROS),发挥细菌耐药作用(吴府丽,2017)。沙门菌中,phs操纵子编码硫代硫酸盐还原酶,由phsA、phsB和phsC等3 段基因组成,是沙门菌产生H2S 的重要功能基因(吴府丽,2017)。我们在T2 菌株的质粒2 上也发现了phsA、phsB和phsC基因,且与 TCDB 数据库中Salmonellatyphimurium 携带的基因一致;此外,在添加沙门氏菌之前,土壤和猪粪中并未检测到能够产生H2S 的菌株。因此,我们推测可能是T2 从添加的沙门氏菌处获得了这3 段基因,从而获得了产生H2S 的能力;这可能暗示着在土壤环境中大肠杆菌能够通过自然转化(细菌从环境中直接摄取游离DNA 并获得相应的遗传特征)或基因转移获得特定功能;也暗示着大肠杆菌从环境中获得基因的潜力可能高于沙门氏菌。
土壤是“One Health”框架下一个极其重要的组成部分,通过与人类和/或动物相关微生物的相互作用,ARGs 逐渐富集并可能最终流入人类食物链(Wang et al.,2022)。尽管我们在3 种土壤中都未发现沙门氏菌从土壤或粪肥处获四环素抗性功能,但是并不排除有其他类型ARGs 发生转移的可能性。此外,未淹水土壤中沙门氏菌初始浓度约为108CFU·g-1干土,因为不能适应土壤环境,其数量在施肥14 d 后逐渐减少,大多数在施入土壤后90 d 内死亡(Peng et al.,2022b),因此本研究只关注了90 d 后仍然存活的沙门氏菌是否获得了耐药性,对于施肥后90 d 内是否有沙门氏菌从土壤获得ARGs 并未关注。Pornsukarom et al.(2017)从施用猪粪7—21 d 的土壤中分离到多株携带IncF1 质粒的耐药沙门氏菌,且它们携带的质粒都可以通过培养体系下的接合转移实验传递给E.coliJM109。考虑到各种生物和非生物因素,肠道病原体在土壤中的存活率是高度可变的,为了避免粪肥中的病原微生物直接或通过食物链间接对人造成感染,应该尽可能地延长施肥和收获间隔,让带入农田的病原微生物数量得到充分削减。
3.2 在纯培养体系下,从土壤中分离的T2 菌株未向病原菌E.coli O157:H7 转移四环素抗性基因
水平基因转移(HGT)在ARGs 的传播中起着重要作用,其中接合转移被认为是ARGs 在环境中传播的主要方式之一。在本研究中,分离到的3 株四环素抗性菌均携带多种ARGs(表3),对其中携带ARGs 种类最多的T2 进行全基因组测序,明确T2 携带了两个质粒并且质粒上携带了多个四环素抗性基因(表5)。为了了解T2 菌株携带的四环素抗性基因的转移潜力,在施加不同浓度四环素的条件下,将其与四环素敏感型E.coliO157:H7 菌株共培养,结果未观察到T2 菌株的四环素抗性基因发生转移。目前,关于耐药基因接合转移的研究多采用的是纯培养的供体和/或受体细菌,研究的质粒以RP4 质粒(Jia et al.,2021;Zhang et al.,2022;Zhai et al.,2022;Liu et al.,2023)或pUC 质粒(Fang et al.,2022;Wang et al.,2022)较为多见。不同的质粒有不同的宿主范围,这在一定程度上可能影响质粒的转移效率,RP4 质粒是一种典型的IncP-1α类质粒,IncP-1 和IncQ-1 类型质粒宿主范围较宽,为泛宿主质粒(broad-host-range plasmids),且常含有抗生素抗性特征,由于它们可以有效的接合转移并在广泛的宿主中稳定复制,因此被认为有助于水平基因转移(Dungan et al.,2020)。所有革兰氏阴性菌都是IncP 和IncW 质粒的潜在宿主(Droge et al.,2000)。本研究分离的T2 携带的两个质粒经过序列比对与数据库中大肠杆菌携带的质粒非常相似(表4),经过PlasmidFinder 分型发现两个质粒分别属于IncX1 和IncY 型,IncX 质粒可追溯到前抗生素时代,是肠杆菌科的狭宿主质粒(narrowhost-range plasmids),它们编码IV 型菌毛,使其自身能够接合转移,并为宿主细菌提供辅助功能,如抗微生物药物耐药性和生物膜形成(Johnson et al.,2012)。IncY 质粒也属于狭宿主质粒(Hawkey,2008),在肠杆菌科中比较常见,如大肠杆菌、沙门氏菌和肺炎克雷伯杆菌等(Zhang et al.,2017)。除了质粒的宿主范围之外,供体细菌携带的质粒转移潜力对接合转移的发生频率也很重要,结合质粒(conjugative plasmid)可编码结合系统控制基因,该基因可以促使细菌自发地进行接合(Li et al.,2022),例如RP4 质粒是一种IncP 类泛宿主质粒,该IncP 质粒的接合转移取决于trfA和trbB基因的产物(Qiu et al.,2012)。有研究报道,IncF、IncA/C、IncL/M、IncI1、IncHI2 和IncN 质粒家族是与肠杆菌科耐药性在全球范围内传播特别相关的质粒家族(Bustamante et al.,2017)。因此,需要进一步研究不同类型质粒的转移潜力及其在环境ARGs 的传播扩散中发挥的作用。
此外,纯培养体系下细菌质粒接合转移的研究方法也有多种,例如Pornsukarom et al.(2017)在接合转移实验中进行了42 ℃热激处理;而其他一些研究则只是将受体和供体菌株在培养液中混合培养(Lin et al.,2016;Zhang et al.,2022;Alvarez-Molina et al.,2023);为了避免细菌繁殖,在另外一些研究中用磷酸盐缓冲液(Wang et al.,2019;Jia et al.,2021;Yan et al.,2023;Li et al.,2023)或生理盐水(Chen et al.,2019;Zhai et al.,2022)替代培养液。接合实验的反应条件,包括初始细菌浓度,供体细菌与受体细菌的比率,养分供应,实验体系的温度和pH,接合时间的长短等都可能对细菌质粒的接合转移频率产生影响。本研究将T2 和E.coliO157:H7 在磷酸盐缓冲液中共培养,并在实验体系中加入不同浓度的四环素,但是均未分离到接合子。因此,需要进一步优化接合转移实验条件,以进一步分析T2 携带的两个质粒的转移潜力及其影响因素。此外,还需要建立合适的ARGs 水平转移实验体系,模拟真实的环境条件,综合利用多种技术对ARGs 的接合、转化和转导进行跟踪和检测。
4 结论
1)向3 种土壤施入含有大量沙门氏菌的猪粪,无论土壤是否处于淹水状态,沙门氏菌均未从粪肥和土壤处获得四环素抗性基因并在土壤中长期存活;说明自然条件下,沙门氏菌通过HGT 从土壤环境获得抗性基因并长期存活的可能性很小。
2)从红壤中分离到3 株具有四环素抗性且能够产生H2S 的细菌,经过生理生化和测序鉴定,最终确定为大肠杆菌;经HT-qPCR 检测,T1、T2 和T3 分别携带38、44 和43 种ARGs,7、11 和10 个MGEs。
3)对T2 进行全基因组测序与分析,结果表明T2 携带了两个长度分别为40.48kb 的IncX1 型和93.31kb 的IncY 型质粒,15 个基因岛,前噬菌体和转座子共197 个;经ARDB 注释获得34 个ARGs,其中包括了2 种四环素抗性基因均位于IncY 型质粒上。
4)将T2 菌体与四环素敏感型E.coliO157:H7菌株共培养,在施加不同浓度四环素的条件下,T2携带的四环素抗性基因没有向E.coliO157:H7 菌株转移。

