48份分蘖洋葱种质资源遗传多样性的ISSR标记和农艺性状分析
金 雪 周新刚 刘守伟 刘淑芹 吴凤芝
(东北农业大学园艺学院,黑龙江哈尔滨 150030)
分蘖洋葱(Allium cepavar.aggregatum)为百合科(Liliaceae)葱属(Allium)一年生栽培草本植物,是洋葱的一个变种(李曙轩 等,1979;崔崇士和傅喜山,2000)。分蘖洋葱营养丰富,药用价值较高,是重要的世界性蔬菜和调味品,在我国东北地区,如黑龙江、吉林等地广泛种植。分蘖洋葱生育期短,病虫害较轻,能有效提高土地复种指数,减少农药使用量,适于发展绿色食品,市场前景较好。但长期以来分蘖洋葱品种的筛选鉴定工作尚不深入,品系分类不清楚,可能存在同物异名现象,不利于分蘖洋葱的推广和应用。
目前同工酶标记(Rouamba et al.,2001;高述民 等,2003;张光花 等,2003)、SSR(Simple sequence repeats)标记技术(Fischer & Bachmann,2000;李慧芝 等,2007;周静 等,2011)、ISSR(Inter-simple sequence repeat)标记技术(徐启江 等,2007;刘宏敏 等,2011)、rDNA原位杂交技术(Ricroch et al.,1992)及形态学与分子生物学相结合的方法(Sangecta et al.,2006)已广泛用于大葱、大蒜、洋葱等葱属作物的遗传多样性研究,并已经取得了较大进展。而分蘖洋葱种质资源的遗传多样行分析尚处于起步阶段(初文娇,2010)。
由Zietkiewicz 等(1994)发展起来的ISSR(Inter-simple sequence repeat)技术具有操作简便、快速、多态性高、重复性好等特点,已在许多作物的亲缘关系分析、遗传多样性研究等方面得到了广泛应用(王武台 等,2011)。本试验搜集了在黑龙江省和吉林省广泛种植的48份分蘖洋葱种质资源,对其进行了田间农艺性状调查和ISSR 标记,以期揭示其遗传多样性及亲缘关系,为分蘖洋葱的品种评价与鉴定、引种栽培等提供科学依据。
1 材料与方法
1.1 试验材料
供试的分蘖洋葱材料共48份(表1),为从黑龙江省和吉林省不同地区搜集的地方品种。
1.2 试验方法
1.2.1 田间农艺性状调查 田间试验于2011年4~7月在东北农业大学试验田进行。48份分蘖洋葱材料每份材料播种1行,行距30 cm,行长9 m,有效播种数为180株,株距5 cm。田间管理按常规进行。调查的农艺性状包括分蘖洋葱的幼叶色、是否抽薹、鳞茎色和单球质量。于种植后30 d(幼苗期)观察分蘖洋葱的幼叶色,将幼叶色划分为3 类:浅绿、翠绿和深绿;种植后90 d(收获前)调察是否抽薹;收获后观察鳞茎色,将鳞茎色划分为4 类:浅棕黄色、棕黄色、黄色和紫黄色。
1.2.2 基因组DNA 提取 植株长至3~4片叶时,取分蘖洋葱嫩叶的新鲜组织并保存于-80℃冰箱。采用TIANGEN 公司柱式植物基因组试剂盒进行基因组DNA 提取。提取的基因组DNA 用1%琼脂糖凝胶电泳检测,并用紫外分光光度计测定其浓度和纯度。
1.2.3 ISSR分析 ISSR所用引物序列见表2(姜福星,2006;徐启江 等,2007;邓文明,2008),由上海生工生物工程有限公司合成。
PCR 反应总体积为50 µL,其中10×Buffer 5 µL,Mg2+3 µL,dNTP 4 µL,DNA 20 ng,Taq酶(TIANGEN 公司)0.8 U,10 μmol·L-1引物2 µL。PCR 扩增反应在Bio-Rad PTC200 PCR 仪(Bio-Rad 公司)上进行,扩增程序为:94℃预变性5 min;35个热循环,每个循环包括94℃变性30 s,58℃退火45 s,72℃延伸90 s;终延伸72℃ 10 min。扩增产物在1.5%琼脂糖凝胶上电泳检测,100 V 电压,150~180 min,并以100~2 000 bp 标准分子量为对照。用Gelred 染色20 min后,用紫外分析仪观察并记录扩增结果。
1.2.4 数据分析 ISSR 电泳结果,对清晰可辨的电泳条带全部用于统计分析,按扩增条带有无记数,当某一扩增带出现时,赋值为“1”,不存在时赋值为“0”,从而把图形资料转换成数据资料。根据Nei 和Li(1979)相似系数法求得品种i和j之间的相似系数(GSij):GSij=2Nij/(Ni+Nj),其中,Nij表示品种i,j共有的条带数目,Ni表示品种i中的条带数目;Nj表示品种j中的条带数目。利用NTSYSpc Version2.0 软件(Rohlf,1987)按照UPGMA法进行聚类分析,并构建树状图。
表型性状数据标准化后用NTSYS 软件分析,按照遗传距离进行UPGMA 聚类分析。

表2 所用引物序列及其多态性
2 结果与分析
2.1 ISSR 多态性分析
从51条ISSR 引物中筛选出了13条能扩增出清晰带型、多态性较好的引物。这13条引物中有7条二核苷酸重复序列,1条三核苷酸重复序列,1条四核苷酸重复序列,4条简并性核苷酸重复序列。二核苷酸重复序列中(CT)n 扩增出的条带数最多,说明分蘖洋葱基因组中含有丰富的CT 重复序列,这为下一步利用ISSR 标记定向开发分蘖洋葱SSR 引物奠定了基础。
用13条引物(表2)对48份分蘖洋葱材料进行PCR 扩增,所扩增的片段大小在100~2 000 bp 之间(图1)。13条引物共扩增出132条条带,其中多态性条带109条,多态百分率(P)为82.6%,平均每条引物扩增出10.15条条带(表2)。其中,引物3A35 扩增出的条带数最多,为18条,多态百分率为94.4%;引物UBC812 扩增出的条带数最少,为3条;多态性最高的引物是UBC840,多态百分率达100%(表2)。说明ISSR 标记扩增效率较高且多态性较好。

图1 引物UBC840 对48份分蘖洋葱材料的ISSR 扩增电泳图谱
2.2 ISSR 标记聚类分析
48份分蘖洋葱的遗传相似系数变化范围在0.59~0.94,平均为0.75,表现出丰富的多样性。其中宾县1 和宾县2,白城1 和双城2 之间的相似系数最大,均为0.94,表明其亲缘关系较近;绥化和阿城、四平和南方毛葱、五常红七社和四六瓣的相似系数最小,均为0.59,表明其亲缘关系较远。吉林省和黑龙江省两省样品的相似系数变化范围分别为0.62~0.90、0.59~0.94,平均相似系数为吉林省0.72<黑龙江省0.74,表明来自黑龙江省的样品遗传多样性低于来自吉林省样品。
从聚类分析树状图(图2)可以看出,在相似系数为0.75(L1)处,48份分蘖洋葱资源可分为3 类:Ⅰ类有6份资源来自吉林省,17份来自黑龙江省,相似系数为0.73~0.94,该类中可继续分为若干亚类和次亚类,遗传多样性较丰富;Ⅱ类只包括1份资源,为来源于黑龙江省的南方毛葱,在相似系数为0.71 的水平上与其他所有材料分开,表明这份材料有特殊的基因,建议进一步试验;Ⅲ类包括24份资源,其中4份来源于吉林省,20份来源于黑龙江省,相似系数为0.81~0.92,遗传差异比较明显。在相似系数为0.81(L2)处,可将Ⅰ类再 分为4个亚类:Ⅰ-a包括1份资源,为吉林;Ⅰ-b 包括18份资源,4份源于吉林省,其余来自黑龙江省;Ⅰ-c 包括五常红七社和1号2份资源;Ⅰ-d 只有1份资源,为长春。
2.3 农艺性状分析
农艺性状观察结果见表3。利用表3中的表型性状数据计算了材料间的遗传相似系数,结果表明,48份分蘖洋葱材料间的遗传相似系数变化范围为0.38~1.00,平均为0.64。利用上述农艺性状观察结果构建的聚类分析树状图(图3)表明:在遗传相似系数为0.60 处可将48份材料分为3 类:Ⅰ类包括西林、独头等18份资源,其中5份来自吉林省;Ⅱ类包括28份资源,其中来自吉林省的内蒙、吉林、农安1、四六瓣、吉林2 等遗传距离较近;Ⅲ类包括2份资源,4号和南方毛葱。
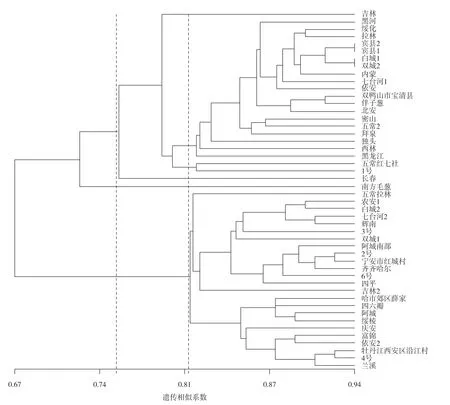
图2 48份分蘖洋葱材料的ISSR 标记聚类分析树状图(UPGMA 法)

表3 48份分蘖洋葱表型性状观察结果
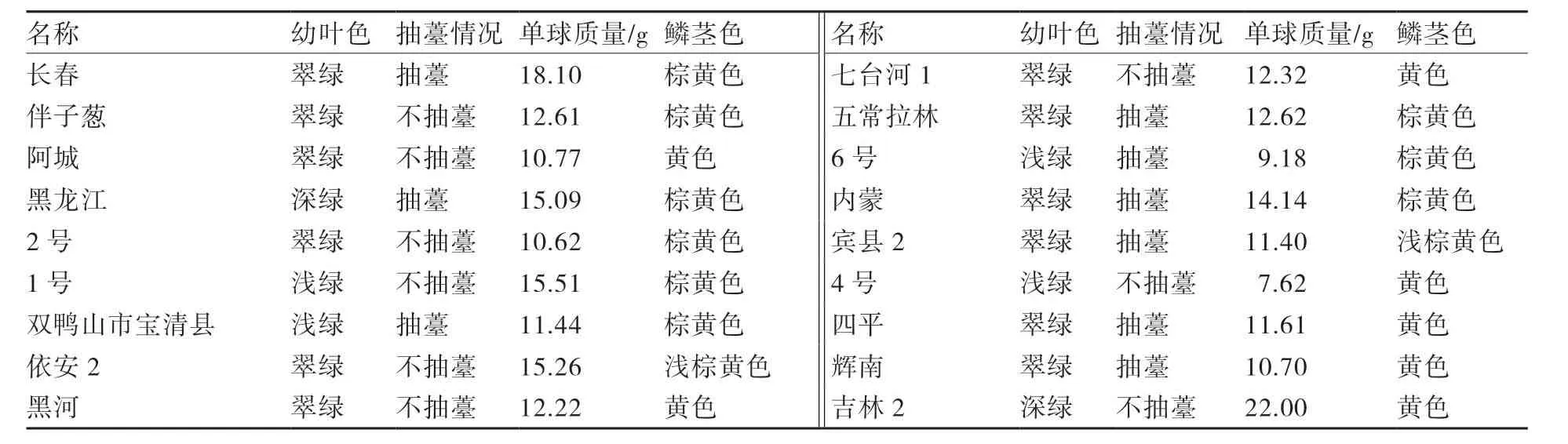
续表

图3 48份分蘖洋葱材料的农艺性状聚类分析树状图(UPGMA 法)
3 结论与讨论
ISSR分子标记结果稳定性高,不受样品形态、基因表达与否及环境因子的限制(Joshi et al.,2000;Pradeep Reddy et al.,2002;Gupta et al.,2010;Tanya et al.,2011),且在葱属作物分类、进化和遗传多样性的研究中应用很多。梁景霞等(2008)应用18个多态性引物对96份有代表性的烟草种质进行PCR 扩增,共获得314条稳定条带,其中多态性条带299条(占95.2%)。刘宏敏等(2011)以96份有代表性的韭菜资源为材料,进行遗传多样性和亲缘关系分析,发现韭菜DNA 的ISSR 扩增多态性比率达94.7%,认为ISSR 是韭菜种质资源研究的一种有效分子标记。
品种间遗传关系研究和遗传多样性分析对于分析作物育种具有十分重要的意义(Schut et al.,1997)。本试验以13条ISSR 引物对48份分蘖洋葱材料共扩增出132条条带,其中多态性条带109条,多态百分率(P)为82.6%,平均每条引物扩增出10.15条条带。供试48份分蘖洋葱的遗传相似系数变化范围在0.59~0.94,其中宾县1 和宾县2、白城1 和双城2 在分子水平上的遗传相似系数达0.94,亲缘关系较近,说明其可能是同物异名的品种;而南方毛葱与其他资源的相似系数较小,在0.59~0.75 之间,可作为资源研究和育种的材料。ISSR 扩增片段多态性较高,遗传相似系数变幅较大,说明分蘖洋葱在分子水平上遗传多样性较丰富。这可能是由于以下几点原因:首先,分蘖洋葱在长期进化过程中,为了适应不同的生态型,分化出了不同的种质资源;其次,分蘖洋葱为无性繁殖,不易发生变异,适应外界环境条件差,只有遗传基础丰富的种质才能存活下来;另外,由于过去对分蘖洋葱研究的投入较少,育种目标的定向选择机会较少,多样性性状丢失也较少。
ISSR分子标记的UPGMA 聚类结果表明,48份分蘖洋葱种质资源可分为三大类,与基于农艺性状的聚类结果基本一致。两者均表明多数种质资源之间的亲缘关系与地理来源具有较大相关性,如来自黑龙江省的大部分种质聚为一类,来自吉林省的四平和吉林2 聚为一类。但也有地理来源差距较大的种质聚为一类,如白城1 和双城2 聚在一起,这可能是近时间内黑龙江省与吉林省之间分蘖洋葱种质资源的频繁交流所致。但是,两种方法的聚类结果也存在一些差别,这可能是由于ISSR 标记体现的是种质资源间分子水平上的差异,是遗传因素的体现,而且检测出的分子水平差异并不都与所观测的农艺性状相关联;而表型性状的差异是基因与环境共同作用的结果(Eivazi et al.,2008)。另外,理想的分子标记应具有多态性高、共显性遗传、遍布整个基因组、检测手段简单快速、成本低廉、重复性好等要求。但是,目前发现的任何一种分子标记均不能满足以上所有要求。例如,扩增片段长度多态性标记技术(AFLP)具有最高的多态性检测效率,但多态信息量不高;简单重复序列标记技术(SSR)标记具有多态性高,遗传信息量大等优点(Ali et al.,2008),如果AFLP位点能够定位在SSR 遗传连锁图谱上,那它将是非常有效的分子标记(Haussmann et al.,2002;Geleta et al.,2006)。应进一步结合SSR、AFLP等分子标记方法进行比较分析,从而更准确地反映不同分蘖洋葱种质资源间的遗传关系。
初文娇.2010.不同品系分蘖洋葱农艺形状比较研究.北方园艺,(17):9-13.
崔崇士,傅喜山.2000.黑龙江省蔬菜常用品种大全.哈尔滨:黑龙江科学技术出版社.
邓文明.2008.岷江百合遗传多样性研究〔硕士论文〕.南京:南京林业大学.
高述民,李凤兰,陆帼,杜慧芳,赵英.2003.中国大蒜(Allium sativumL.)18个品种的酯酶同工酶多态性分析.植物学通报,20(6):723-729.
姜福星.2006.百合种质资源遗传多样性ISSR 研究〔博士论文〕.南京:南京林业大学.
李慧芝,尹燕枰,张春庆,张敏,李建敏.2007.SRAP 在葱栽培品种遗传多样性研究中的适用性分析.园艺学报,34 (4):929-934.
李曙轩,郑光华,蒋先明,李式军.1979.蔬菜栽培学总论.北京:中国农业出版社.
梁景霞,祁建民,方平平,王涛,陈顺辉,周东新,陶爱芬,梁康迳,吴为人.2008.烟草种质资源遗传多样性与亲缘关系的ISSR 聚类分析.中国农业科学,41(1):286-294.
刘宏敏,张明,李延龙,曹秀敏,王贞,乔保健,尹守恒.2011.韭菜种质资源DNA 指纹库构建与聚类分析.河南农业科学,40(8):164-168.
王武台,古瑜,韩启厚,吴锋,孙德岭.2011.芹菜种质资源亲缘关系的 ISSR分析.中国蔬菜,(8):22-27.
徐启江,崔成日,贾铁金.2007.洋葱种质资源多样性的ISSR分析.中国农学通报,6(23):126-130.
张光花,徐玉芳,李春香,张启沛.2003.胡葱与洋葱、葱过氧化物酶同工酶研究及聚类分析.植物遗传资源学报,4(1):47-50.
周静,陈书霞,程智慧,孟焕文.2011.大蒜SSR 体系的建立与优化.西北农业学报,20(11):117-122.
Ali M,Rajewsk J,Baenziger P,Gil K,Eskridge K,Dweikat I.2008.Assessment of genetic diversity and relationship among a collection of US sweet sorghum germplasm by SSR markers.Molecular Breeding,21:497-509.
Eivazi A,Naghavi M,Hajheidari M,Pirseyedi S,Ghaffari M,Mohammadi S,Majidi I,Salekdeh G,Mardi M.2008.Assessing wheat (Triticum aestivumL.) genetic diversity using quality traits,amplified fragment length polymorphisms,simple sequence repeats and proteome analysis.Annals of Applied Biology,152:81-91.
Fischer D,Bachmann K.2000.Onion microsatellites for germplasm analysis and their use in assessing intra- and interspecific relatedness within the subgenusRhizirideum.Theoretical and Applied Genetics,101:153-164.
Geleta N,Labuschagne M T,Viljoen C D.2006.Genetic diversity analysis in sorghum germplasm as estimated by AFLP,SSR and morpho-agronomical markers.Biodiversity and Conservation,15:3251-3265.
Gupta S,Srivastava M,Mishra G,Naik P,Chauhan R,Tiwari S,Kumar M,Singh R.2010.Analogy of ISSR and RAPD markers for comparative analysis of genetic diversity among differentJatropha curcasgenotypes.African Journal of Biotechnology,7:4230-4243.
Haussmann B,Hess D,Seetharama N,Welz H,Geiger H.2002.Construction of a combined sorghum linkage map from two recombinant inbred populations using AFLP,SSR,RFLP,and RAPD markers,and comparison with other sorghum maps.Theoretical and Applied Genetics,105:629-637.
Joshi S,Gupta V,Aggarwal R,Ranjekar P,Brar D.2000.Genetic diversity and phylogenetic relationship as revealed by inter simple sequence repeat (ISSR) polymorphism in the genusOryza.Theoretical and Applied Genetics,100:1311-1320.
Nei M,Li W H.1979.Mathematical model for studying genetic variation endonucleases.Proceedings of the National Academy of Sciences(USA),76:5269-5273.
Pradeep Reddy M,Sarla N,Siddiq E A.2002.Inter simple sequence repeat (ISSR) polymorphism and its application in plant breeding.Euphytica,128:9-17.
Ricroch A,Peffley E,Baker R.1992.Chromosomal location of rDNA inAllium:in situ hybridization using biotin-and fluoresceinlabelled probe.Theoretical and Applied Genetics,83:413-418.
Rohlf F J.1987.NTSYS-pc:microcomputer programs for numerical taxonomy and multivariate analysis.American Statistician,41:330.
Rouamba A.,Sandmeier M,Sarr A,Ricroch A.2001.Allozyme variation within and among populations of onion (Allium cepaL.)from West Africa.Theoretical and Applied Genetics,103:855-861.
Sangecta K M,Vecre G R,Anand L.2006.Analysis of genetic diversity among Indian short-day onion (Allium cepaL.) cultivars using RAPD markers.The Journal of Horticultural Science and Biotechnology,81:774-778.
Schut J,Qi X,Stam P.1997.Association between relationship measures based on AFLP markers,pedigree data and morphological traits in barley.Theoretical and Applied Genetics,95:1161-1168.
Tanya P,Taeprayoon P,Hadkam Y,Srinives P.2011.Genetic diversity among Jatropha and Jatropha-related species based on ISSR markers.Plant Molecular Biology Reporter,29:252-264.
Zietkiewicz E,Rafalske A,Labuda D.1994.Genome fingerprinting by simple sequence repeat (SSR) anchored polymerase chain reaction amplification.Genome,20:178-183.

