长江中下游5 个湖泊黄颡鱼(Pelteobagrus fulvidraco)种群线粒体细胞色素b 基因的遗传变异分析*
钟立强,刘朋朋,潘建林,,王明华,陈友明,秦 钦,边文冀,陈校辉**
(1:江苏省淡水水产研究所,南京 210017)
(2:南京师范大学生命科学学院,南京 210097)
黄颡鱼(Pelteobagrus fulvidraco)属于鲇形目(Siluriformes)、鲿科(Bagridae)、黄颡鱼属(Pelteobagrus),是一种常见的中小型经济鱼类,广泛分布于长江、黄河、珠江及黑龙江等各水域[1].因其肉质细嫩、味道鲜美、营养丰富、无肌间刺等特点,广受消费者欢迎,市场价格一度攀升,已经成为越来越重要的特种养殖品种[2].
线粒体DNA (Mitochondrial DNA,mtDNA)由于其母系遗传特点,复制过程中缺乏修复机制导致序列的累积变异速率远远高于核基因组的变异速率等特点,已成为群体遗传学和系统发育等研究的理想的分子标记[3-4].细胞色素体b(cyt b)为mtDNA 上的蛋白质编码基因,进化速度适中,适合种间到种内水平上的系统发生研究,在鱼类群体遗传结构与系统发育关系中有着广泛的应用[5-7].黄颡鱼在我国各大水系均有分布,通过线粒体DNA 不同区域和基因序列对不同水系黄颡鱼种群的遗传结构和系统演化关系已经进行了一些研究.Watanabe 等[8]、方耀林等[9]和库喜英等[10]分别以 mtDNA D-loop 区、ND1/2 基因和 cyt b 基因为分子标记,对我国多个水系间的黄颡鱼群体进行遗传多样性和遗传结构分析,结果表明我国各水系的黄颡鱼处于中等遗传变异水平,各水系间存在相当的基因流,进化速率相对很低.丁言伟[11]则对我国黄颡鱼属的五种黄颡鱼mtDNA的ND4 基因进行了测序分析,阐明了黄颡鱼属系统发育关系和遗传变异,探讨了黄颡鱼属五种鱼类的种群遗传结构,黄颡鱼的种群遗传多样性最小,黄颡鱼现在的种群可能由一个单一的古老的世系动态扩张而来.而目前对长江水系内部不同湖泊和流域的黄颡鱼遗传结构分析还未见报道.本研究采集长江中下游鄱阳湖、巢湖、滆湖、洪泽湖、太湖5 个湖泊的黄颡鱼种群,通过mtDNA cyt b 基因核苷酸序列的比较分析,探讨5 个湖泊的黄颡鱼种群遗传多样性和进化关系,期望深入了解5 个湖泊黄颡鱼种群的遗传背景和种质资源现状,以利于在黄颡鱼育种中更好地发掘其遗传潜力,为黄颡鱼多性状复合育种技术研究及新品系选育积累基础数据.
1 材料和方法
1.1 研究材料
实验用野生黄颡鱼采集于长江中下游鄱阳湖、巢湖、滆湖、洪泽湖、太湖5 个湖泊,每个种群采集样本数见表1,剪取活鱼部分尾鳍,于无水乙醇中固定,放入4℃冰箱保存.
1.2 DNA提取
基因组DNA 的提取参照Sambrook 等[12]的方法.DNA 浓度、质量用分光光度仪(Unico UV-4802H)和1%琼脂糖凝胶电泳检.总DNA 稀释至100 ng/μl,-20℃储存备用.
1.3 PCR扩增及测序
Cyt b 扩增和测序的引物为通用引物 L14724 和 H15915[13],引物序列为 L14724:5’-GACTTGAAAAACCACCGTTG-3’;H15915:5’-CTCCGATCTCCGGATTACAAGAC-3’,引物由上海捷瑞生物工程有限公司合成.
PCR 反应体系为 50 μl:模板 DNA 50 ng;2× PCR Mix 25 μl(包含 Taq 酶 2.5 U,dNTPs 10 μmol,MgCl20.1 mmol),上、下游引物(10 μmol/L)各1 μl,其余体积用水补足.反应程序为:94℃预变性2 min,94℃变性45 s,60℃退火1 min,72℃延伸1 min,经35 个循环后再72℃延伸10 min.PCR 产物经割胶纯化后,直接送上海捷瑞生物工程有限公司利用正反引物进行双向测序.
1.4 数据分析
DNA 序列用 BioEdit 7.0.5[14]软件比对剪切序列,MEGA 4.1 软件[15]计算碱基含量,构建系统进化树.DnaSP 软件[16]计算变异位点,单倍型数目、单倍型多样性指数(h)、平均核苷酸差异数(K)和核苷酸多态性(π)等,用Arlequin 3.01 软件[17]中的Kimura 2-Parameters 方法构建5 个湖泊种群间的相对遗传距离,用分子方差分析(AMOVA)方法计算遗传分化指数(Fst)并分析遗传变异来源与组成.
2 结果
2.1 序列特征
5 个湖泊的黄颡鱼种群60 个个体的mtDNA cyt b 序列经Bioedit 软件比对剪切后,获得长度为955 bp 的同源序列,MEGA 4.1 软件分析其碱基组成平均为 T (26.0%)、C(32.3%)、A(27.6%)、G(14.1%),A +T含量为53.6%,略高于G+C 含量46.4%.DnaSP 软件分析显示,共有54 个多态性位点,约占核苷酸总数的5.65%,单态突变位点(singleton variable sites)39 个,简约信息位点(parsimony informative sites)15 个;同时还检测到43 个插入或缺失位点,碱基插入或缺失主要以C 或G 单核苷酸重复片段的形式进行.按突变类型分,转换位点33 个,颠换位点17 个,转换和颠换共存位点4 个,转换和颠换之比为1.94.而不计插入/缺失部分,60 个个体中共检出37 种单倍型序列,37 个单倍型序列GeneBank 登录号为:JX424082 ~JX424118,种群共享的单倍型有3 个(表2).5 个种群的单倍型平均多样性指数为0.945±0.018,平均核苷酸多样性指数为0.00419±0.00043,各种群多样性参数见表1.5 个湖泊的黄颡鱼种群中,滆湖和鄱阳湖种群的单倍型多样性指数最高,洪泽湖种群的核苷酸多样性指数最高,而太湖种群的单倍型多样性指数和核苷酸多样性指数均为最低.

表1 5 个黄颡鱼种群的单倍型及遗传多样性参数Tab.1 Parameter summary of haplotypes and genetic diversity of five populations of yellow catfish

表2 黄颡鱼群mtDNA cyt b 序列37 种单倍型变异位点及其在5 个种群中的分布Tab.2 Variation sites of mtDNA cyt b of 37 haplotypes of yellow catfish and their di stribution in five populations
2.2 群体变异和遗传结构
将测序获得的60 个个体的mtDNA cyt b 序列输入Arlequin 软件,使用Kimura 2-Parameters 方法构建5 个湖泊种群间的相对遗传距离和遗传分化系数Fst(表3).从遗传距离来看,太湖种群与滆湖种群间的遗传距离最远为0.00651,遗传距离最近的是鄱阳湖和巢湖种群之间为0.00375.种群内部遗传距离从大到小依次 为:鄱 阳 湖 (0.00640) > 太 湖(0.00521)>洪泽湖(0.00461)>巢湖(0.00446)>滆湖(0.00377).五种群的平均遗传分化系数Fst为 0.0684.5 个湖泊的黄颡鱼种群间存在显著性遗传变异(P <0.05).
为了进一步分析种群间的相互遗传差别,运用Arlequin 软件中AMOVA 分子方差分析法估算5 个种群间的遗传变异结构和来源.分子方差结果显示,5 个黄颡鱼种群内具有很高的遗传变异.种群间的变异占总变异的6.84%,而种群内的变异对总变异的贡献率达到93.16%(表4),群体内未出现遗传分化.分析单倍型数据所得遗传分化系数(Gst)为0.01992,基因流 Nm 为 12.30;而 60 个个体的 Gst为0.13441,基因流 Nm 为1.61.

表3 5 个黄颡鱼种群间K 2-P 遗传距离和遗传分化系数Tab.3 Average pairwise difference and Fst between five yellow catfish populations
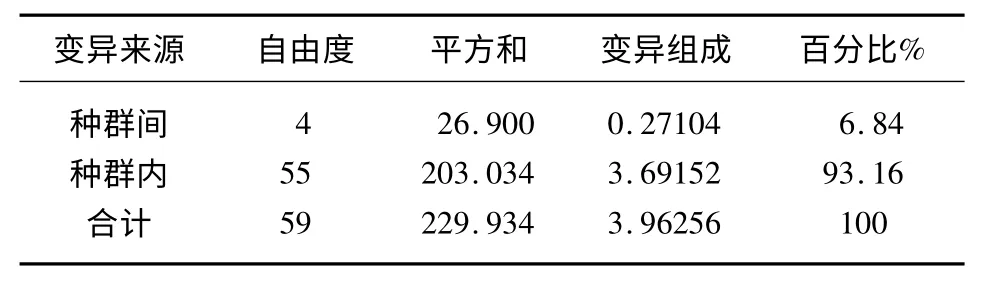
表4 黄颡鱼5 个种群间遗传差异的分子方差分析表(AMOVA)Tab.4 Analysis of molecular variance (AMOVA)of five populations of yellow catfish

图1 5 个黄颡鱼地理种群的 UPGMA 聚类树Fig.1 Dendrogram of five yellow catfish populations by UPGMA method based genetic distance
2.3 聚类分析
将60 个黄颡鱼个体的mtDNA cyt b序列输入MEGA 4.1 软件,对5 个湖泊种群进行聚类分析,得到UPGMA聚类树(图1).5 个黄颡鱼地理种群没有明显聚类,形成了单系类群,长江中游的鄱阳湖与巢湖首先聚为一支,接着分别与下游的滆湖、洪泽湖和太湖聚合,基本与地理分布距离一致.
2.4 种群扩张
用Fu’Fs 中性检验5 个湖泊黄颡鱼种群的显著偏离中性突变,Fu[18]认为:Fs >0,表明种群趋于稳定,Fs <0,表明种群趋于扩张.检验结果显示,种群的整体Fs=-35.277(P <0.05),这表明5 个黄颡鱼种群整体上有扩张趋势.但巢湖、滆湖、鄱阳湖和太湖种群的Fs 值都小于0,而洪泽湖种群Fs 值大于0.用60 个个体的mtDNA cyt b 序列构建两两间的矩阵,进行碱基歧点分布分析,个体间碱基歧点分布曲线见图2,曲线呈现出明显的单峰形,两个结果同时表明长江中下游5 个湖泊的黄颡鱼在历史上可能经历了种群扩张事件.
3 讨论
3.1 遗传多样性

图2 5 个黄颡鱼种群mtDNA cyt b 基因错配碱基分布Fig.2 The mismatch distribution of the Mitochondrial DNA cyt b of five yellow catfish populations
本研究中5 个湖泊黄颡鱼种群 cyt b 序列的碱基组成为 T(26.0%)、C(32.3%)、A(27.6%)、G(14.1%),其中G 的含量显著低于其他碱基的含量,表现出明显的反G 偏倚,显示出细胞色素b 基因的共同特征[19],这也是线粒体 DNA 的一个特点[20].从鱼类线粒体DNA cyt b 基因分析研究的报道可知,福建近海竹荚鱼(Trachurus japonicus)[21]cyt b 基因 A +T 含量为 50.9% 、怒江角鱼(Epalzeorhynchus bicornis)[22]A +T 含量为 62.5% ,鞍带石斑鱼(Epinephelus lanceolatus)[23]A+T 含量为 54.4%,鲌亚科鱼类(Culterinae)[24]A+T 含量为 56.6% ,本研究中黄颡鱼 A+T含量为53.6%,略高于G+C 含量46.4%,符合其他鱼类cyt b序列碱基组成的特点.
本研究所涉及的5 个湖泊黄颡鱼种群60 个个体间的单倍型序列显示,mtDNA cyt b 基因单倍型序列种类丰富,总数达到37 种,占全部序列的61.67%,平均单倍型多样性和核苷酸多样性分别为 0.945 ±0.018 和 0.00419 ±0.00043,高于怒江角鱼[22](0.579 ± 0.069、0.00070 ± 0.00012)、黄河裸体裂尻[25](0.700 ± 0.218、0.0026 ± 0.0019)和青海湖裸鲤[26](0.783 ± 0.053、0.00205 ± 0.00126),与鲤鱼[27](0.637 ±0.055、0.00857 ±0.00200)和福建近海竹荚鱼[21](0.937 ±0.025、0.336 ±0.034)相比,遗传多样性略低.黄颡鱼群体的单倍型多样度高(h=0.945 ±0.018)而核苷酸多样度低(π=0.00419 ±0.00043).Grant等[28]依据单倍型多样度h 和核苷酸多样度π 推测了鱼类群体的4 种进化情景(高h,高π;高h,低π;低h,高π;低h,低π),黄颡鱼种群的状况被认为可能是小的有效种群经过一段时间的稳定后发生了扩张,快速的种群增长有利于提高对新突变的保持力而导致核苷酸多样度降低.Watanabe 等[8]以mtDNA D-loop 区为分子标记,从采自黑龙江水系至珠江水系的60 个黄颡鱼样本中获得8 个单倍型,不同水系间平均遗传距离为0.006.丁言伟[11]研究了我国不同水系60 个黄颡鱼样本的线粒体ND4 序列,获得23 个单倍型,遗传距离在0.001 ~0.007 之间,核苷酸多样度π 为0.0025,与本研究中cyt b 基因序列变异的分析结果高度一致.因此,本研究中黄颡鱼种群遗传多样性低的原因并不是cyt b 基因进化过于保守,更有可能是对黄颡鱼种群遗传多样性现状的真实反映.
AMOVA 对遗传变异的分析表明,全部遗传变异的93.16%来自种群内部,远远大于各种群间的遗传变异,说明黄颡鱼各种群内个体间的遗传多样性较高,遗传变异多来源于种群内,只有较少部分存在于种群间.
3.2 种群遗传结构与基因流
群体遗传学认为,Fst值可以表示群体间的分化程度,一般在0 ~0.05 之间表示分化较弱,0.05 ~0.15 之间表示遗传分化中等,如达到0.15 ~0.25 之间表示遗传分化较大,而超过0.25 表示遗传分化极大.本实验结果显示,5 个湖泊黄颡鱼种群间的遗传分化系数Fst为0.0684,说明5 个黄颡鱼种群间存在中等偏低的遗传分化,不存在显著的系统地理格局.群体间的相对遗传距离能反映群体间的亲缘关系,本研究中种群间的遗传距离在0.00375 ~0.00651 之间,太湖与滆湖种群间的遗传距离最远,鄱阳湖和巢湖种群之间遗传距离最近.滆湖与太湖地理位置相对最近,遗传距离却最远,这可能与滆湖种群采集的样本偏少有关.另外,为了治理太湖水质,太湖的各个入湖和出湖的水道上都建立了大量水利设施,阻断了滆湖与太湖黄颡鱼种群的交流,人为地加大了2 个湖泊黄颡鱼种群间的遗传分化.根据遗传距离构建的UPGMA 进化树显示5 个种群没有分化成不同的分枝谱系,这些结果表明5 个湖泊的黄颡鱼在cyt b 水平上未出现群体遗传分化,提示种群间存在广泛的基因交流.一般认为,由于地理隔离造成鱼类基因交流的完全中断,或同一水域中不同栖息环境的存在部分地限制鱼类的基因交流,是物种形成种群结构甚至分化出亚种的重要因素[29].Wright 认为:当种群基因流系数Nm >1 时,种群间存在一定的基因流动:如果Nm >4,它们就是一个随机的单位;如果Nm 远远小于1,种群会被强烈的分化[30].而本实验中单倍型之间的基因流系数Nm 为12.30,单倍型遗传分化系数(Gst)为0.01992,60 个个体的基因流Nm 为1.61,Gst为0.13441,表明5 个湖泊种群间存在相当大的基因流动.黄颡鱼是一种生态适应性很强的鱼类,不同水系的居群借助于水系的连通而发生基因交流的可能性很大,5 个湖泊种群的黄颡鱼生境差异不明显,在一定程度上为种群间的基因交流提供了有利条件,这可能是造成长江中下游湖泊的黄颡鱼种群缺乏比较明显的地理种群,进化过程中未形成明显的种群遗传结构的重要因素.
黄颡鱼是我国最重要的中小型淡水经济养殖鱼类之一,广泛分布于我国湖泊和河流等水域,种群历史演化检测结果表明,黄颡鱼种群存在扩张趋势.保护好长江及其流域内各支流和湖泊中鱼类的遗传多样性是我国重要的渔业管理政策.本研究对黄颡鱼mtDNA cyt b 基因的遗传背景、遗传结构及遗传变异水平进行探讨,为黄颡鱼的种质资源评估提供了部分基础资料.本研究表明长江中下游5 个湖泊的黄颡鱼种群具有中等的遗传变异,而目前黄颡鱼养殖和繁育的亲本大多来源于野生资源的捕捞,过度捕捞、生态环境的破坏以及人工繁殖数代退化后的种苗流入自然水域,造成野生资源的进一步退化.因此,需要加强对黄颡鱼野生种质资源的保护,有必要建立规范的良种保种和选育基地,在保证种群自然扩张、提高遗传多样性的同时,保护良种的种质资源,维护良种的纯度和遗传稳定性,还能大量供应市场,满足广大人民群众的消费需求.
[1]褚新洛,莫天培.中国动物志,硬骨鱼纲,鲇形目.北京:科学出版社,1999:152-156.
[2]钟立强,陈校辉,蔡永祥等.黄颡鱼DNA 分子标记的研究进展.中国农学通报,2012,28(5):67-75.
[3]Brown WM.Evolution of animal mitochondrial DNA.In:Nei M,Koehn RK eds.Evolution of genes and proteins.Sunderland MA:Sinauer,1983:62-88.
[4]Saccone C,Pesole G,Sbisa E.The main regulatory region of mammalian mitochondrial DNA:structure-function model and evolutionary pattern.J Mol Evol,1991,33(1):83-91.
[5]彭作刚,张耀光,何舜平等.从细胞色素b 基因序列变异分析中国鲇形目鱼类的系统发育.遗传学报,2005,32(2):145-154.
[6]陈合格,刘文彬,李建中等.三种鳖线粒体DNA 细胞色素b 基因序列的比较分析.水生生物学报,2006,30(4):380-385.
[7]程起群,温俊娥,王云龙等.刀鲚与湖鲚线粒体细胞色素b 基因片段多态性及遗传关系.湖泊科学,2006,18(4):425-430.
[8]Watanabe K,Nishida M.Genetic population structure of Japanese bagrid catfishes.Ichthyological Research,2003,50(2):140-148.
[9]方耀林,汪登强,刘绍平等.长江中游湖泊中黄颡鱼线粒体DNA 的遗传变异.中国水产科学,2005,12(1):56-61.
[10]库喜英,周传江,何舜平.中国黄颡鱼的线粒体DNA 多样性及其分子系统学.生物多样性,2010,18(3):262-274.
[11]丁言伟.黄颡鱼属(硬骨鱼纲,鲿科)鱼类分子系统发育及种群遗传结构的研究[学位论文].武汉:华中农业大学,2005.
[12]Sambrook J,Russell DW.Molercular cloning:A laboratory mamual.3rd edition.New York:Cold Spring Harkbor Labroatory Press,2002:463-469.
[13]Xiao WH,Zhang YP,Liu HZ.Molecular systematics of Xenocyprinae (Teleostei:Cyprinidae):Taxonomy,biogeography,and coevolution of a special group restricted in East Asia.Mol Phylogenetics Evol,2001,18(2):163-173.
[14]Hall T.BioEdit v.7.0.5.Biological sequence alignment editor for windows.Ibis Therapeutics a division of Isis pharmaceuticals http://www.mbio.nesu.edu/bioefit.html.2005.
[15]Tamura K,Dudley J,Nei M et al.MEGA4:Molecular evolutionary genetics analysis(MEGA)software version 4.0.Mol Biol Evol,2007,24(8):1596-1599.
[16]Rozas J,Anchez-DelBarrio JC,Esseguer XM et al.DnaSP,DNA polymorphism analyses by the coalescent and other methods.Bioinformatics,2003,19(18):2496-2497.
[17]Excoffier L,Laval G,Schneider S.Arlequin ver 3.01:An integrated software package for population genetics data analysis.Berne:Computational and Molecular Population Genetics Laboratory (CMPG).Switzerland:University of Berne,2006.
[18]Fu YX.Statistical tests of neutrality of mutations against population growth,hitchhiking and background selection.Genetics,1997,147(2):915-925.
[19]Rogers AR,Harpending H.Population growth makes waves in the distribution of pairwise genetic differences.Mol Biol Evol,1992,9(3):552-569.
[20]Wolstenholme DR.Animal mitochondrial DNA:Structure and evolution.In:Wolstenholme DR ed.Mitochondrial genomes.San Diego:Academic Press,1992:173-372.
[21]牛素芳,苏永全,王 军等.福建近海竹荚鱼线粒体DNA 控制区和细胞色素b 遗传多态性.中国水产科学,2011,18(1):66-74.
[22]张东亚,陈 勇,刘绍平等.怒江濒危鱼类角鱼种群遗传结构研究.淡水渔业,2009,39(2):3-7.
[23]区又君,吴 勇,刘楚吾.从细胞色素b 基因序列研究鞍带石斑鱼的分类学地位.热带海洋学报,2008,27(3):45-49.
[24]冯晓宇,谢 楠,冯建彬等.基于细胞色素b 基因序列的鲌亚科鱼类系统发育研究.淡水渔业,2009,39(5):23-27.
[25]赵 凯,杨公社,李俊兵等.黄河裸裂尻群体遗传结构和cyt b 变异分析.水生生物学报,2006,30(2):129-133.
[26]赵 凯,何舜平,彭作刚等.青海湖裸鲤的种群结构和线粒体DNA 变异.青海大学学报,2006,24(4):1-4.
[27]钟立强,张成锋,周 凯等.四个鲤鱼种群ITS-1 序列的遗传变异分析.湖泊科学,2011,23(2):271-276.
[28]Grant WS,Bowen BW.Shallow population histories in deep evolutionary lineages of marine fishes:insights from sardines and anchovies and lessons for conservation.Journal of Heredity,1998,89(12):415-426.
[29]Cassel A,Tammaru T.Allozyme variability in central,peripheral and isolated populations of the scarce heath (Coenonympha hero:Lepidoptera,Nymphalidae);implications for conservation.Conservation Genetics,2003,4(1):83-93.
[30]吕宝忠,钟 扬,高莉萍等译.分子进化与系统发育.北京:高等教育出版社,2002.

