转mapk双链RNA干扰表达载体黄瓜对根际土壤细菌多样性的影响
陈国华 ,弭宝彬,李 莹,李春月
(1.中国农业科学院蔬菜花卉研究所,北京 100081;2.中南大学研究生院隆平分院,长沙 410125;3.北华大学林学院,吉林 132013;4.吉林省白山市靖宇县第一中学,白山 134300)
土壤细菌是土壤中数量最丰富、分布最广泛的微生物类群,它占土壤微生物总量的70%—90%。土壤细菌广泛参与土壤有机质积累、营养元素循环过程,其多样性和活性是保持土壤生态系统稳定的基础。土壤细菌对外界干扰比较敏感,是土壤生态系统变化的预警指标,常用于研究转基因作物对土壤生态系统的影响,评价转基因作物的安全性。目前,很多科学家针对转Bt基因作物对土壤微生物多样性和生物活性的影响进行大量研究[1-4],以揭示Bt蛋白对土壤生态系统的安全性。转基因作物对土壤微生物的影响由转基因作物释放到土壤中的外源蛋白的化学和生物学特性引起的,或者由转基因植株的生理生化特性改变造成的[5]。随着RNAi技术的发展,利用表达双链RNA的RNA干扰作用来防治病虫害的转基因作物越来越多。相对于表达外源蛋白的转基因作物来说,表达双链RNA的转基因植物对土壤微生物的影响可能会通过分泌RNA对土壤微生物起作用。目前,针对表达双链RNA的转基因作物对土壤生态系统安全性的研究未见报道。
陈国华等利用RNAi技术成功地沉默了线虫mapk基因,致使南方根结线虫的生长发育受阻而导致死亡,并成功的构建了表达mapk双链RNA的转基因黄瓜植株,通过黄瓜表达mapk的双链RNA沉默根结线虫的mapk基因,对根结线虫具有良好的防治效果[6]。MAPK信号途径在生物中广泛存在,为了明确mapk双链RNA的转基因黄瓜植株是否对根际土壤细菌具有影响,本研究采用细菌16S rRNA基因克隆文库方法对种植转基因黄瓜根际土壤和非转基因黄瓜根际土壤的细菌群落类群多样性进行分析,以期为转基因植物对土壤微生物的风险评价提供参考。
1 材料与方法
1.1 试验地点及材料
试验田位于中国农业科学院蔬菜花卉研究所温室基地。供试黄瓜品种为新泰密刺,转基因黄瓜材料转入MiMPK1基因片段dsRNA表达载体,非转基因黄瓜为普通新泰密刺。试验田连续种植转基因黄瓜和非转基因黄瓜3a,种植方式为一年两茬轮作。黄瓜正常管理施肥。
1.2 土壤取样方法
土壤取样时间为2012年6月14日和下半年9月14旬,为黄瓜结果期。两次土壤样品混合在一起。土壤取样采用五点采样法,用土壤采样器采集10株黄瓜根系5cm范围内黄瓜根际土壤,采集深度为0—15 cm的土样,混合后用密封袋带回,土样用2 mm孔径筛网过筛,去除颗粒硬物和黄瓜根系。
1.3 土壤微生物DNA提取
采用土壤总DNA提取试剂盒(美国MOBIO公司)提取土壤微生物DNA。按照试剂盒说明书,分别提取转基因黄瓜土壤和非转基因黄瓜土壤DNA,每个处理分别提取5份土壤微生物DNA,分别混合,经电泳和分光光度计检测DNA质量,-20℃保存。
1.4 16S rRNA基因克隆文库构建
采用通用引物27F(5'-AGAGTTTGATCCTGGCTCAG-3')和1492R(5'-TACTTGTTACG ACTT-3')[7]从土壤微生物DNA中扩增16S rRNA基因。PCR反应体系20 μL,具体如下:1 μL DNA模板 (100ng),0.5μmol/L引物,0.2 mmol dNTPs,5 units of EasyTaq DNA聚合酶、1×反应缓冲液,13.7μL dd水。为了减少扩增偏嗜性,采用梯度PCR程序进行扩增,且重复5次,将PCR产物混合。PCR反应程序如下:95℃ 4 min;95℃ 30 s,52℃至58℃ 30 s,72℃ 2 min,30 个循环;72℃ 10 min。
将PCR产物经电泳检测,割胶纯化,连接入PGM-T载体(天根生化科技有限公司),转化Top10感受态细胞(北京全式金生物技术有限公司),在涂有IPTG和X-gal的安苄青霉素LB平板上进行蓝白斑筛选,随机200个挑取白色克隆构成16S rRNA基因文库,将克隆培养于LB液体培养基(安苄青霉素50 μg/mL)中振荡培养8 h,菌液PCR进行阳性克隆鉴定,采用引物为T7和SP6。随机挑取150个阳性克隆送诺塞基因公司测序。
1.5 数据分析
用Chimera Check程序将所得16S rRNA基因序列在RDP(Ribosomal Database Project)数据库进行嵌合体检验,去除嵌合体序列。以97%为划定阈值,用DOTUR软件包对16S序列划分操作分类单元(OTU),并构建稀缺性曲线[8]。
多样性指数依据下列公式进行计算[9]:
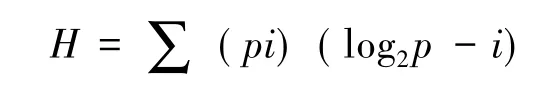
式中,pi代表每个物种样本数量占总样本数量的比例。
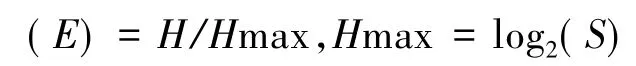
均匀度: 式中,丰富度(S)是样本中物种的数量,这里等同OTU的数量。
2 结果与分析
2.1 转基因黄瓜土壤和非转基因黄瓜土壤细菌类群
非转基因黄瓜根际土壤细菌16S rRNA克隆文库测序150个克隆,以97%划分OTU,获得124个OTU。转基因黄瓜土壤细菌16S rRNA克隆文库测序150个克隆,获得122个OTU。两个文库共同包含的克隆共有115个,这表明两个文库细菌类群比较一致。
转基因黄瓜根际土壤细菌分为 13个类群(图 1A):Acidobacteria、Actinobacteria、Armatimonadetes、Bacteroidetes、candidate division BRC1、Chloroflexi、Firmicutes、Gemmatimonadetes、Nitrospira、Planctomycetes、Proteobacteria、Verrucomicrobia、unclassified_Bacteria。Proteobacteria 为 优 势 种 群,占 24.1%;其 次 为Bacteroidetes,占19.0%,再次为Chloroflexi,占16.8%;Acidobacteria占11.7%,其他种群比例相对较低。这表明转基因黄瓜土壤中,优势细菌类群为Proteobacteria、Bacteroidetes、Chloroflexi和Acidobacteria,而其他细菌类群为非优势菌群。
非转基因黄瓜根际土壤细菌分为14个类群(图1B):Acidobacteria、Actinobacteria、Armatimonadetes、Bacteroidetes、 BRC1、 Chloroflexi、 Cyanobacteria/Chloroplast、 Firmicutes、 Gemmatimonadetes、 Nitrospira、Planctomycetes、Proteobacteria、Verrucomicrobia、unclassified_Bacteria。其中 Proteobacteria 是优势菌群,占22.8%;其次为Bacteroidetes,占19.9%;Chloroflexi占17.6%,Acidobacteria占10.3%,其他类群数量相对较少。这表明在非转基因黄瓜土壤中,优势细菌类群为Proteobacteria、Bacteroidetes、Chloroflexi和Acidobacteria,而其他细菌类群为非优势菌群。
2.2 转基因黄瓜土壤和非转基因黄瓜土壤细菌多样性比较
转基因黄瓜土壤和非转基因黄瓜土壤细菌类群差别不大,13个类群细菌为两种土壤所共有,转基因黄瓜土壤缺少Cyanobacteria/Chloroplast细菌类群,而该类群细菌在非转基因黄瓜土壤比例很低,仅仅为0.7%。在优势细菌类群上,转基因黄瓜土壤和非转基因黄瓜土壤基本一致,Proteobacteria、Bacteroidetes、Chloroflexi和Acidobacteria均为优势细菌类群,但在比例上存在细微差别。转基因黄瓜土壤中,Proteobacteria和Acidobacteria的比例略高于非转基因黄瓜土壤,而Bacteroidetes和Chloroflexi的比例略低于非转基因黄瓜土壤。总体来看,两种土壤细菌类群无显著差异。
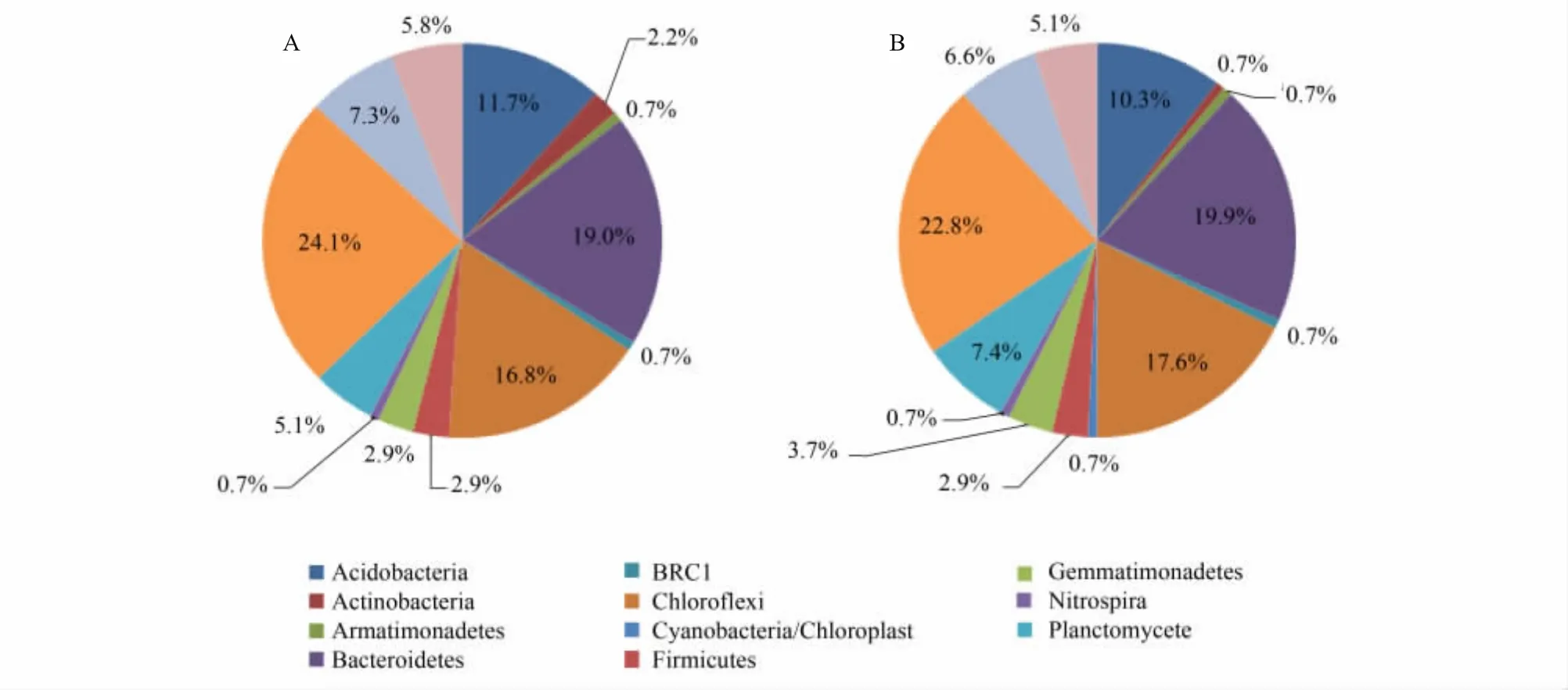
图1 文库各门细菌比例Fig.1 Proportions of bacteria in two libraries at phylum
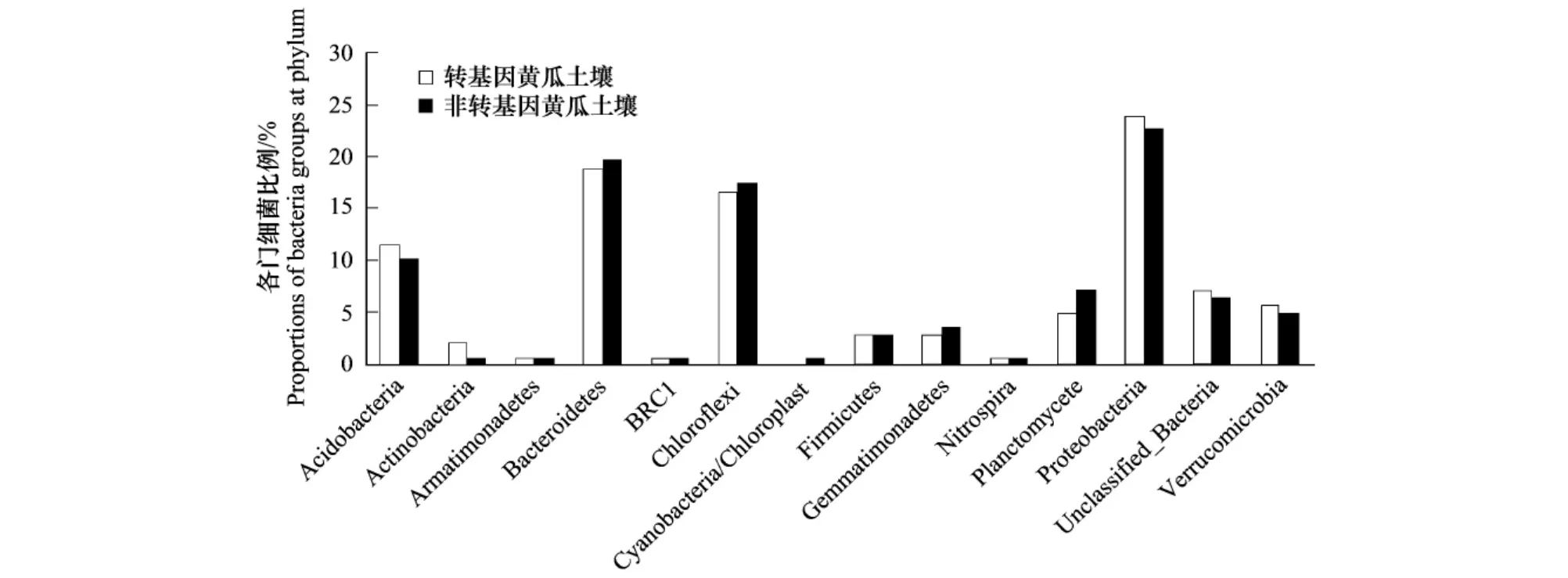
图2 转基因黄瓜土壤和非转基因黄瓜土壤细菌类群比较Fig.2 Comparison of bacteria groups between transgenic cucumber soil and non-transgenic cucumber soil
在纲分类水平上,两种土壤的优势细菌类群差异也不大(图3)。在Acidobacteria门细菌中,两种土壤包含6 个纲的细菌,Acidobacteria_Gp3、Acidobacteria_Gp4、Acidobacteria_Gp5、Acidobacteria_Gp6、Acidobacteria_Gp25和 unclassified Acidobacteria,其中 Acidobacteria_Gp6为优势菌群。在 Bacteroidetes门中,包含Bacteroidetes_incertae_sedis、Flavobacteria、Sphingobacteria 和unclassified Bacteroidetes,其中Sphingobacteria 纲细菌数量最多。在Chloroflexi门细菌中,两种土壤的unclassified Chloroflexi细菌最多,这表明土壤中存在大量的细菌新类群。在Proteobacteria门细菌类群中,两种土壤细菌分属于Alphaproteobacteria、Betaproteobacteria、Deltaproteobacteria和Gammaproteobacteria,其中Betaproteobacteria纲的细菌数量最多,Deltaproteobacteria细菌数最少。
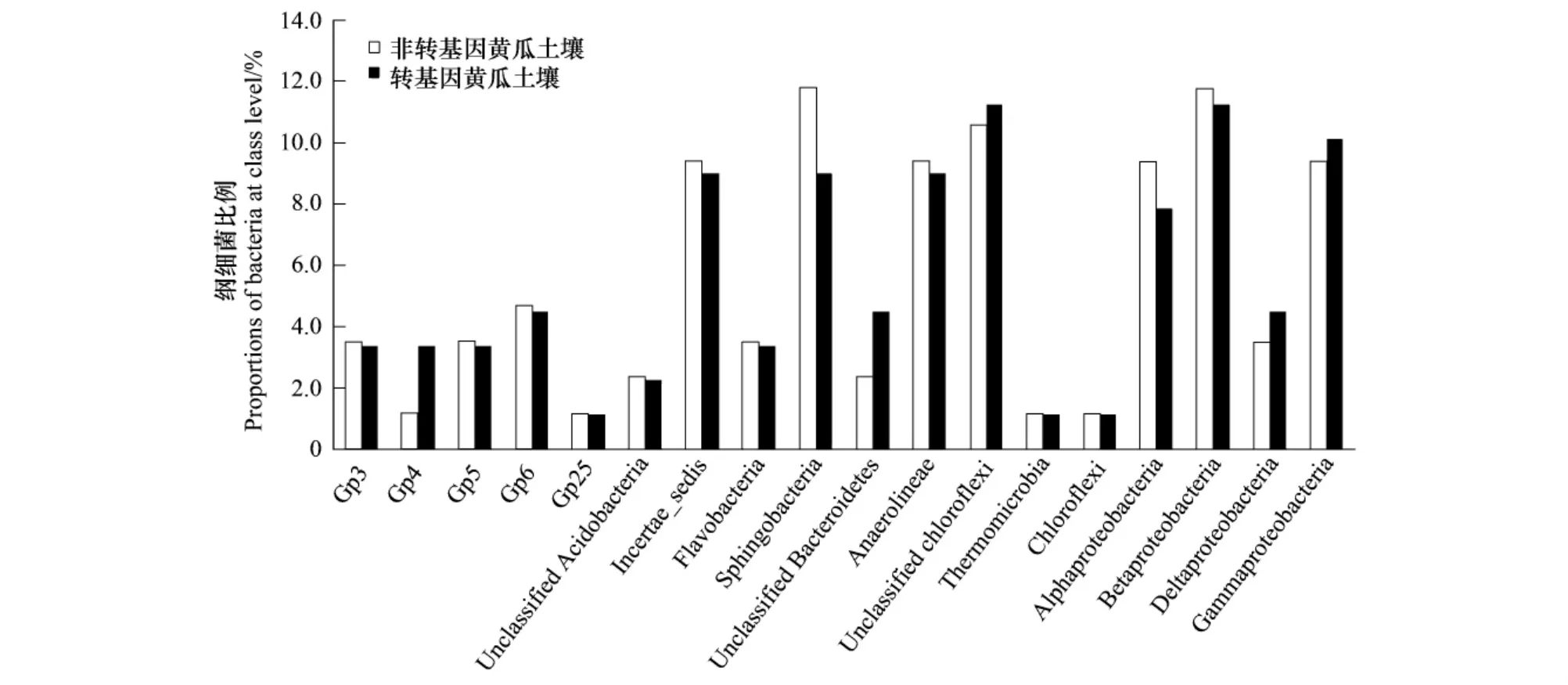
图3 纲分类水平上两种土壤优势细菌类群比较Fig.3 Comparison of the dominant bacteria groups in two libraries at class level
从多样性指数来看(表1),转基因黄瓜土壤和非转基因黄瓜土壤的细菌群落差异不大。Shannon评价一个群落物种多样性,其值越高,表明群落的物种多样性越高。两种土壤的Shannon指数均为4.6,这表明两种土壤细菌多样性无显著差别。Simpson也是描述群落物种多样性的指数,两种土壤细菌的Simpson指数差异不大,也反映出两种土壤细菌的多样性差异不大。Chao是描述群落丰富度的指数,其值越高表明群落物种的丰富度越高。非转基因黄瓜土壤细菌群落的Chao高于转基因黄瓜土壤,这表明非转基因黄瓜土壤细菌的丰度略高于转基因土壤。
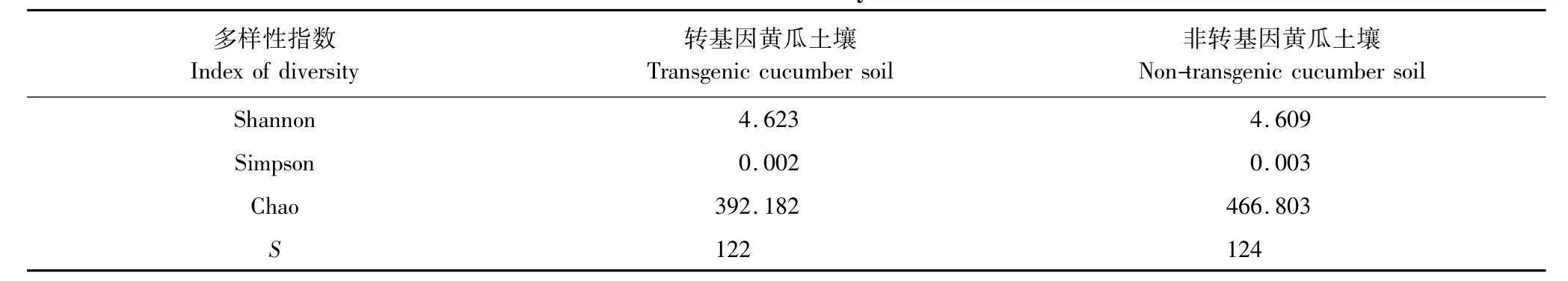
表1 转基因黄瓜土壤和非转基因黄瓜土壤细菌多样性指数Table 1 The index of diversity of two libraries
3 讨论
随着基因工程技术的不断发展,利用RNAi技术培育抗病新品种更是有效的植物病害防治方法。Yadav等将编码根结线虫特异联接因子和整合酶的管家基因的双链RNA表达载体转入烟草,表达dsRNA的转基因烟草对根结线虫具有很高抗性,证实利用RNAi方法控制植物寄生害虫是一个十分有效的策略[10]。本研究采用的转基因黄瓜材料能够表达mapk的双链RNA,能够靶向的抑制根结线虫mapk基因的表达,有效控制根结线虫的侵染,具有广阔的应用前景[6]。然而,针对该转基因植物的生态安全性还未进行研究。
土壤细菌作为土壤中最丰富的微生物,其种群多样性和功能多样性对土壤生态系统的稳定具有重要意义。因此,评价转基因作物的安全性必须考虑转基因产物对土壤细菌的影响。目前,针对转Bt和抗除草剂基因的转基因作物安全性进行了大量研究[11-12],有些研究表明转基因作物对土壤微生物群落产生了显著影响[13],有些研究得出相反的结论[14]。转Bt植物在生长过程中产生的Bt蛋白会以根系分泌物的形式进人到土壤中,并牢牢结合在土壤颗粒中难以降解,最终对土壤微生物造成影响。迄今为止,针对双链RNA在土壤中的残留动态情况和双链RNA转基因植物的生态安全性研究未见报道。
从本研究结果来看,转mapk双链RNA黄瓜并未对根际土壤细菌群落产生明显的影响。造成这种情况,可能基于4个方面的原因:(1)外源的双链RNA表达特异性,它们只能在植物的细胞内表达[15]。未有研究表明双链RNA能够分泌到植物体表。(2)双链RNA发挥作用需要依赖真核生物的Dicer酶[16],而细菌不具有Dicer酶,双链RNA无法对细菌起作用。(3)双链RNA只针对同源性较高的靶基因起RNA干扰作用[16],细菌不具有与mapk同源的基因。(4)土壤环境样品中RNA酶含量丰富,RNA易被降解[17]。即使分泌到植物体外的双链RNA也会很快被土壤RNA酶降解。由于转基因黄瓜的mapk双链RNA特性和土壤环境的影响,使得转基因黄瓜对根际土壤细菌群落的影响较小,而温室的特殊环境对土壤细菌的影响可能更大。
本研究中土壤取自黄瓜连作温室,温室环境特殊,如高温高湿、高有机质含量、高复种指数,这会对土壤微生物群落产生一定的影响。黄瓜连作田土壤微生态环境产生显著改变,导致微生物种群平衡遭受破坏,根际微生态平衡的失调[18],且黄瓜根际细菌种类和数量变化与黄瓜根分泌物密切相关[19]。本研究发现,转基因黄瓜土壤和非转基因黄瓜土壤中Proteobacteria为优势菌群。这与他人的研究结果基本一致[20-22]。另外,在本研究两种土壤中,Bacteroidetes和Acidobacteria的比例也相对较高,也与他人研究结果基本符合[20-22]。此外,Chloroflexi的比例也较高。Janssen通过16S rRNA基因克隆技术发现Chloroflexi在土壤中数量丰富,由于该类细菌生长缓慢,很难用培养法分离获得[23-24]。Chloroflexi是海绵中大量存在,与海绵形成共生体,对于海绵来说具有重要的生态功能[25]。在土壤生态系统中,该类细菌的生态功能还有待深入研究。
总体来看,双链RNA转基因黄瓜并未对土壤细菌群落产生明显影响,但后续的长期观察以及对根际土壤真核生物群落的影响还需深入研究。
[1]Stotzky G.Persistence and biological activity in soil of insecticidal proteins from Bacillus thuringiensis and of bacterial DNA bound on clays and humic acids.Journal of Environmental Quality,2000,29(3):691-705.
[2]Philip J.Dale B C,Eliana M G F.Potential for the environmental impact of transgenic crops.Nature Biotechnology,2002,20:567-574.
[3]Devare M H,Jones C M,Thies J E.Effect of Cry3Bb Transgenic Corn and Tefluthrin on the Soil Microbial Community.Journal of Environmental Quality,2004,33(3):837-843.
[4]Susanne B,Christoph C T.Field studies on the environmental fate of the Cry1Ab Bt-toxin produced by transgenic maize(MON810)and its effect on bacterial communities in the maize rhizosphere.Molecular Ecology,2005,14(8):2539-2551.
[5]Yang Y H.Advances on the effects of genetically modified crops on soil microbial community and main countermeasures of their approaches.Journal of Agricultural Biotechnology,2011,19(1):1-8.
[6]Chen G H,Xiao L,Zhang S Q,Xie B Y.Analysis of silencing MAPK by RNA interference of root-knot nematode:Acta Phytopathologica Sinica,2008,38(5):509-513.
[7]Lane,D.J.16S/23S rRNA sequencing.In:Nucleic acid techniques in bacterial systematics.Stackebrandt,E.,and Goodfellow,M.,eds.,John Wiley and Sons,New York,NY,1991,pp.115-175.
[8]Schloss P D,Handelsman J.Introducing DOTUR,a computer program for defining operational taxonomic units and estimating species richness.Applied and Environmental Microbiology,2005,71(3):1501-1506.
[9]Rani A,Porwal S,Sharma R,Kapley A,Purohit HJ,Kalia V C.Assessing microbial diversity by culture-dependent and independent approaches for efficient functioning of effluent treatment plants.Bioresource Technology,2008,99:7098-7107.
[10]Yadav BC,Veluthambi K,Subramaniam K.Host-generated double stranded RNA induces RNAi in plant-parasitic nematodes and protects the host from infection.Molecular& Biochemical Parasitology,2006,148:219-222.
[11]Lu X M,Zhu L Q,Cao Y P.Recent Advances of Bt Crops and Its Safety Assessment.Journal of Shanghai Jiaotong University(Agricultural Science),2006,24(2):214-220.
[12]Means N E,Kremer R J,Ramsier C.Effects of glyphosate and foliar amendments on activity of microorganisms in the soybean rhizosphere.Journal of Environmental Science and Health,Part B:Pesticides,Food Contaminants,and Agricultural Wastes,2007,42(2):125-132.
[13]Baumgarte S,Tebbe C C.Field studies on the environmental fate of the Cryl Ab Bt-toxin produced by transgenic maize(MON8 10)and its effect on bacterial communities in the maize rhizosphere.Molecular Ecology,2005,14(8):2539-2551.
[14]Shen R F,Cai H,Gong W H.Transgenic Bt cotton has no apparent effect on enzymatic activities or functional diversity of microbial communities inrhizosphere soil.Plant and Soil,2006,285(1/2):149-159.
[15]Urwin P E,Catherine J L,Howard J A.Ingestion of Double-Stranded RNA by Preparasitic Juvenile Cyst Nematodes Leads to RNA Interference.Molecular Plant-Microbe Interactions,2002,15(8):747-752.
[16]Phillip D Z,Thomas T,Phillip A S,David P B.RNAi:double-stranded RNA directs the ATP-dependent cleavage of mRNA at 21 to 23 nucleotide intervals.Cell,2000,101:25-33.
[17]Tom A M,R.Elizabeth S,Penny R H.The detection of Gram-negative bacterial mRNA from soil by RT-PCR.FEMS Microbiology Letters,1998,164(2):369-373.
[18]Hu Y S,Liy Y F,Wu K,Dou H J,Jia X C.Variation of Microbial Community Structure in Relation to Successive Cucumber Cropping Soil.Chinese Journal of Soil Science,2006,37(1):126-129.
[19]Hu Y S,Wu K,Li C X,Jia XC.Effect of Continuous Cropping of Cucumber on Soil Microbial Population Ⅱ — Variation Analysis Based on DGGE Approach.Scientia Agricultura Sinica,2007,40(10):2267-2273.
[20]Liu W Q,Mao Z C,Yang Y H,Xie B Y.Microbial Community Structure in Greenhouse Field Soil Infested with Root-Knot Nematodes.Chinese Journal of Biological Control,2008,24(4):318-324.
[21]Liu W Q,Mao Z C,Yang Y H,Xie B Y.Analysis of soil bacterial diversity by Using the 16S rRNA gene Library.Acta Microbiologica Sinica,2008,48(10):1344-1350.
[22]Schloss PD,Handelsman J.Status of the microbial census.Microbiology and molecular biology Review,2004,68(4):686-691.
[23]Janssen P H.Identifying the dominant soil bacterial taxa in libraries of 16S rRNA and 16S rRNA genes.Applied and Environmental Microbiology,2006,72:1719-1728.
[24]Kathryn E R,Davis,P S,Peter H J.Acidobacteria,Rubrobacteridae and Chloroflexi are abundant among very slow-growing and mini-colonyforming soil bacteria.Environmental Microbiology,2011,13(3),798-805.
[25]Susanne S,Peter D,Faris B,Michael W,Michael W T.Chloroflexi bacteria are more diverse,abundant,and similar in high than in low microbial abundance sponges.FEMS Microbiol Ecol,2011,78:497-510.
参考文献:
[5]杨永华.转基因作物对土壤微生物群落的影响及主要研究策略.农业生物技术学报,2011,l9(l):1-8.
[6]陈国华,肖罗,张双庆,等.南方根结线虫促分裂原活化蛋白激酶(MAPK)基因的RNAi效应分析.植物病理学报,2008,38(5):509-513.
[11]陆小毛,朱路青,曹越平.转Bl基因作物及其安全性研究.上海交通大学学报(农业科学版),2006,24(2):214-220.
[18]胡元森,刘亚峰,吴坤,窦会娟,贾新成.黄瓜连作土壤微生物区系变化研究.土壤通报,2006,37(1):126-129.
[19]胡元森,吴坤,李翠香,贾新成.黄瓜连作对土壤微生物区系影响 Ⅱ——基于DGGE方法对微生物种群的变化分析.中国农业科学,2007,40(10):2267-2273.
[20]刘玮琦,茹振川,杨宇红,谢丙炎.保护地根结线虫发生地土壤微生物群落多样性的研究.中国生物防治,2008,24(4):318-324.
[21]刘玮琦,茆振川,杨宇红,谢丙炎.应用16S rRNA基因文库技术分析土壤细菌群落的多样性.微生物学报,2008,48(10):1344-1350.

