植物MYB蛋白与其靶定DNA结合位点之间的相互作用
李 曼,赵 芊,刘 方,李 浩,宋水山
(1.河北工业大学化工学院,天津300130;2.河北省科学院生物研究所,石家庄050081;3.河北省主要农作物病害微生物控制工程技术研究中心,石家庄050081)
在植物的生长发育中,由于细胞内基因的表达存在着时间和空间的差异从而导致各细胞间出现分化,出现这一现象的主要原因就是转录因子在转录水平上的调控作用。转录因子通常具有模块化结构,即由DNA结合域和转录调节域构成。根据DNA结合功能域的相似性,转录调节因子可以分为家族或超家族,其中MYB转录因子是非常重要的一类,也是植物转录因子中最大的超家族之一[1]。随着研究的深入,在植物中发现MYB转录因子的数量急剧增加,在单一植物中发现的MYB家族成员达到100~200个,并且发现MYB蛋白调控植物新陈代谢以及细胞核发育过程[2]。目前关于动物MYB蛋白和它们相应的DNA结合位点相互作用已经有了深入的了解。但植物中MYB-DNA的相互作用还知之甚少。本文将概述植物MYB蛋白与其靶DNA相互作用的研究进展,并阐述研究植物MYB蛋白与其靶DNA相互作用的新技术。
1 植物MYB蛋白的特性
MYB蛋白首次发现于鸟类成髓细胞血症病毒AMV中,其名称即来源于 AMV病毒的致瘤因子v-MYB[3]。MYB超家族成员具有一个共同特征,即都具有一个高度保守的DNA结合结构域,称为 MYB结构域,该结构域通常由3个包含50~53个氨基酸残基的重复序列(R1、R2、R3)构成(图1),每个重复序列含有多个高度保守的色氨酸残基,由此形成规则的疏水核心[4]。重复序列形成螺旋-转角-螺旋的结构,有利于与DNA的结合。目前发现有些MYB蛋白含有至少4个这样的重复结构。MYB结构域通常位于MYB蛋白的N端,最近也有发现其存在于蛋白质的C端[5-6]。MYB蛋白的C端通常包含一个反式激活结构域和一个负向调控结构域,相较于MYB结构域,C端序列具有非常大的可变性,用以区分不同的MYB蛋白。MYB蛋白的这种结构特征,赋予了MYB超家族成员在结构和功能上的多样性[7]。

图1 MYB转录因子功能域结构示意图Fig 1 Functional domain map of MYB transcription factors structure
植物中存在着大量的MYB超家族蛋白。在双子叶模式植物拟南芥基因组中发现的超过1600个序列特异的转录因子中,大约有10%属于MYB超家族成员[8]。不同于动物,在植物中,具有2个重复序列(R2R3)的MYB蛋白占据主导地位。拟南芥中3重复序列(R1R2R3)的MYB蛋白有5个,而2重复序列(R2R3)的MYB蛋白有126个。单子叶模式植物水稻中经预测有超过110个R2R3-MYB[9]。此外,单重复序列MYB蛋白在植物中也越来越多的被鉴定出来。植物R2R3-MYB蛋白参与调控多种生物学过程,包括初级代谢和次级代谢、调控细胞命运和特性、调控植物生长发育以及植物对生物胁迫和非生物胁迫的响应[10]。
2 植物MYB蛋白的DNA结合位点
植物的R1R2R3-MYB蛋白,例如烟草的MYBA1,MYBA2和MYBB蛋白,通过调控细胞周期蛋白B及其它细胞周期蛋白基因的表达,在细胞周期的G2/M期起重要作用[11]。通过酵母单杂交技术,发现MtMYBA1,NtMYBA2和NtMYBB结合到序列AACGG上。这个共有序列已经在烟草中被证实是有丝分裂M期特异激活子(MSA)元件。烟草中的另外两个R1R2R3结构MYB蛋白NtMYBC1和NtMYBC2经生物信息学预测也是结合到MSA元件上,但这需要进一步实验加以验证。
目前对于植物R2R3-MYB蛋白的DNA结合位点的描述相对来说还比较少,这类蛋白的靶DNA序列是在进行玉米P蛋白的研究中首先被发现的。P蛋白是与类黄酮的生物合成有关的R2R3-MYB蛋白。通过结合位点的选择性实验和电泳迁移率变化分析(EMSAs),显示P蛋白结合的序列为ACC(A/T)ACC(A/C/T)。
MYB蛋白序列来自拟南芥信息资源网站 (TAIR;http://Arabidopsis.org)和美国国家生物技术信息中心(NCBI Entrez;http://www.ncvi.nlm.nih.gov/sites/entrez)。这个系统发育树包含9个三重复结构域的MYB蛋白(R1R2R3-MYB proteins),50个两重复结构域的MYB蛋白(R2R3-MYB proteins)和28个单结构域的MYB蛋白(R1-MYB proteins)。利用快速傅里叶转换的多重对比进行完整的氨基酸序列的对齐。通过运用分子进化遗传分析软件4(MEGA 4)建立遗传树。基于DNA序列的不同,把MYB蛋白标注上不同的颜色,红色、蓝色、绿色、橙色、紫色和灰色。MYB蛋白分别结合到序列 CNGTT(A/G)、ACC(A/T)A(A/C)、TTAGGG、AAAATATCT、GATA和TATCCA上。黑色表示MYB蛋白没有结合到任一个序列上。
许多R2R3-MYB转录因子可以识别富含A、C碱基的DNA序列。一些R2R3-MYB蛋白结合到这些结合位点处,作为转录激活因子发挥作用,另一R2R3MYB蛋白则作为转录抑制因子发挥功能[12-14]。详细的DNA结合位点信息可以在一些数据库如拟南芥基因管理信息数据库(AGRIS)(http://arabidopsis.med.ohio-state.edu/)和转录因子数据库 (TRANSFAC)(http://www.gene-regulation.com/pub/databases.html)中找到。这些数据库中包含许多文献中报道的MYB蛋白DNA结合位点,并且大多数经过了实验的验证(图2)。植物MYB蛋白DNA结合位点一般位于转录起始位点的上游大约500 bp处。和具有两个或三个重复结构的MYB蛋白相比,单独重复结构的MYB蛋白主要的结合在端粒序列TTAGGG,并显示出相似的端粒一致性。单独结合域的MYB蛋白涉及到端粒结合和生物钟调节。羧基端和氨基端的单独结构域的MYB蛋白结合在端粒重复序列TTTAGGG的双链 DNA 上,AtTRB1、AtTRB2、AtTRB3结合在包含至少两个TTTAGGG序列重复的端粒DNA上。可以发现,在MYB结构域的初级序列方面和同源DNA目标结合位点上,这些单独结构域的 MYB蛋白与R2R3-MYB蛋白和R1R2R3-MYB蛋白有所不同。
3 植物MYB蛋白DNA结合的特性
3.1 MYB蛋白DNA结合域和DNA结合特异性之间的关系
基于序列的相似性,不同物种的R2R3-MYB家族成员被划分为A、B和C三个系统发育大类[15]。氨基酸序列的高度相似性导致蛋白对DNA识别模式的相似性,从而使相同类型的MYB蛋白识别的靶DNA序列具有特异性。依据这些分类来分析DNA结合特异性。R2R3-MYB蛋白DNA结合位点的数据主要来源于体外测定的结果。结果显示出A类MYB蛋白的DNA靶序列为 MBSI序列(C(A/C/G/T)GTT(A/G)),B类MYB蛋白的DNA靶序列为MBSI和MBSII序列(G(G/T)T(A/T)GTT(A/G)),大多数C类MYB蛋白的DNA靶序列为MBSIIG序列((C/T)ACC(A/T)A(A/C)C)。例如,C组成员AtMYB6和AtMYB7具有90%的氨基酸序列一致性[16]。AtMYB6和AtMYB7两个都结合MBS类型IIG序列。
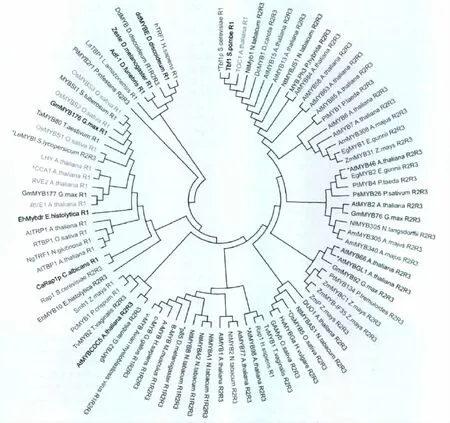
图2 采用邻接法方法对87个MYB超家族成员进行系统发生分类Fig 2 Phylogenetic relationships and subgroup designations for 87 MYB superfamily members using the neighbour-joining method
具有相似结构的MYB超家族成员并不总是有类似的DNA结合位点。尽管Romero等已经阐述过MYB蛋白结构和DNA结合特异性之间的关联,但是有一些MYB家族的成员并不符合他的预想[15]。例如,MYB家族的C组成员一般更倾向于结合MBSIIG序列,但是有2个C类MYB蛋白AtMYB2和AtMYBGL1,以不同的模式结合 DNA。AtMYB2结合 MBSI的序列,而 AtMYBGL1只能结合MBSII序列[17]。这些例子说明即使在结构和功能方面都相似的MYB蛋白也可能结合不同的DNA结合位点。因此针对每个MYB蛋白都进行DNA结合位点实验至关重要。因为很难只是根据同源性来预测一个MYB蛋白的DNA结合位点。
3.2 植物MYB蛋白与DNA结合的本质
迄今为止,由于任何一个多重复结构域的MYB蛋白的晶体结构还未产生,所以关于一些植物MYB蛋白和目的DNA相互作用特性的研究是基于c-MYB蛋白与DNA结合的模型建立起来的。例如,PAP1/AtMYB75就是通过已知的c-MYB的结构数据模拟出其R2R3结构域[18]。在多个MYB蛋白质中找到了一段保守的氨基酸信号([DE]Lx2[RK]x3Lx6Lx3R),用来预测拟南芥蛋白质新的MYB/BHLH互作。通过预测的PAP1/AtMYB75 3D模型的分析显示,该保守基序的氨基酸暴露于R3重复结构的α-螺旋1和2的表面,形成疏水和带电残基模式[19]。这些表满暴露出来的氨基酸被认为用来稳固蛋白质和蛋白质之间的相互作用。这个模型通过突变分析实验得到验证[19]。
大多数的植物MYB蛋白在识别目标位点的能力上显示出很重要的内在多样性。例如,矮牵牛花蛋白MYB.Ph3可以以相同的亲和力结合到MBSI和MBSII两个位点上,并引起靶点DNA相似的弯曲和变形[20]。而其它植物MYB蛋白与这两个DNA结合位点的亲和力存在很大的不同,通常MYB蛋白只能结合其中一种DNA结合位点。
由于植物中存在大量的MYB蛋白,对应于这些蛋白的下游DNA靶标数量也十分庞大。MYB蛋白参与调控的生物学过程也很广泛,这决定了植物MYB蛋白与DNA相互作用的复杂性。至今MYB蛋白与DNA互作机制仍然是世界范围内的一大难题,还需要对此进行更广泛深入的研究。
4 植物MYB蛋白和DNA相互作用研究展望
4.1 体外实验确定MYB蛋白和靶标DNA序列
通过快速和高通量的方法在体外筛选MYB蛋白的DNA结合序列式来分析植物MYB蛋白与其靶标DNA相互作用所必须的。转录因子,包括MYB蛋白,能够与多个目标序列相互作用并启动转录。利用重组的MYB转录因子DNA结合结构域从随机DNA序列文库中筛选靶标DNA的方法已经很好的建立起来。这些方法包括CASTing(cyclic amplification and selection of targets)和SELEX(systematic evolution of ligands by exponential enrichment)[21]。通过这些方法鉴定出了很多MYB转录因子的靶标DNA序列,并且这些方法在一定程度上实现了高通量筛选的目的。基于芯片的技术,如蛋白质结合芯片已经被发展用来鉴定转录因子的序列特异性[22]。通过这一技术鉴定的结合位点序列与通过CHIP体内实验鉴定的转录因子结合的DNA序列是相一致的[23]。
目前有两种蛋白质结合芯片类型:双链DNA芯片和转录因子芯片。在大约240000个寡核苷酸中,双链DNA芯片包含所有的11 bp长度(大约4.2百万个)的序列[24]。来源于转录因子的重组蛋白和双链DNA芯片杂交并且被高浓度的盐类缓冲液冲洗。这个技术仅仅在一级杂交反应中就能精确的检测到所有被转录因子识别的DNA结合位点。
基于蛋白芯片,转录因子DNA结合位点需要捕获大量多重的转录因子并且发现它们的结合位点。采用随机的寡核苷酸文库筛选转录因子,之后用不同浓度的盐离子溶液洗脱。这两种技术都是在短时间内鉴别体外DNA结合蛋白位点高速有效的方法。然而,在这些实验当中有一些局限性。一方面低浓度的盐离子溶液会把结合力比较弱的蛋白-DNA结合物洗脱下去,造成结果有所偏差。另一方面,由于一部分蛋白和芯片杂交使得全部的蛋白并没有结合到优先的DNA上。
4.2 发现植物MYB蛋白目标的新方法和体外分析
重要的是,MYB蛋白在体外的DNA结合位点可能和在体内的结合位点有所不同[25]。这些不同是因为体内存在蛋白-蛋白之间的相互作用和翻译后修饰改变了DNA结合特异性,而且体外重组的MYB蛋白DNA结合结构域的构象和这些结构域在体内的天然构象不同。此外体内转录因子DNA结合位点的有效性还受到染色质内基因组DNA组装的调控。因此,采用在体内实验的方法精确地描述基因组中MYB-DNA的结合位点是必需的。
目前测定体内一个特定的MYB蛋白结合到一个特定的DNA序列的主要方法是基因瞬时表达系统和酵母单杂交系统中完成的。首先分别构建转录因子表达系统和包含假定DNA结合位点序列及报告基因的人工基因系统。将这两个系统导入生物体内,如植物或酵母菌中,通过在体内观察该转录因子的表达是否能够激活报告基因的表达来检测转录因子与假定DNA序列的结合情况。将拟南芥 R2R3-MYB转录因子 AtMYB11,AtMYB12和AtMYB111在拟南芥原生质体中瞬时表达,发现它们的功能类似于与之结构相似的玉米P蛋白,它们都具有相似的靶基因特异性,调控一系列黄酮类物质生物合成基因的表达[26]。而且这些体内被激活的基因的启动子区都存在MYB蛋白的识别元件。
染色质免疫沉淀技术(ChIP)结合全基因组芯片技术(ChIP-chip)或高通量信号测序技术(ChIP-seq)能够检测感兴趣蛋白质靶DNA的异常,在体内的新的靶标DNA序列,从而产生更具有生物学意义的结果[27]。ChIP-chip或ChIP-seq已经被证明是确定在染色质水平序列的特异性转录因子在体内DNA结合位点的有力工具,从而避免了前面提到的需要很多附加说明的技术方法。近年来,应用ChIP技术鉴定出了一些MYB蛋白在体内的DNA结合位点。在植物中,应用ChIP-chip技术鉴定出拟南芥两个R2R3-MYB蛋白的体内DNA结合位点,这两个 MYB蛋白为 FOURLIPS(FLP;AtMYB124)和AtMYB88,它们可以直接结合到细胞周期相关基因的启动子上[28]。ChIP-chip和ChIP-seq技术是目前确定一个转录因子在体内真正靶标位点最有效的方法,但是它需要有转录因子蛋白的抗体才能操作,这限制了它在MYB超家族研究中的应用。
5 结论
MYB类转录因子属于一大类植物转录因子,在植物转录调控和对非生物胁迫的应激反应中发挥重要作用。本实验室中,通过细菌群体感应信号分子AHLs处理野生型拟南芥,发现MYB44基因表达量有所上调,处理TDNA插入基因突变体atmyb44植株,MYB44下游的一些基因表达量下调,可以预测MYB44参与植物感应细菌群体感应信号(数据待发表)。
过去的几十年中我们对于特定序列的转录因子和它们靶目标DNA之间的分子间相互作用的理解已经取得不凡的进展,并且更加具体的了解了MYB蛋白和它们的DNA结合位点之间的关系。然而人们对植物R2R3-MYB蛋白质与其靶标DNA之间的相互作用的分子机制的理解还很不完整,只有某些特定植物中的少量的MYB蛋白与DNA的相互作用的特性在分子水平上得到描述。因此,严格的说在植物R2R3-MYB蛋白调控基因表达从而引起植物表型变化方面的研究还在初期阶段。随着更快速、精确和高通量研究蛋白-DNA间相互作用的新技术的出现,新的植物MYB蛋白与DNA相互作用的特性将会加速分析出来。基于此,在未来十年内有望在将蛋白超家族成员的分子信息转换成整个生物体的应答信息方面为我们提供更加深刻的理解。
[1]Chen Y H,Yang X Y,He K,et al.The MYB transcription factor superfamily of Arabidopsis:expression analysis and phylogenetic comparison with the rice MYB family[J].Plant Mol Biol,2006,60:107-124.
[2]Dubos C,Stracke R,Grotewold E,et al.MYB transcription factors in Arabidopsis[J].Trends Plant Sci,2010,15:573-581.
[3]陈 俊,王宗阳.植物MYB类转录因子研究进展[J].植物生理学与分子生物学学报,2002,28(2):81-88.
[4]王希庆,陈柏君,印莉萍.植物中的MYB转录因子[J].生物技术通报,2003,2(12):71-73.
[5]Stracke R,Werber M,Weisshaar B.The R2R3-MYB gene family in Arabidopsis thaliana[J].Curr Opin Plant Biol,2001,4:447-456.
[6]Linger B R,Price C M.Conservation of telomere protein complexes:shuffling through evolution[J].Crit Rev Biochem Mol Biol,2009,44:434-446.
[7]刘守梅,孙玉强,王慧中.植物MYB转录因子研究[J].杭州师范大学学报(自然科学版),2012,5(3):14-21.
[8]Dubos C,Stracke R,Grotewold E,et al.MYB transcription factors in Arabidopsis[J].Trends Plant Sci,2010,15:573-581.
[9]Sala A,Watson R.B-Myb protein in cellular proliferation,transcription control,and cancer:latest developments[J].Cell Physiol,1999,179:245-250.
[10]Jia L,Clegg M T,Jiang T.Evolutionary dynamics of the DNA-binding domains in putative R2R3 MYB genes identified from rice subspecies indica and japonica genomes[J].Plant Physiol,2004,134:575-585.
[11]Ito M,Iwase M,Kodama H,et al.A novel cis-acting element in promoters of plant B-type cyclin genes activates M phase-specific transcription[J].Plant Cell,1998,10:331-341.
[12]Patzlaff A,McInnis S,Courtenay A,et al.Characterisation of a pine MYB that regulates lignification[J].Plant J,2003,36:743-754.
[13]Yang S C,Sweetman J P,Amirsadeghi S,et al.Novel anther-specific myb genes from tobacco as putative regulators of phenylalanine ammonialyase expression[J].Plant Physiol,2001,126:1738-1753.
[14]Sugimoto K,Takeda S,Hirochika H.MYB-related transcription factor NtMYB2 induced by wounding and elicitors is a regulator of the tobacco retrotransposon Tto1 and defense-related genes[J].Plant Cell,2000,12:2511-2527.
[15]Romero I,Fuertes A,Benito M J,et al.More than 80R2R3 MYB regulatory genes in the genome of Arabidopsis thaliana [J].Plant,1998,14:273-284.
[16]Li S F,Parish R W.Isolation of two novel myb-like genes from Arabidopsis and studies on the DNA-binding properties of their products[J].Plant,1995,8:963-972.
[17]Urao T,Yamaguchishinozaki K,Urao S,et al.An Arabidopsis myb homolog is induced by dehydration stress and its gene product binds to the conserved MYB recognition sequence [J].Plant Cell,1993,5:1529-1539.
[18]Zimmermann I M,Heim M A,Weisshaar B,et al.Comprehensive identification of Arabidopsis thaliana MYB transcription factors interacting with R/B-like BHLH proteins[J].Plant,2004,40:22-34.
[19]Bergholtz S,Andersen T O,Andersson K B,et al.The highly conserved DNA-binding domains of A-,B-and c-Myb differwith respect to DNA-binding,phosphorylation and redox properties[J].Nucleic Acids Res,2001,29:3546-3556.
[20]Solano R,Fuertes A,Sanchez P L,et al.A single residue substitution causes a switch from the dual DNA binding specificity of plant transcription factor MYB.Ph3 to the animal c-MYB specificity[J].Biol Chem,1997,272:2889-2895.
[21]Patzlaff A,Newman L J,Dubos C,et al.Characterisation of PtMYB1,an R2R3 MYB from pine xylem[J].Plant Mol Biol,2003,53:597-608.
[22]Berger M F,Philippakis A A,Qureshi A M,et al.Compact,universal DNA microarrays to comprehensively determine transcriptionfactor binding site specificities[J].Biotechnol,2006,24:1429-1435.
[23]Badis G,Berger M F,Philippakis A A,et al.Diversity and complexity in DNA recognition by transcription factors[J].Science,2009,324:1720-1723.
[24]Godoy M,Franco-Zorrilla J,Prez-Prez M J,et al.Improved proteinbinding microarrays for the identification of DNA-binding specificities of transcription factors[J].Plant,2011,66:700-711.
[25]Verrijdt G,Haelens A,Claessens F.Selective DNA recognition by the androgen receptor as a mechanism for hormone-specific regulation of gene expression[J].Mol Genet Metab,2003,78:175-185.
[26]Stracke R,Ishihara H,Barsch G H A,et al.Differential regulation of closely related R2R3 MYB transcription factors controls flavonol accumulation in different parts of the Arabidopsis thaliana seedling[J].Plant,2007,50:660-677.
[27]Massie C E,Mills I G.ChIPping away at gene regulation[J].EMBO Rep,2008,9:337-343.
[28]Wang Q F,Lauring J,Schlissel M S.c-Myb binds to a sequence in the proximal region of the RAG-2 promoter and is essential for promoter activity in T-lineage cells[J].Mol Cell Biol,2000,20:9203-9211.

