肠道杆菌耐药性及其耐药相关Ⅰ类整合子可变区结构与进化
王 欢,包其郁,孙爱华,赵金方,葛玉梅,严 杰
(1.浙江省人民医院检验科,浙江杭州 310014;2.温州医学院检验医学院和生命科学学院,浙江温州 325035;3.浙江医学高等专科学校,浙江杭州 310053;4.浙江中医药大学附属第一医院检验科,浙江杭州 310006;5.浙江大学医学院病原生物学系,浙江杭州 310058)
近年来我国及东南亚地区临床耐药菌株、尤其是耐多种抗生素的多重耐药菌株的分离率逐年增高,其中大肠埃希菌、阴沟杆菌、不动杆菌等革兰阴性肠道所占比例较大[1-3]。上述细菌主要引发医院感染,包括呼吸道感染、败血症、泌尿系感染、继发性脑膜炎等[4]。
整合子(integron)是细菌基因组中一种介导耐药基因转移的转座元件,其功能与细菌耐药性产生与转移密切相关[5-7]。典型的整合子结构由保守区和可变区两部分组成,前者为整合酶编码基因所在区域活性,后者由串联排列的耐药基因盒组成,迄今发现的整合子相关耐药基因盒至少有数十种,所含耐药基因几乎覆盖了目前临床上使用的所有抗菌药物[8,9]。根据整合酶序列不同,可将整合子分为六型,其中以Ⅰ类整合子最为常见[10-11]。整合子不仅可通过位点特异性重组的方式捕获耐药基因,从而形成多种耐药基因的组合,同时还可介导耐药基因在染色体、质粒及转座子之间移动,导致耐药基因的水平播散[12-13]。本研究中,我们检测了临床标本中分离的大肠埃希菌、阴沟肠杆菌、鲍曼不动杆菌的耐药性,采用PCR及产物测序检测与分析了上述菌株Ⅰ类整合子可变区基因盒,同时以二氢叶酸还原酶基因为靶基因对Ⅰ类整合子系统发生、进化进行分类和比较。
1 材料与方法
1.1 临床菌株来源与鉴定 参照《全国临床检验操作规程》标准,采用法国bioMérieux公司VITEK60全自动细菌检测分析系统及其配套的细菌鉴定卡GPI,从温州医学院附属第一医院临床送检的体液(血液、腹水等)、分泌物(痰液、脓液、伤口分泌液等)标本中分离并鉴定了24株大肠埃希菌(Escherichia coli)、28株阴沟肠杆菌(Enterobacter cloacae)和24株鲍曼不动杆菌(Acinetobacter baumannii)。
1.2 药物敏感试验 采用K-B法并根据CLSI 2008标准判读结果。各类抗菌药物纸片如下:氨苄西林(AMP)、哌拉西林(PIP)、头孢替坦(CTT)、头孢曲松(CRO)、头孢他啶(CAZ)、丁胺卡那(AMI)、庆大霉素(GEN)、妥布霉素(TOB)、环丙沙星(CIP)、左旋氧氟沙星(LVX)、复方新诺明(SMZ)、泰能(TIE)和呋喃妥因(NIT)。质控菌株为大肠埃希菌ATCC25922株,购自卫生部临检中心。
1.3 细菌基因组DNA制备 采用细菌基因组DNA提取试剂盒(GENEray)提取各受试菌株基因组DNA,紫外分光光度法测定其浓度和纯度[14]。
1.4 PCR 以 GenBank中Ⅰ类整合子序列(accession No.:AM939644)为参照,采用Primer Premier 6.0软件在可变区两端保守序列中设计通用引物。引物委托上海Invitrogen公司合成。上游引物序列:5'-GGC ATC CAA GCA GCA AG-3',下游引物序列:5'-AAG CAG ACT TGA CCT GA-3'。采用高保真PCR试剂盒(TaKaRa)扩增各菌株中Ⅰ类整合子可变区片段。反应总体积 50μl,内含:2.5 mol/L dNTP、200 nmol/L 各引物、15 mol/L MgCl2、2.5U EXTaq DNA聚合酶、100 ng DNA模板和1×PCR缓冲液(pH 8.3)。PCR参数:95℃ 5 min;94℃1 min、55℃ 30 s、72℃ 1 min,30 个循环;72℃10 min。采用溴乙锭预染色的1.5%琼脂糖凝胶电泳检测PCR结果。
1.5 扩增产物测序及分析 采用PCR产物纯化试剂盒(BioColor)回收目的扩增片段,采用T-A克隆试剂盒(TaKaRa)将其克隆至pMD-19T质粒中,然后电转化入 E.coli DH5α(Invitrogen)并在LB培养液(Oxoid)中扩增,氨苄西林和蓝白斑双重筛选后委托上海Sangong公司测序[14]。采用BLAST软件对测序结果进行分析和比较。
1.6 进化树分析 以Ⅰ类整合子中最为常见的二氢叶酸还原酶基因为靶基因,先采用ClustalX1.8软件与Swissport库中的不同亚型二氢叶酸还原酶基因序列进行比对,然后采用MEGA系统进化分析软件中的NJ方法,构建Ⅰ类整合子中二氢叶酸还原酶基因的分子进化树并进行系统发生分析。
2 结果
2.1 细菌耐药率检测结果 24株大肠埃希菌、28株阴沟肠杆菌、24株鲍曼不动杆菌药敏试验结果显示:三种细菌对多种抗菌药物耐药,但鲍曼不动杆菌对大多数抗生素耐药率高于大肠埃希菌和阴沟肠杆菌,对哌拉西林(PIP)、头孢曲松(CRO)、头孢他啶(CAZ)及呋喃妥因(NIT)耐药率甚至高达100%,大肠埃希菌和阴沟肠杆菌对头孢曲松头孢替坦(CTT)、(CRO)和头孢他啶(CAZ)耐药率也高达41.6% ~62.5%(表1)。
2.2 Ⅰ类整合子的检出率 24株大肠埃希菌、28株阴沟肠杆菌、24株鲍曼不动杆菌Ⅰ类整合子检出率分别为 62.5%(15/24)、67.9%(19/28)和83.3%(20/24)。见表 2。
2.3 Ⅰ类整合子谱型 54株检出Ⅰ类整合子菌株(15株大肠埃希菌、19株阴沟肠杆菌和20株鲍曼不动杆菌)中,有44株呈单一阳性条带,多数为1.7 kb条带(25株),其余为1.5 kb(10株)、0.75 kb(5 株)和 1.0 kb(4 株)条带(图1);有5株同时检出2条带,分别有0.75/1.70 kb(3 株)、0.75/1.0 kb(1 株)和1.0/1.70 kb(1株)三种组合方式(图2)。
2.4 Ⅰ类整合子可变区基因盒分析结果 在11株大肠埃希菌、15株阴沟肠杆菌和15株鲍曼不动杆菌中有5种不同类型的基因盒:分别为 aac(6')、sad(3″)、aad(2″)、cat(4')和 dfr(含7、A13、15 三个亚型),aac(6')、sad(3″)和 dfr 15能以单基因方式存在,有4种双基因组合方式、1种三基因组合方式(表3)。aac(6')为(Aminoglycoside N(6')-acetyltransferase)基因,sad(3″)为链霉素腺苷酰转移酶(streptomycin 3″-adenylyltransferase)基因,aad(2″)是 (aminoglycoside nucleotidyltransferase)基因,cat(4')是(chloramphenicol acetyltransferase)基因,可分别介导细菌对氨基糖苷类抗生素及氯霉素耐药。dfr 7、dfr A13和 dfr 15为二氢叶酸还原酶(dihydrofolate reductase)基因可赋予细菌对甲氧苄啶的耐药性。然而,上述菌株Ⅰ类整合子中未发现β-内酰胺酶基因。

表1 大肠埃希菌、阴沟肠杆菌和鲍曼不动杆菌耐药率Table 1 Resistance rates of E.coli,E.cloacae and A.baumannii

表2 大肠埃希菌、阴沟肠杆菌和鲍曼不动杆菌I型整合子检出率Table 2 Positive rates of class-Ⅰ integron in E.coli,E.cloacae and A.baumannii
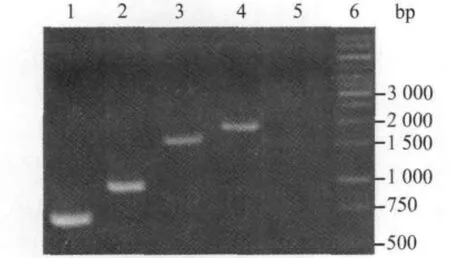
图1 Ⅰ类整合子单条带谱型Fig.1 Spectral patterns of class-Ⅰintegrons with single bands

图2 Ⅰ类整合子双条带谱型Fig.2 Spectral patterns of class-Ⅰ integrons with double bands
3.5 二氢叶酸还原酶基因系统发生分析结果
上述菌株Ⅰ类整合子所携带的二氢叶酸还原酶基因可归纳为4组:①重叠群9(Ctg9)二氢叶酸还原酶基因与15型二氢叶酸还原酶基因接近;②重叠群1和15(Ctg1和Ctg15)二氢叶酸还原酶基因与5型二氢叶酸还原酶基因接近;③重叠群14a和14b(Ctg14a和Ctg14b)二氢叶酸还原酶基因与7型二氢叶酸还原酶基因接近;④重叠群20和21(Ctg20和Ctg21)二氢叶酸还原酶基因与13型二氢叶酸还原酶基因接近(图3),提示上述菌株Ⅰ类整合子有4条不同的系统发生或进化途径。

表3 Ⅰ类整合子基因盒的构成及其分布Table 3 Composition and distribution of gene cassettes in class-Ⅰintegron

图3 Ⅰ类整合子中二氢叶酸还原酶基因系统发生树Fig.3 Phylogenetic tress of dihydrofolate reductase genes in class-Ⅰintegrons
3 讨论
大肠埃希菌、阴沟肠杆菌、鲍曼不动杆菌是临床常见的条件致病菌,且这些细菌普遍存在耐药或多重耐药现象[1,3,15-16]。与欧美国家或地区比较,亚洲地区各种临床分离菌株对抗生素耐药率普遍较高,其中大肠埃希菌对头孢曲松和氧氟沙星耐药率分别为为67.6%和 65.0%,鲍曼不动杆菌对哌拉西林耐药率高达70.5%,阴沟肠杆菌对头孢西丁的耐药率甚至 高 达 93.7%[1,4,17,18]。我 们的药物敏感试验结果显示,鲍曼不动杆菌耐药性最强,对临床常用的三代头孢类抗生素头孢曲松(CRO)、头孢他啶(CAZ)及青霉素类抗生素哌拉西林(PIP)耐药率为100%,对氨苄西林(AMP)耐药率也高达70.8%;阴沟肠杆菌对受试的3种三代头孢类抗生素(CTT、CRO、CAZ)、3 种氨基糖苷类抗生素(AMI、GEN、TOB)和2种喹诺酮类抗生素(CIP、LVX)耐药率高达 46.4% ~71.4%,明显高于大肠埃希菌(表1),表明临床上耐药菌株流行状况已十分严重。细菌的耐药机制十分复杂,仅基因突变和耐药质粒已无法完全解释细菌快速获得耐药性的现象。整合子可捕获耐药基因并通过整合酶将其整合后表达,使细菌产生耐药性,又能介导耐药基因转移,为深入研究细菌耐药及播散机制提供了新方向[9]。整合子分成两大群:可移动整合子和超级整合子[8]。可移动整合子又被分成五类(Ⅰ~Ⅴ),不同整合子类型中基因盒数目不尽相同,也非所有基因盒均可介导耐药性。可移动Ⅰ类整合子广泛地分布于寄生性和腐生性菌株中,在革兰阴性临床菌株中,Ⅰ类整合子更为常见且有较高的耐药基因携带率[10,13,19]。超级整合子基因盒中除耐药基因外,还包含许多参与环境适应的功能蛋白编码基因。我们的实验结果显示,大肠埃希菌、阴沟肠杆菌、鲍曼不动杆菌中Ⅰ类整合子总检出率高达71.1%(54/76),其中鲍曼不动杆菌Ⅰ类整合子检出率(83.3%)高于大肠埃希菌(62.5%)和阴沟肠杆菌(67.9%);各菌株携带的Ⅰ类整合子有高度异质性,表现为不同大小单条带或双条带组合多种谱型,提示这些菌株Ⅰ类整合子功能活跃。
大多数耐药基因盒由Ⅰ类整合子携带,包括β-内酰胺类(青霉素类、头孢菌素类)、喹诺酮类、大环内酯类和喹诺酮类及磺胺类抗性基因[8,9]。然而,我们在实验中仅发现 aac(6')、sad(3″)、aad(2″)、cat(4')、dfr(7、A13 和 15 亚型)等与氨基糖苷类、氯霉素和磺胺类耐药有关基因,未发现β-内酰胺酶基因,但受试的鲍曼不动杆菌对β-内酰胺类抗生素(AMP、PIP、CRO、CAZ、CTT)耐药率高达 54.2% ~100%,阴沟肠杆菌和大肠埃希菌对头孢菌素类抗生素(CRO、CAZ、CTT)耐药率也分别高达46.4% ~50%和41.6% ~62.5%,这提示上述菌株对 β-内酰胺类抗生素的耐药性可能涉及基因突变和耐药质粒等其他耐药机制,与Ⅰ类整合子无关。
在各种抗生素广泛使用的情况下,细菌的耐药性基因也在不断发生变化并逐渐发展出不同的基因型。例如,现已发现超广谱β内酰胺酶即有数百种不同的基因型,仅CTM-X就有近60个型别[21],介导磺胺类耐药的二氢叶酸还原酶基因也有近20个基因型[22]。本研究中I型整合子基因盒主要结构基因二氢叶酸还原酶基因系统发生的分析结果显示,所发现的二氢叶酸还原酶基因可分成4组,分别与15型、5型、7型和13型二氢叶酸还原酶基因接近,提示在药物选择性压力下,Ⅰ类整合子有4条不同的系统发生或进化途径,所携带的二氢叶酸还原酶基因呈现为多样性。
[1]LESTARI E S,SEVERIN J A,VERBRUGH H A.Antimicrobial resistance among pathogenic bacteria in Southeast Asia[J].Southeast Asian J Trop Med Public Health,2012,43(2):385-422.
[2]SUN A H,FAN X L,GU Yi,et al.Predominant porB1A and porB1B genotypes and correlation of gene mutations with drug resistance in Neisseria gonorrhoeae isolates in Eastern China[J].BMC Infect Dis,2010,10:323-331.
[3]CANTÓN R,COQUE T M,BAQUERO F.Multiresistant Gram-negative bacilli:from epidemics to endemics[J].Curr Opin Infect Dis,2003,16(4):315-325.
[4]XIAO YONGhong,SHEN Ping,WEI Zeqing(肖永红,沈 萍,魏泽庆,等),et al.Mohnarin report 2010:Surveillance of bacterial resistance in East China[J].Chinese Journal of Nosocomiology(中华医院感染学杂志),2011,21(23):4896-4902.(in Chinese)
[5]VEEN E L,SCHILDER A G M,TIMMERS T K,et al.Effect of long-term trimethoprim /sulfamethoxazole treatment on resistance and integron prevalence in the intestinal flora:a randomized,double-blind,placebo-controlled trial in children[J].J Antimicrob Chemother,2009,63(5):1011-1016.
[6]PAIRANO G,AGERSO Y,AARESTRUP F M,et al.Occurrence of integrons and resistance genes among sulphonam resistant Shigella spp.from Brazil[J].Antimicrob Agents Chemother,2005,55(3):301-305.
[7]VO A T,van DUIJKEREN E,FLUIT A C,et al.Antibiotic resistance,integrons and Salmonella genomic island l among non-typhoidal Salmonella serovars in the the Netherlands [J].IntJ Antimicrob Agents,2006,28(3):172-179.
[8]FLUIT A C,SCHMITZ F J.Resistance integrons and super-integrons[J].Clin Microbiol Infect,2004,10(4):272-288.
[9]SABAT M,PRATS G.Structure and function of integrons[J].Enferm Infect Microbiol Clin,2002,20(7):341-345.
[10]ROE M T,VEGA E,PILLAI S D.Antimicrobial resistance markers of class 1 and class 2 integronbearing Escherichia coli from irrigation water and sediments[J].Emerg Infect Dis,2003,9(7):822-826.
[11]TURTON J F,KAUFMANN M E,GLOVER J,et al.Detection and typing of integrons in epidemic strains of Acinetobacter baumannii found in the United Kingdom [J].J Clin Microbiol,2005,43(7):3074-3082.
[12]ROWE-MAGNUS D A,MAZEL D.The role of integron in antibiotic resistance gene capture[J].Int J Med Microbiol,2002,292(2):115-125.
[13]DÍAZ-MEJÍA J J,AMÓBILE-CUEVAS C F,ROSA I, et al.An analysis of the evolutionary relationships of integron integrases,with emphasis on the prevalence ofclass 1 integrons in Escherichia coli isolates from clinical and environmental origins.Microbiology,2008,154(Pt 1):94-102.
[14]SAMBROOK J,FRITSCH E F,MANIATIS T.Molecular Cloning:A Laboratory Manual[M].2ndedition,New York:Cold Sping Harbor Labooratory Press,1989,1.21-1.52,2.60-2.80,7.3-7.35,9.14-9.22.
[15]MEZZATESTA ML,GONAF,STEFANIS.Enterobacter cloacae complex:clinical impact and emerging antibiotic resistance [J].Future Microbiol,2012,7(7):887-902.
[16]BEREKET W,HEMALATHA K,GETENET B,et al.Update on bacterial nosocomial infections[J].Eur Rev Med Pharmacol Sci,2012,16(8):1039-1044.
[17]JEANA SS,HSUEH PR.Highburdenof antimicrobial resistance in Asia [J].IntJ Antimicrob Agents,2012,37(4):291-295.
[18]ZHUO Chao,Su Danhong,NI Yuxing(卓 超,苏丹虹,倪 语 星,等),et al.CHINNET 2009 surveillance of antimicrobial resistance in E.coli and Klebsiella spp in China[J].Chinese Journal of Infection and Chemotherapy(中国感染与化疗杂志),2010,10(6):430-435.(in Chinese)
[19]NANDI S,MAUEER J J,HOFACRE C,et al.Gram-positive bacteria are a major reservoir of class 1 antibiotic resistance integrons in poultry litter.Proc Natl Acad Sci USA,2004,101(18):7118-7122.
[20]PLOY M C,DENIS F,COURVALIN P,et al.Molecular characterization of integrons in Acinetobacter baumannii:descrip tion of a hybrid class2 integron [J].Antimicrob Agents Chemother,2000,44(10):2684-2688.
[21]YU Y,JI S,CHEN Y,et al.Resistance of strains producing extended-spectrum beta-lactamases and genotype distribution in China [J].J Infect,2007,54(1):53-57.
[22]DULUCQ S,STONGE G,GAGNé V,et al.DNA variants in the dihydr of olate reductase gene and outcome in childhood ALL [J].Blood,2007,111(7):3692-3700.

