藏绵羊KAP3.2基因的cDNA克隆、序列分析及组织表达的研究
杨珂伟, 胡亮, 罗斌, 刘霜, 郑玉才, 字向东
(西南民族大学生命科学与技术学院, 四川 成都 610041)
藏绵羊KAP3.2基因的cDNA克隆、序列分析及组织表达的研究
杨珂伟, 胡亮, 罗斌, 刘霜, 郑玉才, 字向东
(西南民族大学生命科学与技术学院, 四川 成都 610041)
为研究藏绵羊角蛋白相关蛋白(KAP3.2)基因结构与功能, 揭示该基因的组织特异性表达规律.以藏绵羊为研究对象, 分别提取藏绵羊心、肝、脾、肾及皮肤组织中总RNA, 并以此为模板, 通过RT-PCR技术对藏绵羊KAP3.2基因的cDNA进行克隆、序列分析; 利用Real-time PCR技术进行组织表达研究.结果表明: 藏绵羊KAP3.2基因编码区全长297bp, 编码98个氨基酸; 藏绵羊与普通绵羊、山羊、藏羚羊、草原鼠、家鼠、人的相应核苷酸同源性分别为99%、97%、96%、79%、75%、73%; 藏绵羊KAP3.2基因在皮肤中高表达, 而在其它组织中相对表达量较低, 说明该基因的表达具有组织特异性.
藏绵羊; KAP3.2基因; 克隆; 序列分析; Real-time PCR
角蛋白作为羊毛纤维的主要成分, 是维持皮肤毛囊结构和形成毛囊细胞的主要结构蛋白质, 并且是毛囊中表达最丰富的蛋白质, 它决定了羊毛的结构特性, 同时在毛囊基因表达和毛发的生物学研究中, 其编码基因也是重要的候选基因[1-3].羊毛纤维具有高度的组织结构, 角蛋白的含量达 99%, 平均分子量是 60000[4].羊毛角蛋白共由18 种氨基酸残基构成, 其中半胱氨酸占12%[5].角蛋白家族包括了30种蛋白质分子, 是由多基因家族编码的, 有两种蛋白: 软角蛋白和硬角蛋白[6].组成毛纤维的主要是硬角蛋白中间微丝蛋白(keratin intermediate proteins, IF)和微丝角蛋白相关蛋白(keratin associated proteins, KAP)[7-8].角蛋白相关蛋白基因(KAP3.2)是高硫角蛋白相关蛋白基因家族中的一员, 而高硫角蛋白相关蛋白基因家族在绵羊毛各性状中可能起到重要作用[9-10].
藏绵羊主要分布于我国的青藏高原地区以及与之毗邻的川、甘、滇等高寒地区.正是由于复杂的地域环境,藏绵羊具有丰富多样的生态类型.目前, Mclaren[11]等通过连锁分析对绵羊的KAP家族基因进行了染色体定位,找到了决定羊毛纤维细度的位点, 并认为羊毛性状是受多基因影响的.刘桂芬[12]等研究结果表明在角蛋白相关蛋白的多基因家族中的高硫蛋白相关蛋白中, 位点W08667与羊毛细度的相关性显著(P<0.05), 外显子位点W06933的两种基因型(AA和BB)与羊毛细度之间的相关性显著(P<0.05).有研究把KAP3.2基因作为影响藏绵羊毛长和产毛量性状的分子标记, 以揭示KAP3.2基因与羊毛的光泽度、密度、弹性和强度等性状是否具有相关性.因此, 本研究以藏绵羊的皮肤组织为材料, 运用同源序列克隆技术结合RT-PCR, 对藏绵羊KAP3.2基因的cDNA进行克隆, 并对其进行生物信息学分析; 利用Real-time PCR技术, 研究KAP3.2基因在藏绵羊心、肝、脾、肾和皮肤等组织中表达, 旨在了解藏绵羊KAP3.2基因的结构与功能.
1 材料与方法
1.1 试验动物及样品采集
选取健康藏绵羊6只,屠宰后采取心、肝、脾、肾和皮肤组织样, 迅速放入液氮中带回实验室, -80℃保存备用.
1.2 主要试剂
动物组织总RNA提取试剂盒、DNA Marker DL2000、2×Taq PCR MasterMix 酶购自天根生物科技(上海)有限公司; 克隆载体pMD-19 Vector、氨苄青霉素、X-Gal和IPTG购自大连宝 TaKaRa 生物工程有限公司; 反转录试剂盒购自Fermentas(MBI)公司; DNA凝胶回收试剂盒购自Axygen公司.
1.3 引物设计与合成
根据GenBank中已公布的绵羊KAP3.2基因序列(AY483216), 利用软件Primer Premier5 设计引物PF:5′-ATGGCTTGCTGTGCTCCCCG-3′; PR:5′-AGCAGCACATCACTGAACCC-3′, 扩增片段长363bp.根据上海Invitrogen公司测序得到的藏绵羊KAP3.2基因序列, 设计荧光定量引物PF:5′- ACCACCATCTGCTCCTCTGA-3′; PR: 5′-GTTGGCACATAGGTGTCAGG- 3′, 引物由上海Invitrogen公司合成.
1.4 藏绵羊KAP3.2基因cDNA克隆
按照天根动物组织总RNA提取试剂盒说明书分别提取心、肝、脾、肾及皮肤组织总 RNA.
取1uL总RNA, 采用Fermentas反转录试剂盒进行cDNA合成.以合成的cDNA第一链为模板进行KAP3.2基因扩增, 引物序列为PF:5′-ATGGCTTGCTGTGCTCCCCG-3′; PR:5′-AGCAGCACATCACTGAACCC-3′.25uL反应体系: 上、下游引物(20pmol/L)各1μL, 2×Taq PCR MasterMix 12.5uL, cDNA模板0.5μL和ddH2O 10μL.PCR反应的条件为: 预变性95℃、3min; 变性94℃、30s, 退火60℃、40s, 延伸72℃、90s, 35个循环; 延伸72℃、5min;最后4℃保存.
1.5 基因测序
首先以Axygen DNA凝胶回收说明书进行胶回收, 得到的产物于16℃连接过夜, 连接体系10μL: pMD19-T Vector 0.5μL、胶回收产物5μL、Solution I 4.5μL.然后将连接产物转化到大肠杆菌DH5α感受态细胞中, 并接种于含有氨苄青霉素、X-Gal和IPTG的LB固体培养基上, 37℃培养过夜.挑取单克隆菌落于LB液体培养基中37℃振荡培养12h, 取菌液作PCR鉴定, 经凝胶电泳检测后, 挑选单克隆菌液送由上海Invitrogen公司测序.
1.6 实时荧光定量PCR检测
用提取的藏绵羊6个组织的总RNA, 运用Real-time PCR方法检测不同组织mRNA以及内参GAPDH基因的表达量.设计内参GAPDH基因引物PF:5′-TGCCAAGTATGATGAGAT-3′;PR:5′-GAAGGTAGAAGAGTAGT-3′,反应体系为10uL: 上下游引物各0.8uL、模板dDNA 0.5uL、SYBR Green Supermix 5Ul、ddH2O 3uL.反应条件为: 预变性95℃、3min; 变性95℃、10s, 退火51.2℃、20s, 延伸65℃、5s, 40个循环; 延伸65℃、5min;4℃保存.实验结果用SPSS Statistics V17.0及Excel Viewer 2003 1.0进行统计分析, 用Pfaffi法[13]分析KAP3.2基因在藏绵羊不同组织中的相对表达量, 具体公式为: RE = (1+E ref)Ctref/ (1+E target)Cttarget.其中: E ref和E target分别表示目的基因和内参基因的扩增效率; Ct ref表示对照样本中目的基因的CT值减去实验样本中目的基因CT值, Ct target表示对照样本中内参基因的CT值减去实验样本中内参基因的CT值.
1.7 生物信息学分析
利用生物分析软件DNAMAN将测得的藏绵羊KAP3.2基因序列与GenBank中检索到的绵羊、山羊、藏羚羊、草原鼠、家鼠和人的相应区域进行核苷酸、氨基酸序列多重比对, 并使用Mega 4.0软件构建物种间系统进化树.利用生物在线分析软件预测蛋白质结构.
2 结果与分析
2.1 藏绵羊KAP3.2基因的PCR扩增
以藏绵羊心、肝、脾、肾及皮肤组织总RNA反转录产物(cDNA)为模板, 用特异性引物进行PCR扩增, PCR产物用1%的琼脂糖凝胶电泳检测, PCR产物条带大小为363bp, 与目的片段相近, 结果如图1.
2.2 藏绵羊KAP3.2基因序列测定
利用DNAMAN软件拼接测序结果得到长为363bp的片段, 与预期相符, 所得片段为藏绵羊KAP3.2 cDNA序列(KM102758).序列分析结果表明该编码区共297bp, 编码98个氨基酸, 相对分子质量为10411.0, 理论pI值为5.98; 碱基组成A( 16.5%)、C( 38.0%)、G( 21.2%)和T(24.2%).其中嘧啶碱基占62.3%, 嘌呤碱基占37.7%, GC含量为59.2%.
2.3 藏绵羊KAP3.2基因cDNA序列、氨基酸序列同源性比对
用DNAMAN生物软件, 将藏绵羊KAP3.2基因编码区核苷酸序列与GenBank中普通绵羊(AY483216.1)、山羊(AY510120.1)、藏羚羊(XM-005964804.1)、草原鼠(XM-005368238.1)、小鼠(NM-025720.3)和人(NM-031959.2)6个物种相应基因序列进行比对.结果表明, 藏绵羊与普通绵羊亲缘关系最为接近, 只在第42bp处有一个碱基差异(C-T转换), 同源性99%; 其次为山羊和藏羚羊, 分别有8处、12处核苷酸差异, 同源性大小分别为97%、96%;而与草原鼠、家鼠和人同源性相对较小分别为: 79%、75%、73%(图2).利用BioEdit生物软件对藏绵羊及普通绵羊、山羊、藏羚羊、草原鼠、家鼠、人各物种KAP3.2基因编码区序列进行氨基酸序列推导,计算得出藏绵羊与普通绵羊、山羊、藏羚羊、草原鼠、家鼠、人KAP3.2氨基酸序列间同源性分别为: 99%、94%、93%、72%、73%和71%, 具有一定的保守性.

图1 kap3.2基因电泳检测结果Fig.1 Electrophoresis results of kap3.2 gene

图2 藏绵羊与其他哺乳动物KAP3.2基因核苷酸序列比对Fig.2 Nucleotide sequence alignment of kap3.2 gene in Tibetan sheep and other mammals
采用邻接法(NJ)对藏绵羊及普通绵羊、山羊、藏羚羊、草原鼠、家鼠和人的核苷酸序列分别构建物种间分子系统进化树(图3), 由图可以看出藏绵羊和绵羊亲缘关系最近, 并和山羊、藏羚羊聚为一类, 而草原鼠、家鼠和人聚为另一类, 符合动物分类学的结果.

图3 KAP3.2基因系统发生树Fig.3 Phylogenetic tree of kap3.2 gene in mammals
2.4 藏绵羊KAP3.2基因在组织中的表达
采用RT-PCR检测藏绵羊KAP3.2基因在心、肝、脾、肾及皮肤组织中的相对表达量, 分析结果如图4, 从图中可以看出, 该基因在皮肤组织中的相对表达量最高, 在其他组织中也有所表达, 但表达量相对较低.
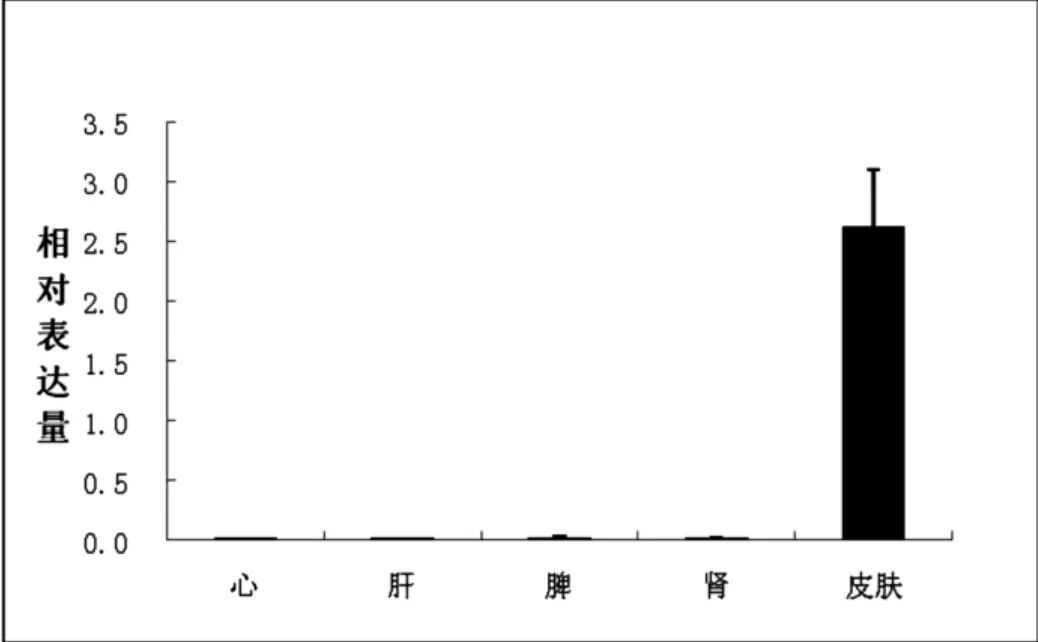
图4 藏绵羊KAP3.2基因在不同组织中的表达水平Fig.4 Expression levels of KAP3.2 gene in different tissues of Tibetan sheep
3 讨论
本研究通过RT-PCR等方法成功克隆出藏绵羊KAP3.2基因CDS区序列, ORF Fingder预测发现KAP3.2基因编码区的长度为297bp, 定位于11号染色体, 无内含子, 共编码98个氨基酸.蛋白质分析结果表明, 该蛋白半衰期为30h, 不稳定指数46.69; 脂肪系数为64.59, 总平均疏水性为0.167, 属于疏水性蛋白; 该蛋白无α螺旋结构, 有折叠和环状结构, 分别占靶蛋白的4.08%、95.92%.另外应用藏绵羊KAP3.2与其他动物氨基酸序列进行系统进化树分析, 从聚类反应图可以看出, 其聚类反应结果与核苷酸同源性比对的结果一致, 符合动物分类学的结果.
Henry[14]、Cockett[15]和Ponz[16]等人通过研究KAP基因和羊毛性状的关系, 发现了控制羊毛纤维直径、羊毛纤维直径变异系数和羊毛长的QTL.王志有[17]和张亚妮[18]等认为KAP基因与绒山羊和细毛羊部分羊毛性状有一定的相关性.本研究通过KAP3.2基因与其他哺乳动物相应核苷酸序列分析表明, 该基因在核苷酸水平上有差异,而且在相应的蛋白水平亦有一定差异.这可能是不同物种在不同环境下进化的结果, 要确定这种结果, 需要再做进一步的研究.
KAP3.2基因与羊毛产量和羊毛长性状之间存在一定的关系, 研究中可作为藏绵羊产毛量和毛长性状的分子标记.本研究荧光定量结果显示, 该基因在皮肤组织中的高表达与预测结果一致, 而在其他组织中也有所表达,但表达量相对较低.说明该基因主要在皮肤组织中发挥重要作用, 这也与其生物学功能相符合.
4 结论
本试验采用实时RT-PCR技术, 以藏绵羊皮肤组织为材料克隆KAP3.2基因的CDS区序列(GenBank NO: KM102758).通过Real-time PCR检测KAP3.2基因在所测组织中均有表达, 但在皮肤组织中高表达, 说明该基因与羊毛性状具有重要作用.
[1]王志有, 陈玉林, 徐秋良, 等.藏系绵羊 KAP3.2 基因多态性及其对部分经济性状的影响[J].畜牧兽医学报, 2011, 42(2): 284-288.
[2]许汉峰.角蛋白关联蛋白基因(KAP和KRT)与绵羊毛品质的相关性研究[D].石河子大学, 2008.
[3]姚毅, 雷承志, 君亮, 等.新疆细毛羊和陕北细毛羊羊毛细度候选基因的PCR-SSCP分析[J].西北农林科技大学学报, 2006, 34(12): 6-10.
[4]赵宗胜.绵羊产毛性能的细胞及分子生物学调控机理研究[D].南京: 南京农业大学, 2005.
[5]管峰,石国庆,刘守仁.角蛋白家族及其对羊毛生长发育的调控[J].生命的化学, 2007, 27(1): 92-93.
[6]SHIBUYA K, KUDON J, OBAYASHI A, et al.Comparative genomics of the keratin- associated protein (K AP) gene clusters in human, chimpanzee, and baboon [J].Mamm Genome, 2004, 15(3): 179-192.
[7]PARRY DA, SMITH TA, ROGERS MA, et al.Human hair keratin- associated proteins: Sequence regularities and structural implications [J] .J Struct Biol, 2006, 155(2): 361-369.
[8]KARIYA N, SHIMOMURA Y, ITO M.Size polymorphisms in the human ultrahigh sulfur hair keratin-associated protein 4, KA P4, gene family [J].J Investig Dermatol, 2005, 124(6): 1111-1118.
[9]YAHAQI S, SHIBUYA K, OBAYASHI I, et al.Identification of two novel clusters of ultrahigh-sulfur keratin-associated protein genes on human chromosome11 [J].Biochem Biophys Res Commun, 2004, 318(3): 655- 664.
[10]尹俊, 扈庭茂, 李金泉,等.与绵羊KAP6-1相似的6个绒山羊全长cDNA的克隆与序列分析[J].遗传学报, 2004, 31(5): 502-507.
[11]MCLAREN RJ, ROGERS GR, DAVIES KP, et al.Linkage mapping of wool keratin and keratin-associated protein genes in sheep [J].Mamm Genome, 1997, 8( 12) : 938-940.
[12]刘桂芬, 田可川, 张恩平, 等.优质细毛羊羊毛细度的候选基因分析[J].遗传, 2007, 29(1): 70-74..
[13]PFAFFL MW.A new mathematical model for relative quantification in real-time RT-PCR [J].Nucleic Acids Res, 2001, 29(9):e45.
[14]HENRY HM, DODDSS KG, WULIJI T, et al.A genome screen for QTL for wool traits in a Merinox Romney backcross flock [J].Mamm Genome, 1998, 8: 938-940.
[15]COCKETT NE, SHAY TL, SMIT M.Analysis of the sheep genome [J] .Physiol Genomics, 2001, 7(2): 69-78.
[16]PONZ R, MORENO C, ALLAIN D, et al.Assessment of genetic variation explained by markers for wool traits in sheep via asegment mapping approach[J].Mamm Genome, 2001, 12(7): 569-572.
[17]王志有, 祁全青, 陈玉林.高原型藏绵羊KAP基因多态性与部分产毛性状的关系研究[J].中国农学通报, 2010, 26(14): 18-22.
[18]张亚妮, 张恩平, 吴迪, 等.内蒙古绒山羊KAP基因与经济性状关系的研究[J].畜牧兽医学报, 2007, 38(5): 447-451.
Cloning and tissue expression of KAP3.2 gene in Tibetan sheep
YANG Ke-wei, HU Liang, LUO Bin, LIU Shuang, ZHENG Yu-cai, ZI Xiang-dong
(School of Life Science and Technology, Southwest University for Nationalities, Chengdu 610041, P.R.C.)
This study was carried out to obtain and analyze sequence of Tibetan sheep KAP3.2 gene by RT-PCR, and to determine its mRNA expression in different tissues by real-time PCR. Total RNA was extracted from the heart, liver, spleen, kidney and skin tissues of the Tibetan sheep. The results showed that the coding region of Tibetan sheep KAP3.2 gene was 297bp in length, encoding 98 amino acids. The homologies of nucleotide sequences of the coding region of KAP3.2 gene between Tibetan sheep and sheep, goat, chiru, prairie vole, mouse and human were 99%, 97%, 96%, 79%, 75% and 73%, respectively. The KAP3.2 gene was highly expressed in the skin tissue and relatively was low in other tissues studied. The expression level of KAP3.2 mRNA is tissue specific.
Tibetan sheep; KAP3.2 gene; cloning; sequence analysis; real-time PCR
S826
A
1003-4271(2014)06-0809-05
10.3969/j.issn.1003-4271.2014.06.02
2014-09-25
字向东(1963-), 男, 白族, 云南云龙人, 教授, 研究方向: 动物遗传育种与繁殖; E-mail: zixd@sina.com.
国家863计划课题(2013AA102506); 西南民族大学优秀学生培养工程项目(2014ZYXS58)

