锌指蛋白及人工锌指蛋白对微生物代谢影响的研究进展
刘卓,张飞,赵心清,白凤武
大连理工大学生命科学与技术学院,辽宁 大连 116024
锌指结构域是真核细胞中最常见的 DNA结合域,含有锌指结构域的蛋白被称为锌指蛋白。锌指蛋白在真核生物中分布广泛,参与 DNA、RNA和蛋白质的识别、调控基因转录,并参与蛋白质之间的相互作用[1]。锌指蛋白对生物体的生长、发育及代谢具有重要的调控作用,例如在植物中,锌指蛋白可调控花和叶的发育,并在植物抗逆反应中起重要的调控作用[2-3],锌指蛋白对动物的发育和疾病发生过程也有重要的影响[4]。锌指蛋白对细胞生长和代谢具有重要的调节作用,但目前对锌指蛋白的研究多集中在植物和哺乳动物,对微生物锌指蛋白的研究相对较少。人工锌指蛋白是人工转录因子中研究较多的调控技术,近年来在微生物菌株选育中有很多成功的应用,但使用的锌指结构域多来自人类或哺乳动物基因组。本文综述了近年来微生物中,尤其是原核生物细胞中发现的锌指蛋白及其对细胞基因表达和代谢功能的作用,并对人工锌指蛋白技术在微生物菌株选育中的应用进行了评述和展望。
1 锌指结构域
锌指结构域不仅存在于转录因子蛋白中,还出现在酶、金属硫蛋白等细胞内重要的大分子结构中[5-7]。自 1983年首次发现调控基因转录的锌指蛋白 TFIIIA以来[8],新的锌指蛋白不断被发现。锌指蛋白类转录因子主要包括C2H2、C4、C6、C4HC3、C3HC4、C2HC、C3H 以及联合型等多种类型,其中以 C2H2型最为常见[1]。典型的C2H2型锌指由30个左右氨基酸组成,含有一组反向平行的 β折叠与一个含有DNA识别位点的α螺旋,通过疏水作用组装而成。β折叠上的 2个Cys残基与 α螺旋上的 2个 His残基结合一个 Zn2+离子稳定其结构,由于其结构类似手指状,因此而得名。C2H2型锌指在锌离子存在时能够紧密折叠形成ββα结构,其中锌离子夹叠在 α螺旋和 2股反向平行的 β折叠中。锌指结构既能与RNA和DNA结合,又能与DNA-RNA双链分子以及同类蛋白结合[9]。
锌指蛋白的DNA结合域具有与DNA双螺旋互补的特殊表面结构,依靠其指形结构伸入到DNA双螺旋的大沟内,通过α螺旋中氨基酸的侧链与DNA碱基以氢键特异性结合。锌指与DNA发生结合时必须满足如下条件:1) 锌指蛋白的 α螺旋位于 DNA大沟内;2) 锌指蛋白携带正电荷的区域接近 DNA磷酸骨架;3) 锌指间的接头结构相对固定[10]。与锌指特异性结合相关的主要氨基酸位点是α螺旋上的第1、2、3、6位残基 (图1A)。每个锌指单元能够特异性地识别3 bp DNA序列。与其他DNA识别结构域如亮氨酸拉链结构域、αβα结构域等不同的地方是锌指结构域能够识别非对称序列。因此,将一个或多个锌指单元串联并与转录激活或抑制域结合,可构建以锌指蛋白作为特异的DNA结合结构域的转录因子,称之为人工锌指蛋白转录因子(Artificial zinc-finger protein transcription factor)。锌指蛋白转录因子可以结合在启动子周围的特定区域内,它们所连接的激活或者抑制域可以直接或者间接作用于RNA聚合酶,对后续的转录过程进行调节 (图1B)。将这一类人工锌指蛋白转录因子导入细胞中表达,根据锌指结构域的识别序列,特异性地结合在启动子区域,调控靶基因的转录水平,通过定向筛选可获得所需的表型。
2 微生物锌指蛋白及其对细胞代谢的作用
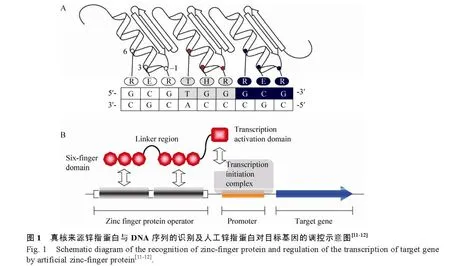
存在于真核生物转录因子中的锌指结构域受到很多关注,但近年来在微生物转录因子、酶蛋白等重要的生物大分子中也发现了锌指结构域的存在,并证明这些蛋白在与 DNA、底物及金属离子的识别过程中起着重要的作用。例如,在丝状真菌中存在与胞内代谢紧密相关的锌指蛋白,包括黄曲霉Aspergillus flavus中与乙醇代谢相关的 C6型锌指蛋白 AlcR,粗糙脉孢菌Neurospora crassa中参与糖代谢调节的Cre-1锌指蛋白等[5-6]。植物病原性霉菌玉米黑粉菌Ustilago maydis中的Rual蛋白含有2个C2H2型锌指结构域,能够调控黑粉菌酸 (Ustilagic acid)的生物合成。在碳源贫瘠的环境中,Rua1蛋白的锌指结构域与黑粉菌酸合成基因簇的启动子区中的保守序列结合,产生具有广谱抗细菌抗真菌活性的糖脂,从而应对环境压力[13]。此外,酿酒酵母Saccharomyces cerevisiae中的Yml081W是C2H2型锌指蛋白,能调节ALD4和ALD6基因的 mRNA水平,从而影响乙酸的合成[14]。Gis2、Mgs1锌指蛋白也在酿酒酵母的 DNA的复制、修复及RNA加工等过程中发挥着重要的作用[15-16]。近期微生物中报道的部分锌指蛋白及其功能如表1所示。
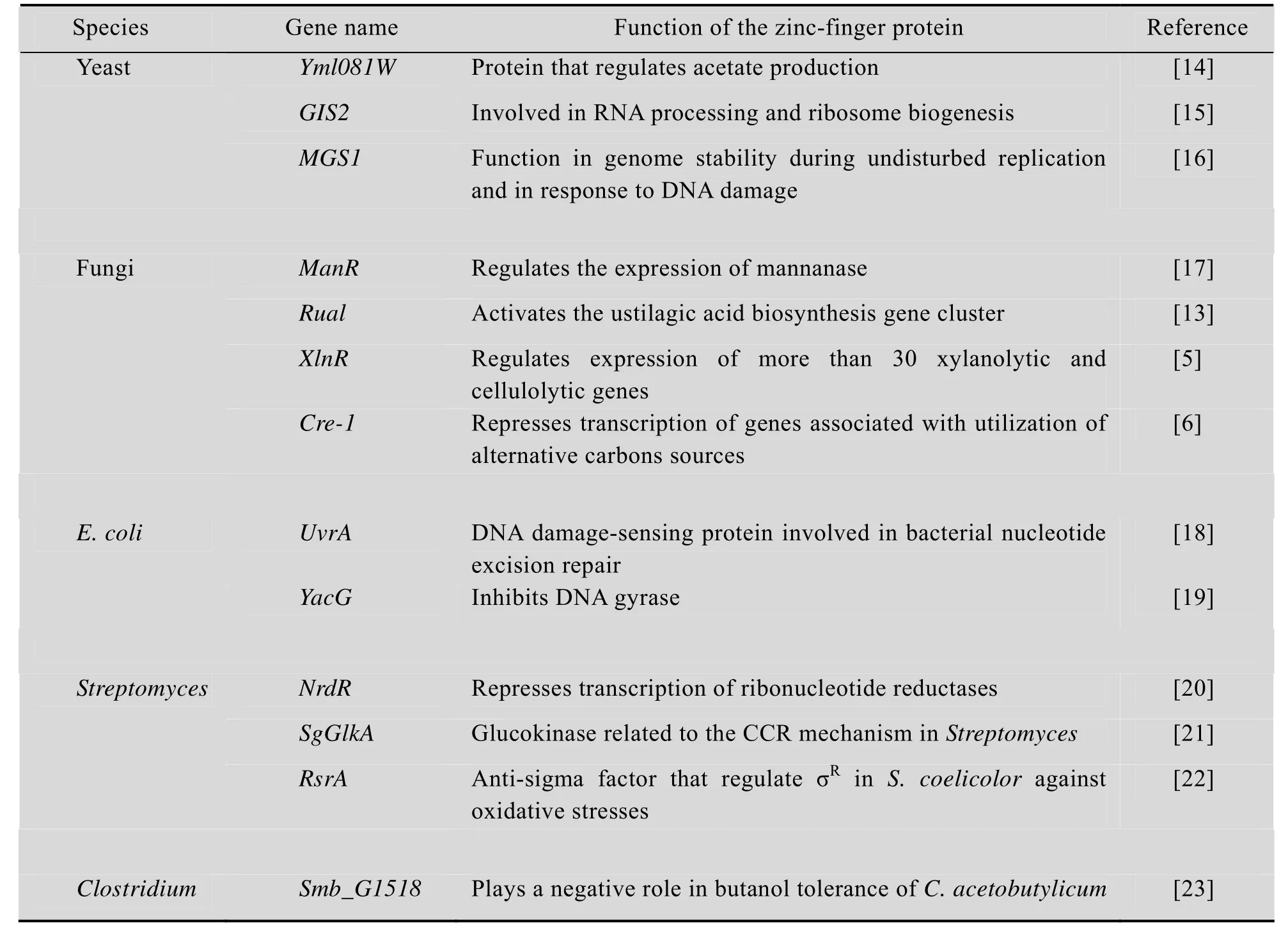
表1 部分微生物锌指蛋白Table 1 Characterized zinc-finger proteins in microbial strains
首次在原核细胞中发现的C2H2型锌指蛋白是根癌农杆菌Agrobacterium tumefaciens中的Ros蛋白[24]。Ros蛋白大小为 15.5 kDa,由A.tumefaciens中的染色体基因ros表达,能够抑制致病基因和位于 Ti质粒上致癌基因的转录。Ros蛋白含有一个锌指结构域,能够特异性地结合在致病基因上游大约9 bp的反向重复序列上 (称为Ros box)。随着蛋白中所含的锌指单元数量的增加,会增强与靶基因识别的特异性和亲和性。
在天蓝链霉菌Streptomyces coelicolorA3(2)中,NrdR蛋白能够抑制两种核苷酸还原酶(RNRsI,II) 的表达。NrdR蛋白由大约 167个氨基酸组成,N端含有一个C4型锌指结构域与ATP锥域 (ATP cone domain)。当ATP/dATP与ATP锥域结合时,能够改变锌指结构域的构象,使之与靶基因上游序列 (NrdR box) 特异性结合,调控目标基因的表达[20]。这种锌指单元与ATP锥域的组合系统能够迅速地检测胞内物质变化,灵活地调控基因表达量。
原核细胞中一些与代谢活动相关的酶与金属硫蛋白中也含有锌指结构域,这些生物大分子通过锌指结构域与胞内小分子物质结合来调控胞内代谢活动。在微生物的碳代谢物阻遏(Carbon catabolite repression, CCR) 过程中,葡萄糖激酶发挥着重要的作用。灰色链霉菌Streptomyces griseus中的葡萄糖激酶SgGlkA属于ROK葡糖激酶家族,SgGlkA含有两段保守序列。其中一段含有由3个Cys结合一个锌离子构成的C3H型锌指结构域,它能够与葡萄糖中C1、C2位的羟基形成氢键,在葡糖激酶识别底物的过程中起着重要的作用[21]。金属硫蛋白普遍存在于许多真核细胞中,尽管在原核细胞中存在这种蛋白的报道较少,但大肠杆菌中和蓝藻SynechococcusPCC 7942都存在SmtA类锌离子结合金属硫蛋白,每分子蛋白含有4个Cys组成锌指结构域,能够结合一个锌离子,大肠杆菌中的 SmtA类似蛋白更倾向于在氧化应激环境下释放锌离子,从而改变大分子间的相互作用[7]。
蛋白激酶C (Protein kinase C, PKC) 在调控醇耐受性中起着重要的作用,PKC通过亚基C1A与C1B上的甘油二酯/佛波酯基识别乙醇分子,N端含有的锌指结构域与其他调控蛋白结合以抑制细胞代谢活动。基于这一机理,我国学者利用PKC中识别乙醇分子的高度保守序列与丙酮丁醇梭菌Clostridium acetobutylicum蛋白组进行比对,发现氨基端序列相似度 30%以上并含有锌指结构域的Smb_G1518蛋白,并推测该蛋白与C. acetobutylicum的丁醇耐受性相关。通过敲除或抑制Smb_G1518基因,得到在1%丁醇浓度下生长速度加快,菌体终浓度 (A600)提高70%的突变菌株,从而证明了Smb_G1518蛋白的确与丁醇胁迫耐受相关[23],这是首次关于梭菌中锌指蛋白的报道。
3 人工锌指蛋白技术及其在微生物代谢工程中的应用
多种生物过程如发育、分化和疾病发生等都受细胞内转录因子的调节完成。为实现细胞代谢的定向调控,可使用人工设计的转录因子定向调控细胞内相关靶基因的表达[11-12]。结合在特定基因启动子区域的人工转录因子可以实现生物体内靶基因的表达调节,并且可根据需要实现不同强度的微调节。人工转录因子的结构包括一个DNA结合域,一个转录调控域或效应区。其中DNA结合域负责结合特定的基因上游序列,而效应域可以实现上调或下调不同靶基因的表达。因此可以设计DNA结合域和效应区,创建所需要的人工转录因子。与其他DNA结合域相比,锌指结构域结构上相对保守,与DNA作用相对简单,识别 DNA序列具有高度特异性,能够在转录和翻译水平上调节和控制基因表达。因此在人工转录因子的构建中,锌指结构域被认为是最合适的DNA结合域。
目前,人工锌指蛋白技术已经成功应用于多个领域,例如细胞分化、逆境抗性、表面抗原及新型药物筛选[11-12]。人工锌指蛋白转录因子有以下优点:第一,人工锌指蛋白转录因子应用范围广泛,包括哺乳动物细胞、植物细胞、酵母等真核细胞,以及细菌等原核细胞。第二,通过测定转录组的变化水平,能够找到影响目标基因表达水平的相关基因,从而进一步针对性地研究。第三,根据转录因子的结合位点不同,分别转入同种细胞,能够获得不同的表型。例如温度耐受性、渗透胁迫耐受性、溶剂耐受性等。第四,与传统的过表达和敲除相比,人工锌指蛋白转录因子技术能够在胞内引起全局基因表达微小的变化,因此对细胞生长代谢影响较小,致死率低。下面将针对性地对人工锌指蛋白技术在菌种改造中的应用进行介绍。
3.1 人工锌指蛋白技术对酵母菌的代谢调控
当控制性状的基因未知或性状受多基因控制时,利用人工锌指蛋白文库技术结合适当的筛选方法能够获得优良性状。韩国Jin-Soo Kim研究小组从人类基因组中克隆25种不同的锌指单元的基因,以 4个为一组,构建了 358种能够识别 12 bp核苷酸序列的锌指蛋白文库。与Gal4转录激活功能域或Ume6抑制功能域结合,在半乳糖诱导型启动子调控下在S. cerevisiae中表达,筛选得到酮康唑 (Ketoconazole) 抗性、耐热性或具有渗透胁迫耐受性质的酵母菌株[25]。与获得突变菌株的传统方法相比,人工锌指蛋白技术更简易,避免了繁重的多轮筛选。例如,随机突变法至少需要107–109个单克隆,核酶文库的多样性要达到107,而人工锌指蛋白文库技术只需筛选105–106个转化子[26]。除了改变内源性基因的表达,人工锌指蛋白还能够调节外源性重组蛋白的表达量。Jin-soo Kim研究小组在酿酒酵母中异源表达人类生长激素 (hGH),利用锌指蛋白文库筛选得到了 hGH产量提高 2.5倍的转化菌株[26]。
人工锌指蛋白可代替天然转录因子,对细胞代谢起到调控作用。利用酵母HIS3筛选系统从人工锌指蛋白文库中筛选得到能够代替 Gal4转录因子的C2H2型人工锌指蛋白。该实验中删去了GAL1启动子区域中Gal4转录因子的结合序列,将残余启动子序列作为锌指结构域的识别位点;文库中的锌指蛋白结构域与Gal4转录因子激活域相连,在半乳糖筛选平板中得到了3株转化菌株。Real-time PCR实验结果表明,3株转化菌株中HIS3基因表达量提高了10倍,证明人工锌指蛋白可有效激活目标基因的转录[27]。
对人工锌指蛋白在酵母菌代谢工程改造中的作用机理进行研究发现,人工锌指蛋白技术改造后获得的菌株性状通常是多基因表达量共同变化的结果。将人工锌指蛋白文库转入酿酒酵母生长缺陷型菌株S. cerevisiaesec14ts中表达,定向筛选得到生长缺陷表型消除的转化子SG1、SG9 和 SG23[28]。已报道NET1、CSR1、PRD17、SFH5和PIK1基因转录量的上调或PCT1、CKI1和CPT1基因的下调能够克服该生长缺陷表型。测定转化子SG1和SG9中的转录组水平发现,上述基因均呈下调状态。在转化子SG9中过表达PCT1基因,只能部分恢复生长缺陷的表型;转化子SG23中的NTE1基因的表达量呈上调,因此研究人员认为该锌指转录因子与NTE1基因的结合引起了表达量的上调,进而利用ChIP-qPCR技术测定人工锌指蛋白的结合位点,结果发现锌指蛋白并不与NTE1基因结合,而是通过与其他基因的结合,从而导致NTE1基因的表达量增多[28]。以上结果证明,锌指蛋白对细胞代谢的调控在一般情况下是具有多基因全局调控能力的,相对于单基因的表达调控,利用人工锌指蛋白可实现受多基因控制的复杂表型的调控。
人工锌指蛋白在细胞内的作用靶点,与预期的来源锌指蛋白的保守结合位点可能有很大不同。在上述研究中获得的锌指蛋白转化子SG1,胞内含有的锌指转录因子预测能够与12 bp序列特异性结合。在原始菌株基因组中发现 18个基因内含有预测的结合位点,但是这些基因的转录水平均未见变化,说明无法通过预测的结合位点来表征基因的表达水平,但是正是这种低特异性造就了转化子表型的多样性。
本课题组近期与韩国学者合作,在酿酒酵母中转入利用人工锌指蛋白文库,成功获得了乙醇耐性和乙醇发酵性能改善的工业酿酒酵母[29],显示了人工锌指蛋白可与酿酒酵母细胞内的多种功能调控元件相结合,调控细胞的代谢和生理功能。
3.2 人工锌指蛋白技术对大肠杆菌的代谢调控
2000年麻省理工学院的研究学者证明了将锌指蛋白与大肠杆菌RNA聚合酶的α亚基融合表达的蛋白能够结合在报告基因上游并激活目标基因的转录[30]。这一发现首次证明了锌指蛋白能够在原核细胞中发挥调控作用,从此开始了人工锌指蛋白技术在原核菌种改造中的研究。将人类基因组来源的40种不同的锌指单元组成三锌指蛋白,与 cAMP repressor protein(CRP) 转录功能域融合表达,CRP为大肠杆菌环化腺嘌呤核苷酸受体蛋白,能够调控大肠杆菌细胞内多于 200种不同启动子的转录,三锌指蛋白于CRP融合后构成了人工锌指转录因子文库,库容6.4×104。将该文库转入E. coli中表达进行表型筛选,获得了热激耐受性,渗透压胁迫耐受性及抗低温的转化子[31]。此外该实验还研究了CRP功能域对锌指蛋白调控效果的影响,无CRP功能域的锌指转录因子只能抑制目标基因的表达,抑制效果为含有CRP功能域的转录因子的1/4;基于锌指结构域的识别位点不同,CRP功能域能够产生不同的调控效果。当识别位点位于核苷酸序列–80与–30之间时,该人工转录因子激活靶基因表达;当识别位点位于–30下游时,抑制靶基因的表达。因此,人工锌指蛋白连接的功能域在其发挥调控过程中起着重要作用。
利用通过人工锌指蛋白技术获得的转化子,可进一步深入挖掘与菌株表型相关的基因。例如,获得耐热性大肠杆菌锌指蛋白转化子T9后,研究者利用免疫共沉淀方法找到与锌指蛋白可能的结合序列并分析该基因的表达量变化。实验结果表明,T9锌指蛋白的结合抑制了ubiX基因的表达量;敲除ubiX基因后,转化菌株仍保持耐热性状。因此推测,ubiX基因与大肠杆菌菌株耐热性相关[32]。
目前,人工锌指蛋白技术在原核微生物菌株改造中的应用较少,主要集中于菌株对环境胁迫耐性方面的研究。近期韩国学者利用大肠杆菌人工锌指蛋白文库筛选出丁醇耐受性提高的转化菌株 (丁醇耐受浓度从1%提高至1.5%),转录组分析结果表明sdhCDAB、flu和ybgD基因的转录水平急剧上调,其中sdhCDAB基因参与细胞的氧化应激反应,已报道与细胞的丁醇胁迫耐受性相关。在野生型大肠杆菌中过表达以上 3个基因,菌株仍表现出较好的丁醇耐受性,证明基因sdhCDAB、flu和ybgD与菌株的丁醇耐受性相关。此外,丁醇耐受性菌株还表现出较好的温度耐受性[33]。但该研究并未深入研究在耐性提高的突变体中人工锌指蛋白与哪些靶点基因结合。
除了上述构建人工锌指转录因子与多个靶点基因的结合来通过细胞全局的代谢调控改造菌株性状外,还可以利用锌指结构域与DNA结合的特性,对菌株特定代谢物的生产进行调控。韩国学者利用锌指结构域构建DNA脚手架系统(DNA scaffold system) 以提高大肠杆菌中L-苏氨酸的产量。将L-苏氨酸合成途径中的3种关键酶分别与识别不同DNA序列的3种锌指蛋白融合表达,这些融合蛋白能特异性地识别脚手架质粒 (Scaffold plasmid),并按照一定顺序排列在DNA脚手架上进行酶促反应。这一技术缩小了酶促反应的作用空间,避免中间产物由于扩散而造成的浓度降低或耗时长等问题。更重要的是,这一技术能够通过简单地改造脚手架质粒来调整酶蛋白间距和作用角度,以及限速酶的结合位点数。实验结果表明,大肠杆菌的L-苏氨酸生产时间缩短了50%,而且由于有害中间产物的迅速转化,突变菌株的生长状态更加良好[34]。
4 存在问题及展望
目前国内学者利用人工锌指蛋白技术主要进行的是调控哺乳动物细胞的的基因表达及基因治疗方面的研究,但在微生物代谢工程领域的应用依然较少。尽管人工锌指蛋白转录因子在提高微生物菌种耐性的研究中得到广泛应用。但是目前还存在以下几点有待探索:1) 对锌指识别DNA、RNA及蛋白间相互作用的分子机制的研究,并在此基础上,设计出更有效的人工锌指来调节不同微生物基因转录,优化多酶催化体系;2) 目前该技术主要集中于提高菌株胁迫耐受性,例如高温和丁醇耐受性。但是根据其调控基因表达量的机理,该技术有望应用于提高目标菌株的代谢产物,例如抗生素和酶等的生产;3) 目前用于构建人工转录因子的锌指结构域主要是C2H2型锌指,但是在原核微生物中目前发现的天然锌指结构域大多为C4和C6型。因此以多种锌指类型为骨架,设计新型锌指结构域,有助于人工锌指蛋白在原核菌株中的应用;4) 目前锌指蛋白技术主要应用于酵母和大肠杆菌等菌株的改造,未来将有望应用于梭菌、放线菌等其他具有工业生产价值的微生物菌株。
近年来,国内外对微生物,尤其是原核微生物中锌指蛋白的研究越来越多,锌指蛋白作用机制的深入探究,将有助于理解锌指蛋白对细胞代谢调控的分子机制。随着微生物内源锌指蛋白调控机理的进一步阐明,人工锌指蛋白技术将具有更广阔的应用前景,成为对微生物进行代谢工程改造的有力工具。
[1]Li XB, Zhang JW. Structure and function of zinc finger proteins in Eucaryon. Chin J Biochem Mol Biol, 2009, 25(3): 206–212 (in Chinese).
李晓波, 张俊武. 真核生物中锌指蛋白的结构与功能. 中国生物化学与分子生物学报, 2009,25(3): 206–211.
[2]Song B, Hong Y, Wang PW, et al. Advances on plant C2H2-type zinc finger protein. Genome Appl Biol, 2010, 29(5): 1133–1141 (in Chinese).
宋冰, 洪洋, 王丕武, 等. 植物 C2H2型锌指蛋白的研究进展. 基因组学与应用微生物学, 2010,29(5): 1133–1141.
[3]Xiang JH, Li LZ, Chen XB. Progress in the study of abiotic stress-related zinc finger protein genes in plants. J Nuclear Agri Sci, 2012, 26(4): 666–672(in Chinese).
向建华, 李灵之, 陈信波. 植物非生物逆境相关锌指蛋白基因的研究进展. 核农学报, 2012,26(4): 666–672.
[4]Qin ZH, Yang QC, Chao YH, et al. Study of CCCH-type zinc finger protein. Biotech Bull, 2010,8: 1–6 (in Chinese).
秦智慧, 杨青川, 晁跃辉, 等. CCCH型锌指蛋白研究进展. 生物技术通报, 2010, 8: 1–6.
[5]Chang PK, Ehrlich KC. Genome-wide analysis of the Zn (II) 2Cys6 zinc cluster-encoding gene family inAspergillus flavus. Appl Microbiol Biotechnol,2013, 97(10): 4289–4300.
[6]Sun J, Glass NL. Identification of the CRE-1 cellulolytic regulon inNeurospora crassa. PLoS ONE,2011, 6(9): e25654.
[7]Blindauer CA, Harrison MD, Robinson AK, et al.Multiple bacteria encode metallothioneins and SmtA-like zinc fingers. Mol Microbiol,2002,45(5): 1421–1432.
[8]Roeder RG. Multiple forms of DNA-dependent RNA polymerases inXenopus laevis, properties,purification and subunit structure of class III RNA polymerases. J Biol Chem, 1983, 258: 1932–1941.
[9]Brown RS. Zinc finger proteins: getting a grip on RNA. Curr Opin Struct Biol,2005, 15(1): 94–98.
[10]Zhao N, Zhao F, Li YH. Advances in research on zinc finger protein. Lett Biotech, 2009, 20(1):131–134 (in Chinese).
赵楠, 赵飞, 李玉花. 锌指蛋白结构及功能研究进展. 生物技术通讯, 2009, 20(1): 131–134.
[11]Sera T. Zinc-finger-based artificial transcription factors and their applications. Adv Drug Deliver Rev, 2009, 61(7/8): 513–526.
[12]Khalil AS, Lu TK, Bashor CJ. A synthetic biology framework for programming eukaryotic transcription functions. Cell, 2012, 150(3): 647–658.
[13]Teichmann B, Liu L, Schink KO, et al. The C2H2zinc finger transcription factor Rua1 activates the ustilagic acid biosynthesis gene cluster inUstilago maydis. Appl Environ Microbiol,2010, 76(8):2633–2640.
[14]Walkey CJ, Luo Z, Madilao LL, et al. The fermentation stress response protein Aaf1p/Yml081Wp regulates acetate production inSaccharomyces cerevisiae. PLoS ONE,2012,7(12): e51551.
[15]Scherrer T, Femmer C, Schiess R, et al. Defining potentially conserved RNA regulons of homologous zinc-finger RNA-binding proteins.Genome Biol,2011, 12(1): R3.
[16]Saugar I, Parker JL, Zhao S, et al. The genome maintenance factor Mgs1 is targeted to sites of replication stress by ubiquitylated PCNA. Nucleic Acids Res,2012, 40(1): 245–257.
[17]Ogawa M, Kobayashi T, Koyama Y. ManR, a novel Zn(II)2Cys6 transcriptional activator, controls the β-mannan utilization system inAspergillus oryzae.Fungal Genet Biol,2012, 49: 987–995.
[18]Wagner K, Moolenaar GF, Goosen N. Role of the insertion domain and the zinc-finger motif ofEscherichia coliUvrA in damage recognition and ATP hydrolysis. DNA Repair,2011, 10(5):483–496.
[19]Sengupta S, Nagaraja V. YacG fromEscherichiacoliis a specific endogenous inhibitor of DNA gyrase. Nucleic Acids Res,2008, 36(13):4310–4316.
[20]Grinberg I, Shteinberg T, Gorovitz B, et al. TheStreptomycesNrdR transcriptional regulator is a Zn ribbon/ATP cone protein that binds to the promoter regions of class Ia and class II ribonucleotide reductase operons. J Bacteriol,2006, 188(21):7635–7644.
[21]Miyazono KI, Tabei N, Morita S, et al. Substrate recognition mechanism and substrate-dependent conformational changes of an ROK family glucokinase fromStreptomyces griseus. J Bacteriol,2012, 194(3): 607–616.
[22]Bae JB, Park JH, Hahn MY, et al. Redox-dependent changes in RsrA, an anti-sigma factor inStreptomyces coelicolorzinc release and disulfide bond formation. J Mol Biol,2004, 335(2):425–435.
[23]Jia K, Zhang Y, Li Y. Identification and characterization of two functionally unknown genes involved in butanol tolerance ofClostridium acetobutylicum. PLoS ONE,2012, 7(6): e38815.
[24]Bouhouche N, Syvanen M, Kado CI. The origin of prokaryotic C2H2zinc finger regulators. Trends Microbiol,2000, 8(2): 77–81.
[25]Park KS, Lee D, Lee H, et al. Phenotypic alteration of eukaryotic cells using randomized libraries of artificial transcription factors. Nat Biotechnol,2003, 21(10): 1208–1214.
[26]Park KS, Seol W, Yang HY, et al. Identification and use of zinc finger transcription factors that increase production of recombinant proteins in yeast and mammalian cells. Biotechnol Prog,2005,21(3): 664–670.
[27]Park KS, Kim JS. Engineering of GAL1 promoter-driven expression system with artificial transcription factors. Biochem Biophys Res Commun,2006, 351(2): 412–417.
[28]Lee SW, Kim E, Kim JS, et al. Artificial transcription regulator as a tool for improvement of cellular property inSaccharomyces cerevisiae.Chem Eng Sci,2013, 103: 42–49.
[29]Ma C, Zhao XQ, Li Q, et al. Breeding of robust industrial ethanol tolerant Saccharomyces cerevisiae strain using artificial zinc finger protein library. Chin J Biotech, 2013, 29(5): 612–619 (in Chinese).
马翠, 赵心清, 李倩, 等. 利用人工锌指文库选育高乙醇耐受性工业酵母菌株. 生物工程学报,2013, 29(5): 612–619.
[30]Joung JK, Ramm EI, Pabo CO. A bacterial two-hybrid selection system for studying protein-DNA and protein-protein interactions. Proc Natl Acad Sci USA,2000, 97(13): 7382–7387.
[31]Lee JY, Sung BH, Yu BJ, et al. Phenotypic engineering by reprogramming gene transcription using novel artificial transcription factors inEscherichia coli. Nucleic Acids Res,2008, 36(16):e102.
[32]Park KS, Jang YS, Lee H, et al. Phenotypic alteration and target gene identification using combinatorial libraries of zinc finger proteins in prokaryotic cells. J Bacteriol,2005, 187(15):5496–5499.
[33]Lee JY, Yang KS, Jang SA, et al. Engineering butanol-tolerance inEscherichia coliwith artificial transcription factor libraries. Biotechnol Bioeng,2011, 108: 742–749.
[34]Lee JH, Jung SC, Kang KH, et al. Improved production of L-Threonine inEscherichia coliby use of a DNA scaffold system. Appl Environ Microbiol, 2013, 79(3): 774–782.

