我国西花蓟马线粒体DNA-COⅠ基因变异及群体遗传结构分析
张桂芬, 乔玮娜, 古君伶, 闵 亮, 万方浩
1中国农业科学院植物保护研究所,植物病虫害生物学国家重点实验室,北京100193;2农业部外来入侵生物预防与控制研究中心,北京100193; 3北京市顺义区植保植检站,北京101300
西花蓟马Frankliniellaoccidentalis(Pergande),又称苜蓿蓟马alfalfa thrips,属蓟马科Thripidae花蓟马属Frankliniella,原产于加拿大西部和美国北部,最早记载于1895年,是美国加利福尼亚州的常见害虫(Beshear, 1983)。随着加州花卉产业的发展,西花蓟马随花卉及其产品的销售,扩散蔓延至宾夕法尼亚州(1966年)、堪萨斯州(1971年)、北卡罗来纳州(1977年)、路易斯安那州(1983年)等地(任洁, 2006)。迄今,西花蓟马已遍布美国50个州(Rugman-Jonesetal., 2010)。同时,随着国际贸易往来的日趋频繁,该虫随花卉和蔬菜的远距离运输迅速向世界各地扩散。1983年,西花蓟马首次在荷兰温室的非洲堇SaintpauliaionanthaWendl上被发现,而后迅速在欧洲南部地区建立种群(Brodsgaard, 1993; Kirk & Terry 2003; Vierbergen, 1999)。之后,该蓟马又先后在以色列(1987年)(Argamanetal., 1989)、南非(1987年)(Giliomee, 1989)、马来西亚(1989年)(Fauziah & Saharan, 1991)、日本(1990年)(Hayase & Fukuda, 1991)、澳大利亚(1993年)(Malipatiletal., 1993)、韩国(1994年)(Chungetal., 2000)等国发现,并在局部地区造成严重危害。目前,西花蓟马已成为世界范围内严重制约蔬菜、花卉、果树等产业发展的重要害虫之一(Gerinetal., 1994; Kirk & Terry, 2003)。
早在1997年,我国就将西花蓟马列入《中华人民共和国进境植物检疫潜在危险性病、虫、杂草名录(试行)》[动植检植字(1997)33号]。我国于2003年7月在北京郊区的温室辣椒上发现西花蓟马(张友军等,2003),之后又在云南(2005年)(徐家菊等, 2005)、贵州(2006年)(袁成明等, 2008)、山东(2007年)(郑长英等, 2007)、黑龙江(2007年)(武晓云等, 2009)、湖南(2008年)(刘佳等, 2010)、南京(2008年)(严丹侃等, 2010)、新疆(2010年)(杨华等, 2010)、西藏(2012)(王海鸿等, 2013)等地相继有西花蓟马发生的报道。
以往的研究显示,西花蓟马有2个品系,即温室品系(glasshouse strain)和羽扇豆品系(lupin strain)(Brunner & Frey, 2010; Rugman-Jonesetal., 2010)。其中,温室品系源于美国加利福尼亚州,20世纪80年代,西花蓟马从美国传入荷兰温室蔬菜和花卉集散地后,迅速向整个欧洲和非洲南部蔓延,在当地形成抗药性较强的温室品系(Kirk & Terry, 2003)。羽扇豆品系源于新西兰,早在19世纪30年代从美国加利福尼亚州随羽扇豆传入新西兰(Kirk & Terry, 2003; Martin & Workman, 1994),只危害羽扇豆类植物,对其他作物没有危害(Martin & Workman, 1994);20世纪90年代,羽扇豆品系传至整个美国北部和加拿大南部(Kirk & Terry 2003; Rugman-Jonesetal., 2010)。有研究显示,西花蓟马的2个品系在体色、抗寒性、抗药性等方面均有很大区别(Fellandetal.,1993; Kirk & Terry, 2003; Martin & Workman, 1994; Tsumukietal., 2007)。
入侵害虫的遗传多样性及其扩张趋势研究,是入侵生物学的重要研究内容之一(万方浩等, 2005)。Fangetal.(2005)利用AFLP(amplified fragment length polymorphisim)技术分析以色列不同地理种群西花蓟马的遗传结构,发现各种群内部具有很高的遗传多样性。张治军(2007)利用该技术研究西花蓟马7个不同地理种群(北京、云南、新西兰、荷兰、美国、澳大利亚和日本),分析结果表明,不同地区的西花蓟马种群之间存在明显的遗传分化;聚类分析结果显示,北京种群与荷兰种群聚为一支,推测北京的西花蓟马可能来自荷兰。沈荣登等(2011)应用核糖体DNA中的内转录间隔区ITS2(internal transcribed spacer 2)基因序列对云南各地理种群西花蓟马的遗传结构及其分化进行初步研究,结果表明各地理种群间的遗传分化程度很低,种群间存在基因交流。
本研究针对西花蓟马在我国传播扩散迅速但其遗传结构尚不明晰的问题,以采自我国13个地理区域的西花蓟马为靶标,采用COⅠ分子标记技术,研究分析我国西花蓟马不同地理种群的遗传结构和遗传多样性,研究结果对深入探讨我国西花蓟马的入侵来源、传播扩散途径及其有效阻截具有重要意义。
1 材料与方法
1.1 供试虫源
根据入侵物种传播扩散的可能途径以及我国各省/直辖市工农业发展的特点,以我国经济比较发达的沿海城市、旅游胜地以及矿产资源比较丰富的地区为主要采集区域,进行西花蓟马的系统采集。本研究中,供试西花蓟马样品的采集信息如表1所示。

表1 不同地理种群西花蓟马采集信息Table 1 Collecting information of F.occidentalis from different geographical populations
*中国主要外来入侵昆虫DNA条形码识别系统。
*Database of invasive alien species in China (http:∥www.chinaias.cn).
1.2 总DNA提取
西花蓟马总DNA的提取参照Moritzetal.(2001)的方法并稍加改进。用软毛毛笔轻轻挑取单头蓟马,放在滴有20 μL提取缓冲液(50 mmol·L-1Tris-HCl, 1 mmol·L-1EDTA, 1% SDS, 20 mmol·L-1NaCl, pH 8.0)的Parafilm膜上,以0.2 mL PCR管底部作为匀浆器,充分研磨,匀浆液移入1.5 mL离心管;用50 μL缓冲液清洗匀浆器和Parafilm膜4次,移入同一离心管;加入5 μL蛋白酶K(20 mg·mL-1),充分混匀后于60 ℃水浴1 h(中途混匀1次);加入220 μL氯仿/异戊醇(v∶v=24∶1),轻柔混匀数十次后,冰浴30 min;以4 ℃、12000 r·min-1离心20 min,取上清液(约200 μL)并加入440 μL预冷无水乙醇,轻轻混匀后在-20 ℃冰箱放置30 min;取出后,于4 ℃、12000 r·min-1离心20 min,弃去上清液;加入440 μL预冷75%乙醇洗涤,4 ℃、12000 r·min-1离心15 min,弃去上清液。将离心管倒扣于洁净滤纸上,自然干燥20 min,每管加入20 μL超纯水,充分溶解后于-20 ℃保存备用。以生物分光光度计测定DNA浓度,以D260 nm/D280 nm值确定其是否被污染。
1.3 PCR扩增、电泳检测以及序列测定
COⅠ基因序列扩增所使用的DNA条形码标准引物LCO1490(5′-GGTCAACAAATCATAAAGATATTGG-3′)和HCO2198(5′-TAAACTTCAGGGTGACCAAAAAATCA-3′)(Folmeretal., 1994),由生工生物工程(上海)股份有限公司合成。PCR反应体系25 μL,其中模板DNA 2 μL(约500 ng),10×buffer(含Mg2+)2 μL,dNTPs(0.2 mmol·L-1)0.5 μL,上游引物和下游引物(5 pmol·L-1)各0.5 μL,Taq DNA聚合酶(1.0 U)0.2 μL。反应条件:94 ℃预变性5 min;30个循环:94 ℃ 30 s,48 ℃ 50 s,72 ℃ 50 s;最后72 ℃延伸7 min。取5 μL PCR扩增产物,加2 μL上样缓冲液(0.25%溴酚兰,40%蔗糖水溶液),以DNA Marker为参照,在含有染色剂GoldView的1.5%的琼脂糖凝胶上进行电泳分离(电泳液为0.5×TBE),85 V电泳45 min后,以GelDoc Universal Hood Ⅱ型凝胶成像系统分析结果。将经电泳检测验证合格的PCR产物送北京三博远志生物技术有限责任公司测序(单向测序)。根据标本数量及PCR产物的质量,每个地理种群检测西花蓟马5~25头(表1),共计检测175头,并将175条序列录入中国主要外来入侵昆虫DNA条形码识别系统(http:∥www.chinaias.cn)(表1)。
1.4 序列分析
1.4.1 序列校对及人工比对 Bioedit软件读取COⅠ基因序列,对每条序列的碱基进行人工读取和反复校对。校正后的碱基序列在NCBI上进行同源性比对,以确保所获得的序列为目的基因片段。
1.4.2 遗传多样性分析 应用DnaSP 5.0(Librado & Rozas, 2009)和MEGA 4.0软件(Kumaretal., 2008)分析西花蓟马各地理种群COⅠ基因的序列特征及单倍型多样性(haplotype diversity,Hd)。
序列特征分析主要包括序列的多态性位点(variable/polymorphic sites, V)、碱基组成(base composition)、转换(transition, Ti)和颠换(transversion, Tv)位点数、转换/颠换偏倚率(Ti/Tv bias, R)。多态性位点包括自裔位点(singleton variable sites, Si)和简约信息位点(parsimony informative sites, Pi)。在DNA序列中,碱基恒定的位点和只出现一次变异的位点即为自裔位点;至少有2种变异类型在该位点出现2次或2次以上的,称为简约信息位点。碱基组成主要是指,DNA序列中4种碱基A、T、C、G的相对含量。碱基的替换方式有2种,即转换和颠换,A与G之间或者C与T之间的替换称为转换,A、G与C、T之间的替换称为颠换。
单倍型多样性是指在给定群体中随机抽取到2个不同单倍型的频率,单倍型多样性高的种群说明其遗传多样性高,遗传资源丰富。核苷酸多样性(nucleotide diversity,π)是指在给定群体中随机选取的COⅠ基因序列间的平均每个位点的核苷酸差异数目,是衡量一给定群体遗传多态性的重要指标,π值越大,种群的多样性程度越高(彭奕欣和黄诗笺, 1997)。应用DnaSP 5.0软件分析各地理种群的单倍型多样性和核苷酸多样性(Librado & Rozas, 2009),同时进行Tajima′s D中性检验,以明确其是否遵循中性模型(Tajima, 1989)。
1.4.3 遗传分化和基因流分析 固定指数(genetic fixations index,Fst)表示不同种群间等位基因频率的变异,是反映种群进化历史的重要参数,可在一定程度上揭示种群间基因流和遗传漂变的程度。基因流(gene flow,Nm)指基因在种群内和种群间的运动(Grant, 1985),包括基因从一个种群到另一种群成功运动的所有机制(Slatkni, 1985)。基因流可揭示群体间可能的基因渗透及影响遗传分化的遗传现象。基因流越大,群体间的相似性越大。固定系数与基因流之间的关系为Nm=1/(Fst+1)(Hmailton, 1999)。应用DnaSP 5.0软件(Librado & Rozas, 2009)计算各地理种群间的固定指数和基因流。
固定指数Fst和基因流Nm是表示种群遗传分化程度的重要指标。Fst值越大,表明遗传分化越明显,Nm值与之相反,Nm值越大说明种群间基因交流越充分,遗传分化越小。0
分子变异分析(analysis of molecular variance, AMOVA)是在分子水平上分析种群遗传结构的工具(Excoffieretal., 2005),根据单倍型或基因型间的进化距离(evolutionary distance)对各种群间的遗传差异进行等级划分,AMOVA具有分子数据便于直接计算离差方的优点,本研究应用Arlequin 3.0软件进行分子变异分析(Excoffieretal., 2005)。
1.4.4 系统发育树的构建 采用MEGA 4.0软件按照Kimura双参数模型计算系统发育重建所需的遗传距离(Kumaretal., 2008),以邻接法(neighbor-joining method, NJ)构建单倍型系统发育树,发育树分支的置信度采用自展法(bootstrap analysis, BP)重复检测1000次。
2 结果与分析
2.1 PCR扩增、序列测定及同源性分析
以每个地理种群的单头西花蓟马DNA为模板,通用型引物LCO1490/HCO2198进行PCR扩增,电泳检测结果显示,每个地理种群的西花蓟马均可扩增出清晰的靶标片段(长度约为690 bp)(图1)。对电泳检测验证合格的PCR产物进行纯化和序列测定,得到西花蓟马13个地理种群共计175条COⅠ基因序列。将所有序列采用ClustalX软件比对,修剪成长度为631 bp的标准片段进行分析。利用NCBI中的BLAST程序进行同源性比对,结果显示与数据库中已公开的西花蓟马COⅠ基因序列的同源性为99%~100%,表明所获得的西花蓟马的COⅠ基因序列准确可靠。
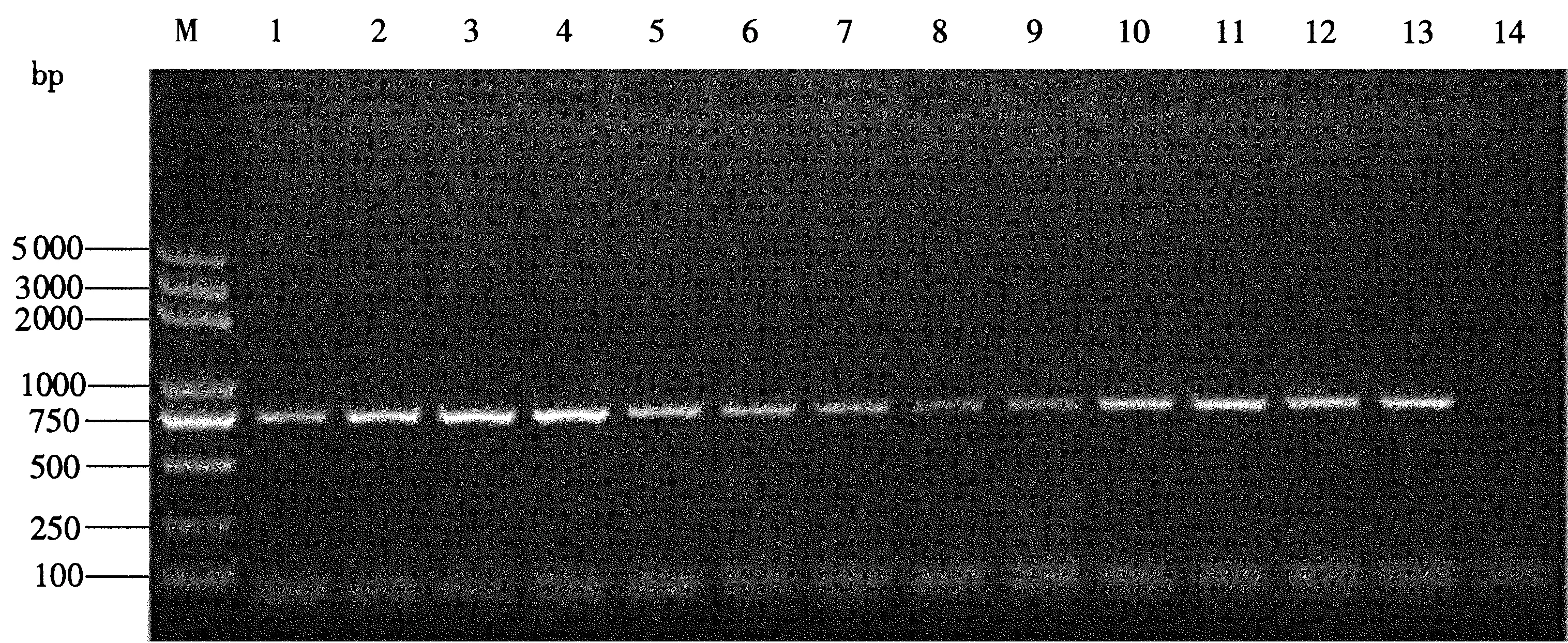
图1 引物LCO1490/HCO2198对不同地理种群西花蓟马COⅠ基因扩增结果电泳检测图Fig.1 Amplification pattern of the COⅠ gene of F.occidentalis from 13 geographical populations using the universal primers LCO1490/HCO2198M:DNA分子质量标准; 1~13:西花蓟马不同地理种群,编号同表1; 14:阴性对照(超纯水)。M: DNA ladder marker; 1~13: Different geographical populations, same as in table 1; 14: Negative control (ultra-pure water).
2.2 西花蓟马COⅠ基因序列变异分析
对西花蓟马不同地理种群共计175条COⅠ基因序列进行变异分析(表2),共检测到596个保守位点,35个多态性位点,多态性位点数占序列总长的5.55%,其中含有12个自裔位点,23个简约信息位点。自裔位点分别位于第22、41、93、101、102、125、126、414、444、565、567和585位;简约信息位点分别位于第84、111、183、213、255、270、294、357、366、378、385、399、411、447、499、504、531、543、558、588、598、606和612位。所有序列的碱基平均含量分别为,A:31.1%,T:38.1%,C:14.5%,G:16.3%,A+T:69.2%。所有序列的总突变数(total number of mutations,η)为70,其中发生转换的位点数为57,发生颠换的位点数为13,转换/颠换偏倚率为4.093(表2)。
2.3 单倍型分布及遗传多样性分析
在所检测的西花蓟马13个地理种群175条序列中,共检测到13种单倍型(haplotype, H)(H1~H13)。其中,H2为优势共享单倍型,在13个种群中均有分布,共享序列共计80条,占序列总数的45.7%;其次为单倍型H3,除河北石家庄SJZ和山东青岛QD种群外,在其他11个种群中均有分布,占序列总数的30.3%;此外,河北石家庄SJZ、黑龙江哈尔滨HEB和宁夏银川YC,共享单倍型H4,所占比例为8.6%;山西太原TY、山东青岛QD和云南昆明KM,共享单倍型H6,所占比例为5.7%;另有9种单倍型,即H1,H5,H7,H8,H9,H10,H11,H12和H13,分别为北京丰台FT、河北石家庄SJZ、吉林四平SP、黑龙江哈尔滨HEB、山东青岛QD(H9,H10)、河南洛阳LY、甘肃兰州LZ和宁夏银川YC等种群所特有(表3)。
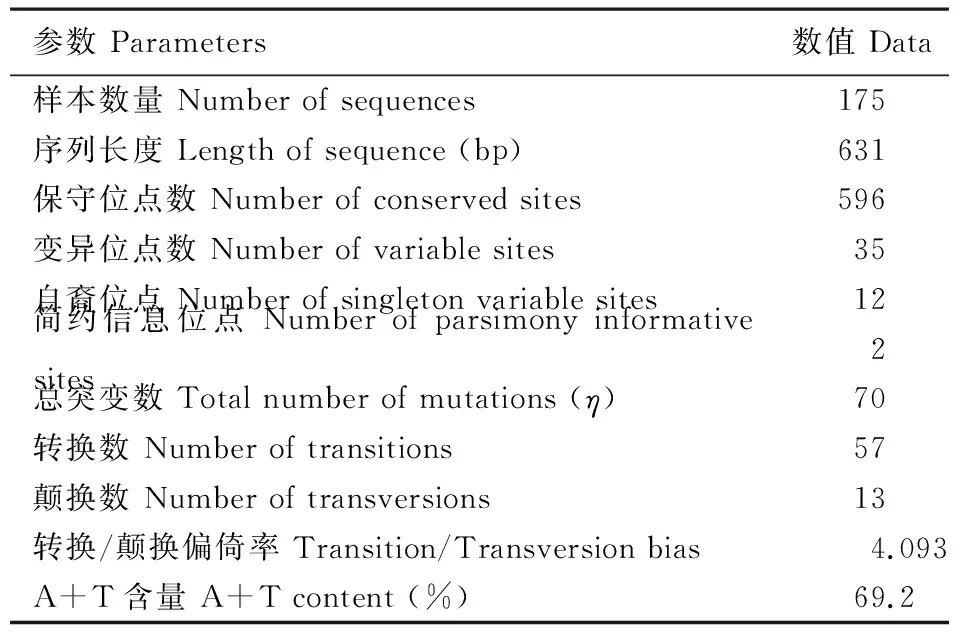
表2 不同地理种群西花蓟马的mtDNA COⅠ基因序列变异分析Table 2 Results of mtDNA COⅠ gene sequences of F.occidentalis from different geographical populations
对单倍型多样性Hd和核苷酸多样性π进行分析,结果表明,总体单倍型多样性Hd0.691,不同地
理种群单倍型多样性范围0.425~0.732,单倍型多样性最高和最低的种群分别为黑龙江哈尔滨HEB(Hd=0.732)和甘肃兰州LZ(Hd=0.425);总体核苷酸多样性π0.00652,不同地理种群核苷酸多样性范围0.00070~0.01594,核苷酸多样度最高和最低的种群分别为宁夏银川YC(π=0.01594)和云南昆明KM(π=0.00070)。对各种群单倍型多样性Hd和核苷酸多样性π进行综合分析,结果表明各种群在地域上存在明显的种群多样性差异,其中,宁夏银川YC(Hd=0.689,π=0.01594)和黑龙江哈尔滨HEB种群(Hd=0.732,π=0.01506)具有较高的多态性,而内蒙古包头BT和陕西宝鸡BJ种群(Hd=0.476,π=0.00075)的多态性较低(表4)。Tajima′s D中性进化模型检验结果显示,除河北石家庄SJZ、黑龙江哈尔滨HEB和宁夏银川YC 3个种群外,其他种群的Tajima′s D检验结果均未达到显著水平。该结果表明,除上述3个种群以外的其他种群的mtDNA COⅠ基因的进化都遵循中性模型。

表3 各单倍型在不同地理种群西花蓟马中的分布Table 3 Haplotypes of mtDNA COⅠ gene in different geographical populations of F.occidentalis
FT:北京丰台; SJZ:石家庄; TY:太原; BT:包头; SY:沈阳; SP:四平; HEB:哈尔滨; QD:青岛; LY:洛阳; KM:昆明; BJ:宝鸡; LZ:兰州; YC:银川。
FT: Fengtai, Beijing; SJZ: Shijiazhuang; TY: Taiyuan; BT: Baotou; SY: Shenyang; SP: Siping; HEB: Harbin; QD: Qingdao; LY: Luoyang; KM: Kunming; BJ: Baoji; LZ: Lanzhou; YC:Yinchuan.
2.4 遗传分化及基因流分析
将所有种群作为一个整体进行分析,其总体固定系数Fst0.24359,变异范围-0.05604~0.55271。总体基因流Nm0.78,变异范围-40.04~35.75(表5)。综合分析显示,我国西花蓟马各地理种群之间可能已发生一定的分化。其中,黑龙江哈尔滨HEB种群与北京丰台FT、山西太原TY、内蒙古包头BT、辽宁沈阳SY、吉林四平SP、山东青岛QD、河南洛阳LY、云南昆明KM、陕西宝鸡BJ和甘肃兰州LZ等10个种群之间可能存在明显分化;宁夏银川YC种群与北京丰台FT、山西太原TY、内蒙古包头BT、辽宁沈阳SY、吉林四平SP、山东青岛QD、河南洛阳LY、云南昆明KM、陕西宝鸡BJ和甘肃兰州LZ等10个种群之间可能存在明显分化;北京丰台FT种群与山西太原TY、黑龙江哈尔滨HEB、山东青岛QD、云南昆明KM、陕西宝鸡BJ、甘肃兰州LZ和宁夏银川YC等7个种群之间可能存在明显分化,与河北石家庄SJZ种群之间存在中度分化;山东青岛QD种群与北京丰台FT、内蒙古包头BT、辽宁沈阳SY、吉林四平SP、黑龙江哈尔滨HEB、河南洛阳LY和宁夏银川YC等7个种群之间可能存在明显分化,与陕西宝鸡BJ、云南昆明KM和甘肃兰州LZ等3个种群之间可能发生了中度分化(表5)。

表4 西花蓟马不同地理种群COⅠ基因单倍型、核苷酸多样性分析及中性检验Table 4 Haplotype diversity, nucleotide diversity and Tajima′s D neutrality test of F.occidentalis from different geographic populations
**P<0.01. FT:北京丰台; SJZ:石家庄; TY:太原; BT:包头; SY:沈阳; SP:四平; HEB:哈尔滨; QD:青岛; LY:洛阳; KM:昆明; BJ:宝鸡; LZ:兰州; YC:银川。
**P<0.01. FT: Fengtai, Beijing; SJZ: Shijiazhuang; TY: Taiyuan; BT: Baotou; SY: Shenyang; SP: Siping; HEB: Harbin; QD: Qingdao; LY: Luoyang; KM: Kunming; BJ: Baoji; LZ: Lanzhou; YC: Yinchuan.

表5 西花蓟马种群间固定指数Fst (下三角形)和基因流Nm (上三角形)Table 5 Genetic fixation index (Fst) (lower triangular) and gene flow (Nm) (upper triangular) between geographical populations of F.occidentalis
FT:北京丰台; SJZ:石家庄; TY:太原; BT:包头; SY:沈阳; SP:四平; HEB:哈尔滨; QD:青岛; LY:洛阳; KM:昆明; BJ:宝鸡; LZ:兰州; YC:银川。
FT: Fengtai, Beijing; SJZ: Shijiazhuang; TY: Taiyuan; BT: Baotou; SY: Shenyang; SP: Siping; HEB: Harbin; QD: Qingdao; LY: Luoyang; KM: Kunming; BJ: Baoji; LZ: Lanzhou; YC: Yinchuan.
2.5 AMOVA分子变异分析
AMOVA分析结果显示,不同种群间的变异占总体变异的12.4%,同一种群内不同个体间变异占87.6%,表明引起种群总体变异的主要因素为种群内个体间的变异(表6)。
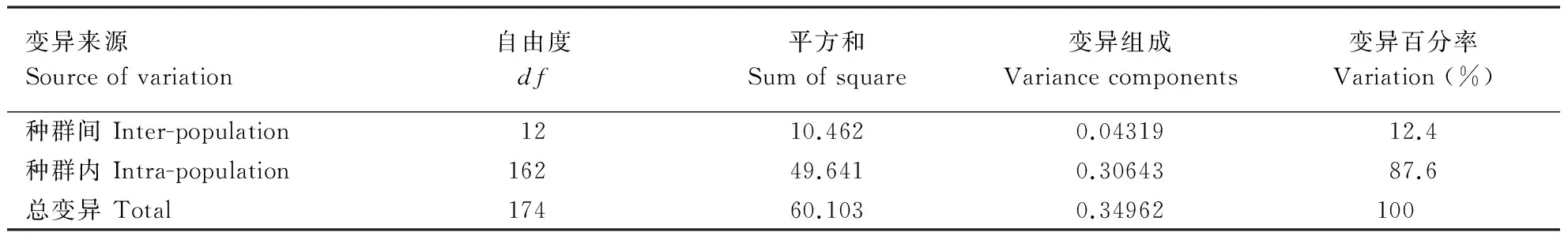
表6 西花蓟马各地理种群 mtDNA COⅠ 基因序列间的分子变异分析Table 6 Analysis of molecular variance among different geographical populations of F.occidentalis
2.6 西花蓟马各地理种群的系统进化分析
2.6.1 基于本研究的西花蓟马13个地理种群的系统进化分析 采用邻接法构建我国西花蓟马各地理种群不同单倍型的系统发育树,并对系统发育树显示的拓扑结构进行分析。结果显示,13个单倍型并没有按照地理分布形成明显的分支格局,各单倍型分散在不同的地理种群中,没有形成明显的、系统的地理种群结构(图2)。整体显示,系统发育树分为明显的两大支。第一支:共享单倍型H2的地理种群和共享单倍型H5的地理种群聚在一起,13个地理种群都共享单倍型H2,其中河北石家庄SJZ种群还共享单倍型H5。此外,共享单倍型H1、H3、H7、H11和H12的地理种群聚在一起;其中,内蒙古包头BT、吉林四平SP、云南昆明KM、山西太原TY、宁夏银川YC、河南洛阳LY、北京丰台FT、甘肃兰州LZ、辽宁沈阳SY、陕西宝鸡BJ和黑龙江哈尔滨HEB等11个种群共享单倍型H3,北京丰台FT种群共享单倍型H1,吉林四平SP种群共享单倍型H7,河南洛阳LY种群共享单倍型H11,甘肃兰州LZ种群共享单倍型H12。同时,共享单倍型H6、H9、H10的地理种群聚在一起;其中,山东青岛QD、山西太原TY和云南昆明KM等种群共享单倍型H6,且山东青岛QD种群还共享单倍型H9和H10(图2)。第二支:共享单倍型H4、H8、H13的地理种群聚在一起,其中河北石家庄SJZ、宁夏银川YC和黑龙江哈尔滨HEB等3个种群共享单倍型H4,黑龙江哈尔滨HEB种群还共享单倍型H8,宁夏银川YC种群共享单倍型H13(图2)。
2.6.2 结合数据库中已公开的西花蓟马不同地理种群的系统进化分析 基于本研究已获得的我国西花蓟马各地理种群不同单倍型COⅠ基因序列,结合GenBank中已公开的国内外不同地理种群西花蓟马的高质量COⅠ基因序列(片段大小为433~680 bp)(表7)[GenBank中已公开的西花蓟马序列多为广泛存在的温室品系序列,只有新西兰的6条序列(EF555794~EF555799)为羽扇豆品系序列](Rugman-Jonesetal., 2010),共同构建系统发育树;同时,进行单倍型(表7)和系统发育关系分析。结果显示,系统发育树分成了明显的两大支(图3)。
第一大支,包括4个分支。其中,第一分支:山西太原TY(H2,n=4)、甘肃兰州LZ(H2,n=12)、吉林四平SP(H2,n=1)、山东青岛QD(H2,n=14;H9,n=6)、北京丰台FT(H2,n=4)、河南洛阳LY(H2,n=4)、黑龙江哈尔滨HEB(H2,n=6)、宁夏银川YC(H2,n=8)、云南昆明KM(H2,n=12)、陕西宝鸡BJ(H2,n=5)、辽宁沈阳SY(H2,n=2)、内蒙古包头BT(H2,n=2)和河北石家庄SJZ(H2,n=6;H5,n=1)等13个种群与GenBank中的北京Beijing(Beijing-2,n=3)和云南Yunnan(Yunnan-2,n=4;Yunnan-3,n=7)种群,以及国外的荷兰Holland(Holland-5,n=3;Holland-3,n=8;Holland-6,n=3)、澳大利亚Australia(Australia-2,n=9)、美国加利福尼亚California(California-3,n=6)和堪萨斯Kansas(Kansas-2,n=6;Kansas-1,n=2)、新西兰New Zealand(New Zealand-4,n=5)和日本Japan(Japan-1,n=7)等种群聚在一起(图3)。第二分支:吉林四平SP(H3,n=3;H7,n=1)、黑龙江哈尔滨HEB(H3,n=7)、内蒙古包头BT(H3,n=5)、陕西宝鸡BJ(H3,n=2)、山西太原TY(H3,n=2)、河南洛阳LY(H3,n=3;H11,n=1)、甘肃兰州LZ(H3,n=3;H12,n=1)、北京丰台FT(H3,n=13;H1,n=3)、辽宁沈阳SY(H3,n=3)、宁夏银川YC(H3,n=3)和云南昆明KM(H3,n=9)等11个种群与GenBank中的北京Beijing(Beijing-4,n=4)和云南Yunnan种群(Yunnan-4,n=7),以及国外的荷兰Holland(Holland-2,n=1)、澳大利亚Australia(Australia-1,n=1)、美国加利福尼亚California(California-1,n=2)和肯尼亚Kenya(Kenya-1,n=1)等种群聚在一起(图3)。第三分支:宁夏银川YC(H4,n=1;H13,n=1)、黑龙江哈尔滨HEB(H4,n=8;H8,n=1)、河北石家庄SJZ(H4,n=1)等种群与GenBank中的北京Beijing(Beijing-1,n=1)和云南Yunnan(Yunnan-1,n=1)种群,以及国外的新西兰New Zealand(New Zealand-2,n=1;New Zealand-3,n=5)和荷兰Holland(Holland-4,n=1)等种群聚在一起(图3)。第四分支:山东青岛QD(H6,n=5;H10,n=2)、云南昆明KM(H6,n=4)和山西太原TY(H6,n=1)等种群与GenBank中的北京Beijing种群(Beijing-3,n=3),以及国外的新西兰New Zealand(New Zealand-1,n=1)、荷兰Holland(Holland-1,n=1)、美国的加利福尼亚California(California-2,n=4)和纽约New York(New York-1,n=1;New York-2,n=2)及英国UK(UK-1,n=1)等种群聚在一起(图3)。
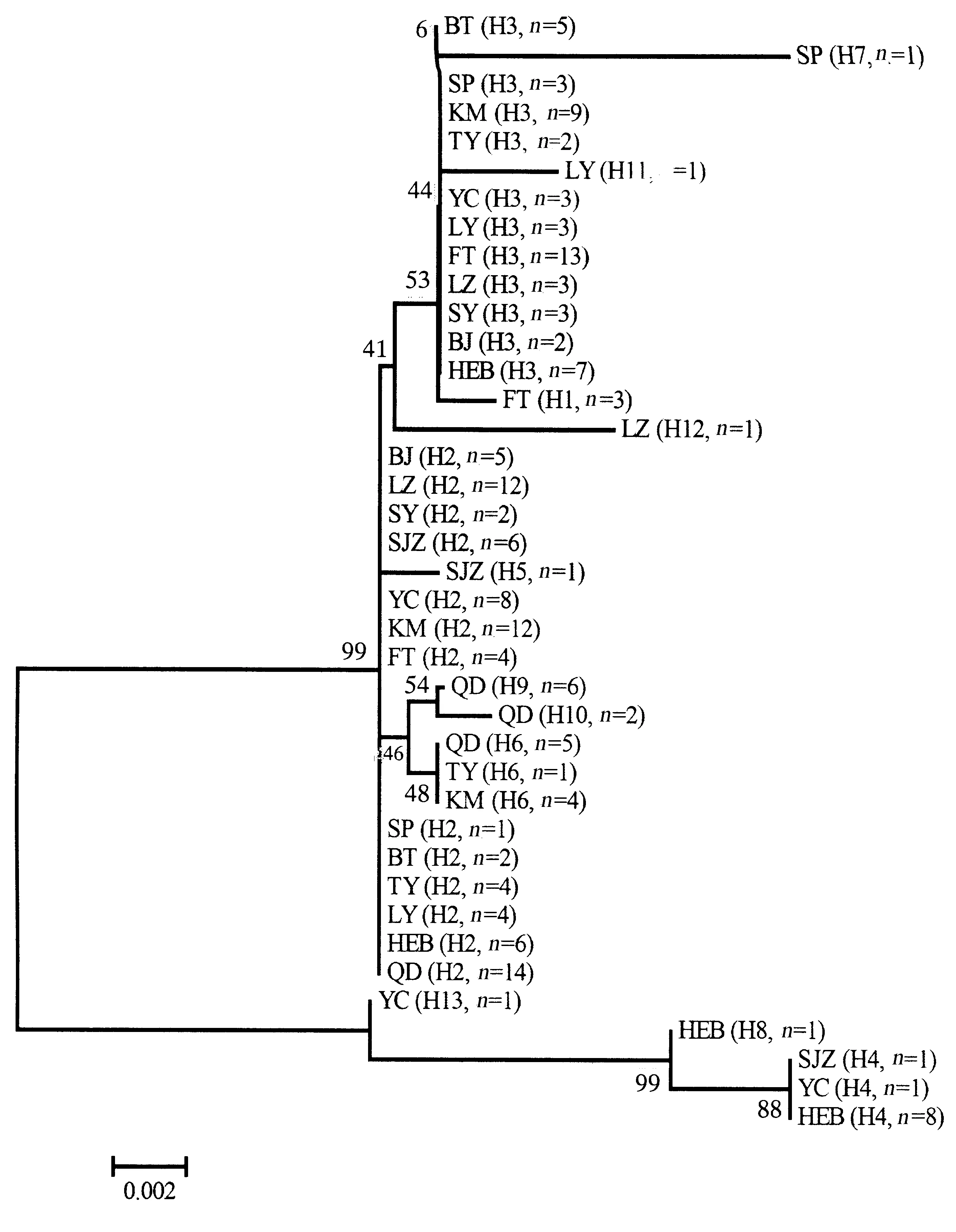
图2 邻接法构建的我国西花蓟马各地理种群不同单倍型系统发育树Fig.2 Neighbor-joining tree based on the analysis of the 13 haplotypes of F.occidentalis from different geographical populations in China分支处上方数值表示重复1000次后的自展值 (>50%); 标尺示遗传距离。BT:内蒙古包头; SP:吉林四平; KM:云南昆明; TY:山西太原; LY:河南洛阳; YC:宁夏银川; LZ:甘肃兰州; BJ:陕西宝鸡; HEB:黑龙江哈尔滨; SY:辽宁沈阳; QD:山东青岛; FT:北京丰台; SJZ:河北石家庄。Numbers on branches are bootstraped values (>50%) under 1000 replicates, the scale bar represents genetic distance.BT: Baotou, Neimenggu; SP: Siping, Jilin; KM: Kunming, Yunnan; TY: Taiyuan, Shanxi; LY: Luoyang, Henan; YC: Yinchuan, Ningxia; LZ: Lanzhou, Gansu; BJ: Baoji, Shaanxi; HEB: Harbin, Heilongjiang; SY: Shenyang, Liaoning; QD: Qingdao, Shandong; FT: Fengtai, Beijing; SJZ: Shijiazhuang, Hebei.
第二大支,南非South Africa(South Africa-1,n=1;South Africa-2,n=2)和意大利Italy种群(Italy-1,n=1;Italy-2,n=1)聚在一起,但未包含本研究的任一种群(图3)。
在国外的所有序列中,只有单倍型编号为New Zealand-2(GenBank登录号:EF555799)和New Zealand-3(GenBank登录号:EF555794~EF555798)的序列为新西兰西花蓟马的羽扇豆品系序列(Rugman-Jonesetal., 2010),其余为广泛分布的温室品系序列。聚类分析还显示,宁夏银川YC种群的2条序列(H4,n=1;H13,n=1)、黑龙江哈尔滨HEB种群的9条序列(H4,n=8;H8,n=1)、河北石家庄SJZ种群的1条序列(H4,n=1)以及GenBank中北京Beijing种群的1条序列(Beijing-1,n=1)和云南Yunnan种群的1条序列(Yunnan-1,n=1),与新西兰西花蓟马的羽扇豆品系序列(GenBank登录号:EF555794~EF555799)聚在一起,表明我国的北京、云南以及宁夏银川、黑龙江哈尔滨、河北石家庄等部分地区已存在一定数量的西花蓟马羽扇豆品系。此外,荷兰的1条序列(Holland-4,n=1)也与新西兰的羽扇豆品系序列聚在一起,表明荷兰亦存在西花蓟马的羽扇豆品系(图3)。本研究中13个地理种群的其余序列,均为温室品系序列,其可能的入侵来源推测如下:河北石家庄SJZ种群的序列与荷兰、澳大利亚、美国加利福尼亚、美国堪萨斯、新西兰、日本等种群的序列聚在一起,推测该种群可能来源于上述6个国家或地区(图4A);北京丰台FT、河南洛阳LY、黑龙江哈尔滨HEB、辽宁沈阳SY、吉林四平SP、宁夏银川YC、陕西宝鸡BJ、内蒙古包头BT、山西太原TY、甘肃兰州LZ等10个种群的序列与荷兰、澳大利亚、美国加利福尼亚、美国堪萨斯、新西兰、日本、肯尼亚等种群的序列聚在一起,推测我国上述10个区域的西花蓟马可能来源于这7个国家或地区(图4B);山东青岛QD种群的序列与荷兰、澳大利亚、美国加利福尼亚、美国堪萨斯、新西兰、日本、美国纽约、英国等种群的序列聚在一起,推测该种群可能来源于上述这8个国家或地区(图4C);云南昆明KM种群的序列与荷兰、澳大利亚、美国加利福尼亚、美国堪萨斯、新西兰、日本、肯尼亚、美国纽约、英国等种群的序列聚在一起,推测该种群可能来源于上述这9个国家或地区(图4D)。此结果说明,我国西花蓟马温室品系的各地理种群存在多个入侵来源。

表7 NCBI数据库中西花蓟马不同地理种群高质量COⅠ基因序列登录号Table 7 Accession numbers of partial COⅠ gene sequences of different geographical populations of F.occidentalis from GenBank

图3 邻接法构建的西花蓟马不同地理种群COⅠ基因序列系统发育树Fig.3 Neighbor-joining tree based on analysis of COⅠ gene sequences of F.occidentalis from different geographical populations分支处上方数值表示重复1000次后的自展值(>50%); 标尺示遗传距离。BT:内蒙古包头; SP:吉林四平; KM:云南昆明; TY:山西太原; LY:河南洛阳; YC:宁夏银川; LZ:甘肃兰州; BJ:陕西宝鸡; HEB:黑龙江哈尔滨; SY:辽宁沈阳; QD:山东青岛; FT:北京丰台; SJZ:河北石家庄; Beijing:北京; Yunnan:云南; New Zealand:新西兰; Australia:澳大利亚; Japan:日本; Holland:荷兰; South Africa:南非; Italy:意大利; Kenya:肯尼亚; UK:英国; California:加利福尼亚; Kansas:堪萨斯; New York:纽约。 Numbers on branches are bootstraped values (>50%) under 1000 replicates, the scale bar represents genetic distance.BT: Baotou, Neimenggu; SP: Siping, Jilin; KM: Kunming, Yunnan; TY: Taiyuan, Shanxi; LY: Luoyang, Henan; YC: Yinchuan, Ningxia; LZ: Lanzhou, Gansu; BJ: Baoji, Shaanxi; HEB: Harbin, Heilongjiang; SY: Shenyang, Liaoning; QD: Qingdao, Shandong; FT: Fengtai, Beijing; SJZ: Shijiazhuang, Hebei.
3 小结与讨论
本研究利用mtDNA COⅠ基因序列分析我国西花蓟马的种群多样性和遗传结构,并以此推测西花蓟马的来源和传播扩散途径。研究涉及我国西花蓟马13个地理种群。研究结果显示,无论是西花蓟马种群的覆盖度还是每一种群的样本数量,均符合研究物种种群遗传关系分析的基本需求(Zhangetal., 2010)。
对西花蓟马13个地理种群共计175条COⅠ基因碱基序列进行变异分析,共检测到596个保守位点,35个多态性位点,其中自裔位点12个,简约信息位点23个。所有序列中A+T含量为69.2%,表现出明显的A/T碱基偏向性,与昆虫线粒体基因组的碱基组成特征基本一致(Simonetal., 1994)。Kinght & Mindell(1993)研究指出,转换/颠换偏倚率的临界值为2,当小于2时表明基因突变已达到饱和状态,大于2时表明基因突变尚未达到饱和状态。本研究中13个西花蓟马地理种群所有序列的总突变数η为70,发生转换和颠换的位点数分别为57和13,转换/颠换偏倚率为4.093,说明我国西花蓟马COⅠ基因碱基序列的突变尚未达到饱和状态。

图4 西花蓟马河北石家庄SJZ (A)、北京丰台FT、河南洛阳LY、黑龙江哈尔滨HEB、辽宁沈阳SY、吉林四平SP、宁夏银川YC、陕西宝鸡BJ、内蒙古包头BT、山西太原TY、甘肃兰州LZ (B)、山东青岛QD (C)和云南昆明KM (D)等种群入侵来源地示意图Fig.4 Diagram showing the sources of F.occidentalis of Shijiazhuang, Hebei (A), Fengtai, Beijing; Luoyang, Henan; Harbin, Heilongjiang; Shenyang, Liaoning; Siping, Jilin; Yinchuan, Ningxia; Baoji, Shaanxi; Baotou, Neimenggu; Taiyuan, Shanxi; and Lanzhou, Gansu (B), Qingdao, Shandong (C) and Kunming, Yunnan (D) populations绿色圆圈表示河北石家庄SJZ种群; CA:加利福尼亚; KS:堪萨斯; NY:纽约; UK:英国; NL:荷兰; KE:肯尼亚; JP:日本; AU:澳大利亚; NZ: 新西兰。Green circle indicates population Shijiazhuang, Hebei; CA: California; KS: Kansas; NY: New York; UK: United Kingdom; NL: Holland; KE: Kenya; JP: Japan; AU: Australia; NZ: New Zealand.
单倍型多样性Hd和核苷酸多样性π是衡量一个种群线粒体DNA遗传多样性的2个重要指标。本研究对13个西花蓟马地理种群、175条COⅠ基因序列的单倍型多样性和核苷酸多样性的检测结果显示,13个地理种群共有13种单倍型。进一步的综合分析显示,其总体单倍型多样性较高,为0.691;但核苷酸多态性较低,仅为0.00652。其中,黑龙江哈尔滨HEB、宁夏银川YC、云南昆明KM、山东青岛QD、河南洛阳LY、山西太原TY、辽宁沈阳SY等种群均表现出较低的单倍型多样性;而核苷酸多样性较高的种群只有黑龙江哈尔滨HEB和宁夏银川YC种群,从而可能会使这2个种群与我国目前发现的83%以上的种群发生遗传分化。因此,我国西花蓟马可能是由一较小的有效种群迅速增长而来,虽然通过变异积累了一定的单倍型多样性,但却尚未积累足够的核苷酸序列多样性(Avise, 1998)。
固定指数Fst和基因流Nm是衡量种群遗传分化的重要指标。本研究对西花蓟马13个地理种群总体以及各种群之间的固定指数Fst和基因流Nm的分析结果显示,各地理种群间可能存在一定程度的分化,其中黑龙江哈尔滨HEB、宁夏银川YC、北京丰台FT、山东青岛QD等种群与其他种群之间可能出现了明显分化。而不同种群间出现遗传分化的原因,一方面可能是由于不同的地理分布区域,其温度、湿度、寄主植物等环境因素不同,进而导致西花蓟马在适应不同的环境条件过程中可能出现一定的遗传分化;另一方面可能与西花蓟马的入侵来源地不同有关;而后者所起作用可能更为重要。
武晓云等(2009)以COⅠ基因通用型引物mtD-7.2F/mtD-9.2R扩增北京、云南昆明、黑龙江哈尔滨等三地,共计29头西花蓟马的COⅠ基因片段(长度为433 bp,包含在本研究的650 bp靶标序列之中,对齐位置约从200 bp开始),通过聚类分析显示,上述三地的部分COⅠ基因序列与数据库中已公开的新西兰西花蓟马的羽扇豆品系序列(EF555794~EF555799)聚在一起,表明上述三地的种群都存在一定数量的羽扇豆品系,其来源地可能是新西兰。本研究以我国西花蓟马不同地理种群单倍型序列和国外不同地理种群单倍型序列共同构建系统发育树,聚类分析结果显示,我国西花蓟马亦存在2个品系,即羽扇豆品系和温室品系。我国的北京丰台、云南昆明、宁夏银川、黑龙江哈尔滨、河北石家庄种群中已存在一定数量的羽扇豆品系,上述区域的部分西花蓟马可能来源于新西兰或荷兰。而广泛存在于我国的不同地理种群的西花蓟马温室品系存在多个入侵来源地。
随着国际贸易以及国内地区之间水果、蔬菜、花卉及其种苗的调运或展览展示,可能导致同一地理种群的西花蓟马存在多个入侵来源,或同一来源的多次入侵,或多个来源的多次入侵。该研究结果对西花蓟马的有效阻截及其种群扩张趋势监测意义重大。
刘佳, 张林, 卢焰梅, 张宏瑞. 2010. 湖南外来入侵害虫西花蓟马初步调查. 安徽农业科学, 38(25): 13800-13801.
彭奕欣, 黄诗笺. 1997. 进化生物学. 长沙: 武汉大学出版社.
任洁. 2006. 西花蓟马Frankliniellaoccidentalis(Pergande)生物学特性及其控制技术研究. 杨凌: 西北农林科技大学.
沈登荣, 张宏瑞, 李正跃, 董坤, 和绍禹. 2011. 云南西花蓟马rDNA-ITS2遗传多态性及其种群扩张. 应用昆虫学报, 48(3): 504-512.
万方浩, 郑小波, 郭建英. 2005. 重要农林外来入侵物种的生物学与控制. 北京: 科学出版社.
王海鸿, 雷仲仁, 李雪, 代安国, 陈翰秋. 2013. 西藏发现重要外来入侵害虫——西花蓟马. 植物保护, 39(1): 187-189.
武晓云, 程晓非, 张仲恺, 桂富荣, 李正跃. 2009. 西花蓟马 (Frankliniellaoccidentalis) rDNA ITS2和COⅠ基因5′末端序列的克隆与比较分析. 浙江大学学报: 农业与生命科学版, 35(4): 355-364.
徐家菊, 韦丽莉, 禹崇云, 包丽琼, 林兴华, 彭贵清, 赵满云. 2005. 临沧市新发现外来有害生物——西花蓟马. 植物检疫, 19(5): 23-24.
严丹侃, 汤云霞, 贺子义, 孙雷, 王鸣华, 薛晓峰, 范加勤. 2010. 南京地区西花蓟马发生调查及其分子检测. 南京农业大学学报, 33(4): 59-63.
杨华, 崔元玗, 张升, 孙晓军. 2010. 危险性外来入侵生物——西花蓟马在新疆发生危害. 新疆农业科学, 47(11): 2252-2253.
袁成明, 郅军锐, 李景柱, 张勇. 2008. 贵州省蔬菜蓟马的种类、分布及综合防治. 湖北农业科学, 47(12): 1442-1444.
张友军, 吴青君, 徐宝云, 朱国仁. 2003. 危险性外来入侵生物——西花蓟马在北京发生危害. 植物保护, 29(4): 58-59.
张治军. 2007. 西花蓟马生物学特性及不同地理种群遗传分化研究. 北京: 中国农业大学.
郑长英, 刘云虹, 张乃芹, 赵希丽. 2007. 山东省发现外来入侵有害生物西花蓟马. 青岛农业大学学报: 自然科学版, 24(3): 172-174.
Allendorf F W. 1983. Isolation, gene flow and genetic differentiation among populations∥Schonewald-Cox C M, Chambers S M, MacBryde B and Thomas W L.GeneticsandConservation:AReferenceforManagingWildAnimalandPlantPopulations. Benjamin/Cummings, New York, 51-65.
Argaman Q, Klein Z, Ben-Dov Y and Mendel Z. 1989.Frankliniellaoccidentalis(Pergande) (Thysanoptera: Thripidae), an injurious intruder.Hassadeh, 69: 1268-1269.
Avise J C. 1998. The history and preview of phylogeograhy: a personal reflection.MolecularEcology, 7: 371-379.
Balloux F and Lugon-Moulin N. 2002. The estimation of population differentiation with microsatellite markers.MolecularEcology, 11: 155-165.
Beshear R J J. 1983. New records of thrips in Georgia (Thysanoptera, Terebrantia, Tubulifera).JournaloftheGeorgiaEntomologicalSociety, 18: 342-344.
Brodsgaard H F. 1993. Cold hardiness and tolerance to submergence in water inFrankliniellaoccidentalis(Thysanoptera: Thripidae).EnvironmentalEntomology, 22: 647-653.
Brunner P C and Frey J E. 2010. Habitat-specific population structure in native western flower thripsFrankliniellaoccidentalis(Insecta, Thysanoptera).JournalofEnvironmentalBiology, 23: 797-804.
Chung B K, Kang S W and Kwon J H. 2000. Chemical control system ofFrankliniellaoccidentalis(Pergande) (Thysanoptera: Thripidae) in greenhouse eggplant.JournalofAsia-PacificEntomology, 3: 1-9.
Excoffier L, Laval G and Schneider S. 2005. Arlequin (version 3.0): an integrated software package for population genetics data analysis.EvolutionaryBioinformaticsOnline, 1: 47-50.
Fang J, Kritzman A, Yonash N, Gera A, Pollak N and Lavi U. 2005. Genetic variation of thrips populations assessed by amplified fragment length polymorphism (Thysanoptera: Thripidae).AnnalsoftheEntomologicalSocietyofAmerica, 98: 351-358.
Fauziah I and Saharan H A. 1991.ResearchonThripsinMalaysia. Asian Vegetable Research and Development Center Publication.
Felland C M, Hull L A, Teulon D A J and Cameron E A. 1993. Overwintering of western flower thrips (Thysanoptera: Thripidae) in Pennsylvania.TheCanadianEntomologist, 125: 971-973.
Folmer O, Black M, Hoeh W, Lutz R and Vrijenhoek R. 1994. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit Ⅰ from diverse metazoan invertebrates.MolecularMarineBiologyandBiotechnology, 3: 294-299.
Gerin C, Hance T and Van Impe G. 1994. Demographical parameters ofFrankliniellaoccidentalis.JournalofAppliedEntomology, 118: 370-377.
Giliomee J H. 1989. First record of western flower thripsFrankliniellaoccidentalis(Pergande) (Thysanoptera: Thripidae) in South Africa.JournaloftheEntomologicalSocietyofSouthernAfrica, 52: 179-180.
Grant V. 1985.TheEvolutionaryProcess:ACriticalReviewofEvolutionaryTheory. New York: Columbia University Press.
Hayase T and Fukuda H. 1991. Occurrence of the western flower thrips,Frankliniellaoccidentalis(Pergande), on the cyclamen and its identification.PlantProtection, 45: 59-61.
Hmailton M B. 1999. Tropical tree gene flow and seed dispersal.Nature, 401: 129-130.
Kinght A and Mindell D P. 1993. Substitution bias, weighting of DNA sequences evolution, and the phylogenetic positions of fea′s viper.SystematicBiology, 42: 18-31.
Kirk W D J and Terry L I. 2003. The spread of the western flower thripsFrankliniellaoccidentalis(Pergande).AgriculturalandForestEntomology, 5: 301-310.
Kumar S, Nei M, Dudley J and Tamura K. 2008. MEGA: A biologist-centric software for evolutionary analysis of DNA and protein sequences.BriefingsinBioinformatics, 9: 299-306.
Librado P and Rozas J. 2009. DnaSP v5: A software for comprehensive analysis of DNA polymorphism data.Bioinformatics, 25: 1451-1452.
Malipatil M B, Postle A C, Osmelak J A, Hill M and Moran J. 1993. First record of western flower thripsFrankliniellaoccidentalis(Pergande) (Thysanoptera: Thripidae) in Australia.JournaloftheAuatralianEntomologicalSociety, 32: 378.
Martin N A and Workman P J. 1994. Confirmation of a pesticide-resistant strain of western flower thrips in New Zealand∥Proceedingsofthe47thNewZealandPlantProtectionConference,HorticulturalCrops, 144-148.
Millar C L and Libby W J. 1991. Strategies for conserving clinal, ecotypic, and disjunct population diversity in widespread species∥Fald D A and Holsinger K E.GeneticsandConservationofRarePlants. New York: Oxford University Press, 149-170.
Moritz G, Paulsen M, Delker C, Picl S and Kumm S. 2001. Identification of thrips using ITS-RFLP analysis∥Marullo R and Mound L A.ThripsandTospoviruses:Proceedingsofthe7thInternationalSymposiumonThysanoptera. Canberra: Australian National Insect Collection CSIRO Publishing, 365-367.
Rugman-Jones P F, Hoddle M S and Stouthamer R. 2010. Nuclear-mitochondrial barcoding exposes the global pest western flower thrips (Thysanoptera: Thripidae) as two sympatric cryptic species in its native California.JournalofEconomicEntomology, 103: 877-886.
Simon C, Frati F, Beckenbach A, Crespi B, Liu H and Flook P. 1994. Evolution, weighting, and phylogenetic utility of mitochondrial gene sequences and a compilation of conserved polymerase chain reaction primers.AnnalsoftheEntomologicalSocietyofAmerica, 87: 651-701.
Slatkin M. 1985. Gene flow in natural populations.AnnualReviewofEcologyandSystematics, 16: 393-430.
Tajima F. 1989. Statistical method for testing the neutral mutation hypothesis for DNA polymorphism.Genetics, 123: 585-595.
Tsumuki H, Ishida H, Yoshida H, Sonoda S, Izumi Y and Murai T. 2007. Cold hardiness of adult western flower thrips,Frankliniellaoccidentalis(Pergande) (Thysanoptera: Thripidae).AppliedEntomologyandZoology, 42: 223-229.
Vierbergen G. 1999. Risks of Thysanoptera detected on imported plant products: the Dutch experience∥Vierbergen G and Tunç I.Proceedings6thInternationalSymposiumonThysanoptera. Akdeniz University, Antalya, Turkey, 99-106.
Zhang A B, He L J, Crozier R H, Muster C and Zhu C D. 2010. Estimating sample sizes for DNA barcoding.MolecularPhylogeneticsandEvolution, 54: 1035-1039.

