α-淀粉酶在不同地衣芽孢杆菌宿主菌中分泌表达的对比分析
陈敬帮,周银华,赵新宇,2,陈守文,魏雪团,2,*
α-淀粉酶在不同地衣芽孢杆菌宿主菌中分泌表达的对比分析
陈敬帮1,周银华1,赵新宇1,2,陈守文1,魏雪团1,2,*
(1.华中农业大学 农业微生物学国家重点实验室,湖北 武汉 430070;2.华中农业大学食品科学技术学院,湖北 武汉 430070)
研究不同地衣芽孢杆菌(Bacillus licheniformis)宿主菌对α-淀粉酶分泌表 达的影响。以Bacillus subtilis的P43启动子,B. licheniformisWX-02α-淀粉酶基因的信号肽、编码区和终止子序列为表达原件,构建了α-淀粉酶分泌表达载体pP43SAT。将pP43SAT分别转入3株B. licheniformis宿主菌:WX-02(ΔamyL)、BL9和BL10,获得3株α-淀粉酶基因工程菌WX-02(ΔamyL,pP43SAT)、BL9(pP43SAT)和BL10(pP43SAT),并将构建的3株工程菌进行发酵对比分析。BL9(pP43SAT)和BL10(pP43SAT)的淀粉酶发酵活力分别达到94.01 U/mL和101.94 U/mL,比原始宿主菌WX-02(ΔamyL,pP43SAT)分别提高了21%和31%,这说明BL9和BL10新型宿主菌有利于淀粉酶的分泌表达。本研究证明多蛋白酶基因缺失可显著提高α-淀粉酶的表达,为α-淀粉酶的高效表达提供了新型宿主菌和新策略。
α-淀粉酶;地衣芽孢杆菌;宿主菌;分泌表达
α-淀粉酶可水解淀粉,产生糊精、低聚糖和单糖,在食品、医药和纺织品领域应用广泛[1-2]。基因工程技术常用来提高目标蛋白的产量,李金霞等[3]通过大肠杆菌宿主菌实现了α-淀粉酶的表达,但大肠杆菌表达系统易形成包涵体,且外膜含有对人和动物有毒性的内毒素脂多糖,产物分离纯化比较困难[4]。相比之下,地衣芽孢杆菌为食品级微生物,具有较高的安全性,且最适生长温度高,生长速度快,细胞壁组成简单,蛋白分泌能力强[5]。王凤寰等[6]通过同源整合技术,构建了含透明颤菌血红蛋白基因的地衣芽孢杆菌宿主菌,显著提高了α-淀粉酶的发酵活性。牛丹丹等[7]采用同源介导α-淀粉酶基因扩增技术,提高了基因拷贝数,显著提高了α-淀粉酶的发酵活性。杨艳华等[8]通过过量表达调控基因degQ,显著提高了α-淀粉酶的表达水平。Ghang等[9]选择不同酿酒酵母表达α-淀粉酶基因,最大淀粉酶活性提高了10倍,而Galdino等[10]发现α-淀粉酶在酿酒酵母中的表达活力仅为3.93 U/mL。由此可见,宿主菌对外源蛋白的表达影响很大,宿主菌的改造对α-淀粉酶的发酵生产至关重要。
宿主菌的胞外蛋白酶可降解目标蛋白,从而降低目标蛋白的表达量。因此,缺失其胞外蛋白酶可能抑制目标蛋白的降解,从而提高目标蛋白的表达量。Ignatova等[11]采用不同的大肠杆菌宿主菌表达青霉素酰化酶,发现多蛋白酶基因缺失可显著提高青霉素酰化酶的表达量。Nguyen等[12]利用8蛋白酶基因缺失的枯草芽孢杆菌WB800为宿主菌,显著提高了纳豆激酶的表达量。WX-02为本课题组前期筛选的一株地衣芽孢杆菌[13],可高产聚谷氨酸[14-15]、2,3-丁二醇[16]和乙偶姻[17],在WX-02的基础上,通过叠加敲除的手段分别构建了多蛋白酶基因缺失的新型宿主菌,前期研究结果证实多基因缺失宿主菌可显著提高纳豆激酶的表达量[18]。多蛋白酶基因缺失的宿主菌还未应用于α-淀粉酶的表达,无法理解宿主菌蛋白酶缺失对α-淀粉酶的影响,阻碍该策略在α-淀粉酶表达中的应用。本研究以前期构建的多基因缺失宿主菌BL9和BL10为宿主菌进行α-淀粉酶的分泌表达,评价多蛋白酶基因缺失对α-淀粉酶表达的影响,为α-淀粉酶的高效表达提供新思路。
1 材料与方法
1.1 材料与试剂
1.1.1菌株
表1为实验所用菌株。其中BL9缺失7个蛋白酶基因(mpr、vpr、aprX、epr、bpr、wprA、aprE、amyL)和1个鞭毛蛋白基因(hag),同时为了排除自身基因组中α-淀粉酶基因对α-淀粉酶表达的影响,还缺失了α-淀粉酶基因(amyL),BL10是在BL9的基础上进一步敲除了蛋白酶基因bprA。
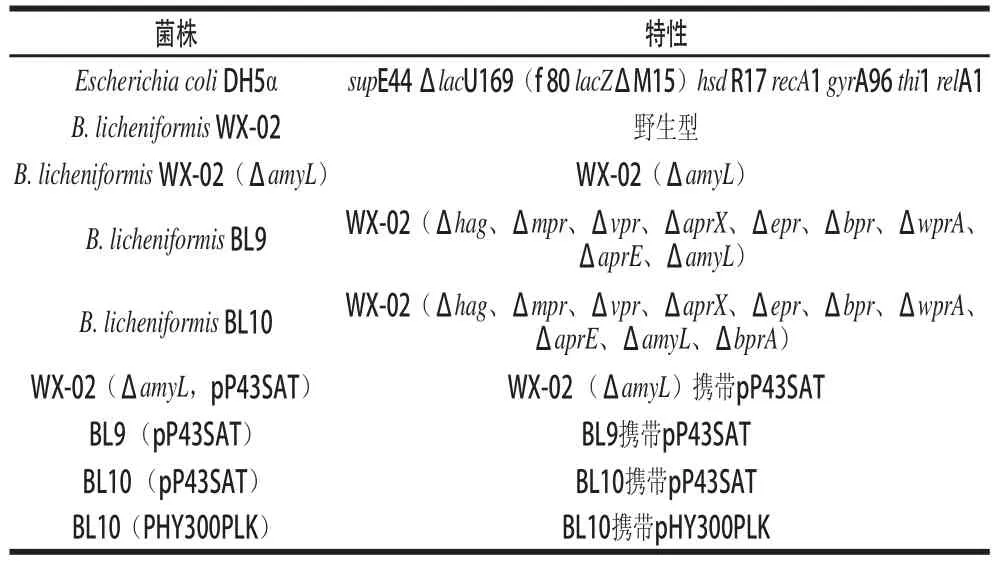
表1 实验所用菌株Table1 A list of the strains used in this study
1.1.2试剂
DNA聚合酶、dNTPs、各种DNA限制性内切酶、T4 DNA连接酶、RNA酶、蛋白质标准Marker、DNA Marker日本TaKaRa公司;DNA回收试剂盒 武汉思特进科技发展有限公司;琼脂糖 法国Biowest公司;氨苄青霉素(ampicillin,Amp)、四环素(tetracycline,Tet)、蛋白胶配制所需试剂 美国Sigma公司;其他主要生化试剂购自国药集团化学试剂有限公司。
1.1.3培养基
LB培养基:蛋白胨10 g/L、酵母浸出粉5 g/L、NaCl 10 g/L,pH 7.0~7.2,固体培养基加琼脂15 g/L。
芽孢杆菌感受态制备培养基:生长培养基为含0.5 mol/L山梨醇的LB培养基;洗涤培养基为0.5 mol/L山梨醇,0.5 mol/L甘露醇,10%甘油;恢复培养基为含0.5 mol/L山梨醇和0.38 mol/L甘露醇的LB培养基。
淀粉酶发酵培养基:玉米浆干粉5 g/L、酵母粉5 g/L、蛋白胨10 g/L、柠檬酸钠12 g/L、K2HPO4·H2O 1 g/L、MgSO4·7H2O 0.5 g/L、CaCl2·2H2O 0.15 g/L,pH 7.0~7.2。
抗生素质量浓度:大肠杆菌克隆筛选和培养使用Amp质量浓度为100μg/mL;芽孢杆菌转化子筛选使用Tet质量浓度为20μg/mL,重组菌株的培养使用Tet质量浓度为25μg/mL。
1.2仪器与设备
凝胶成像系统 美国Alpha公司;GenePulserTM电脉冲转化仪 美国Bio-Rad公司;HQL300B恒温大幅振荡摇床 武汉中科科仪技术发展有限责任公司;高压灭菌锅 日本三洋公司;电子分析天平(万分之一)德国Sartorius Analytic公司。
1.3方法
1.3.1α-淀粉酶分泌表达载体构建
α-淀粉酶分泌表达载体构建参考本课题组先前报道的方法[19],构建步骤如图1所示。根据B. licheniformisWX-02基因组序列中的amyL基因序列(MUY_00877),设计一对引物PamyLF1(5’-GAGAGGAATGTACA CATGAAATGAAACAACAAAAACGGCTT-3’)和PamyTR(5’-GAAGATCTCGCAATAATGCCGTCGCAC TG-3’),以B. licheniformisWX-02的总DNA为模板,聚合酶链 式反应(polymerase chain rea ction,PCR)扩增得到α-淀粉酶基因的编码区(包括信号肽)片段和终止子片段amyL+TamyL,长度约2 040 bp。 根据B. subtilis168基因组上P43启动子的序列设计一对引物P43F(5’-GG AATTCTGATAGGTGGTATGTTTTCG-3’)和P43R1(5’-AAGCCGTTTTTGTTGTTTCATTTCATGTGTACA TTCCTCTC-3’),PCR扩增得到启动子P43片段,长度约305 bp,将这两个片段经DNA纯化试剂盒纯化回收后作为模板,用重叠延伸PCR法(splicing overlap extension PCR,SOE-PCR)连接在一起,得到片段SAT,总长度约2 345 bp,纯化回收后,用EcoRⅠ和BglⅡ双酶切,纯化回收片段SAT。然后将pHY300PLK空质粒用同样的酶双酶切,纯化回收后与前面得到的片段连接。连接产物转化E. coliDH5α,转化子经菌落PCR验证、质粒抽提和双酶切验证,获得的重组α-淀粉酶分泌表达质粒命名为pP43SAT,长度约4 870 bp。
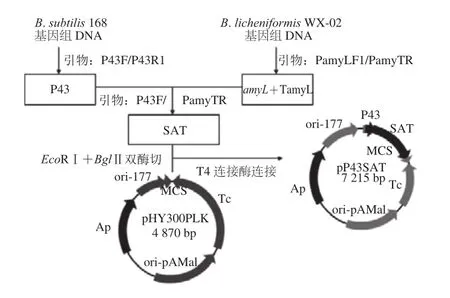
图 1 1 α-淀粉酶表达载体pP43SAT的构建过程Fig.1 Construction process of α-amylase expression vector (pP43SAT)
1.3.2α-淀粉酶重组表达菌株的构建
重组表达菌株参考本课题组先前报道的方法构建[18]。分别接种B. licheniformisBL9和BL10于LB培养基,37℃、180 r/min过夜培养,转接至电转化生长培养基中,250 r/min、37℃培养至OD600nm为0.8~0.9,冰浴10 min,5 500 r/min离心6 m in,收集菌体,用电转化洗涤培养基洗涤菌体3次,重悬于洗涤培养基中,即为感受态细胞。取感受态细胞加入5μLpP43SAT质粒DNA(50 ng/μL),转至预冷的电转化杯(0.2 cm)中,冰浴1~1.5 min,用电脉冲转化仪2.5 kV电击1次,迅速加900μL电转化恢复培养基,37℃水浴1 h,100 r/min恢复培养2 h,涂含有20μg/mL的四环素抗性平板,37℃培养20 h,挑取转化子,经菌落PCR验证、质粒抽提验证,获得阳性转化子,通过该方法构建重组表达菌株B. licheniformisBL9(pP43SAT)和BL10(pP43SAT),并构建含有空质粒pHY300PLK的对照表达菌株。
1.3.3重组发酵菌株的发酵培养
将B. licheniformisBL9(pP43SAT),BL10(pP43SAT)和对照菌株接种于5 mL的含四环素(20μg/mL)的LB培养基中,37℃、180 r/min培养过夜。再以4%的接种量转接到50 mL新鲜的LB培养基中,直到OD600nm为1.0时,以1%的接种量接种到50 mL新鲜的淀粉酶发酵培养基中,37℃、180 r/min振荡培养。每隔4 h取样一次,测定 其OD600nm和淀粉酶活力,取发酵终点的发酵液用于十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate-polyacrylamide gel electrophoresis,SDS-PAGE)分析,鉴定淀粉酶的表达。
1.3.4表达蛋白的鉴 定
取0.9 mL发酵上清液,加入0.1 mL 100 g/100 mL的三氯乙酸(trichloroacetic acid,TCA)溶液,颠倒10次混匀,4℃过夜放置,13 000 r/min离心10 min,弃上清,加入0.2 mL无水乙醇洗涤,13 000 r/min离心10 min,弃上清,依此重复洗涤3次,37℃吹干,加入30μL包含8 mol/L尿素和2 mol/L硫脲的溶液溶解蛋白 样品,再加入30μL2×SDS-PAGE上样缓冲液和3μL1 mol/L二硫苏糖醇(DL-dithiothreitol,DTT),100℃水浴10 min溶解沉淀。SDS-PAGE凝胶参考《分子克隆实验指南》进行配制[20]。将溶解好的蛋白离心后取20μL上样,用80 V的电压电泳,当溴酚蓝指示剂从浓缩 胶进入分离胶后,电压调到120 V,继续电泳直到溴酚蓝指示剂抵达分离胶底部,断开电源停止电泳。电泳结束后,于考马斯亮蓝R250染色液 中处理1~2 h,最后于脱色液中处理数次直至条带清晰。
1.3.5α-淀粉酶活力测定
发酵液经1 000 r/min离心10 min,取上清液,稀释即为待测样品。吸取20.0 mL 2 g/mL可溶性淀粉溶液于50 mL离心管中,加入5 mL磷酸盐缓冲液(pH 6.0)摇匀后置于60℃恒温水浴锅中预热8 min。加入1 mL待测样品,摇匀,反应10 min。吸取1.00 mL反应液至预先盛有0.5 mL 0.1 mol/L盐酸溶液和5 mL稀碘液的离心管中,摇匀,并以0.5 mL盐酸溶液和5 mL稀碘液为空白,在660 nm波长处迅速测定其吸光度(A)。淀粉酶活力单位为U/mL,1 U定义为:在60℃、pH 6.0的条件下1 min水解1 mg淀粉所需的酶量[21]。
2 结果与分析
2.1α-淀粉酶表达质粒pP43SAT的构建
以设计在pHY300PLK载体上PHY-F/PHY-R为引物,通过菌落PCR初步挑选阳性克隆子。挑取候选阳性克隆子过夜培养,抽质粒,酶切验证结果如图2所示,pP43SAT质粒经过BglⅡ和EcoRⅠ双酶切后,得到片段大小约为4 870 bp和2 345 bp的两个片段,分别与空质粒pHY300PLK和片段SAT大小相等,说明pP43SAT质粒构建成功。
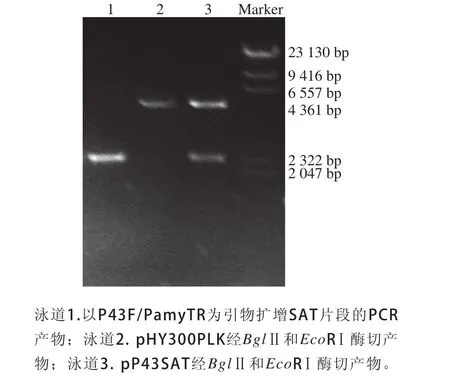
图2 表达质粒pP43SAT的酶切鉴定Fig.2 Identification of expression vector pP43SAT by restriction enzyme digestion
2.2α-淀粉酶表达蛋白鉴定

图3 不同基因工程菌胞外蛋白的SDS-PAGE图谱分析结果Fig.3 SDS-PAGE analysis of extracellular proteins from different genetically engineered strains
将构建好的α-淀粉酶表达载体pP43SAT分别转入BL9和BL10,得到工程菌株BL9(pP43SAT)和BL10(pP43SAT),并在BL10中转入空质粒pHY300P LK得到对照菌BL10(pHY300PLK),抽质粒进行验证,经过液体发酵,取发酵24 h时的发酵液样品,在相同条件下进行SDS-PAGE分析,结果如图3所示。与空白对照相比,BL9(pP43SAT)和BL10(pP43SAT)菌株发酵液中含有强烈的α-淀粉酶条带,大小约58 kD,与先前报道的B. licheniformis α-淀粉酶的分子质量相一致[22],说明α-淀粉酶在BL9和BL10中实现了分泌表达。从α-淀粉酶条带强度可以看出,BL10宿主菌具有更高的α-淀粉酶表达量。BL10是在BL9的基础上敲除了bprA基因,说明宿主菌缺失蛋白酶BprA可进一步提高α-淀粉酶的表达。
2.3不同宿主菌对α-淀粉酶发酵过程的影响
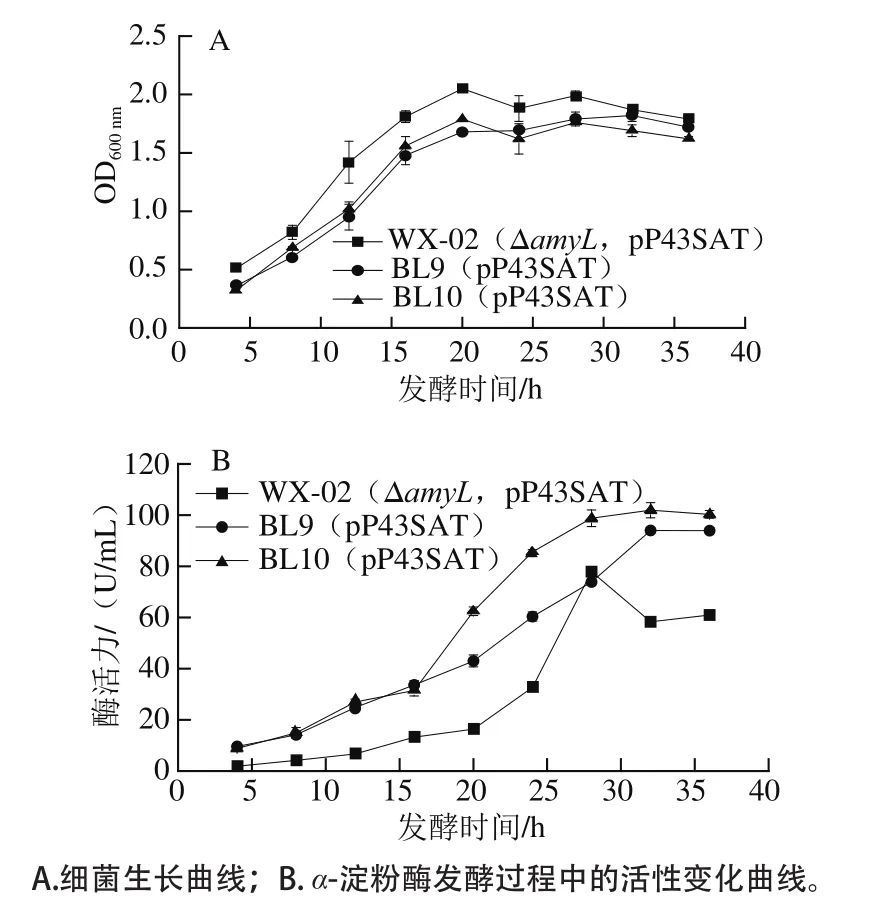
图4 不同宿主菌对α-淀粉酶发酵过程的影响Fig.4 Effects of different host strains on the fermentation process of α-amylase
为了对比BL9、BL10和野生菌WX-02的α-淀粉酶表达效果,并排除B. licheniformis自身α-淀粉酶对蛋白表达的影响,将pP43SAT表达载体转入缺失α-淀粉酶基因的WX-02(ΔamyL)宿主菌中,构建了B. licheniformisWX-02(ΔamyL,pP43SAT)。WX-02(ΔamyL,pP43SAT)、BL9(pP43SAT)和BL10(pP43SAT)的淀粉酶发酵过程如图4所示。与WX-02(ΔamyL,pP43SAT)相比,BL9(pP43SAT)和BL10(pP43SAT)菌体生长相对缓慢,最终生物量微弱下降(图4A),这可能是由于BL9和BL10菌株分别缺失9个和10个基因,对菌体的生长具有微弱的损伤作用,这与本课题先前发现的现象相似[18]。
微弱降低的菌体量并没有降低淀粉酶的表达量,如图4B所示,BL9(pP43SAT)和BL10(pP43SAT)淀粉酶发酵活力分别达到94.01 U/mL和101.94 U/mL,比WX-02(ΔamyL,pP43SAT)的发酵活力(77.88 U/mL)提高了21%和31%,这说明BL9和BL10新型宿主菌有利于α-淀粉酶的表达,这与先前报道的纳豆激酶变化趋势相符合[18]。B. licheniformis宿主菌缺失蛋白酶基因后,可降低自身蛋白酶对目标蛋白的降解作用,这可能是淀粉酶表达活性增加的原因。同时,BL10(pP43SAT)的α-淀粉酶表达活性相对于BL9(pP43SAT)提高了8%,这与SDS-PAGE的蛋白质浓度变化趋势相一致,进一步说明BprA蛋白酶的缺失有利于α-淀粉酶的表达。
3 结 论
本研究构建了α-淀粉酶表达载体pP43SAT,实现了α-淀粉酶在地衣芽孢杆菌宿主菌WX-02(ΔamyL)、BL9和BL10中的成功表达。与宿主菌WX-02(ΔamyL)相比,BL9和BL10可显著提高α-淀粉酶的表达,分别提高了21%和31%。目前,以枯草芽孢杆菌蛋白酶缺失株为宿主表达外源蛋白的研究较多,如6蛋白酶缺失株B. subtilisWB600可高效表达纳豆激酶[23]和枯草蛋白酶[24],8蛋白酶缺失株B. subtilisWB800可高效表达木聚糖酶[25]和磷脂酶C[26],而多蛋白酶缺失的地衣芽孢杆菌宿主菌报道较少,本课题组前期证明BL10有利于纳豆激酶的表达,本研究证明胞外蛋白酶基因缺失可显著提高α-淀粉酶的表达,进一步证实多蛋白酶缺失宿主菌的应用价值。更重要的是,本研究为α-淀粉酶的增强表达提供了新的思路,提供了一种高效表达α-淀粉酶的地衣芽孢杆菌工程菌BL10(pP43SAT),相信经过进一步的发酵工艺优化和中试放大研究,该菌株有望应用于α-淀粉酶的工业化生产。
[1] 丁皓, 王冠, 徐丽, 等. α-淀粉酶的应用研究进展[J]. 饲料博览, 2012(8)∶ 12-15.
[2] SOUZA P M, MAGALH☒ES P O. Application of microbial α-amylase in industry∶ a review[J]. Brazilian Journal of Microbiology, 2010, 41∶850-861.
[3] 李金霞, 蔡恒, 路福平, 等. 地衣芽孢杆菌耐高温α-淀粉酶基因在大肠杆菌中的克隆、表达及其产物的分泌[J]. 食品与发酵工业, 2013, 30(3)∶ 70-73.
[4] S☒RENSEN H P, MORTENSEN K K. Advanced genetic strategies for recombinant protein expression in Escherichia coli[J]. Journal of Biotechnology, 2005, 115(2)∶ 113-128.
[5] 孙柏欣, 刘长远, 陈彦, 等. 基因表达系统研究进展[J]. 现代农业科技, 2008(2)∶ 205-209.
[6] 王凤寰, 杨建国, 汪兵. 透明颤菌vgb基因在地衣芽孢杆菌中表达用于改良α-淀粉酶生产菌株[J]. 中国食品学报, 2012, 12(8)∶ 11-16.
[7] 牛丹丹, 石贵阳, 王正祥. 通过同源介导α-淀粉酶基因扩增改良地衣芽孢杆菌α-淀粉酶生产菌株[J]. 生物工程学报, 2009(3)∶ 375-380.
[8] 杨艳华, 牛丹丹, 石贵阳, 等. 过量表达DegQ有利于地衣芽孢杆菌表达高温α-淀粉酶[J]. 微生物学杂志, 2009(2)∶ 21-24.
[9] GHANG D M, YU L, LIM M H, et al. Efficient one-step starch utilization by industrial strains of Saccharomyces cerevisiae expressing the glucoamylase and alpha-amylase genes from Debaryomyces occidentalis[J]. Biotechnology Letters, 2007, 29(8)∶ 1203-1208.
[10] GALDINO A S, ULHOA C J, MORAES L M, et al. Cloning, molecular characterization and heterologous expression of Amy1, an alpha-amylase gene from Cryptococcus fl avus[J]. FEMS Microbiology Letters, 2008, 280(2)∶ 189-194.
[11] IGNATOVA Z, MAHSUNAH A, GEORGIEVA M, et al. Improvement of posttranslational bottlenecks in the production of penicillin amidase in recombinant Escherichia coli strains[J]. Applied and Environmental Microbiology, 2003, 69(2)∶ 1237-1245.
[12] NGUYEN T, QUYEN T, LE H. Cloning and enhancing production of a detergent- and organic-solvent-resistant nattokinase from Bacillus subtilis VTCC-DVN-12-01 by using an eight-protease-gene-deficient Bacillus subtilis WB800[J]. Microbial Cell Factories, 2013, 12(1)∶ 79. doi∶ 10.1186/1475-2859-12-79.
[13] WEI Xuetuan, JI Zhixia, CHEN Shouwen. Isolation of halotolerant Bacillus licheniformis Wx-02 and regulatory effects of sodium chloride on yield and molecular sizes of poly-gamma-glutamic acid[J]. Applied Biochemistry and Biotechnology, 2010, 160(5)∶ 1332-1340.
[14] LI Xin, LONG Dan, JI Jian, et al. Sample preparation for the metabolomics investigation of poly-gamma-glutamate-producing Bacillus licheniformis by GC-MS[J]. Journal of Microbiological Methods, 2013, 94(1)∶ 61-67.
[15] LI Xin, GOU Xiangyong, LONG Dan, et al. Physiological and metabolic analysis of nitrate reduction on poly-gamma-glutamic acid synthesis in Bacillus licheniformis WX-02[J]. Archives of Microbiology, 2014, 196(11)∶ 791-799.
[16] QI Gaofu, KANG Yanfang, LI Lu, et al. Deletion of meso-2,3-butanediol dehydrogenase gene budc for enhanced D-2,3-butanediol production in Bacillus licheniformis[J]. Biotechnology for Biofuels, 2014, 7(16)∶ 1-17.
[17] LIU Yongfeng, ZHANG Shuling, YONG Yangchun, et al. Efficient production of acetoin by the newly isolated Bacillus licheniformis strain Mel09[J]. Process Biochemistry, 2011, 46(1)∶ 390-394.
[18] WEI Xuetuan, ZHOU Yinhua, CHEN Jingbang, et al. Efficient expression of nattokinase in Bacillus lichenifo rmis∶ host strain construction and signal peptide optimization[J]. Journal of Industrial Microbiology and Biotechnology, 2015, 42(2)∶ 287-295.
[19] TIAN Guangming, FU Juntao, WEI Xuetuan, et al. Enhanced expression of pgdS gene for high production of poly-γ-glutamic aicd with lower molecular weight in Bacillus licheniformis WX-02[J]. Journal of Chemical Technology and Biotechnology, 2014, 89(12)∶1825-1832.
[20] 萨姆布鲁克, 拉塞尔. 分子克隆实验指南[M]. 3版. 北京∶ 科学出版社, 2005∶ 1713-1720.
[21] 武利庆, 谢宝民, 王晶. α-淀粉酶活性测定方法研究及测量结果不确定度评估[J]. 计量学报, 2007, 28(3)∶ 250-255.
[22] HMIDET N, BAYOUDH A, BERRIN J G, et al. Purification and biochemical characterization of a novel α-amylase from Bacillus licheniformis NH1∶ cloning, nucleotide sequence and expression of amyN gene in Escherichia coli[J]. Process Biochemistry, 2008, 43(5)∶499-510.
[23] PENG Yong, HUANG Qing, ZHANG Renhuai, et al. Purification and characterization of a fibrinolytic enzyme produced by Bacillus amyloliquefaciens DC-4 screened from douchi, a traditional Chinese soybean food[J]. Comparative Biochemistry and Physiology B-Biochemistry and Molecular Biology, 2003, 134(1)∶ 45-52.
[24] XIAO Lu, ZHANG Renhuai, PENG Yong, et al. Highly efficient gene expression of a fibrinolytic enzyme (subtilisin DFE) in Bacillus subtilis mediated by the promoter of α-amylase gene from Bacillus amyloliquefaciens[J]. Biotechnology Letters, 2004, 26(17)∶ 1365-1369.
[25] PHUONG N D, JEONG Y S, SELVARAJ T, et al. Production of Xynx, a large multimodular protein of Clostridium thermocellum, by protease-deficient Bacillus subtilis strains[J]. Applied Biochemistry and Biotechnology, 2012, 168(2)∶ 375-382.
[26] DURBAN M A, SILBERSACK J, SCHWEDER T, et al. High level expression of a recombinant phospholipase C from Bacillus cereus in Bacillus subtilis[J]. Applied Microbiology and Biotechnology, 2007, 74(3)∶ 634-639.
Comparative Analysis on the Effects of Different Host Strains of Bacillus licheniformis on the Expression and Secretion of α-Amylase
CHEN Jingbang1, ZHOU Yinhua1, ZHAO Xinyu1,2, CHEN Shouwen1, WEI Xuetuan1,2,*
(1. State Key Laboratory of Agricultural Microbiology, Huazhong Agricultural University, Wuhan 430070, China;2. College of Food Science and Technology, Huazhong Agricultural University, Wuhan 430070, China)
In the present study, effects of different host strains ofBacillus licheniformison the expression and secretion ofα-amylase were investigated. Firstly, theα-amylase expression vector, named pP43SAT, was constructed based on the promoter P43 fromBacillus subtilisand the signal peptide, coding region and terminator ofα-amylase gene fromB. licheniformisWX-02. The pP43SAT plasmids were then transformed into theB. licheniformishost strains WX-02 (ΔamyL), BL9 and BL10B, respectively, to obtain theα-a mylase genetic engineering strainsWX-02 (ΔamyL;pP43SAT), BL9 (pP43SAT) and BL10 (pP43SAT), respectively. Theα-amylase fermentation processes of these engineered strains were compared.Theα-amylase activities ofstrains BL9 (pP43SAT) and BL10 (pP43SAT) reached 94.01 and 101.94 U/mL, respectively, which were increased by 21% and 31%, respectively, as compared with that of WX-02 (ΔamyL;pP43SAT). These results showed that the novel host strains BL9 and BL10 contributed to the secretion and expression ofα-amylase. It was proved that the deletion of multiple proteases genes could enhanceα-amylase expression obviously, and this st udy provided the novel host strains and novel strategies for efficient expression ofα-amylase.
α-amylase;Bacillus licheniformis; host strain;secretion and expression
Q812
1002-6630(2015)17-0196-05
10.7506/spkx1002-6630-201517037
2015-04-10
湖北省自然科学基金项目(2014CFB943);华中农业大学自主科技创新基金项目(2662015QC035)
陈敬帮(1990—),男,硕士研究生,研究方向为食品酶学。E-mail:1176154594@qq.com
*通信作者:魏雪团(1984—),男,讲师,博士,研究方向为食品微生物学。E-mail:weixuetuan@mail.hzau.edu.cn

