考虑到隐性感染人群的潜伏期和发病期均传染的SLICAR模型*
陈慧林董慧茹郑一男唐欣然侯雅文陈 征△陈平雁
考虑到隐性感染人群的潜伏期和发病期均传染的SLICAR模型*
陈慧林1董慧茹1郑一男1唐欣然1侯雅文2陈 征1△陈平雁1
目的提出一种包含隐性感染者并且含潜伏期均传染的流行病模型。方法基于已有的SEIR模型,加入隐性感染者分箱得到SLICAR模型,并以2009年春季的甲型H1N1和1918年秋季的Spanish流感数据为实例分析。结果H1N1流感数据模型拟合的基本繁殖数R0值为2.174(决定系数R2=0.802)以及Spanish数据疫情17天拟合R0值为2.636和疫情结束时拟合值为3.675。结论SLICAR模型考虑到了隐性感染者以及潜伏期患者的传染性,为基本繁殖数R0的估计提供了一种较为全面的算法;也为疫情防控提供了更为全面的信息。
流行病学模型 基本繁殖数 甲型H1N1流感 隐性感染
对于流行病传播情况的研究,Kermack[1]提出了无潜伏期的模型-SIR模型,此模型并未考虑到感染病毒后的潜伏期因素,当流行病的潜伏期较长时,是有必要考虑带有时滞效应的潜伏期人群对疾病传播的影响,于是Aron和Schwartz[2]提出了SEIR模型。这两个经典流行病动力学模型已被广泛而深入的研究[1-5]。董霖等[6]研究了一类总人口可变,具有非线性传染率且潜伏期和发病期均传染的模型。Aaron[7]提出了包含隐性感染者的流行病学模型,具有传染性的隐性感染者对疾病的传播不可忽视。隐性感染(asymptomatic or inapparent infection)是指病原体侵入人体后,仅引起机体产生特异性的免疫应答,不引起或仅引起轻微的组织损伤,在临床上不显出任何症状、体征,甚至生化改变,只能通过免疫学检查才能发现。Yang等[8]研究指出,对于像甲流等此类流行病,存在隐性感染并且该类人群具有一定的传染能力。当该类人群所占比重较大时,其对疾病的传播会产生不容忽视的作用,如Longini[9]在研究中假定隐形传染者占到33%。而对于用以反映流行病传播能力的重要参数——基本繁殖数R0(basic reproduction number)[10]的估计,现有的动力学模型均未同时考虑到具有传染的隐性感染和潜伏期对R0估算结果的影响。
本文将这类疾病作为研究对象,以SEIR模型为基础,建立考虑到隐性感染人群并且潜伏期和染病期均传染的SLICAR(susceptible latent infected confirmed asymptomatic recovered)模型。运用最小二乘思想拟合模型参数,更好地刻画并预测甲流的实际传播情况,为决策与评价提供更加丰富的信息。
SLICAR模型
1.各类人群的转化过程
各类人群的转化过程如图1所示。SLICAR模型主要包含六类人群:易感人群S(susceptible),潜伏期人群L(latent),显性感染人群I(infected),实验室确诊人群C(confirmed),隐性感染人群A(asymptomatic)及移出人群R(recovered)。N表示t时刻的有效总人口数[6],则有S+L+I+C+A+R=N;β为日平均接触数;ε、δ分别为潜伏期者和隐性感染者的有效传染人数比;1/k为潜伏期;p为显性感染者占全体感染者的人数比例;1/α为显性感染者平均传染期;1/γ为平均报道延迟期,即显性感染者从被确诊到被报道的延迟天数;1/η为隐性感染者传染期;q为实验室确诊病例校正系数[11]。由于监控机构无法检测出实际的所有患者,因此一般公开的数据为实验室确诊病例数。于是将实验室确诊病例数作为实验室确诊人群C分箱加入模型,引入实验室确诊病例校正系数q,用以反应监控数据对疫情情况反映的真实度。该系数由显性感染人群I转化为实验室确诊人群C中五个过程的率乘积决定[11]:流感样病人就诊率、就诊采样率、采样送检率、RT-PCR法的检出率、实报率。
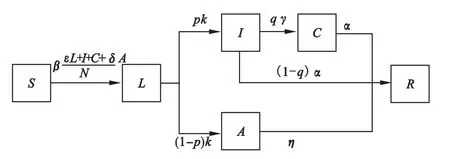
图1 SLICAR模型状态转移图
2.SLICAR模型
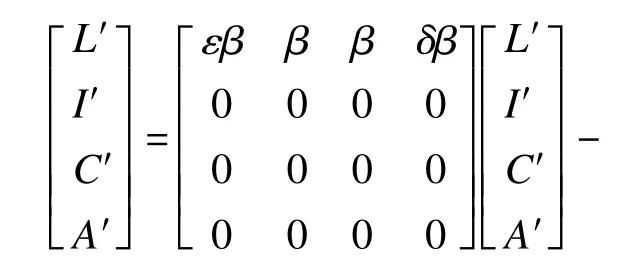
模型假设:(1)人口数在研究区域内呈均匀分布;(2)患者治愈后获得免疫能力,不会被二度感染。(3)由于模拟的时间较短,人的出生率与自然死亡率忽略,迁出率与迁入率也暂不作考虑。设S(t),L(t),I(t),C(t),A(t),R(t)分别表示各类人群在t时刻的人数,且随时间连续变化。假设传染期间人群总数保持不变,以及各人群变化数等于该人群的转入量减去转出量,可得到SLICAR模型的微分方程组:

3.基本繁殖数(R0)
R0为一个病人在其具有传染性的期限内预期直接传播的新病例数,亦即直接传播的第二代病例数。其临界值为1,当R0<1时,就是说一个病例在传染期限内,不能传播一个新病例,疾病传播将趋于终止。根据微分方程组(1),通过转化有:

实例分析
1.甲型H1N1流感 数据来源于2009年4月18日至8月28日美国CDC(http://www.cdc.gov/)每周公布的甲型H1N1流感实验室确诊病例数。美国CDC进行数据统计时,将美国分为十个区进行数据的汇总及报告,这里选取纽约州的数据为例,总人口数28172958。根据数据特点,利用模型估算了该周累计的病例数,用来与实际公布的每周数据进行拟合分析。
根据某些前期研究和发现[12],固定参数的取值展现在表1中,其中假设隐性感染者几何均数传染期与显性感染者几何均数传染期相同,即1/η和1/α取9.3天,1/γ取1天;由于处于潜伏期最后一天的人群具有传染性[12],而潜伏期中位天数为4.3天,所以潜伏期有效传染人数比ε取值为1/4.3;甲流无症状患者中亦具有一定传染力,此处取1。此外,Reed等[11]研究指出,由Monte-Carlo模拟可得到实验室确诊病例校正系数q中位值为1.760×10-2(90%置信区间:1.071×10-2~2.722×10-2)。该参数取倒数后为57,说明每报道1例甲流确诊病例,就代表实际有57例显性感染者。甲流爆发初期是以个别城市为中心,而非全国同时爆发。本研究包含了诸多城市,面积广阔。因此,并不能将整个区的全体人口作为甲流爆发初期的易感人群,而易感人群所在的区域应为以疫情发展严重的城市为中心向四周辐射的一定范围。由于此范围的影响因素繁多,如:人口密度、年龄构成、人口流动性等,每个区都有其特点,没有统一的标准。因此引入总人口调整系数θ,假定该系数在甲流爆发初期保持不变,运用最小二乘法拟合θ,进而将区域内的总人口数N调整为有效总人口数Ne(=θN)。
图2展示每周实验室确诊病例拟合图,其中柱状图代表实验室确诊病例数据,实线为实验室确诊病例校正系数q取中位数1.760×10-2时的拟合结果,上下虚线分别为q取90%置信区间上下限时的拟合结果。使用模型预测实验室确诊病例数在2009年6月13日至6月19日期间达到峰值331人,截止至8月28日实验室确诊病例共1806人,总人口调整系数θ值为0.001,求得R0的值为2.174,拟合优度决定系数为0.802。

表1 固定参数取值表

图2 每周实验室确诊病例拟合图
2.Spanish流感 数据来源于美国旧金山1918年H5N1[13],该地区总人口数为550000。总发病人数28310,假设该处的显性感染者都被及时确诊为实验室病人,即q=1。其他参数见表2。
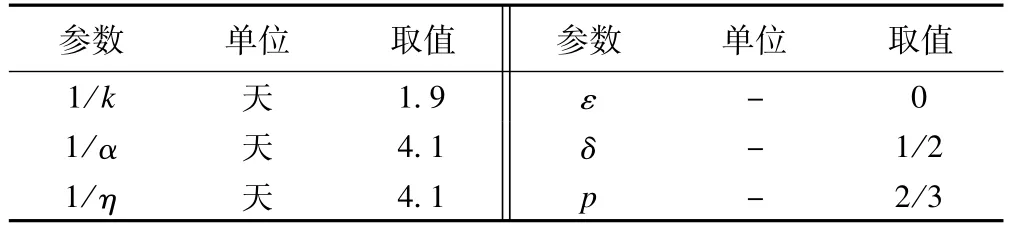
表2 固定参数取值表
根据SLICAR模型计算得出疫情17天时R0为2.636。整个疫情的R0为3.675,与疫情17天比较偏高。从整个疫情数据来看[13],10月22日发病人数为1143人,到23日为2058人,以及接下来三四天维持在2000人左右之后骤然下降到1400人,这可能导致R0估计值的偏高。
讨 论
本模型引入了隐性感染者分箱A,并考虑到隐性感染人群的潜伏期和染病期均具有传染性的情况,能够较准确地预测疾病爆发高峰期及感染人数,估计合理的R0值,对于防控工作具有一定参考价值。由于传统的流行病动力学模型一般未考虑具有传染性的隐性感染者和有传染性的潜伏期患者对疫情发展的影响,可能会低估疫情发展的严重程度。而本模型考虑到这一群体的影响,能够更为详实地反映疾病的传播情况。从实例模拟效果来看,总人口调整系数θ的引入可以正确、简便地确定易感人群的初始值,使模型较好地适用于人口基数较大的情况;同时,模型引入病例校正系数q,较好地解决了政府难以及时准确地报告真实疫情这一客观问题,为防控力度的调整提供更加准确的参照。
本模型假定平均日接触率β及实验室确诊病例调整系数q为定值。而β值在疾病初期会随着病情的发展而逐渐增大,之后随着政府的防控力度与人们防范意识的增强,β会随之减小。同时,在疫情发展的不同阶段,需要进行大量的调查分析以确定较准确的q值。尽管此过程较为繁琐,但q值的准确度对模型能否准确模拟实际疫情发展具有至关重要的作用。
SLICAR模型考虑到了隐性感染者和潜伏期患者均具有传染性的因素,一般的流行病学模型未同时考虑到这两种因素对基本繁殖数R0的影响,因此可能会低估了R0的估计值。SLICAR模型同时将该两因素纳入模型中,对模型的预测以及疫情防控提供了更为全面的信息。
1.Kermack WO,McKendrick AG.Contributions to the mathematical theory of epidemics-I.Bulletin of Mathematical Biology,1991,53(1-2):33-55.
2.Aron JL,Schwartz IB.Seasonality and period-doubling bifurcations in an epidemic model.Journal of Theoretical,1984,110(4):665-679.
3.Jaime M,Hethcote WH.Dynamic models of infectious diseases as regulators of population sizes.Journal of Mathematical Biology,1992,30(7):693-716.
4.Busenberg S,Driessche P.Analysis of a disease transmission model in a population with varying size.Journal of Mathematical Biology,1990,28(3):257-270.
5.Fred B,Driessche P.Models for transmission of disease with immigration of infectives.Mathematical Biosciences,2001,171(2):143-154.
6.董霖,邱亚林.一类潜伏期和染病期均传染的SEIS模型.龙岩学院学报,2007,25(06):8-10.
7.Aaron AK,Edward L,Mercedes P,et al.Inapparent infections and cholera dynamics.Nature,2008,454:877-880.
8.Yang J,Yang F,Huang F,et al.Subclinical Infection with the Novel Influenza A(H1N1)Virus.Clinical Infectious Diseases,2009,49(10):1622-1623.
9.Longini IM,Nizam A,Xu S,et al.Containing pandemic influenza at the source.Science,2005,309(5737):1083-1087.
10.Dietz K.The estimation of the basic reproduction number for infectious diseases.Statistical Methods in Medical Research,1993,2(1):23-41.
11.Reed C,Angulo FJ,Swerdlow DL,et al.Estimates of the prevalence of pandemic(H1N1)2009,United States,April-July 2009.Emerging Infectious Diseases,2009,15(12):2004-2007.
12.Tuite AR,Greer AL,Whelan M,et al.Estimated epidemiologic parameters and morbidity associated with pandemic H1N1 influenza.CMAJ,2010,182(2):131-136.
13.Chowell G,Nishiura H,Bettencourt LM.Comparative estimation of the reproduction number for pandemic influenza from daily case notification data.Journal of The Roal Society Interface,2007,4(12):155-166.
(责任编辑:郭海强)
An Epidemic Model Named SLICAR for Transmission of Disease with Asymptomatic Infection and Infective Latent Period
Chen Huilin,Dong Huiru,Zheng Yinan,et al.(Department of Biostatistics,School of Public Health and Tropical Medicine,Southern Medical University(510515),Guangzhou.)
ObjectiveTo provide a model including latent and onset periods of infectiousness in asymptomatic patients.MethodsSLICAR model was constructed by combining asymptomatic patients and SEIR model.We analyzed and checked the model using pandemic influenza A(H1N1)and Spanish flu data.ResultsThe basic reproduction number(R0)of H1N1 was 2.174(coefficient of determinationR2=0.802).2.636 and 3.675 represented 17-days and entire-epidemicR0,respectively.ConclusionSLICAR model takes consideration of the infectious of asymptomatic patients in latent and onset periods.It is a comprehensive way for evaluatingR0,thus abundant information is available for taking measures.
Epidemic model;Basic reproduction number;Pandemic influenza A(H1N1);Asymptomatic patients
国家自然科学基金(81202288);广州市科技计划(2012J5100023);高等学校博士学科点专项科研基金(20114433120010);广东省科技计划(2010B031600100)
1:南方医科大学公共卫生与热带医学学院广东省热带病研究重点实验室生物统计学系(510515)
2:暨南大学经济学院统计学系
△通信作者:陈征,zchen@smu.edu.cn

