MBR工艺处理螺旋霉素制药废水过程中抗生素耐药菌与抗性基因的研究
覃彩霞,张俊亚,佟娟,*,申佩弘,魏源送,3
1. 中国科学院生态环境研究中心 环境模拟与污染控制国家重点联合实验室,北京 10085 2. 广西大学生命科学与技术学院,南宁 530005 3. 江西省科学院能源研究所,南昌 330096
MBR工艺处理螺旋霉素制药废水过程中抗生素耐药菌与抗性基因的研究
覃彩霞1,2,张俊亚1,佟娟1,*,申佩弘2,魏源送1,3
1. 中国科学院生态环境研究中心 环境模拟与污染控制国家重点联合实验室,北京 10085 2. 广西大学生命科学与技术学院,南宁 530005 3. 江西省科学院能源研究所,南昌 330096
抗生素废水含有大量的抗生素耐药菌(antibiotic resistant bacteria, ARB)与抗性基因(antibiotic resistance genes, ARGs),处理排放后可能增强受纳环境的微生物抗性,因此有必要深入研究抗生素废水处理过程中ARB与ARGs的削减效果及其影响因素。本研究采用膜生物反应器(membrane bioreactor, MBR)工艺处理螺旋霉素制药废水,考察了不同水力停留时间(hydraulic retention time, HRT)对ARB与ARGs削减效果的影响。结果表明,虽然在HRT=30 h时MBR对COD与氨氮的去除率略高于HRT=40 h,但HRT=40 h时,不仅总异养菌与肠球菌的去除效果更佳,出水肠球菌耐药率及携带的抗性基因检出率也更低,而且MBR对废水中ermB、ermF、ermX、mefA、ereA、mphB和转移元件ISCR1、Tn916/1545相对丰度的削减效果更好。这表明长HRT更有利于MBR工艺削减螺旋霉素废水的耐药菌与抗性基因。
耐药菌;抗性基因;螺旋霉素制药废水;膜生物反应器;水力停留时间
抗生素耐药菌(antibiotic resistant bacteria, ARB)与抗性基因(antibiotic resistance gene, ARGs)已成为全球关注的热点,目前已经在废水、地表水、地下水、沉积物、土壤等环境中检测到ARB与ARGs的存在[1-3],这对环境和人体健康构成了巨大的潜在危害。我国是世界上最大的抗生素生产与消费国,然而抗生素制药废水不仅有机物浓度高,难于处理,对环境造成了严重的危害[4],并且抗生素废水中含有高浓度的抗生素残余物[5-6],在处理过程中可能会产生大量抗生素耐药菌,导致高浓度抗生素和抗生素耐药菌随着出水排放不断进入环境。近年来,医药废水分离出来的大肠杆菌中超过75%为高耐药水平的超广谱耐药菌,丰度甚高,严重威胁人类的健康[7],且医药废水中细菌的耐药率也明显高于相邻水体中的细菌[8-9]。因此,抗生素制药废水造成的抗性污染不容忽视。
膜生物反应器工艺(membrane bioreactor, MBR)是膜技术与污水生物处理技术有机结合的一种新型、高效的废水处理工艺,广泛应用于污水处理与回用。Munir等[10]调研了美国5个利用不同处理工艺的城市污水处理厂对抗生素耐药菌与抗性基因的去除情况,研究结果发现MBR工艺具有显著的ARB与ARGs去除效果(2.57~7.06 logs),优于传统活性污泥工艺(2.37~4.56 logs)。但是MBR运行过程中,不同工艺操作参数对其中ARB与ARGs去除效果的影响鲜见相关报道,水力停留时间(HRT)作为MBR运行过程中最重要的参数之一,值得进行深入探讨。
因此,本文以江苏无锡某螺旋霉素制药废水为研究对象,重点考察和比较不同水力停留时间对MBR工艺去除抗生素耐药菌和抗性基因的影响,并初步探讨了其影响机制。
1 材料与方法 (Materials and methods)
1.1 样品采集
江苏省无锡市某螺旋霉素制药厂废水处理站废水处理流程为:调节池-厌氧池-缺氧池-好氧池-二沉池。本研究采集该厂缺氧池出水作为实验室MBR装置的进水。在HRT=40 h时,每半个月取样1次,取样点为MBR进水、MBR活性污泥、MBR出水,共取样4次;然后将MBR装置的HRT调整为30 h,在相同取样点每半个月取样1次,共取样3次。
1.2 MBR装置及其操作参数
实验室规模一体式MBR污水处理装置采用SINAP-10-PVDF平板膜组件(上海斯纳普公司),有效膜面积为0.1 m2;膜组件外形尺寸:宽×高×厚=220 mm ×320 mm×6 mm;膜孔径为0.1 μm;运行膜通量为8 L·(m2·h)-1。生物反应器的有效容积30 L,平板膜所需冲刷空气6 L·(min·片)-1由位于膜组件下方的曝气管提供,活性污泥所需的曝气量设置为0.2 m3·h-1。反应器进水由液位继电器(JYB-714,欣灵电气公司)控制,平板膜的出水通过蠕动泵(BT600,保定兰格公司)的连续抽吸来实现,装置的工艺流程见图1。MBR内接种污泥为该螺旋霉素制药废水处理站的二沉池浓缩污泥。课题组以往研究结果表明,采用MBR工艺处理该螺旋霉素废水时,HRT=30~36 h可取得较好效果[11],据此,本研究中HRT分别设定为40 h和30 h,考察水质指标处理效果相近的情况下,不同HRT对出水肠球菌耐药性及抗性基因的影响。实验室MBR系统共运行7个月,污泥停留时间(sludge retention time, SRT)均为30 d。设置初始HRT=40 h,在满足MBR设备运行时间达到1个污泥龄以上,并且出水COD、氨氮等指标稳定在20 d以上,视为系统达到稳定运行状态,可以取样研究。
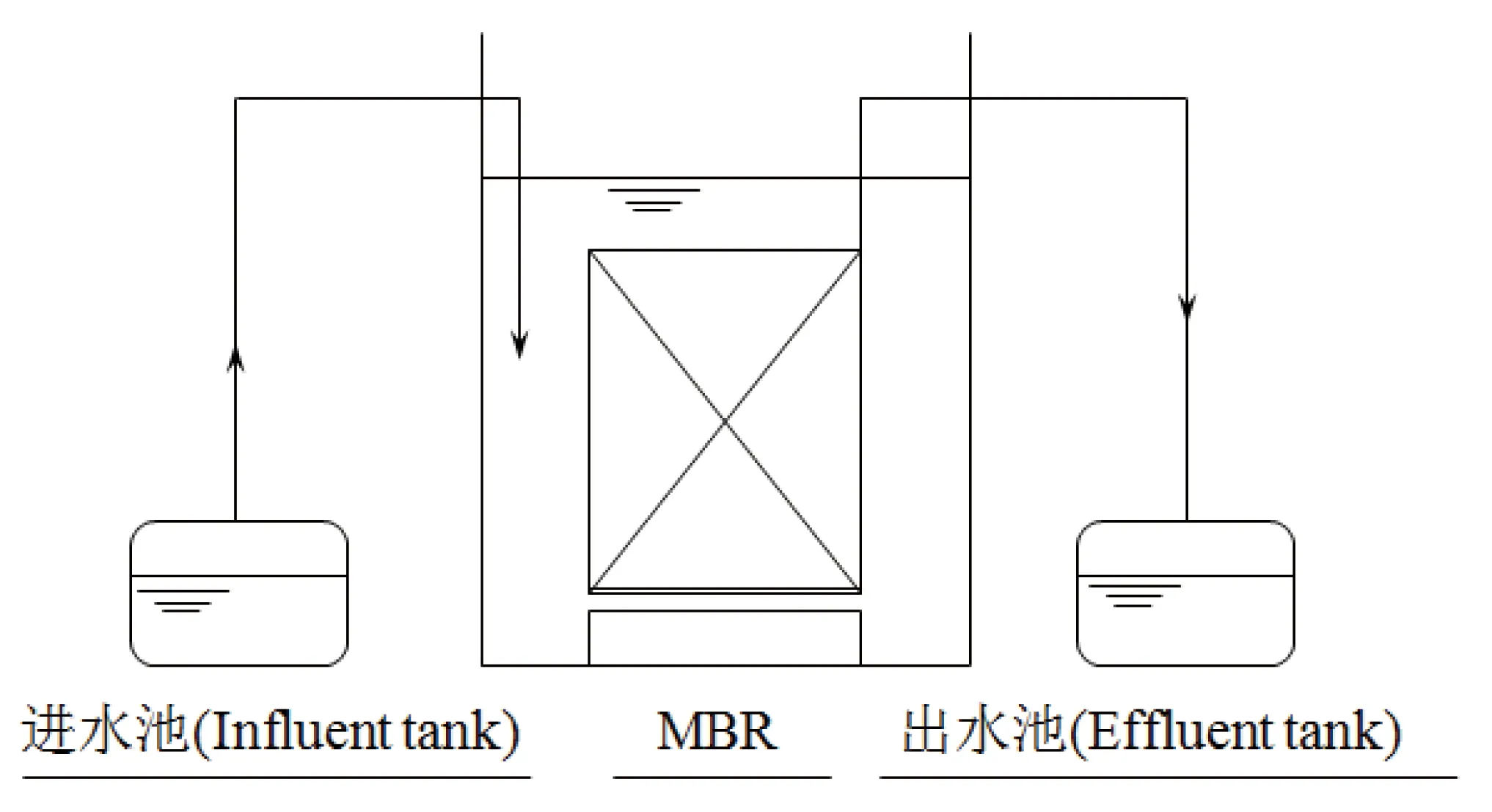
图1 板式MBR处理螺旋霉素废水试验的工艺流程图
1.3 水质分析
根据国家标准方法[12]对常规指标(混合液悬浮固体浓度(MLSS)、混合液挥发性悬浮固体浓度(MLVSS)、COD、氨氮)进行检测。
1.4 样品总DNA提取
取MBR进水与出水提取基因组DNA。由于污水样品为泥水混合物,为了使泥水能够分离,同时又不会把水样中的微生物沉淀下去,并考虑到泥水混合物中大颗粒物与微生物的沉降速度不同,故通过低速离心使其分离,根据MBR进水与出水的水质不同分别取20~100 mL经过0.22 μm滤膜过滤,将滤膜剪碎,采用试剂盒Fast DNA Spin Kit for soil (USA)对滤膜上截留的生物量提取基因组总DNA。提取的DNA溶液在Nanodrop分光光度计(Nanodrop,US)上测定浓度和质量,并进行琼脂糖凝胶电泳(TBE缓冲液,质量体积比1%)。
1.5 样品中总异养菌和肠球菌的检测
异养菌总数采用R2A培养基进行培养计数[10],用1 mL灭菌移液吸管吸取污水样品1 mL,缓慢注入含有9 mL灭菌PBS缓冲液的试管内,震荡试管混合均匀,制成10-1稀释菌液。同法依次连续稀释至10-2、10-3、10-4、10-5稀释菌液。每个采样点水样选择3个适宜稀释度,每个稀释梯度做3个平行样,吸取0.1 mL稀释菌液,加在已经倒入灭菌培养基并凝固备用的平板上。涂布法涂布均匀,37 ℃恒温生化培养箱中培养2 d后计数,以上无菌操作均在超净台中进行。
肠球菌数采用滤膜法测定[13],污水样品同上经过稀释,不同采样点的样品采用PBS缓冲液稀释成适宜的稀释液,用孔径0.45m醋酸纤维滤膜过滤,将滤膜以截留面朝上贴附在灭菌滤膜肠球菌培养基并凝固备用的平板上,平板倒置于生化培养箱中,37 ℃恒温培养2~5 d,根据菌落生长情况确定培养时间(t)。计数每个平板上的菌落数,并根据稀释倍数计算样品中肠球菌的丰度。挑取乳白色菌落划线于胆盐七叶苷琼脂培养基上,挑取有棕色光泽的菌落保存进行下一步实验。
1.6 肠球菌耐药性分析
通过药敏实验分析肠球菌耐药性,采用美国临床和实验室标准协会(CLSI)推荐的Kirby-Bauer纸片法[14],将待检菌株菌液的浊度用无菌生理盐水调节至0.5麦氏浊度后,用棉签蘸取菌液涂布于Muller-Hinton琼脂(Oxoid)平板上,贴上4种抗生素药敏纸片(Oxoid):阿奇霉素(AZI)、红霉素(ERY)、螺旋霉素(SP)、克拉霉素(CLR)。将平板倒置于生化培养箱中,37 ℃恒温培养24 h,测定抑菌圈直径,采用CLSI标准判断菌株的抗性,本研究以ATCC25922菌株为质控菌株。
1.7 抗性基因和转移元件的检测
1.7.1 抗性基因的定量检测
采用SYBR-Green实时定量PCR方法对基因组DNA的ermB、ermF、ermX、mefA、ereA、mphB和转移元件ISCR1、intI1、Tn916/1545进行定量分析,PCR所用引物见表1。检测仪器为StepOne型荧光定量PCR仪(ABI,7500,美国)。PCR产物经过克隆测序确认后,使用生工质粒提取试剂盒SK1131从阳性克隆子中提取质粒,用作标准曲线。使用NanoDrop微量分光光度计(Nanodrop 2000-Thermo,美国)测定质粒浓度。制作标准曲线时按照10倍梯度稀释构建好的各质粒,于90L稀释液中加入10L质粒,做4~6个点,通过预试验选取合适标准品用于制备标准曲线。标准质粒、环境样品、阴性对照均做3个平行,取平均值进行计算。
载体(pMD 18-T)碱基数 = 2 692 bp,载体(pGEMX-T Easy)碱基数 = 3 023 bp
1.7.2 肠球菌携带抗性基因的检测
采用菌液PCR的方法检测肠球菌携带的抗性基因ermB、mefA和转移元件intI1,基因引物见表1。菌液PCR采用20L反应体系:10L Taq PCR Master Mix (Biomed, Beijing),0.5L引物(F),0.5L引物(R),菌液0.5L,加ddH2O至20L。反应程序为:94 ℃,15 min;94 ℃,30 s,55 ℃,30 s,72 ℃,1 min,34次循环;72 ℃,5 min,并于4 ℃保存。每次运行使用无菌水做阴性对照,PCR产物以1%的琼脂凝胶电泳分析。
2 结果与讨论(Results and discussion)
2.1 不同HRT时MBR的运行效果
不同HRT条件下,MBR的常规检测指标如表2所示。由于本研究是基于课题组以往对于MBR处理螺旋霉素废水优化后的工艺条件[11],出水水质相对较为稳定。由表中数据可知,在HRT分别为40 h与30 h的运行过程中,MBR内进出水水质变化不大,出水COD差值仅为11 mg·L-1,氨氮差值小于3 mg·L-1。HRT为30 h时,COD与氨氮的去除率分别为74.3%和97.7%,略高于HRT为40 h时的72.1%与96.2%。污泥浓度MLVSS在HRT为30 h比40 h高出9.5%,这主要是由于短HRT条件下,污泥有机负荷更高(HRT为30 h和40 h时污泥负荷分别为0.05 kgCOD·(kgVSS·d)-1和0.04 kgCOD·(kgVSS·d)-1,更有利于微生物的增殖。
2.2 HRT对总异养菌和肠球菌的去除效果的影响 HRT=40 h时,进水与出水中异养菌分别为5.60×108CFU·L-1和7.02×107CFU·L-1,肠球菌分别为1.13×104CFU·L-1和1.00×102CFU·L-1(见图2)。MBR对总异养菌和肠球菌有较好的去除效果,去除率分别达到87.5%和99.1%。HRT=30 h时,进水中总异养菌和肠球菌的含量分别为2.29×108CFU·L-1和7.73×107CFU·L-1,出水中异养菌和肠球菌的浓度则为9.06×103CFU·L-1和1.60×102CFU·L-1,总异养菌和肠球菌去除率分别为66.2%和98.2%。Novo等[19]对比了不同HRT的城市污水处理厂(处理工艺分别为活性污泥法、滴滤池、淹没式曝气滤池),发现长HRT的污水处理厂对进水中的总异养菌、肠球菌、耐药肠球菌的去除效果均优于短HRT的污水厂,认为长HRT更利于微生物菌群的均匀分布,因此总细菌和所有耐药菌的去除效果均优于短HRT。本研究结果与之相似,长HRT更有利于MBR工艺去除螺旋霉素废水中的异养菌与肠球菌。
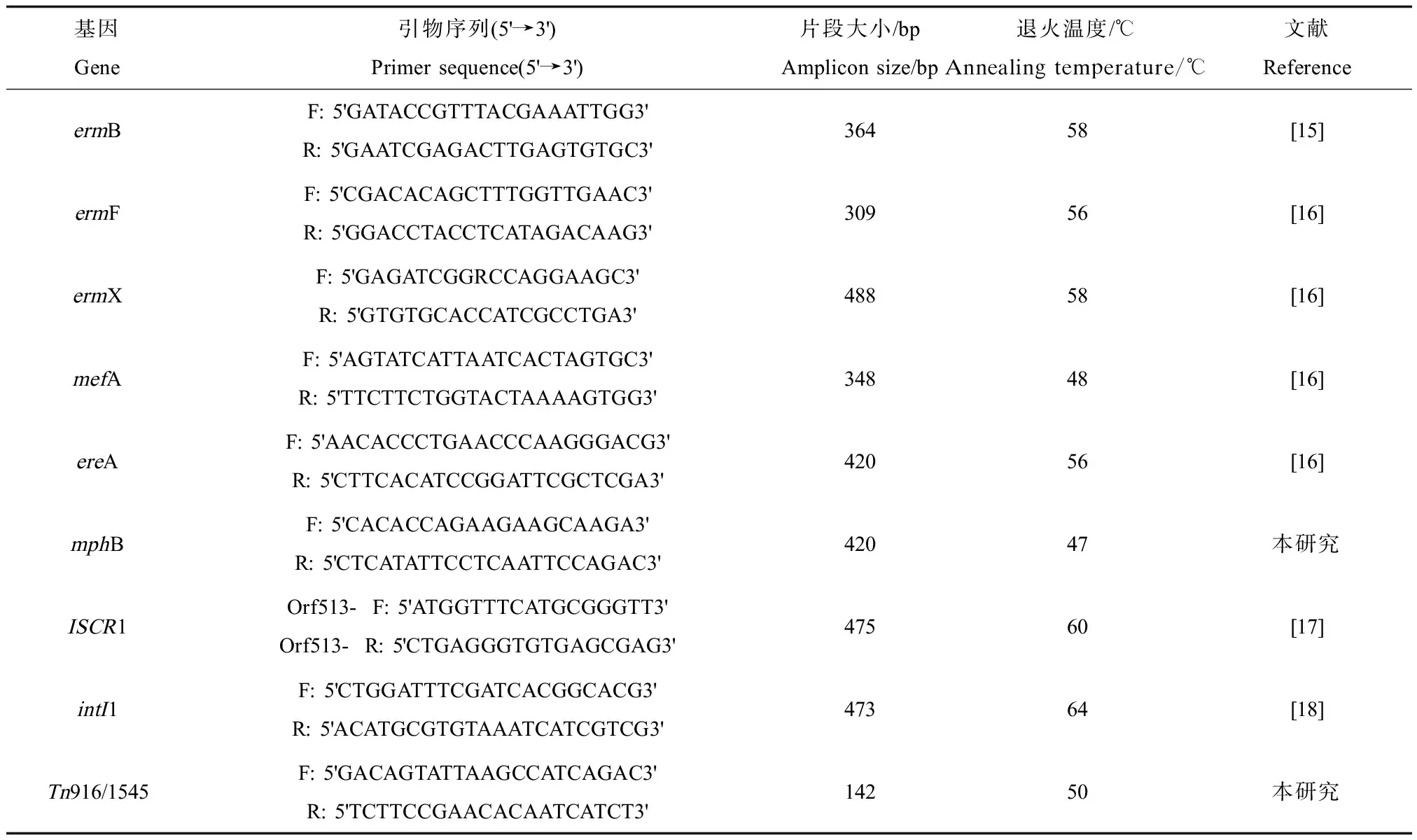
表1 抗性基因和转移元件所用引物序列Table 1 Primers for resistance genes and mobile elements
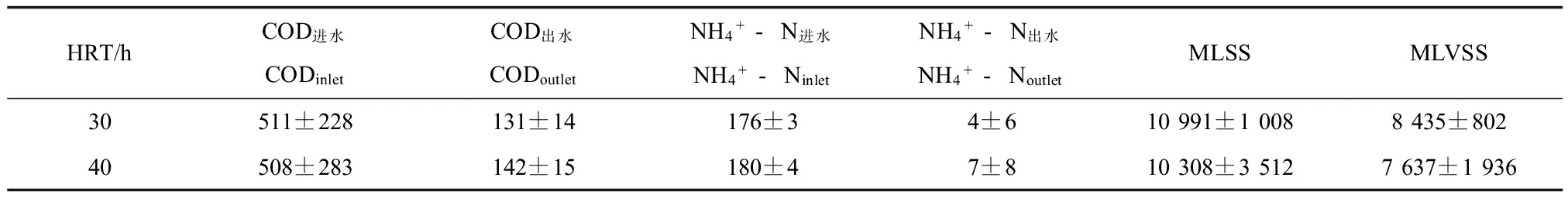
表2 MBR进出水水质特征(平均值±标准差)Table 2 Characteristics of influent and effluent of MBR (means±SD) (mg·L-1)
注:MLSS为混合液悬浮固体浓度,MLVSS为混合液挥发性悬浮固体浓度。
Note: MLSS stands for mixed liquor suspended solids, MLVSS stands for mixed liquor volatile suspended solids.
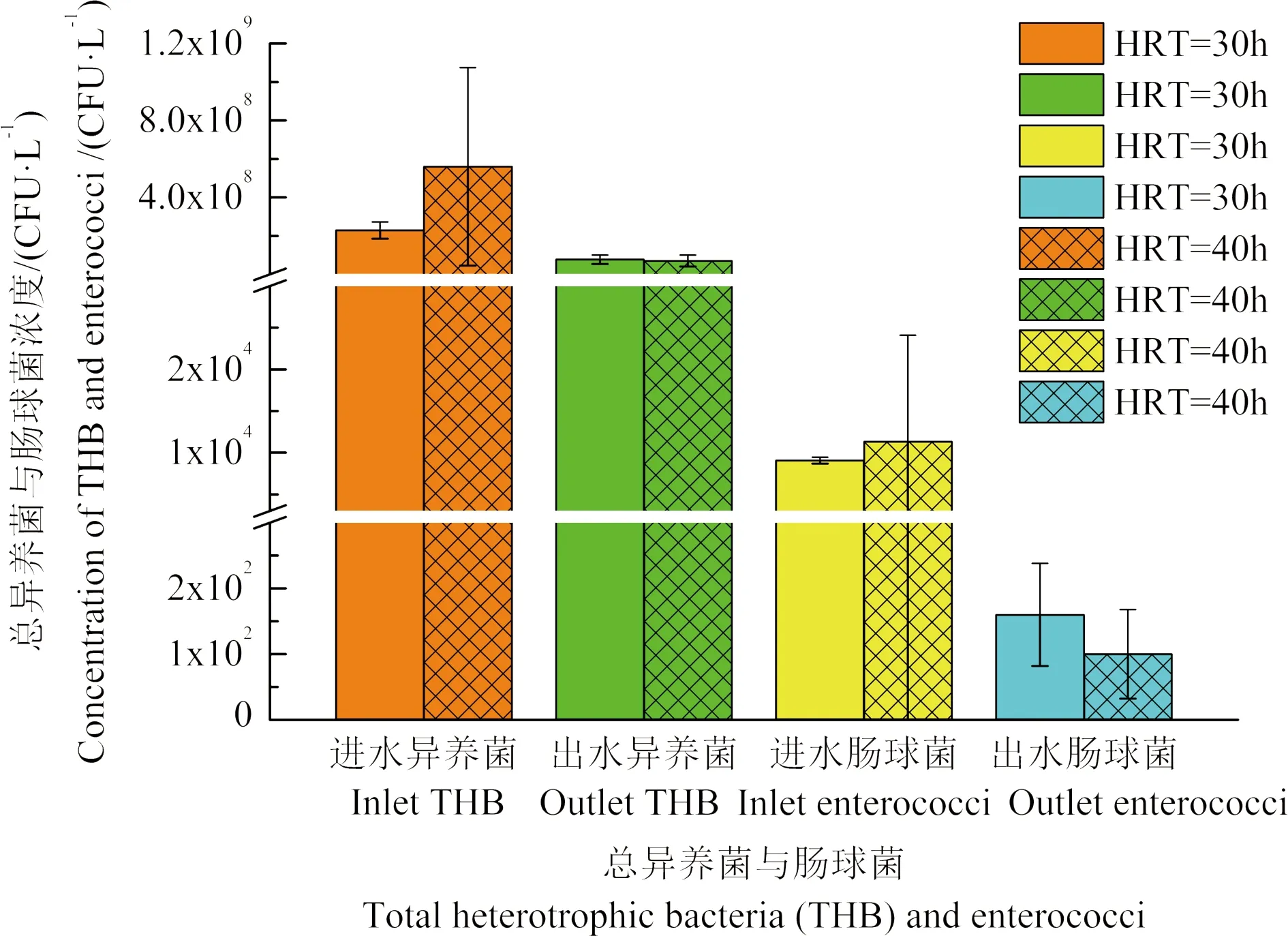
图2 MBR工艺处理过程中异养菌和肠球菌的浓度变化
2.3 HRT对肠球菌耐药率的影响
当HRT=40 h时,从样品中分离出95株肠球菌(进水44株,活性污泥样品20株,出水31株)分别对大环内酯类抗生素阿奇霉素、克拉霉素、红霉素、螺旋霉素进行药敏实验。进水中肠球菌对大环内酯类4种抗生素的耐药率分别为98%、93%、93%、75%,出水中耐药率都有明显的下降(见表3),耐药率分别为51%、54%、52%、39%。与进水肠球菌耐药率相比,MBR装置的活性污泥中肠球菌对红霉素和螺旋霉素的耐药率分别降低了18%和10%,对阿奇霉素和克拉霉素的耐药率则变化不大,仅分别降低了3%和升高了2%。当HRT=30 h时,共分离出了101株肠球菌(进水50株,活性污泥样品19株,出水32株)。MBR进水中肠球菌对阿奇霉素、克拉霉素、红霉素、螺旋霉素4种抗生素的耐药率均为100%,活性污泥样品中肠球菌的耐药率与进水肠球菌耐药率近似,为98%~100%,出水中肠球菌的耐药率分别为78%、79%、80%、82%。
由研究结果可知,不同水力停留时间的MBR处理工艺对大环内酯类4种抗生素的耐药率均有显著的削减效果。但在HRT为40 h时,MBR工艺出水中肠球菌耐药率下降了36%~47%;HRT降到30 h时,出水肠球菌的耐药率只比进水下降了18%~22%。此外,MBR活性污泥中的肠球菌在长HRT条件下对红霉素与螺旋霉素的耐药率更低。根据Kim等[20]的研究结果,活性污泥的有机负荷增加可导致四环素耐药菌的增加。本研究中短HRT条件下MBR内的污泥负荷更高,更有利于MBR内耐药肠球菌量增加,从而导致出水肠球菌耐药率相对较高。
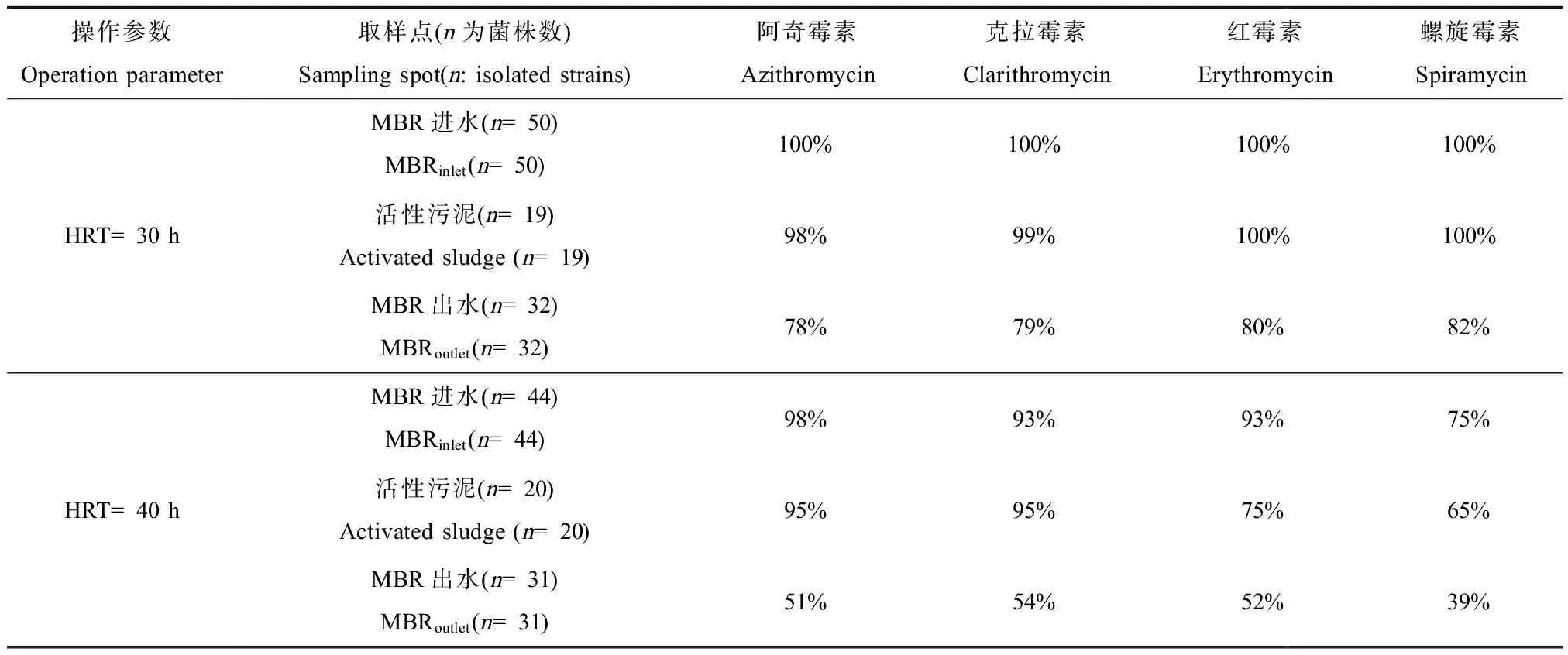
表3 HRT对肠球菌耐药率的影响Table 3 The resistance rate of enterococci at different HRT
2.4 HRT对肠球菌携带的抗性基因削减效果影响
不同水力停留时间下分离出的肠球菌携带的抗性基因如表4所示。已有文献报道ermB抗性基因的检出率高于MLS其他抗性基因的检出率[16,21]。本研究中分离出来的肠球菌ermB的携带率较高,而PCR结果均对mefA和intI1呈阴性。与Luna等[22]在临床分离出的肠球菌中检测到抗性基因mefA不同,我国的研究者在临床分离的肠球菌中未检测出mefA[23-24],或mefA检出率非常低[25],本研究也未检测出携带mefA的肠球菌,mefA检出率低可能是我国环境中肠球菌的一个抗性污染特征。当HRT=40 h时,MBR进水、活性污泥、MBR出水样品中肠球菌携带的抗性基因ermB检出率分别为88.5%、90.0%、48.4%,而HRT=30 h时的检出率分别为86.0%、89.4%、56.3%。出水肠球菌中ermB的检出率分别下降了40.2%和29.8%,当HRT缩短后,出水中肠球菌携带的ermB检出率明显增加。
2.5 HRT对基因组DNA中抗性基因相对丰度的影响
通过将各抗性基因归一化到16S rRNA,考察了抗性基因在MBR工艺处理螺旋霉素制药废水过程中的相对丰度变化(见图3)。由图3可知,进水中ermB和ermF的相对丰度最高。已有文献报道,ermB是在肠球菌中检出率较高的MLS抗性基因[26-27],并且肠球菌获得ermB基因是导致其对红霉素高水平耐药的主要原因[21]。ermB和ermF的相对丰度高,一个原因是宿主广泛,覆盖了革兰氏阳性菌(G+)和革兰氏阴性菌(G-)、好氧菌和厌氧菌[28],另外也可能是由于这些基因常常与结合型或非结合型转座子和质粒等转移元件易于传播有关[29]。
当MBR工艺以HRT=30 h运行时,抗性基因的丰度变化如图3(a)所示,ereA、ermX、mefA、mphB和转移元件Tn916/1545、ISCR1的丰度下降了15.2%~94.3%,而ermB、ermF和intI1的丰度在MBR处理之后均出现了反弹,增加了19.5%~38.0%。MBR工艺为HRT=40 h时(见图3(b)),除intI1的丰度增加25.2%之外,其他的抗性基因和转移元件丰度均成下降趋势,下降范围为24.8%~99.0%。整体来说,MBR工艺在长HRT条件下运行对抗性基因的削减效果更好。这可能是由于短HRT条件下,MBR内污泥浓度与污泥负荷更高,有利于耐药菌的增殖,从而导致出水抗性基因相对长HRT时更多。
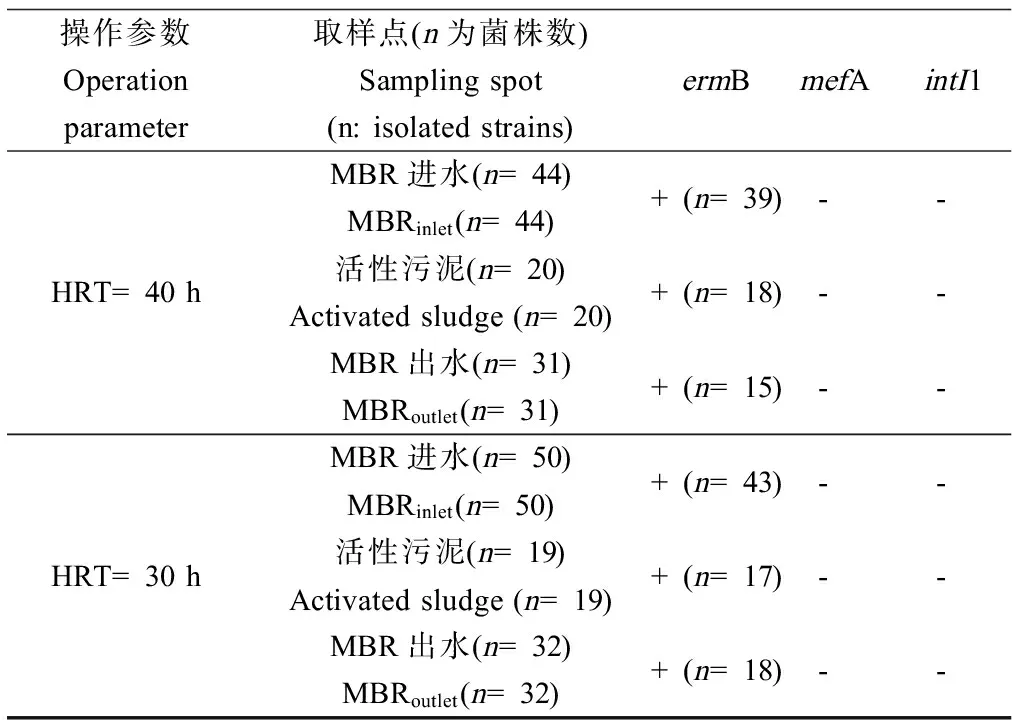
表4 肠球菌携带抗性基因的检测结果Table 4 Detection of antibiotic resistance genes in enterococci
注:“+”表示阳性,“-”表示阴性。
Note:+ positive; - negative.
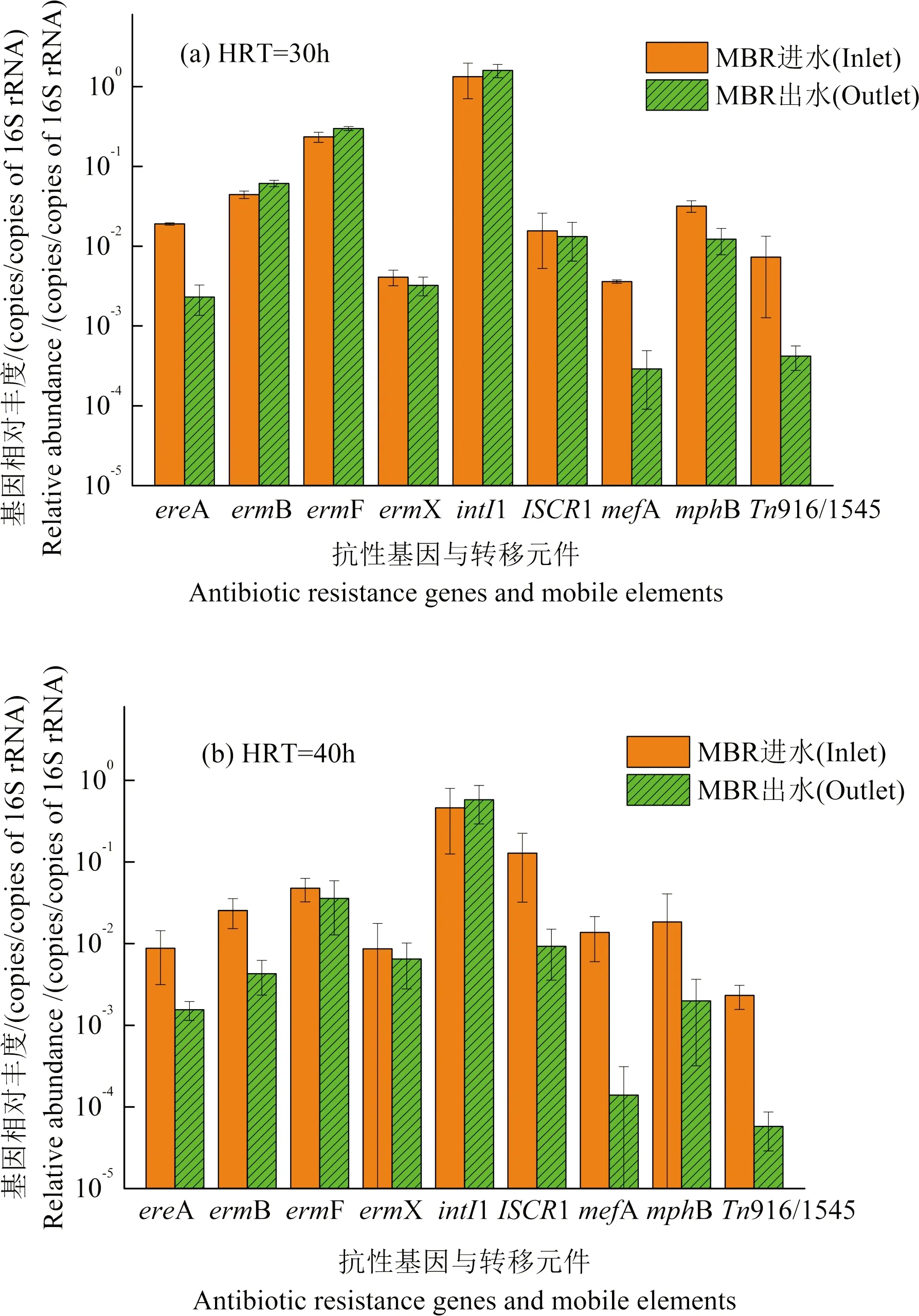
图3 不同HRT时抗性基因和转移元件的相对丰度变化
虽然目前对于污水厂出水并无耐药菌与抗性基因的要求,但是考虑到抗性污染传播将给人类健康带来的巨大风险,建议在今后实际污水处理过程中,应综合考虑出水常规指标与抗性污染控制来选择工艺操作参数,在经济条件允许的情况下,选择能够达到出水常规指标要求,又能减少出水中耐药菌与抗性基因的工艺条件,以期控制抗性污染传播。
本研究考察了不同HRT条件下MBR处理螺旋霉素废水过程中对肠球菌耐药性与抗性基因的去除效果,结论如下:
(1)当HRT分别为40 h与30 h时,MBR出水水质较为接近,HRT为30 h时,COD与氨氮的去除率分别为74.3%和97.7%,略高于HRT为40 h时的72.1%与96.2%。
(2)长HRT更有利于去除螺旋霉素废水中的总异养菌和肠球菌,出水中肠球菌耐药性、肠球菌携带抗性基因的检出也更低。此外,出水中6种抗性基因ermB、ermF、ermX、mefA、ereA、mphB和3种转移元件ISCR1、intI1、Tn916/1545在长HRT条件下也得到更好的去除。
(3)短HRT时MBR内污泥浓度与污泥负荷更高,有利于耐药菌的增殖,可能是导致出水抗性基因和耐药菌比长HRT时更多的原因之一。
[1] Chen H, Zhang M. Occurrence and removal of antibiotic resistance genes in municipal wastewater and rural domestic sewage treatment systems in Eastern China [J]. Environment International, 2013, 55: 9-14
[2] Pitondo-Silva A, Martins V V, Tonelli Fernandes A F, et al. High level of resistance to aztreonam and ticarcillin in Pseudomonas aeruginosa isolated from soil of different crops in Brazil [J]. Science of the Total Environment, 2014, 473: 155-158
[3] Wang C, Dang H, Ding Y. Incidence of diverse integrons and beta-lactamase genes in environmental Enterobacteriaceae isolates from Jiaozhou Bay, China [J]. World Journal of Microbiology & Biotechnology, 2008, 24(12): 2889-2896
[4] 朱开宪, 唐嘉. 抗生素废水的研究进展[J]. 广东化工, 2010(11): 121-122
Zhu K X, Tang J. Theresearch of treating antibiotic wastewater in an anaerobic fluidized bed reactor [J]. Guangdong Chemical Industry, 2010(11): 121-122 (in Chinese)
[5] Fick J, Soederstrom H, Lindberg R H, et al. Contamination of surface, ground, and drinking water from pharmaceutical production [J]. Environmental Toxicology and Chemistry, 2009, 28(12): 2522-2527
[6] Larsson D G J, De Pedro C, Paxeus N. Effluent from drug manufactures contains extremely high levels of pharmaceuticals [J]. Journal of Hazardous Materials, 2007, 148(3): 751-755
[7] Gundogdu A, Jennison A V, Smith H V, et al. Extended-spectrum beta-lactamase producing Escherichia coli in hospital wastewaters and sewage treatment plants in Queensland, Australia [J]. Canadian Journal of Microbiology, 2013, 59(11): 737-745
[8] Reinthaler F F, Posch J, Feierl G, et al. Antibiotic resistance of E. coli in sewage and sludge [J]. Water Research, 2003, 37(8): 1685-1690
[9] Schwartz T, Kohnen W, Jansen B, et al. Detection of antibiotic-resistant bacteria and their resistance genes in wastewater, surface water, and drinking water biofilms [J]. FEMS Microbiology Ecology, 2003, 43(3): 325-335
[10] Munir M, Wong K, Xagoraraki I. Release of antibiotic resistant bacteria and genes in the effluent and biosolids of five wastewater utilities in Michigan [J]. Water Research, 2011, 45(2): 681-693
[11] Wang J X, Li K, Wei Y S, et al. Performance and fate of organics in a pilot MBR-NF for treating antibiotic production wastewater with recycling NF concentrate [J]. Chemosphere, 2015, 121: 92-100
[12] 国家环境保护总局, 水和废水监测分析方法编委会. 水和废水监测分析方法(第四版)[M]. 北京: 中国环境科学出版社, 2002: 105-108, 210-213, 276-281
[13] Da Silva M F, Tiago I, Verissimo A, et al. Antibiotic resistance of enterococci and related bacteria in an urban wastewater treatment plant [J]. FEMS Microbiology Ecology, 2006, 55(2): 322-329
[14] Clinical and Laboratory Standards Institute (CLSI). Performance standards for antimicrobial susceptility testing; twenty-second informational supplement, M100- S22. [S]. Wayne, PA: Clinical and Laboratory Standards Institute, 2012
[15] Chen J, Yu Z, Michel F C Jr, et al. Development and application of real-time PCR assays for quantification of erm genes conferring resistance to Macrolides-Lincosamides-Streptogramin B in livestock manure and manure management systems [J]. Applied and Environmental Microbiology, 2007, 73(14): 4407-4416
[16] Sutcliffe J, Grebe T, Taitkamradt A, et al. Detection of erythromycin-resistant determinants by PCR [J]. Antimicrobial Agents and Chemotherapy, 1996, 40(11): 2562-2566
[17] Quiroga M P, Andres P, Petroni A, et al. Complex class 1 integrons with diverse variable regions, including aac(6 ')-Ib-cr, and a novel allele, qnrB10, associated with ISCR1 in clinical enterobacterial isolates from Argentina [J]. Antimicrobial Agents and Chemotherapy, 2007, 51(12): 4466-4470
[18] Stokes H W, Nesbo C L, Holley M, et al. Class 1 integrons potentially predating the association with Tn402-like transposition genes are present in a sediment microbial community [J]. Journal of Bacteriology, 2006, 188(16): 5722-5730
[19] Novo A, Manaia C M. Factors influencing antibiotic resistance burden in municipal wastewater treatment plants [J]. Applied Microbiology and Biotechnology, 2010, 87(3): 1157-1166
[20] Kim S, Jensen J N, Aga D S, et al. Fate of tetracycline resistant bacteria as a function of activated sludge process organic loading and growth rate [J]. Water Science and Technology, 2007, 55(1-2): 291-297
[21] Portillo A, Ruiz-Larrea F, Zarazaga M, et al. Macrolide resistance genes in Enterococcus spp [J]. Antimicrobial Agents and Chemotherapy, 2000, 44(4): 967-971
[22] Luna V A, Coates P, Eady E A, et al. A variety of Gram-positive bacteria carry mobile mef genes [J]. Journal of Antimicrobial Chemotherapy, 1999, 44(1): 19-25
[23] 李爽, 张正. 粪肠球菌与屎肠球菌药敏表型和耐药基因的比较[J]. 临床检验杂志, 2005(3): 174-176
Li S, Zhang Z. Detection for phenotype and genetype comparation of Enterococcus faecalis with Enterococcus faecium [J]. Chinese Journal of Clinical Laboratory Science, 2005(3): 174-176 (in Chinese)
[24] 黄支密, 石晓霞, 糜祖煌, 等. 肠球菌抗生素耐药基因检测[J]. 中华医院感染学杂志, 2006(1): 1-5
Huang Z M, Shi X X, Mi Z H, et al. Detection on antibiotics-resistant genes in enterococci [J]. Chinese Journal of Nosocomiology, 2006(1): 1-5 (in Chinese)
[25] 吕萍, 徐樨巍, 宋文琪, 等. 儿童肠球菌耐药表型和耐药基因检测分析[J]. 中国实用儿科杂志, 2009(12): 937-939
Lv P, Xu X W, Song W Q, et al. Detection for phenotype and genetype of the enterococci in children [J]. Chinese Journal of Practical Pediatrics, 2009(12): 937-939 (in Chinese)
[26] Klare I, Konstabel C, Badstübner D, et al. Occurrence and spread of antibiotic resistances in Enterococcus faecium [J]. International Journal of Food Microbiology, 2003, 88: 269-290
[27] Koike S, Aminov R I, Yannarell A C, et al. Molecular ecology of Macrolide-Lincosamide-Streptogramin B methylases in waste lagoons and subsurface waters associated with swine production [J]. Microbial Ecology, 2010, 59(3): 487-498
[28] Roberts M C, Sutcliffe J, Courvalin P, et al. Nomenclature for Macrolide and Macrolide-Lincosamide-Streptogramin B resistance determinants [J]. Antimicrobial Agents and Chemotherapy, 1999, 43(12): 2823-2830
[29] 方小龙, 陈群. 肠球菌耐药机制研究进展[J]. 医学综述, 2006, 12(19): 1165-1168
Fang X, Chen Q. Progress in the drug fast mechanism of enterococcus[J]. Medical Recapitulate, 2006, 12(19): 1165-1168 (in Chinese)
◆
Reduction of Antibiotic Resistant Bacteria and Genes in Spiramycin Wastewater Treated by Membrane Bioreactor (MBR) Process
Qin Caixia1,2, Zhang Junya1, Tong Juan1,*, Shen Peihong2, Wei Yuansong1,3
1. State Key Joint Laboratory of Environmental Simulation and Pollution Control, Research Center for Eco-Environmental Sciences, Chinese Academy of Sciences, Beijing 100085, China 2. College of Life Science and Technology, Guangxi University, Nanning 530005, China 3. Institute of Energy, Jiangxi Academy of Sciences, Nanchang 330096, China
Received 19 September 2015 accepted 11 December 2015
Antibiotics production wastewater contains large amounts of antibiotic resistant bacteria (ARB) and antibiotic resistance genes (ARGs), and the treated wastewater would improve the antibiotic resistance of the environmental bacteria. It is necessary to investigate the reduction of ARB and ARGs during the treatment of antibiotics production wastewater. In this study, the spiramycin production wastewater was treated by membrane bioreactor (MBR), and the removal of ARB and ARGs under two hydraulic retention times (HRT) were investigated. The results revealed that the COD and NH4+-N removal performances were slightly higher at HRT 30 h than at 40 h, however, the removals of total heterotrophic bacteria and enterococci were better at HRT 40 h, as well as the antibiotic resistance rate of enterococci, and the detection of ARGs carried by enterococci were lower at HRT 40 h. Furthermore, the relative abundances of ARGs (ermB, ermF, ermX, mefA, ereA and mphB) and mobile elements (ISCR1 and Tn916/1545) were reduced more at HRT 40 h than at 30 h. These results indicated that longer HRT for MBR treating spiramycin wastewater would achieve better removal efficiencies of the ARB and ARGs.
membrane bioreactor (MBR); antibiotic resistant bacteria (ARB); antibiotic resistance genes (ARGs); spiramycin wastewater; hydraulic retention time (HRT)
国家水体污染控制与治理科技重大专项(2012ZX07203-002);国家自然科学基金项目(21207147);公益性行业(农业)科研专项(201303091)
覃彩霞(1990-),女,硕士研究生,研究方向为资源与环境微生物,E-mail: qincaixia1990@163.com
*通讯作者(Corresponding author),E-mail: hittj@163.com
10.7524/AJE.1673-5897.20150919001
2015-09-19录用日期:2015-12-11
1673-5897(2015)5-100-08
X171.5
A
佟娟(1977—),女,博士,助理研究员,主要研究方向为污水/污泥处理与资源化利用技术,污水/污泥处理过程中的抗性污染研究。
覃彩霞, 张俊亚, 佟娟, 等. MBR工艺处理螺旋霉素制药废水过程中抗生素耐药菌与抗性基因的研究[J]. 生态毒理学报,2015, 10(5): 100-107
Qin C X, Zhang J Y, Tong J, et al. Reduction of antibiotic resistant bacteria and genes in spiramycin wastewater treated by membrane bioreactor (MBR) process [J]. Asian Journal of Ecotoxicology, 2015, 10(5): 100-107 (in Chinese)

