东海陆架表层沉积物细菌群落结构及地理分布研究*
刘明华 王健鑫 俞凯成 蒋 然 刘晓辉 王帅兵 刘雪珠
(浙江海洋学院海洋微生物生态与应用实验室 舟山 316022)
海洋占据了地球总面积的71%, 拥有丰富的生物资源。微生物在海洋环境中扮演着重要角色, 包含细菌、真菌、放线菌及病毒在内的海洋微生物不仅为地球提供了近一半的初级生产力, 为现代工业生产提供了重要的天然药物和酶资源(Arrigo, 2005),同时参与物质和能量循环(Simmonset al, 2008), 影响全球气候变化(Azamet al, 2004)。海洋沉积物是地球上面积最大的覆盖层, 具有低温、高压、寡营养和无光照等极端环境特征, 孕育了大量特殊生理特性的微生物类群, 成为地球上最复杂的微生物栖息地, 并在整个海洋生态系统的生物地球化学循环过程中发挥着重要的作用(Kösteret al, 2008)。因此开展海洋沉积环境中微生物资源及其多样性研究是海洋微生物从资源走向应用的关键(刘玉娟等,2014)。
东海是西太平洋构造活动带中一个大型边缘海,处于 21º54′—33º17′N, 117º05′—131º03′E 之间, 是西太平洋沟-弧-盆体系的典型发育地区(李家彪, 2008)。东海海域的水文状况及沉积动力环境较为复杂, 碎屑沉积物的搬运和沉积作用主要取决于沉积动力条件。东海海流系统主要由黑潮、台湾暖流、东海沿岸流, 即外来的黑潮流及其分支和当地生成的沿岸流等组成(李万超, 2008)。典型的地质构造和多重的海流组成使得东海陆架不仅是研究生物地球化学循环的重要区域, 也是研究海底各种沉积作用和微生物群落多样性的有利场所。关于东海微生物多样性的研究, 早期多集中在表层海水环境和可培养微生物(郑国兴等, 1982; 宋志刚等, 2006; 卢婧雯等,2012), 近年来关于陆架沉积物微生物的研究成为新的热点领域(Parket al, 2008; 张东声, 2011; 杜萍等,2012; 王健鑫等, 2012; 有小娟等, 2013), 当然采样区域、时间、方法以及研究对象、技术手段、结果都各有侧重。
海洋环境中绝大多数微生物都是处于未可培养的状态(Arakakiet al, 2010), 影响了对环境微生物的深入研究。随着近几年分子生物学技术的普及, 从DNA水平研究微生物的多样性已成为当前研究最主要的技术手段, 并推动了微生物海洋学的快速发展(Venteret al, 2004)。本文采用16S rRNA 基因克隆测序, 对东海陆架五个站点(DH6, DH9, DH16, DH17,DH21)沉积物样品进行细菌种群多样性、群落结构和地理分布规律进行初步探讨, 以期为东海陆架沉积环境微生物分子生态学研究提供参考。
1 材料与方法
1.1 样品采集
海洋沉积物样品采集自 2007年 4—5月海洋调查项目, 五个采样站点的位置信息如表 1和图 1所示。沉积物柱芯样品采集后立即分装于无菌样品袋中, −20°C保存于科考船冰柜中, 后置于实验室−80°C超低温冰箱中长期保存, 沉积物主要以中细砂为主。
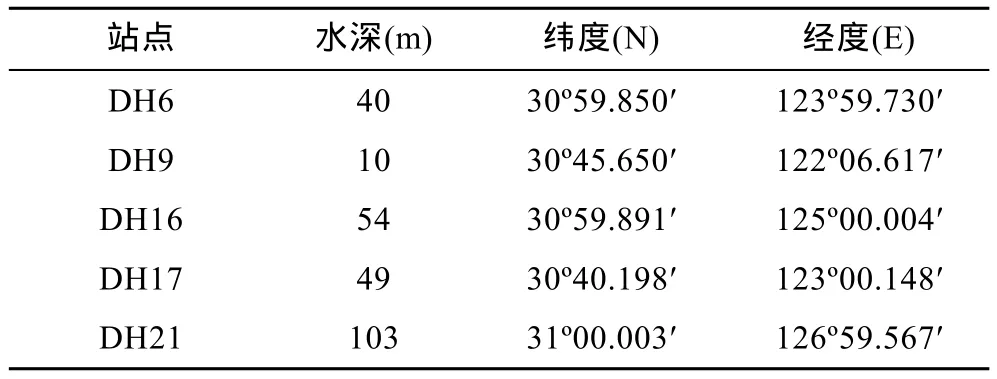
表1 各采样点信息Tab. 1 The information of five sites

图1 各采样站点分布示意图Fig.1 Map of five sampling sites
1.2 样品总DNA的提取与纯化
利用FastPrep®-24快速核酸提取仪(MP Biomedicals公司)和 Fast DNA spin kit for soil 试剂盒进行沉积物样品DNA提取, 核酸蛋白检测仪(Bio-Rad公司)测定DNA浓度和纯度,纯化后用于 PCR扩增。
1.3 PCR扩增和产物回收
将样品总DNA在梯度PCR仪(Biometra公司)中进行扩增,所用引物为 341F (CCTACGGGAGGCAG CAG)和 907R (CCGTCAATTCMTTTRAGTTT)。50μL PCR 扩增体系: DNA 模板 1μL, 反应引物 341F(10μmol/L)和 907R (10μmol/L)各 1μL, 10×PCR buffer(含 MgCl21.5mmol/L) 5μL, dNTP (2.5mmol/L) 5μL,rTaq 聚合酶(5U/μL) 0.5μL, ddH2O 36.5μL。PCR 扩增循环条件为: 94°C预变性5min; 94°C变性30s, 55°C退火40s, 72°C延伸40s, 35个循环; 最后72°C延伸7min。1.2%琼脂糖凝胶电泳检测 PCR扩增结果, 对条带(500—750bp)进行割胶, 用 DNA 胶回收试剂盒(QIAGEN公司)回收产物。
1.4 克隆子筛选和测序
PCR割胶回收产物连接pMD-18T(TaKaRa)载体,转化于Escherichia coliDH5α感受态细胞。经蓝白斑筛选, 挑取白斑进行液体培养过夜, 再通过菌液PCR进行扩增, 扩增 10μL 体系为: Extaq 5μL, ddH2O 4.1μL, 341F (10μmol/L)和 M13R (10μmol/L)各 0.2μL,菌液0.5μL。PCR扩增循环条件为: 95°C预变性5min;94°C 变性 1min, 58°C 退火 40s, 72°C 延伸 40s, 35个循环; 最后72°C延伸10min。电泳检测, 选取500—750bp之间的克隆送苏州金唯智生物科技有限公司进行检测。
1.5 序列分析
将序列提交到 RDPⅡ数据库, 利用在线工具CHECK-CHIMERA检测嵌合体; 应用 BLASTN程序搜索相似性序列, 进行系统发育分析。采用ClustalX (Version 1.8)进行序列比对分析, 通过MEGA 5软件里Neighbor-joining法构建系统发育树(Kumaret al, 2004)、利用 DOTUR软件确定分类单位(OTU)(Schlosset al, 2005)。细菌16S rDNA序列在GenBank核苷酸数据库中的接受号为 KR086422-KR086717。
2 结果与分析
2.1 细菌16S rDNA文库数据分析
对公司返回的序列进行去载体和嵌合体处理,共得到 508有效克隆子, 通过 DOTUR 软件分析,将相似度大于97%的有效克隆子归为一个OTU, DH6、DH9、DH16、DH17和DH21五个站点的OTU分别为47、82、36、69和86, 并获得五个站点各自的Biascorrected Chao 1、ACE丰富度指数、香农指数(H′)和辛普森指数(D)(如表2所示)。利用生成的Rarefaction数据, 制作稀释性曲线(图2)。
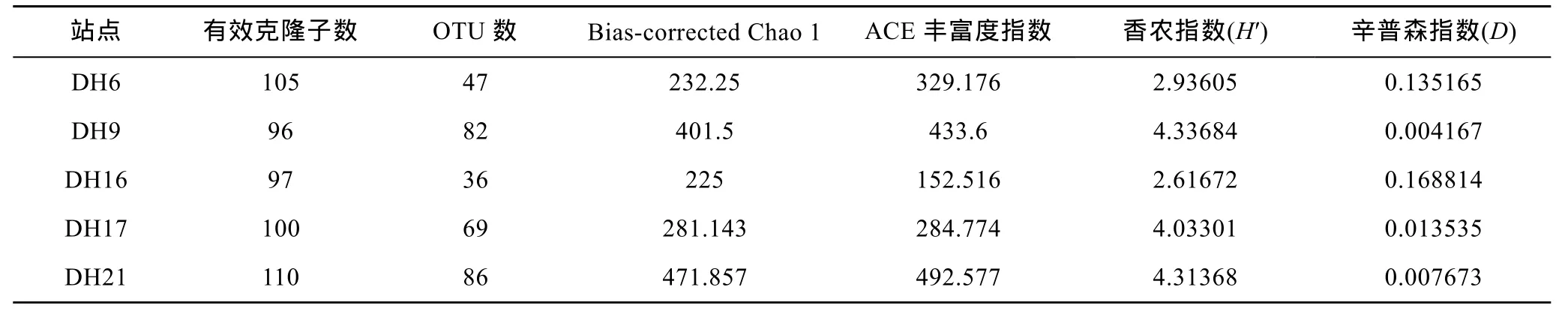
表2 东海陆架表层沉积物细菌多样性指数Tab. 2 The bacterial diversity index from the surface layer sediments of the East China Sea

图2 东海站点克隆文库稀释曲线Fig.2 Rarefaction curve from five sampling sites of the East China Sea
文库分析结果表明: DH21和 DH9两个站点的Bias-corrected Chao 1和ACE丰富度指数显著高于其它三个站点; 而辛普森指数(D)显著低于其它三个站点, 可见DH9和DH21站点多样性较高, DH17站点次之, DH6和DH16两个站点的多样性最低。从稀释曲线上来看, 五个站点多样性由高到低排列依次为:DH9>DH21>DH17>DH6>DH16, 与指数分析结果基本一致; 随着克隆子数量的增加, 稀释曲线的斜率有一定程度的减小, 但DH9、DH21和DH17三个站点的斜率仍然较大, 说明克隆子和 OTU的数量还没有覆盖站点细菌多样性的真实范围。
2.2 细菌群落结构分析
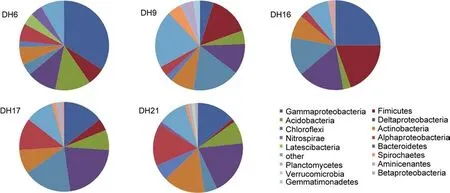
图3 东海各站点表层沉积物菌群组成Fig.3 Composition of clone libraries for surface layer sediments in five sites of the East China Sea
对返回的DNA序列参考NCBI Taxonomy上的分类方法, 变形菌门以“纲”为分类单位, 其它细菌以“门”为分类单位, 共得到包括变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、酸杆菌门(Acidobacteria)、绿弯菌门(Chloroflexi)、放线菌门(Actinobacteria)、硝化螺旋菌门(Nitrospira)、WS3、拟杆菌门(Bacteroidetes)、螺旋体门(Spirochaetes)、OP8、浮霉菌门(Planctomycetacia)、疣微菌门(Verrucomicrobiae)和芽单胞菌门(Gemmatimonadetes)等 13个类群, 其中变形菌门包含 α-变形菌纲(Alpha-proteobacteria)、β-变形菌纲 (Beta-proteobacteria)、 γ-变 形 菌 纲 (Gammaproteobacteria)和 δ-变形菌纲(Delta-proteobacteria),五个站点的细菌多样性及群落组成结构如图3所示。
由图3可知, 变形菌门是东海陆架沉积物中的优势菌群, DH6、DH9、DH16、DH17和DH21五个站点变形菌门的 OTU数占总 OTU的百分比分别为51%、23%、44%、48%和50%。在变形菌门中, γ-变形菌和 δ-变形菌是两个主要的典型类群, 但在不同的站点, 其丰度也存在一定的差异: DH6和DH16站点 γ-变形菌丰度高于 δ-变形菌, 而 DH9、DH17和DH21站点的δ-变形菌丰度又明显高于γ-变形菌。α-变形菌在各个站点也均有分布, DH21站点的α-变形菌丰度最高(含13个OTU, 占比为15.1%), DH17站点的 α-变形菌丰度次之(8个 OTU, 占比为 11.6%),DH6、DH9和DH16站点丰度较低。β-变形菌丰度最低, 只有在DH9和DH21两个站点有分布, 分别占总OTU的3.7%和1.1%。
除了优势菌群外, 厚壁菌门、绿弯菌门、酸杆菌门和放线菌门在各个站点均有分布, 其丰度和多样性差异较大。厚壁菌门在 DH16站点的丰度最高(占16.4%), DH9站点次之(12个 OTU, 占比 14.6%), 其它三个站点均在7%以下。绿弯菌门在DH17站点的丰度最高(占比17.4%), DH9和DH16站点也有较高的丰度(分别为 17.1%和13.9%), 而DH6和DH21站点绿弯菌所含比例仅为 4.2%和 4.7%。另外, 酸杆菌门在各个站点的丰度并不高, 除 DH6站点的占比达到12.8%外, 其它站点丰度均在 10%以下; 而放线菌门在 DH21站点(14个 OTU)丰度最高, 占总 OTU的15.1%。
还有一些门类并不是所有站点都有分布, 且丰度普遍较低。如硝化螺旋菌门只在DH6、DH9和DH21站点分布, 占比分别为2.1%、2.4%和5.8%; WS3只存在于 DH6站点, 占比为 4.3%; 拟杆菌门只存在于DH6和DH21两个站点中, 占比分别为4.2%和1.2%;螺旋体门和浮霉菌门存在于DH9、DH17和DH21站点, 都只有唯一的OTU分布; OP8在DH16和DH17站点各有一个OTU分布; 芽单胞菌门只在DH9站点有一个 OTU分布; 疣微菌门也只在 DH21站点有一个OTU分布。另外5个站点还有8.3%—20.7%的分类地位不确定的微生物。
2.3 16S rRNA基因序列的系统进化分析
2.3.1 DH6站点系统发育树分析 DH6站点的105个有效克隆子可分为11个细菌类群(图4), 其中γ-变形菌纲占47个OTU的36%, 是该站点的优势菌群, 以基因型DH61B3(38个克隆子)为代表的序列与爱琴海沉积物嗜冷杆菌(Psychrobactersp. 3BM13Y12)同源性较高(99%), 基因型DH61B46与东海陆架沉积物未培养γ-变形菌DH133B19克隆有高同源性(99%),基因型 DH62B33与大洋地壳不可培养 γ-变形菌P0X4b3H08克隆有高同源性(99%), DH61B56序列和澳大利亚大堡礁附近沉积物未培养 γ-变形菌 R76-92克隆相似度达 99%。δ-变形菌在 DH6站点也有一定分布, 基因型 DH61B39与比塞大瀉湖(突尼斯)沉积物未培养 δ-变形菌 2C48克隆有高同源性(99%),DH62B39序列与冲绳中部槽伊平屋村北场深海热液喷口附近沉积物未培养δ-变形菌IBP10m-6克隆有高同源性(99%)。α-变形菌则以基因型DH62B34为代表,其序列与来自海洋死亡区(最小含氧区)的未培养细菌SGSX1079克隆同源性最高(100%)。
酸杆菌门有6个OTU, 代表基因型DH61B48与南大西洋海岸线沙滩的未培养细菌LC3-26克隆高同源性(99%)。拟杆菌门在东海含量较低(2个克隆子),代表基因型DH62B28序列和南太平洋东劳扩张中心(ELSC)的深海热液区未培养 Bacteroidetes bacterium clone 44有高同源性(99%)。放线菌代表基因型DH62B38序列与西南印度洋中脊沉积物中未培养细菌T13J-B77克隆同源性较高(99%)。绿弯菌门代表基因型DH62B36序列与东太平洋中国结核区的未培养细菌ES0303-B80克隆同源性高(99%)。厚壁菌代表基因型DH61B16(2个克隆子)与青藏高原湖泊细菌未培养细菌Fi09-23克隆具有高相似性(99%)。硝化螺旋菌在该区有少量分布, DH61B60序列与香港维多利亚港沉积物未培养Nitrospirae bacterium VHS-B4-29克隆有较高的同源性(99%)。WS3是DH6站点特有的类群, 其 DH62B29序列与法属圭亚那海岸的未培养细菌克隆5_64具有很高的同源性(99%)。
2.3.2 DH9站点系统发育树分析 从DH9站点的系统发育树中可以发现, 变形菌门虽丰度不高, 但覆盖了 4个纲(图 5所示)。δ-变形菌其代表基因型DH94B1与西班牙西北海岸谢斯群岛漏油区域未培养细菌 ANOX-036克隆序列有高同源性(99%), 基因型 DH94B15序列与马洛卡岛(地中海附近海域)原油污染响应的未培养硫酸盐还原菌(SRB)MS-B135克隆相似度较高(99%), 这与 δ-变形菌纲一些种类可以在厌氧条件下还原硫酸盐的性质相符。β-变形菌代表基因型DH92B6与黄瓜根际未培养细菌HG-J0184克隆同源性高(99%), 另外基因型 DH91B8与长江口崇西湿地芦苇根际未培养细菌 P-B269克隆同源性较高(98%)。γ-变形菌和 α-变形菌在该站点只有很少的类群存在, 其中代表基因型 DH94B16序列与椒江口沉积物未培养 γ-变形菌克隆 C6_54同源性高(99%),DH91B9序列与法属圭亚那海岸沉积物未培养 α-变形菌克隆4_165相似性较高(99%)。

图4 DH6站点系统发育树Fig.4 Phylogenetic tree for DH6 site
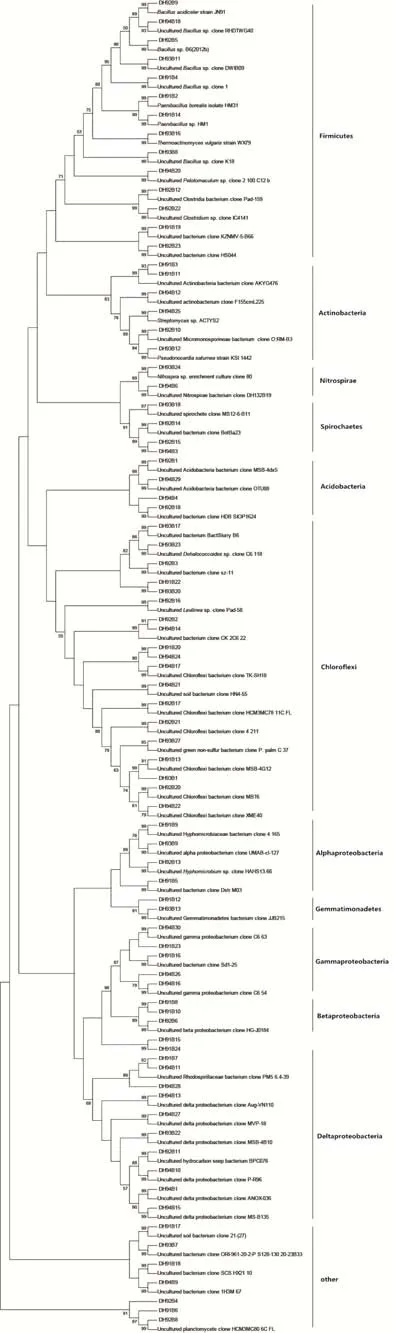
图5 DH9站点系统发育树Fig.5 Phylogenetic tree for DH9 site
绿弯菌门和厚壁菌门是 DH9站点的两个主要类群, 分别占 17%和 14%。绿弯菌门类群中的基因型DH94B17与非洲中部坦噶尼喀湖沉积物的未培养细菌TK-SH18克隆同源性较高(99%), 基因型DH92B21与法属圭亚那海岸沉积物未培养绿弯菌 4_211克隆具有较高亲缘性(99%), 基因型DH92B17与希腊克里特岛海域沉积物未培养细菌HCM3MC78_11C_FL克隆同源性较高(99%)。厚壁菌门类群中代表基因型DH92B22序列(2个克隆子)与斯瓦尔巴特群岛Austre Lovénbreen冰川样品的未培养细菌IC4141克隆同源性较高(99%), DH94B20序列与含烃蓄水层的Pelotomaculumsp. clone 2_100_C12_b有高同源性(相似度99%)。
硝化螺旋菌门共有两个 OTU, 基因型 DH93B24序列与地中海瀉湖石油污染沉积物中的可培养Nitrospirasp. clone 80同源性较高(99%), DH94B6序列与同样来自东海沉积环境的未培养细菌 DH132B19克隆同源性达99%。螺旋体门中的DH 92B14序列与珠江口沉积物未培养细菌 BotBa23克隆有很高的同源性(99%)。酸杆菌门在该站点有分布, 其中基因型94B29序列与南沙海湾养殖区沉积物的未培养细菌 OTU88克隆同源性较高(99%)。芽单胞菌门是DH9站点特有的菌群, 其代表基因型 DH91B12与黄海大陆架沉积物Gemmatimonadetes bacterium clone JJB215相似度达99%。
2.3.3 DH16站点系统发育树分析 由图 6可知,DH16站点中 γ-变形菌纲含量最高, 其中代表基因型DH161B1(38个克隆子)与椒江口沉积物未培养细菌C5B01克隆的同源性最高(100%), 基因型DH162B12与挪威斯瓦尔巴特群岛水样中的未培养军团菌目克隆Legionellales bacterium SSIM-D10具有很高的同源性(99%), 基因型 DH162B56与南大西洋拉普拉塔河的沉积物未培养细菌克隆 γ-proteobacterium ARTE12_226的亲缘性比较近(99%)。另外还有少量的δ-变形菌克隆子, 其代表基因型DH162B19 (3个克隆子)与马洛卡沙性沉积物中的未培养细菌 MS-K53克隆具有很高亲缘性(100%)。
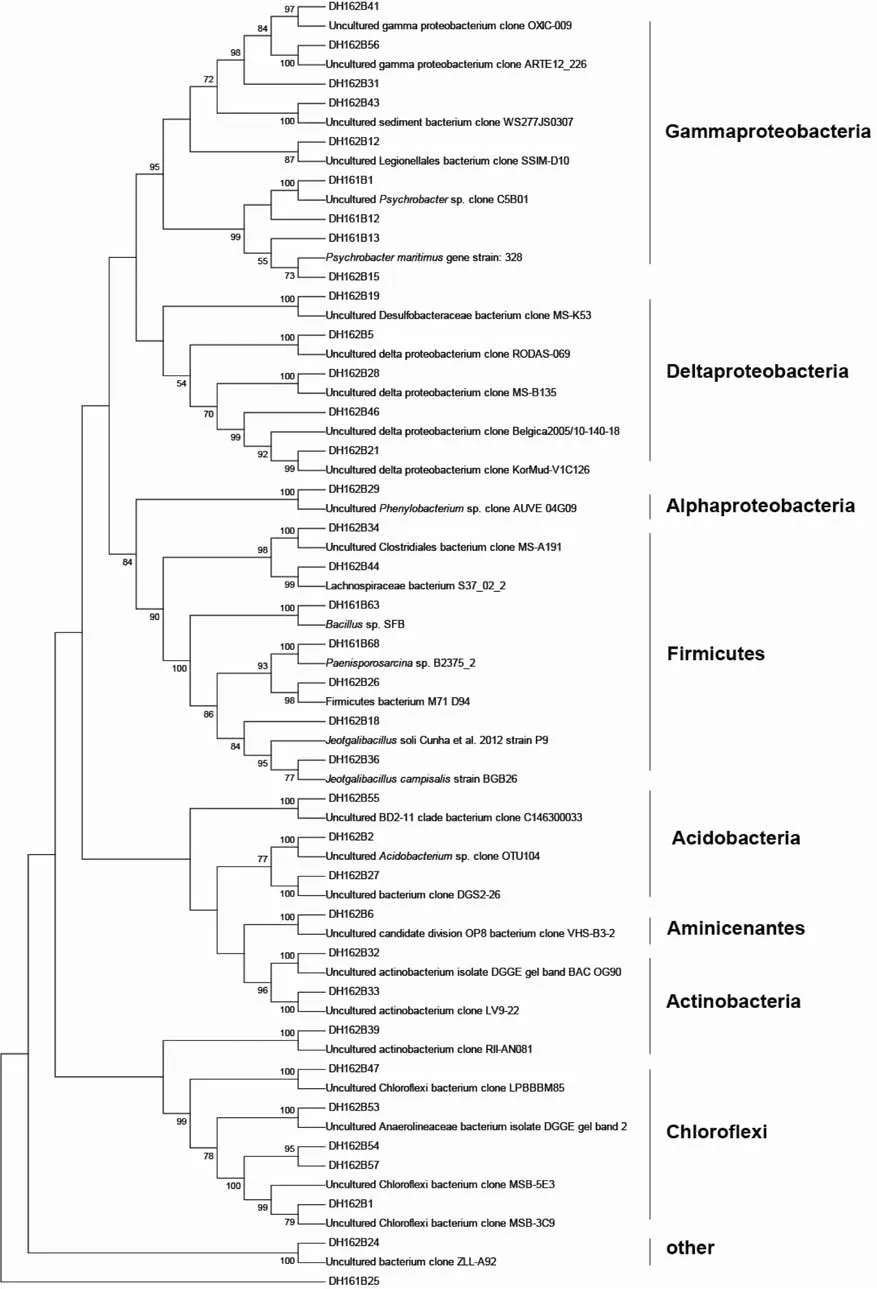
图6 DH16站点系统发育树Fig.6 Phylogenetic tree for DH16 site
厚壁菌门的含量仅次于 γ-变形菌纲(7个 OTU),该类群的基因型 DH162B34序列与地中海马洛卡岛沿岸附近的沉积物层未培养梭菌目克隆 Clostridiales bacterium MS-A191的相似度最高(100%), DH162B26序列则与地中海东部深海未培养克隆子 Fimicutes bacterium M71_D94表现出高同源性(99%)。放线菌在该区域只有3个OTU分布, 其中DH162B32序列与北海区域的沉积物未培养放线菌 BAC-OG90相似度最高(100%)。DH162B2序列与南沙海湾的网箱养殖区域沉积物酸杆菌克隆子Acidobacteriumsp.OTU104的同源性高达 100%。绿弯菌门在该站点有分布, 其中DH162B53序列与摩卡岛浅甲烷冷泉附近未培养克隆子Anaerolineaceae bacterium isolate DGGE gel band 2有高的同源性(100%)。DH16站点还有独特的OP8类群分布, 其基因型DH162B6与香港维多利亚港未培养沉积物细菌candidate division OP8 bacterium VHS-B3-2克隆同源性为100%。
2.3.4 DH17站点系统发育树分析 γ-变形菌纲和δ-变形菌纲是DH17站点两个主要的类群(图7), 其中γ-变形菌纲的代表基因型 DH172B7(5个克隆子)意大利里窝那港海底沉积物未培养细菌isolate 1.2, clone 140有较高同源性(98%), DH173B23序列和墨西哥Alchichica盐碱湖的未培养细菌 Alchichica_AQ1_1_1B_07克隆同源性比较高(99%); δ-变形菌纲中的基因型 DH171B20(3个克隆子)序列与意大利里窝那港海底沉积物的未培养细菌isolate 2.1, clone 59b克隆有较高同源性(99%), 基因型 DH173B46与南沙海湾养殖区沉积物的未培养细菌 OTU146克隆有很高亲缘性(99%)。α-变形菌纲类群中 DH173B41序列与甲基杆菌Methylobacterium komagataen23e-3序列亲缘性比较高(99%), DH173B12(3个克隆子)序列与 α-变形菌纲中的未培养醋杆菌 Acetobacteraceae bacterium AMIG7克隆相似度为99%。

图7 DH17站点系统发育树Fig.7 Phylogenetic tree for DH17 site
绿弯菌门在该站点的含量比较丰富, 其中代表基因型DH171B9(6个克隆子)与坦噶尼喀湖得到的未培养细菌 TK-SH18克隆具有较高的同源性(97%),DH172B23序列与墨西哥大陆坡海湾的未培养细菌IODP1320B2H7.13克隆具有高同源性(99%), 基因型DH173B15序列则和厦门红树林沉积物未培养细菌XME13克隆相似度为99%。酸杆菌门类群中的基因型 DH172B10序列与日本竹富岛海底热液环境中的未培养细菌 pItb-vmat-12克隆具有高度同源性(99%)。厚壁菌门的代表基因型DH171B13序列与地中海马洛卡岛沿岸附近的沉积物未培养梭菌目 MSC192克隆的相似度最高(99%)。螺旋体门和浮霉菌门在 DH17站点分布较少, 螺旋体门代表基因型DH172B3序列与日本下北半岛的海底沉积物中未培养细菌 C9001C_B01_1_C047克隆具有高同源性(99%); 浮霉菌门代表基因型 DH172B14与南沙海湾养殖区沉积物的未培养细菌 OTU97克隆相似性达99%。OP8在该站点也有分布, 其基因型 DH172B15与法属圭亚那海岸沉积物未培养细菌 1_145克隆具有很高亲缘性(99%)。
2.3.5 DH21站点系统发育树分析 由图 8可知,DH21站点的细菌多样性非常丰富。δ-变形菌纲在该站点的丰度较高, 代表基因型DH211B18(3个克隆子)与胶州湾未培养沉积物细菌JBS_E404克隆高度同源(100%), 基因型DH216B6(2个克隆子)和DH213B2(5个克隆子)序列分别与西班牙西北海岸谢斯群岛漏油区域获得的未培养细菌OXIC-088和RODAS-105克隆高度同源(100%)。α-变形菌纲中的代表基因型DH212B2(6个克隆子)与盐碱土壤未培养生丝微菌克隆Hyphomicrobiumsp. HAHS13.66同源性很高(99%),DH211B16序列与印度西部阿拉伯海岸坎贝湾沿岸土壤细菌ONGS204克隆有高同源性(98%), DH215B2序列与南大西洋海岸线的海洋沙滩未培养细菌 LC1-34克隆高度同源(100%)。γ-变形菌纲的代表基因型DH215B12(3个克隆子)与意大利里窝那港海底沉积物未培养细菌isolate 1.2,clone 147同源性高达100%,DH213B14序列和同样来自西班牙西北海岸谢斯群岛漏油区域沉积物的未培养细菌 PET-057克隆高度同源(100%)。β -变形菌在该站点分布很少, 且得到的序列DH216B13与地中海海绵区未培养细菌Nitrosospirasp. Msd8克隆高度同源(100%)。

图8 DH21站点系统发育树Fig.8 Phylogenetic tree for DH21 site
放线菌门在该站点比较丰富, 代表基因型 DH214B16与希腊南部克里特岛的附近好氧沉积物未培养细菌HCM3MC80_9D_FL克隆具有很高的亲缘性(100%),另外基因型 DH216B10相似度很高的序列来自北海(54°4'N/4°E)区域沉积物, 与未培养细菌 isolate DGGE gel band BAC_OG90克隆同源性高达100%。酸杆菌门在 DH21站点分布同样较多, 基因型DH215B6序列与西班牙西北海岸谢斯群岛漏油区域沉积物的未培养细菌 PET-051克隆同源性最高(100%), 基因型 DH215B13与来自东海陆架 DH-17站点表层沉积物未培养细菌 DH132B07克隆同源性较高(98%)。拟杆菌门的代表基因型DH214B14序列与南汤加弧火山 1号口的浅层热液喷口的未培养细菌 V1B07b75克隆具有很高的同源性(99%)。硝化螺旋菌门以DH214B11为代表, 有5个克隆子, 其序列与西班牙西北海岸谢斯群岛漏油区域沉积物的未培养细菌ANOX-094克隆同源性高达100%。浮霉菌门和螺旋体门在 DH21站点都只有一个 OUT分布, 其中浮霉菌代表序列DH211B11与墨西哥Alchichica盐碱湖的未培养细菌 Alchichica_AL52_2_1B_131克隆同源性高达 100%; 螺旋体门代表基因型 DH216B15序列与埃布罗、卡马格三角洲未培养细菌 LH042克隆亲缘性很高(100%)。疣微菌是DH21站点的特有类群, 其基因型 DH212B9序列与普罗旺斯 Etangde-Berre瀉湖沉积物未培养细菌13bis T0h-oil克隆高度同源(100%)。
3 讨论
3.1 典型优势菌群丰度和地理分布
变形菌门一直被认为是海洋表层沉积环境中的优势细菌类群, 一般都超过微生物生物量的 50%以上(Ravenschlaget al, 2001; Bowmanet al, 2003), 但在不同海域表层沉积物基因文库中所占的比例差异较大, 如卡斯卡底古陆边缘次表层沉积物中变形菌门含量占文库比例高达 95% (Marchesiet al, 2001),而在日本Nankai海槽1176站位次表层沉积物中, 变形菌门仅占基因文库的22% (Kormaset al, 2003)。关于变形菌在东海陆架表层沉积物中是否为典型优势菌群, 王健鑫等(2012)报道变形菌门占东海陆架表层沉积物细菌文库的 41.5%; 张东声(2011)研究长江口及邻近海域的沉积物微生物中变形菌门占该海域基因文库的 47.2%; 郑艳玲等(2012)研究崇明东滩表层沉积物变形菌门占文库的比例在 22.4%—34.6%; 本研究五个站点中有4个站点的变形菌门所占OTU数的比例接近或超过 50%, 说明变形菌门在东海区域是主要的细菌类群。
变形菌门不同纲的菌群在海洋沉积物中的丰度变化也是海洋微生物群落结构研究中的一个重要指标。王健鑫等(2012)研究表明γ-变形菌纲是优势类群,占东海 DH-13站点区域基因文库的 52%; Feng等(2009)对长江口和东海沿岸研究也认为沉积物中变形菌门以 γ-变形菌纲为主; 李友训等(2008)对东太平洋深海沉积物的研究也认为 γ-变形菌纲是该沉积物的优势菌群, 这些结果与本研究 DH6和 DH16相一致。但李涛等(2008)对南海西沙海槽地区的表层沉积物细菌多样性的研究发现 δ-变形菌纲为优势类群;郭建丽等(2013)对双台子河口沉积物中细菌多样性研究表明δ-变形菌纲占基因文库的60%, 为绝对优势菌群, 这些结果与本研究DH9、DH17和DH21站点相一致。另外有研究表明δ-变形菌丰度的增加可能是与重金属、石油烃等环境污染影响相关(Orcuttet al,2010; 李新伟, 2012; 郭建丽等, 2013), 本研究的DH9、DH17站点研究结果也与这一观点相符。从地理位置看, DH9和DH17站点位于舟山市嵊泗列岛附近, 周围渔船较多, 人为活动也相对频繁, 这些都可能引起重金属和油污污染。
绿弯菌门是20世纪80年代才被认可的一个新的系统发育分支(Woeseet al, 1987), 研究表明其在深海的冷泉、海底深部玄武岩、热液区等环境都有分布(Lysneset al, 2004; Reedet al, 2006), 被认为在低氧和厌氧环境中发挥重要作用。绿弯菌门在 DH9站点含量最高, 有着绝对优势, 在 DH17站点含量次于最优势菌δ-变形菌纲。分析两个站点发现, DH9(绿弯菌门共14个OTU, 表层占3个)和DH17(绿弯菌门共12个 OUT, 表层占 2个)表层含有绿弯菌门比例分别为21.4%和 16.7%, 这似乎也表明绿弯菌适合生活在含氧量较低的沉积物环境中, 与陈明娜(2007)的研究结果相一致。
3.2 特殊类别细菌丰度和地理分布
疣微菌门是革兰氏阴性细菌, 在海洋动物、南极沿岸沉积物和海水等环境中存在(Bowmanet al, 2003,2004; Sullivanet al, 2004), 能在厌氧条件下进行亚硝化作用(Freitaget al, 2003), 根据16S rRNA序列差异可将疣微菌门分为7个亚门(Schlesneret al, 2006), 但关于疣微菌门的研究还是很少。Freitas等(2012)对疣微菌门在全球海洋环境的分布和多样性的研究发现,疣微菌在海洋沉积物细菌群落中占1.4%, Ⅰ型和Ⅳ型亚门是沉积环境中丰度最高的类群, 对海洋中碳的生物地球化学循环有着重要作用, 同时对沿岸多个站点的研究表明, 陆地径流输出不是影响沿岸海域疣微菌门丰度的主要原因。本实验室前期研究发现疣微菌门在东海 DH-13站点的含量为 3.9%(王健鑫等,2012), 本研究表明疣微菌门是离岸最远的 DH21站点的特有菌群(占比1.1%), 其丰度和地理分布基本和Freitas等(2012)的结果相一致。
芽单胞菌门(Gemmatimonadetes)是土壤环境微生物的9大类群之一, 丰度占到土壤中细菌16S rRNA基因文库的 0.2%—6.5%, 平均含量为 2.2% (Janssen,2006; DeBruynet al, 2011), 但目前正式命名的仅为一属, 即芽单胞菌属(Gemmatimonas), 代表菌种为Gemmatimonas aurantiacastrain T-27, 是一类革兰氏阴性细菌, 通过出芽方式繁殖(Zhanget al, 2003)。芽单胞菌门在海洋沉积环境的研究并不多, Heijs等对地中海东部海底碳酸盐风化壳的研究发现有Gemmatimonadetes序列存在; Durbin等(2011)对南太平洋深渊沉积环境的微生物群落进行研究, 发现Gemmatimonadetes在表层沉积环境丰度较高(7%—16%), 但随着深度增加, 丰度快速减少; 另外根据16S rRNA序列将芽单胞菌门划分为4个亚门, 其中Ⅱ型和Ⅳ型亚门偏好存在于海洋和盐沼环境。芽单胞菌在本研究中存在于离岸最近的 DH9站点, 可能是由于长江和钱塘江等径流冲刷陆地土壤后进入海洋沉积环境的结果。
Candidate division使用最初的来源地或最先命名的克隆子来命名各分支, 如OPx (x为数字)是根据黄石公园热泉黑曜岩池塘(Obsidian Pool)而命名的(Hugenholtzet al, 1998)。Farag等(2014)通过 16S rRNA基因全序列分析, 将OP8分为4纲8目, 并发现该类群在烃类污染的环境中丰度较高, 其次是海洋生境(特别是热液喷口和珊瑚礁), 水, 非海洋生境(尤其是地面泉水和地下水样品); 陈明娜(2007)和李涛等(2008)分别对东西太平洋和南海的沉积物研究,均有发现该菌群, 本研究在DH16和DH17站点中也检测到同源序列, 说明 OP8在不同海域沉积物中还是广泛存在的。另一类Candidate division (WS3)只存在于 DH6站点, 早期研究认为该菌群与产甲烷环境有关(Dojkaet al, 1998), 王永霞(2014)在程海湖沉积物细菌多样性研究也有报道, Lin等(2015)研究发现,WS3一些类群能够形成硫复铁矿磁小体, 在铁和硫循环过程中扮演着未知的角色。
3.3 细菌群落结构与地质区域影响
东海陆架沉积环境错综复杂, 郑国兴等(1982)将东海陆架划分为现代长江水下三角洲沉积区、现代浅海沉积区、滨海残留沉积区、混合沉积区等不同地质区域。本研究的五个站点中, DH9和DH17位于现代长江水下三角洲沉积区, 且两个站点都位于人类活动密集的区域, 受人为影响比较多; DH6和DH16位于滨海残留沉积区, DH21站点位于混合沉积区。
随着东部沿海地区经济的发展和人类活动影响的加大, 陆源输入使得东海的富营养化程度增加和环境污染加剧(王新等, 2010), 比如重金属污染、化学物质污染、油气田的开发、近海船坞的建设等都在持续影响着东海的近海生态环境平衡, DH9和DH17受其影响比较多, 细菌多样性和丰度均较高, 有较多同源序列都与重金属、化学物质和油气污染相关。DH6和DH16位于滨海残留沉积区, 该区域是黑潮暖流与台湾暖流之间的过渡带, 为外洋海水所控制, 沉积速率低, 营养不够丰富, 故站点菌群的丰度不是很高,这与郑国兴等(1982)得出的在滨海残留沉积区菌量最低的研究结果相一致。DH21站点是本研究离海岸最远的站点, 其细菌多样性和丰度都比较高(尤其是放线菌门), 可能与郑国兴等(1982)研究中的 G8023站点(离岸最远海区)一样, 受较高浓度的有机质、总氮含量影响, 同时该站点部分同源序列与油烃污染环境有关。
王 新, 李志江, 郑天凌, 2010. 海洋浮游细菌在东海赤潮高发区的分布与活性. 环境科学, 31(2): 287—295
王永霞, 2014. 云南高原程海湖沉积物中的细菌多样性研究.昆明: 云南大学博士学位论文, 1—146
王健鑫, 许贤恩, 周链链等, 2012. 东海陆架表层沉积物微生物多样性初步研究. 海洋与湖沼, 43(4): 805—813
卢婧雯, 张心齐, 杜丽丽等, 2012. 中国东海及南海近海 4采样点海水可培养细菌的多样性研究. 浙江大学学报(理学版), 39(4): 443—449
有小娟, 李秋芬, 张 艳等, 2013. 象山港内西沪港海域沉积环境细菌群落结构的时空变化及其主要环境影响因子.应用与环境生物学报, 19(5): 742—749
刘玉娟, 田新朋, 黄小芳等, 2014. 中国南海沉积环境可培养细菌多样性研究. 微生物学通报, 41(4): 661—673
杜 萍, 刘晶晶, 沈李东等, 2012. Biolog和PCR-DGGE技术解析椒江口沉积物微生物多样性. 环境科学学报, 32(6):1436—1444
李 涛, 王 鹏, 汪品先, 2008. 南海南部陆坡表层沉积物细菌和古菌多样性. 微生物学报, 48(3): 323—329
李万超, 2008. 东海泥质区沉积物部分化学成分的组成特征及其对环境因素的响应. 青岛: 中国海洋大学硕士学位论文,1—77
李友训, 李富超, 秦 松等, 2008. 东太平洋深海沉积物中DNA的提取及细菌多样性初步分析. 海洋科学, 32(12):69—74
李家彪, 2008. 东海区域地质. 北京: 海洋出版社, 1—105
李新伟, 2012. 胶州湾近岸沉积物中细菌群落对石油和铜污染的响应特征. 青岛: 中国海洋大学硕士学位论文, 1—70
宋志刚, 许强芝, 鲁心安等, 2006. 中国东海海洋微生物种群多样性初步研究. 微生物学通报, 33(1): 63—67
张东声, 2011. 长江口及其邻近海域微生物的多样性和生态分布特征研究. 杭州: 浙江大学博士学位论文, 1—131
陈明娜, 2007. 东、西太平洋深海沉积物细菌多样性研究. 青岛: 中国科学院研究生院(海洋研究所)硕士学位论文,1—65
郑国兴, 史君贤, 陈忠元等, 1982. 长江口及邻近陆架海区细菌与沉积物相互关系的初步探讨. 海洋学报, 4(6):743—752
郑艳玲, 侯立军, 陆 敏等, 2012. 崇明东滩夏冬季表层沉积物细菌多样性研究. 中国环境科学, 32(2): 300—310
郭建丽, 樊景凤, 付 慧等, 2013. 双台子河口沉积物中细菌多样性分析. 微生物学通报, 49(9): 1550—1559
Arakaki A, Shibusawa M, Hosokawa Met al, 2010. Preparation of genomic DNA from a single species of uncultured magnetotactic bacterium by multiple-displacement amplification. Applied and Environmental Microbiology,76(5): 1480—1485
Arrigo K R, 2005. Marine microorganisms and global nutrient cycles. Nature, 437(7057): 349—355
Azam F, Worden A Z, 2004. Microbes, molecules, and marine ecosystems. Science, 303(5664): 1622—1624
Bowman J P, McCuaig R D, 2003. Biodiversity, community structural shifts, and biogeography of prokaryotes within Antarctic continental shelf sediment. Appl Environ Microbiol, 69(5): 2463—2483
Bowman J P, Nowak B, 2004. Salmonid gill bacteria and their relationship to amoebic gill disease. J Fish Dis, 27(8):483—492
DeBruyn J M, Nixon L T, Fawaz M Net al, 2011. Global biogeography and quantitative seasonal dynamics of Gemmatimonadetes in soil. Appl Environ Microbiol, 77(17):6295—6300
Dojka M A, Hugenholtz P, Haack S Ket al, 1998. Microbial diversity in a hydrocarbon-and chlorinated-solventcontaminated aquifer undergoing intrinsic bioremediation.Appl Environ Microbiol, 64(10): 3869—3877
Durbin A M, Teske A, 2011. Microbial diversity and stratification of South Pacific abyssal marine sediments. Environ Microbiol, 13(12): 3219—3234
Farag I F, Davis J P, Youssef N Het al, 2014. Global Patterns of Abundance, Diversity and Community Structure of theAminicenantes(Candidate Phylum OP8). PLoS One, 9(3):e92139, http://journals.plos. org/plosone/article?id=10.1371/journal.pone.0092139
Feng B W, Li X R, Wang J Het al, 2009. Bacterial diversity of water and sediment in the Changjiang estuary and coastal area of the East China Sea. FEMS Microbiol Ecol, 70(2):236—248
Freitag T E, Prosser J I, 2003. Community structure of ammonia-oxidizing bacteria within anoxic marine sediments.Appl Environ Microbiol, 69(3): 1359—1371
Freitas S, Hatosy S, Fuhrman J Aet al, 2012. Global distribution and diversity of marineVerrucomicrobia. ISME J, 6(8):1499—1505, doi:10.1038/ismej.2012.3
Hugenholtz P, Pitulle C, Hershberger K Let al, 1998. Novel division level bacterial diversity in a Yellowstone hot spring.J Bacteriol, 180(2): 366—376
Janssen P H, 2006. Identifying the dominant soil bacterial taxa in libraries of 16S rRNA and 16S rRNA genes. Appl Environ Microbiol, 72(3): 1719—1728
Kormas K A, Smith D C, Edgcomb Vet al,2003. Molecular analysis of deep subsurface microbial communities in Nankai trough sediments (ODP Leg 190, Site 1176). FEMS Microbiol Ecol, 45(2): 115—125
Köster M, Wardenga R, Blume M, 2008. Microscale investigations of microbial communities in coastal surficial sediments. Mar Ecol, 29(1): 89—105
Kumar S, Tamura K, Nei M, 2004. MEGA3: Integrated software for molecular evolutionary genetics analysis and sequence alignment. Brief Bioinform, 5(2): 150—163
Lin W, Pan Y, 2015. A putative greigite-type magnetosome gene cluster from the candidate phylumLatescibacteria. Environ Microbiol Rep, 7(2): 237—242, http://onlinelibrary.wiley.com/doi/10.1111/1758-2229.12234/abstract?deniedAccessC ustomisedMessage=&userIsAuthenticated=false
Lysnes K, Thorseth I H, Steinsbu B Oet al, 2004. Microbial community diversity in seafloor basalt from the Arctic spreading ridges. FEMS Microbiol Ecol, 50(3): 213—230
Marchesi J R, Weightman A J, Cragg B Aet al,2001.Methanogen and bacterial diversity and distribution in deep gas hydrate sediments from the Cascadia margin as revealed by 16S rRNA molecular analysis. FEMS Microbiol Ecol,34(3): 221—228
O’Sullivan L A, Fuller K E, Thomas E Met al, 2004.Distribution and culturability of the uncultivated ‘AGG58 cluster’ of theBacteroidetesphylum in aquatic environments.FEMS Microbiol Ecol, 47(3): 359—370
Orcutt B N, Joye S B, Kleindienst Set al, 2010. Impact of natural oil and higher hydrocarbons on microbial diversity,distribution, and activity in Gulf of Mexico cold-seep sediments. Deep Sea Res Part II, 57(21—23) : 2008—2021,doi:10.1016/j.dsr2.2010.05.014
Park S-J, Park B-J, Pham V Het al, 2008. Microeukaryotic diversity in marine environments, an analysis of surface layer sediments from the East Sea. J microbiol, 46(3):244—249
Ravenschlag K, Sahm K, Amann R, 2001. Quantitative molecular analysis of the microbial community in marine Arctic sediments (Svalbard). Appl Environ Microbiol, 67(1) :387—395
Reed A J, Lutz R A, Vetriani C, 2006. Vertical distribution and diversity of bacteria and archaea in sulfide and methane-rich cold seep sediments located at the base of the Florida Escarpment. Extremophiles, 10(3): 199—211
Schlesner H, Jenkins C, Staley J T, 2006. The Phylum Verrucomicrobia: a phylogenetically heterogeneous bacterial group. In: Dworkin M, Falkow S, Rosenberg E eds. The Prokaryotes. New York: Springer, 881—896
Schloss P D, Handelsman J, 2005. Introducing DOTUR, a computer program for defining operational taxonomic units and estimating species richness. Appl Environ Microbiol,71(3): 1501—1506
Simmons T L, Coates R C, Clark B Ret al, 2008. Biosynthetic origin of natural products isolated from marine microorganism-invertebrate assemblages. Proc Natl Acad Sci U S A, 105(12): 4587—4594
Venter J C, Remington K, Heidelberg J Fet al, 2004.Environmental genome shotgun sequencing of the Sargasso sea. Science, 304(5667): 66—74
Woese C R, 1987. Bacterial evolution. Microbiol Rev, 51(2):221—271
Zhang H, Sekiguchi Y, Hanada Set al, 2003.Gemmatimonas aurantiacagen. nov., sp. nov., a Gram-negative, aerobic,polyphosphate-accumulating micro-organism, the first cultured representative of the new bacterial phylumGemmatimonadetesphyl. Nov. Int J Syst Evol Microbiol,53(4): 1155—1163

